真核生物mRNA应激状态下的帽-非依赖性翻译
刘子昂, 金 焰, 吴 楠*
(1)中国遗传资源保护与疾病防控教育部重点实验室(哈尔滨医科大学), 哈尔滨 150081;2)哈尔滨医科大学医学遗传学研究室, 哈尔滨 150081)
在真核生物中,mRNA的结构可分为5个部分:m7GpppN帽结构、5′非翻译区(untranslated region,UTR)、编码序列(coding sequence,CDS)、3′非翻译区(untranslated region,UTR)和多腺嘌呤尾(Poly(A) tail)。这5个部分共同维持着mRNA翻译的起始、延伸和终止。其中,翻译的起始阶段最为重要且复杂,它决定了mRNA能否被有效翻译成蛋白质[1]。根据mRNA的5′端的m7G帽结构在翻译起始中的不同作用,可以将翻译起始机制分为2种:mRNA帽-依赖性翻译和帽-非依赖性翻译。在帽-依赖性翻译中,m7G帽结构对于翻译的起始是必需的,没有它翻译无法启动。而在帽-非依赖性翻译中,m7G帽结构并不是翻译起始所必需的,即使mRNA的5′端的帽结构缺失或不完整,翻译仍然可以进行[2]。传统的帽-依赖性翻译起始机制,在真核生物mRNA中占据主导地位,而当外部环境的刺激或细胞自身的改变使细胞处于应激状态时,传统的帽-依赖性翻译被抑制或下调,替代机制帽-非依赖性翻译得以启动[3]。
m7G帽结构依赖性翻译起始机制是真核生物进行翻译起始的主要形式。在真核生物中,翻译的起始阶段是组成有延伸能力的80S核糖体的过程。这个过程需要多种因子和结构的协同作用,包括翻译起始因子(eukaryotic initiation factor,eIF)[4, 5](Table 1)、m7GpppN帽结构、多聚腺苷酸尾、Kozak序列(Kozak consensus sequence)等。这些因子和结构能够影响mRNA与核糖体小亚基的结合、扫描到起始密码子的速度和准确性、核糖体大亚基的加入等过程,从而调节翻译起始的效率和精准性[1](Fig.1)。
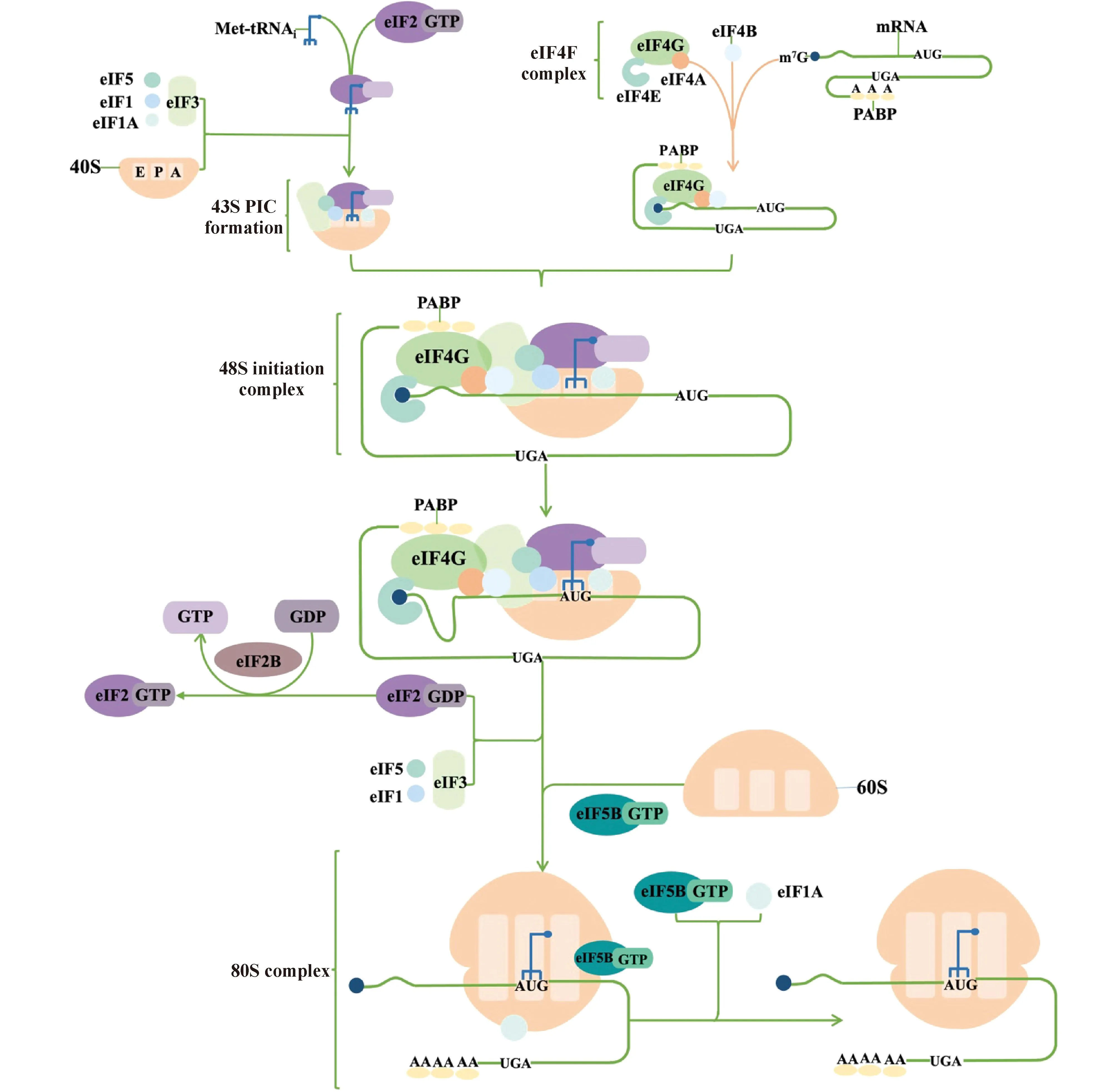
Fig.1 A model of the initiation mechanism of traditional cap-dependent translation in Eukaryotes Eukaryotic mRNA cap-dependent translation initiation is a multi-step, rate-limited process involving the assembly of multiple translation initiation factors and complexes, including the eIF4F complex, 43S preinitiation complex (PIC), 48S initiation complex, and 80S complex. (1) Assembly of eIF4F complex: eIF4A, eIF4E and eIF4G were assembled together to form eIF4F complex, which was bound to mRNA m7G cap structure; (2) Assembly of 43S preinitiation complex (PIC) : eIF2-GTP was first combined with tRNAiMet to form a ternary complex (eIF2-GTP-tRNAiMet), which was then combined with eIF1, eIF1A, eIF3, eIF5 and 40S ribosome small subunits to form a 43S preinitiation complex (PIC). (3) Assembly of 48S initiation complex: The 43S preinitiation complex (PIC) and eIF4F complex bound to the m7G cap structure of mRNA were promoted by eIF4B to co-assemble into 48S initiation complex, scanning the 5′untranslated region (UTR); (4) Assembly of the 80S complex: when the start codon was scanned, the translation initiation factor was dissociated and the large subunit of the ribosome was added, forming the 80S complex with extensibility. (5) The hydrolyzed eIF2-GDP will regain GTP under the action of eIF2B and participate in a new round of translation initiation. One of the core mechanisms of translation initiation is that eIF2 cycles between its GTP and GDP binding states
细胞应激状态是指细胞在受到压力刺激时所产生的一种特殊的生理状态,此时的细胞会通过mRNA翻译的可塑性及时、动态地应对各种刺激,例如低氧和营养匮乏[1]。在细胞应激条件下,替代翻译机制——帽-非依赖性翻译会被激活,以促进蛋白质的生产,维持细胞的生存和恢复。已经发现多种机制可以启动帽-非依赖性翻译,例如,内部核糖体进入位点(internal ribosome entry site,IRES)能通过招募40S核糖体小亚基特异性翻译拥有这种位点的mRNA;N6-甲基腺苷(m6A)修饰也可介导帽-非依赖性翻译,单个5′UTR中的m6A残基可直接招募43S预起始复合物以启动翻译;帽-非依赖性翻译增强子(cap-independent translation enhancer,CITE)招募翻译起始因子以结合43S预起始复合物,自mRNA的5′端开始扫描。本文在总结传统帽-依赖性翻译的基础上(Table 1,Fig.1),着重介绍了IRES、m6A和CITE所启动的帽-非依赖性翻译,旨在为更好地探索细胞应激状态下的翻译机制提供参考。
1 帽-非依赖性翻译起始机制
在一些压力因素下,例如缺氧、营养限制、药物刺激和分化等,均会导致细胞处于应激状态[1]。此时,细胞会通过调节mRNA翻译的可塑性以应对各种刺激。其中,在应激状态下,高能耗的mRNA翻译过程会被抑制或下调,以节省能量,这一过程主要是通过控制真核翻译起始因子4E(eukaryotic initiation factor 4E,eIF4E)与mRNA 5′端帽子结合来实现[6]。
eIF4E是翻译起始的限速因子之一,也是翻译控制的主要靶点。在正常情况下,eIF4E和真核翻译起始因子4E结合蛋白(eukaryotic translation initiation factor 4E-binding protein, 4E-BP)紧密结合。激活的雷帕霉素靶蛋白1(mechanistic target of rapamycin complex 1,mTORC1)可以直接诱导4E-BPs的磷酸化[7],减弱其与eIF4E的结合,从而释放eIF4E,并促进帽-依赖性翻译。已有研究发现,mTORC1的活性在营养缺乏、细胞周期阻滞、氧化应激、DNA损伤等应激条件下被抑制[7],使得4E-BPs不能被磷酸化进而继续结合eIF4E,导致帽-依赖性翻译被抑制,此时的mRNAs便可通过内部核糖体进入位点(IRES),或N6-甲基腺嘌呤(m6A)修饰来启动帽-非依赖性翻译。

Table 1 Eukaryotic translation initiation factor
1.1 IRES启动的翻译
真核生物的一些mRNA可以通过在5′UTR区域形成特殊的二级结构,使得翻译复合物在不依赖m7G帽结构的情况下能够进行识别和结合,从而实现帽-非依赖性翻译。其中,最为典型的RNA结构是内部核糖体进入位点,它可以在不依赖m7G帽结构的情况下,直接招募40S核糖体小亚基,从而实现帽-非依赖性翻译[8]。
最初发现IRES是在小核糖核酸病毒科的病毒中,例如脊髓灰质炎病毒[9]和脑心肌炎病毒[10]。后来发现,除了病毒mRNA外,在细胞mRNA中也存在IRES。其中,许多带有IRES的mRNA编码应激反应所需的蛋白质,包括DNA损伤、氨基酸饥饿、缺氧、内质网应激以及生理刺激,例如细胞分化或突触网络形成等[11-13]。因此,IRES在维持细胞应对各种应激状态下蛋白质翻译中发挥了重要作用。
1.1.1 IRES结构与分类 IRES位于mRNA的5′UTR区域,大约有10%的哺乳动物mRNA含有IRES[14]。与病毒IRES不同,真核细胞中的IRES通常缺乏序列和结构的相似性,因此,很难根据IRES的序列和结构将真核细胞中的IRES分为不同的组别。根据核糖体招募的机制,真核细胞IRES可以大致分为两种类型:I型IRES具有完整的结构,能与“IRES反式作用因子”(IRES trans-acting factor,ITAF)结合。这些ITAF直接与40S核糖体小亚基相互作用,或通过结合翻译起始因子共同招募40S核糖体小亚基[12, 13];而II型IRES包含短序列元件,可以与18S rRNA进行碱基互补配对来直接招募40S核糖体小亚基[15](Fig.2)。

Fig.2 A model of the initiation mechanism of cap-independent translation by IRES in Eukaryotes (1) Type I IRES: ITAF acts as an RNA chaperone to reconstruct the RNA structure around the IRES, allowing the 40S ribosome small subunits to bind to the IRES, or as a cohesive protein interacting with other translation initiation factors to co-recruit the 40S ribosome small subunits. (2) Type II IRES: Short sequence elements on type II IRES are directly complementary paired with 18S rRNA on the 40S ribosome small subunit to recruit the 40S ribosome small subunit
1.1.2 I型IRES ITAFI型IRES的活性由特定的蛋白质控制,称为“IRES反式作用因子”(ITAF),多数IRES都需要几个ITAF的协助来招募核糖体进行翻译启动。几乎所有的ITAF都是由IRES元件招募的RNA结合蛋白质,以促进核糖体组装到mRNA上。但ITAF如何促进依赖于IRES翻译的确切机制尚不清楚。有研究表明,它们可以作为RNA伴侣来重塑IRES周围的RNA结构,允许40S核糖体小亚基与IRES结合,或者作为衔接蛋白质与其他翻译起始因子相互作用共同招募40S核糖体小亚基[16-18]。从体外分析判断,如果不添加ITAF,大多数IRES的体外活性较弱,这证明了ITAF可以提高同源IRES的翻译效率[19]。由于ITAF的表达水平不同,IRES介导的翻译效率在不同的细胞类型或组织中普遍存在差异。例如,多嘧啶束结合蛋白(polypyrimidine tract binding protein,PTB)是一种经典的ITAF,在细胞应激过程中,PTB的表达大幅增加并与相关基因mRNA上的IRES的活性增强密切相关[20, 21]。
1.1.3 II型IRES 在许多动植物的mRNA中,发现了与18S rRNA不同区域互补的序列。其中,包括许多IRES[22, 23]。这表明,mRNA与rRNA的碱基互补配对可能会影响某些真核生物mRNA的翻译,并在特定的IRES活性下发挥作用。有研究采用16S rRNA与细菌中SD(shine-dalgarno)序列相互作用类似的标准,检测出mRNA 5′端的9个核苷酸序列与18S rRNA存在碱基配对的交互作用[23]。在先前的研究中,该序列被定义为IRES[22],后来证明它可以增强单链mRNA的翻译[23]。
1.1.4 IRES与疾病 IRES启动的帽-非依赖性翻译与多种疾病相关。在应对细胞压力时,P53肿瘤抑制蛋白可以通过其5′UTR中的IRES招募40S核糖体小亚基来促进其翻译。但是某些突变可能会导致IRES介导的P53合成受阻,从而影响其功能,导致一些疾病的发生,例如食管癌、小细胞肺癌和鳞状细胞癌[24]。此外,在胰腺癌细胞中,BZW1(basic leucine zipper and w2 domain-containing protein 1)蛋白的高表达促进了真核翻译起始因子2α亚单位(eukaryotic translation initiation factor 2 alpha,eIF2α)的磷酸化,抑制一般的全细胞翻译,但它并不影响IRES启动的翻译,导致与肿瘤相关的基因例如HIF1-α(hypoxia-inducible factor 1-alpha)和C-MYC(c-myc proto-oncogene)的翻译增加,从而增加了细胞增殖能力与胰腺癌的危险程度[25]。在乳腺癌细胞中,诱导ELP3(elongation protein 3)的稳定表达可以增强tRNA的修饰程度,影响LEF1(lymphoid enhancer-binding factor 1)mRNA的IRES结构,从而启动LEF1 mRNA的IRES依赖性翻译,促进乳腺癌的转移和生长[26]。
IRES启动的翻译除了在肿瘤发生发展中的重要作用外,在一些其他疾病中的功能同样不可忽视。例如,通过增强IRES依赖性翻译的相关蛋白质表达,可以降低肌肉细胞中代谢异常所致的炎症程度,改善肌肉功能和减少肌肉损伤,从而有效地减轻肌萎缩病患者的症状[27]。此外,有研究使用特殊的基因工程技术,将IRES和IL-10(interleukin 10)mRNA相连,制备了IRES-IL-10 mRNA,然后将这种IRES-IL-10 mRNA送入外泌体中,以此在响应炎症的情况下特异性向靶细胞递送,并且能够在体内通过细胞吞噬的方式靶向性向动脉壁细胞递送,从而减轻了动脉粥样硬化的病理损伤[28]。
这些研究表明,IRES启动的帽-非依赖性翻译在多种疾病的发生、发展和治疗中都具有重要的作用,并且可能成为治疗这些疾病的一个重要靶点。
1.2 N6-甲基腺嘌呤(m6A)修饰启动的翻译
N6-甲基腺嘌呤(m6A)是真核生物mRNA中最丰富的内部修饰[29]。这是一种可逆的碱基修饰,存在于5′非翻译区(UTR)、编码序列(CDS)和3′非翻译区(UTR)[29, 30]。在哺乳动物细胞中,m6A修饰的不对称分布使得5′非翻译区(UTR)相对于其他区域来说甲基化较少[30]。有研究发现,在m6A甲基转移酶METTL3(methyltransferase-like 3)缺失的情况下,含有5′UTR m6A修饰的mRNA的翻译效率大幅降低,而在其他区域含有m6A修饰的mRNA则未显示出这种效应[31]。通过检测细胞应激反应中全局5′UTR m6A修饰分布的变化,发现5′UTR m6A修饰与细胞应激状态相关,并提供了一种绕过m7G帽结构的mRNA翻译机制,实现了不依赖m7G帽结构的翻译起始模式[31]。
1.2.1 5′UTR中,m6A修饰的翻译起始机制 5′UTR内的m6A残基可以作为m6A诱导的核糖体结合位点,促进mRNA的帽-非依赖性翻译。一个单独的5′UTR m6A直接与真核翻译起始因子3(eukaryotic translation initiation factor 3,eIF3)结合,足以在缺少帽结合因子eIF4E的情况下招募43 S预起始复合物来启动翻译[32, 33](Fig.3)。目前,已发现多种细胞应激诱导了m6A在转录组水平上的重新分布,导致含有5′UTR m6A的mRNA数量增加,而腺苷甲基化的抑制选择性地降低了含有5′UTR m6A修饰的mRNA的翻译[31]。例如,在热休克应激反应中,新合成的mRNA 5′UTR内的某些腺苷被优先甲基化。进一步发现,新转录物的5′UTR m6A甲基化是由应激诱导的核定位YTHDF2(yth domain family protein 2)蛋白在细胞核内通过限制m6A去甲基化酶FTO(fat mass and obesity-associated protein)的结果[34]。以m6A形式增加的5′UTR甲基化,促进了帽-非依赖性翻译的启动,为热休克应激下的选择性mRNA翻译提供了一种机制。
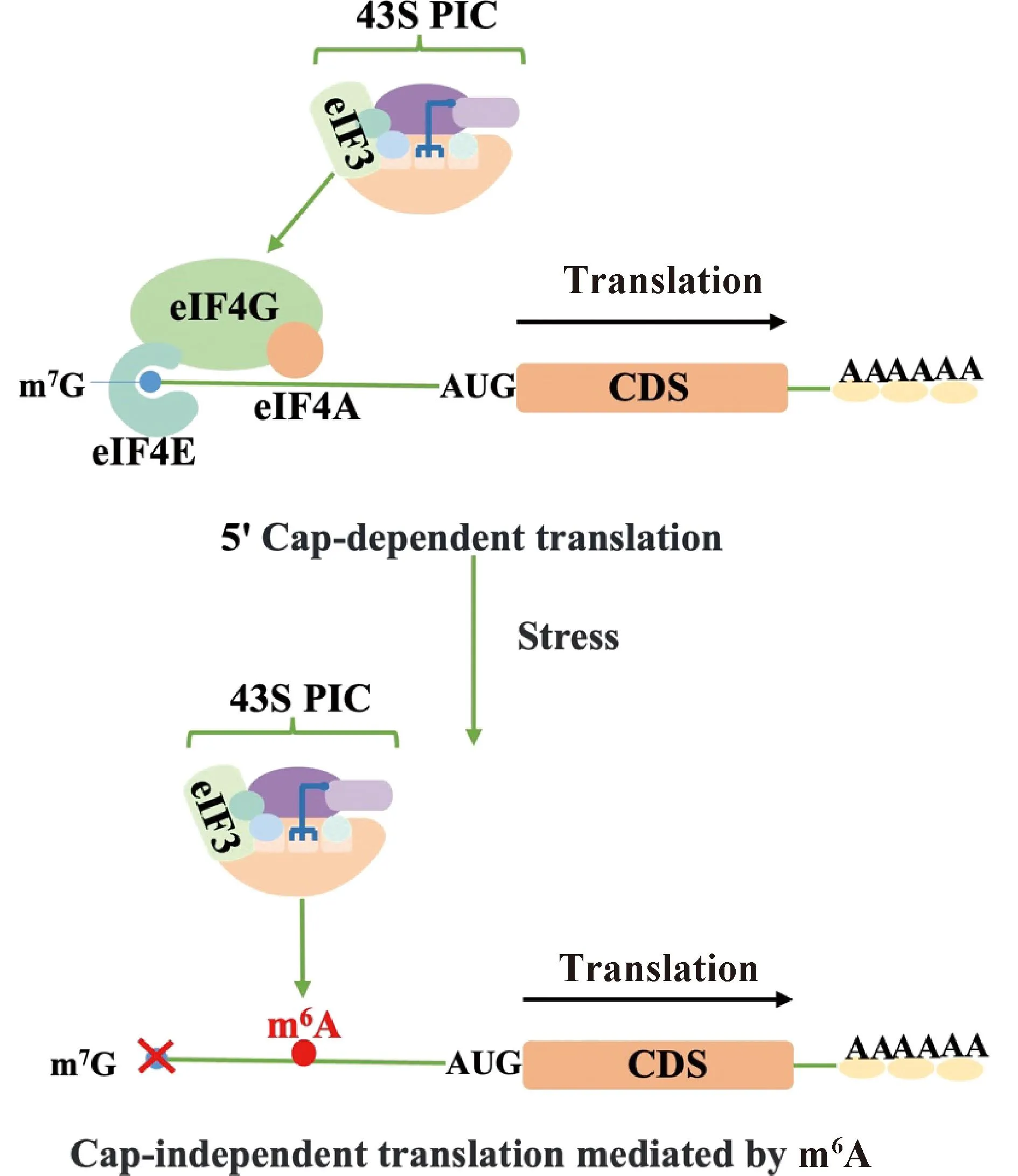
Fig.3 A model of the initiation mechanism of cap-independent translation by m6A in Eukaryotes Under stress, m6A residues within the 5′UTR directly bind to eukaryotic translation initiation factor 3 (eIF3), recruiting a 43S preinitiation complex to initiate translation
1.2.2 不同区域m6A修饰启动翻译机制的差异 m6A修饰对mRNA具有多种影响,这些效应主要归因于不同区域的m6A修饰启动翻译机制的差异。位于终止密码子周围或在3′UTR中的m6A修饰通过结合YTHDF1(yth domain family protein 1)蛋白,提高mRNA的翻译效率[35]。YTHDF1进一步结合多种蛋白质,包括eIF3和其他与核糖体相关的蛋白质,这些蛋白质被认为招募到3′UTR中以影响翻译的启动[32, 35]。这与位于5′UTR中的m6A修饰所启动的翻译不同。5′UTR中的m6A残基直接招募eIF3,并在5′UTR中组装翻译起始复合物,而不需要结合其他蛋白质[31]。5′UTR中的m6A残基通过一种独立于YTHDF1的机制促进帽-非依赖性翻译。
1.2.3 5′UTR中的m6A修饰与疾病 mRNA 5′UTR中的m6A修饰已经被证实与多种疾病发展相关。在乳腺癌发生和发展过程中,YTHDF3(yth domain family protein 3)蛋白结合到自身mRNA 5′UTR中的m6A残基,同时有助于eIF3A与同样位于mRNA 5′UTR上m6A残基结合,以调节其mRNA的翻译,从而增加自身蛋白质的表达水平[36]。此外,过多表达的YTHDF3蛋白通过多个步骤促进乳腺癌向脑部转移[36]。在宫颈癌和肝癌的肿瘤生长和进展中,PDK4(pyruvate dehydrogenase kinase 4)mRNA 5′UTR中的m6A残基通过与YTHDF1/eEF-2复合物和IGF2BP3(insulin-like growth factor 2 mrna binding protein 3)结合,正向调节其翻译延伸和mRNA稳定性,从而影响癌细胞的糖酵解和ATP产生[37]。在肝癌患者的组织标本中,YTHDF2蛋白通过结合OCT4(octamer-binding transcription factor 4)mRNA 5′UTR的m6A修饰来调节OCT4蛋白的表达,从而促进癌症干细胞表型和癌症转移的发生[38]。最近的研究表明,甲基转移酶(methyltransferase-like 5,METTL5)引导C-MYC的翻译能够促进胰腺癌细胞的增殖、迁移、入侵和肿瘤形成。在METTL5的特定翻译调控中,C-MYCmRNA的5′UTR m6A修饰发挥了关键作用[39]。
mRNA 5′UTR中m6A修饰在肿瘤外的许多相关疾病中也发挥着重要作用。在大脑皮层中,5′UTR中m6A修饰的mRNA具有调节皮质发育的独特功能和机制,与神经系统疾病密切相关[40]。此外,核输出蛋白1 mRNA 5′UTR中,m6A修饰会改变其蛋白质翻译,进而调节肠道炎症反应[41]。
1.2.4 5′UTR中m6A修饰的重要意义 在生物体应对特定形式的应激刺激(例如热休克刺激和紫外线刺激[31, 34])过程中,5′UTR中的m6A修饰具有重要意义。这种m6A修饰的应激响应,证明了5′UTR中m6A修饰的重要性及其可能存在的机制:帽-非依赖性翻译起始模式可以通过mRNA的5′UTR中腺苷残基的可逆甲基化来打开或关闭[31]。未来的研究对于了解应激途径在mRNA的5′UTR中如何增加m6A修饰并重塑RNA甲基组非常重要。此外,分析不同的应激反应途径如何利用这些上调的5′UTR m6A残基参与翻译反应也是至关重要的。
1.3 CITE启动的翻译
帽-非依赖性翻译也可以受到帽-非依赖性翻译增强子(cap-independent translation enhancer,CITE)的诱导。一些先前被认为含有IRES的mRNA未通过严格的翻译内部启动测试,表明它们可能采用了其他机制进行翻译起始[42]。此后,人们注意到mRNA 5′UTR中存在的CITE。
CITE位于真核生物mRNA的5′UTR区域,能够招募关键的翻译起始因子,并促进翻译起始复合物的组装。最初,CITE在一些植物病毒的mRNA非翻译区域(5′UTR和3′UTR)[43]被发现,可以帮助天然无帽的病毒mRNA实现高水平的翻译[44, 45]。随后,在真核生物的mRNA 5′UTR区域也发现了CITE元件,它能介导一种基于5′端扫描的翻译过程,而不依赖于m7G帽结构的存在[46]。
1.3.1 CITE的翻译起始机制 CITE的翻译起始机制涉及将翻译起始因子直接招募到靠近mRNA 的5′端结构化CITE上,随后与43S预起始复合物结合以启动扫描。无论mRNA的m7G帽结构是否存在,招募到的43S预起始复合物不是被主动地转移到mRNA的5′端,就是在内部位置捕获了5′端,然后在该位置启动扫描,最终扫描到AUG起始密码子以实现帽-非依赖性翻译的启动[47, 48]。
1.3.2 CITE和IRES的相似性与差异性 CITE和IRES在功能和位置上存在一定的相似性。已有研究表明,将病毒IRES中的1个与eIF4G结合的元件插入到哺乳动物细胞的强帽-依赖性mRNA的5′UTR中,其可以发挥CITE的作用,降低对mRNA 5′m7G帽结构的依赖,从而实现帽-非依赖性翻译[48]。与IRES相同的是,CITE也能绕过对5′m7G帽结构的依赖并抵抗eIF4F失活。然而,与IRES不同的是,CITE仍然需要mRNA的5′端才能实现帽-非依赖性翻译[47, 48]。
1.3.3 CITE启动的帽-非依赖性翻译 CITE启动的帽-非依赖性翻译机制已在人类APAF-1(apoptotic protease-activating factor-1)mRNA中被证实。研究表明,当eIF4E帽结合因子受到抑制时,可以通过该机制启动APAF-1 mRNA的翻译[46]。APAF-1 mRNA的5′UTR区域能够介导一种不依赖m7G帽结构,但依赖于5′端扫描的翻译,这种翻译方式甚至在细胞发生凋亡时也是有效的[46]。APAF-1在DNA损伤诱导的细胞凋亡中扮演核心角色,其缺失可能促进肿瘤的恶性转化[49]。此外,APAF-1基因的失活与某些恶性肿瘤的疾病进展以及对化疗的耐药性相关,例如转移性黑色素瘤[50]。因此,CITE介导的翻译在维持APAF-1蛋白水平以及在应激条件下调节其作为抑制肿瘤蛋白质的活性方面可能发挥重要作用。
2 其他帽-非依赖性翻译起始机制
已有研究报道,模式诱导免疫(pattern-triggered immunity,PTI)信号转导途径揭示了一种帽-非依赖性翻译机制,促进应激诱导的转录后调控[51]。PTI诱导mRNA脱帽以抑制一般的翻译,但具有富含嘌呤元素(“R-motif”)的mRNA,利用R-motif作为核糖体进入位点进行选择性的帽-非依赖性翻译[51]。
在真核生物中,除了常规的mRNA,帽-非依赖性翻译也会发生在非编码RNA(non-coding RNA,ncRNA)中[52],其中包括circRNA。circRNA是一种在真核生物中广泛存在的非编码RNA[53]。近期的研究表明,一些细胞质中的circRNA可以被有效翻译成可监测到的肽,这揭示了circRNA在细胞生理功能中的重要性[54, 55]。由于circRNA缺乏5′端和3′端,它们的翻译只能通过帽-非依赖性翻译机制启动。IRES和m6A修饰介导的帽-非依赖性翻译起始被认为是circRNA翻译的潜在机制[53-57]。已有研究发现,内源性circRNA中存在类似IRES的短元件,可借助这些短元件启动circRNA的翻译[55, 58]。m6A修饰也已被证明可以促进人类细胞中circRNA的有效翻译启动[53, 54]。研究发现,circRNA中存在丰富的m6A修饰,单个m6A位点就足以推动翻译的启动[53]。这种m6A驱动的翻译需要翻译起始因子eIF4G2和m6A阅读蛋白YTHDF3的参与[59],并受到甲基转移酶METTL3/14的增强和去甲基化酶FTO的抑制[54]。在热休克应激时,这种m6A驱动的翻译被上调[54]。
3 问题与展望
本文着重介绍了IRES、m6A和CITE所启动的帽-非依赖性翻译。其中,IRES通过招募40S核糖体小亚基特异性的翻译拥有这种IRES序列的mRNA;而单个5′UTR中的m6A残基可以直接招募43S预起始复合物以启动翻译;CITE则招募翻译起始因子以结合43S预起始复合物,自mRNA的5′端开始扫描。据报道,任何长度超过50nt的片段都可能偶然包含类似IRES的短元素[60],表明许多序列都可以驱动与帽无关的翻译,尤其是在应激条件下抑制与帽相关的翻译时。这一发现还意味着,单个mRNA中可以编码多个ORF,一些ORF可以通过帽-非依赖性翻译进行内部翻译。类似于替代剪接,这种“替代翻译”可以作为增加人类基因组编码蛋白质组复杂性的新机制。
随着对真核生物mRNA翻译起始模式的研究,帽-非依赖性翻译机制的探索越来越受到重视。未来,研究方向可以从以下几个方面展开:(1)探索帽-非依赖性翻译机制中的未知因素和机制。目前已经发现,帽-非依赖性翻译机制中存在多种不同的启动机制,但仍有许多未知的因素和机制需要进一步探索和研究。(2)挖掘更多的翻译起始机制。揭示生物体在不同环境条件下,特别是在响应环境压力或病理状态时,不同的翻译方式是如何交替发挥作用的,对深入认识基因在翻译水平上的表达调控有重要意义。(3)将帽-非依赖性翻译机制应用到临床实践中。如何利用帽-非依赖性翻译机制来治疗一些疾病或病理状态,是一个值得研究的方向。最后,细胞应激状态下的翻译机制研究还可以结合其他生物学领域,例如基因表达调控和蛋白质合成等,从而推动更全面和深入的研究。
- 中国生物化学与分子生物学报的其它文章
- 青蒿素通过TIGIT/CD155信号轴调控4T1乳腺癌细胞转移与凋亡
- 丝裂原活化蛋白激酶15纳米抗体的制备及其在B16-F10黑素瘤细胞生长过程中的抑制作用
- 外套膜转录物组分析揭示尿素在贻贝耐受海水酸化中的作用
- KLF4 Protects VSMCs from Damage of Foam Cell-like Phenotypic Transformation by Maintaining Cellular Homeostasis through Supporting PPARγ Signaling Pathway
- 《中国生物化学与分子生物学报》2022~2023年度优秀编委
- 2023年封面汇总

