禾谷炭疽菌中内吞相关蛋白找寻及特征解析
张凯强, 韩长志,2*
(1.西南林业大学生物多样性保护学院, 昆明 650224; 2.云南省森林灾害预警与控制重点实验室, 昆明 650224)
受体介导的内吞作用作为诸多生物体内大多数细胞与环境相互作用的重要途径之一[1-2],通过内吞相关蛋白作用,实现对于特定生物大分子的摄取[3]。文献[4]对于模式真菌(酿酒酵母、构巢曲霉等)中的内吞作用相关蛋白展开了诸多研究工作,明确含有NPFxD基序以及DPFxD基序的蛋白,且基序蛋白通常参与内吞靶向信号过程。对于植物病原丝状真菌而言,其内吞过程与菌丝细胞的生长、分化以及发育等关系密切[5]。
禾谷炭疽菌[Colletotrichumgraminicola(Cesati) Wilson]是一种半活体营养型植物病原丝状真菌,可以侵染玉米、小麦、高粱等禾本科植物而引起炭疽病,给世界各国农业生产造成巨大的经济损失[6-7]。20世纪以来,学者们对植物病原丝状真菌的研究主要集中在遗传转化系统[8]、细胞凋亡[9]、异源蛋白表达[10]等方面。随着2012年该菌全基因组序列的释放[11],促进了学术界对于其促分裂原活化蛋白激酶(mitogen-activated protein kinase,MAPK)信号转导途径[12]和G蛋白信号调控因子(regulators of G-protein signaling, RGS)[13]以及Rab蛋白[14]等研究工作。本研究小组前期对该菌的G蛋白信号通路相关蛋白[15-16],碳水化合物活性酶类蛋白[7]、RGS蛋白[13]、效应分子motif序列[17]等开展了诸多研究报道,并且对该菌NPFxD基序蛋白进行了找寻及特征解析[18]。NPFxD基序以及DPFxD基序蛋白作为模式真菌中均含有的内吞相关蛋白,对于植物病原丝状真菌来说,以上两种蛋白的找寻及特征解析有助于后续有针对性的开展生物学试验对其功能进行研究,但目前学术界对于植物病原丝状真菌生长发育过程中DPFxD基序蛋白的报道较为少见,且中外学者对禾谷炭疽菌中存在具有DPFxD基序的蛋白情况尚未见学术报道。
鉴于此,基于文献[19]报道,利用模式真菌构巢曲霉Aspergillusnidulans中具有DPFxD基序的蛋白氨基酸序列,利用同源序列比对及关键词搜索等方法,对禾谷炭疽菌中全蛋白进行分析,找寻具有DPFxD基序的蛋白,并对上述蛋白的保守结构域、跨膜结构域、亚细胞定位、理化性质等特征进行分析,为进一步开展生物学的试验验证工作打下坚实的理论基础。
1 材料与方法
1.1 数据来源
通过NCBI数据库下载禾谷炭疽菌M1.001全基因组序列(https://www.ncbi.nlm.nih.gov/genome/?term=Colletotrichum+graminicola)。
1.2 DPFxD基序蛋白序列找寻方法
利用EMBOSS fuzzpro[20]预测获取具有DPFxD保守结构域的蛋白质序列,对上述所获得序列进行汇总分析,最终明确该菌中DPFxD基序蛋白登录号、功能等相关信息。
1.3 DPFxD基序蛋白特征分析
1.3.1 跨膜区结构预测
利用在线跨膜区结构预测网站HMMTOP version 2.0[21]和TMHMM Server v.2.0[22]等对具有DPFxD基序的蛋白进行预测分析。
1.3.2 保守结构域预测
利用在线保守结构域特征分析软件SMART[23]对具有DPFxD基序的蛋白进行分析。
1.3.3 蛋白亚细胞定位分析
利用亚细胞定位分析软件ProtComp v9.0[24]对具有DPFxD基序的蛋白进行预测进行并绘制其的定位情况。
1.3.4 理化性质分析
利用理化性质测定程序Protscale[25]对具有DPFxD基序的蛋白进行预测。
1.3.5 信号肽预测
利用蛋白质转运肽线在分析软件TargetP 2.0 Server[26]对具有DPFxD基序的蛋白进行预测,利用蛋白质信号肽在线分析软件SignalP 5.0 Server[22]对具有DPFxD基序的蛋白进行预测。
1.3.6 二级结构预测
采用蛋白质二级结构预测在线分析软件Phyre[27]对具有DPFxD基序的蛋白进行预测。
1.3.7 系统进化树构建
在NCBI数据库中在线进行Blastp搜索获取同源序列,并利用ClustalX[28]和MEGA X软件[29]分别对其进行多重比对分析和构建系统进化树。
2 结果与分析
2.1 禾谷炭疽菌中DPFxD基序蛋白找寻
文献[19]对构巢曲霉中的全基因组开展DPFxD基序蛋白的搜索工作,利用EMBOSS fuzzpro软件包进行预测[20],通过对禾谷炭疽菌全基因组序列进行分析,共获得了48条含有DPFxD基序的蛋白序列,如表1所示。
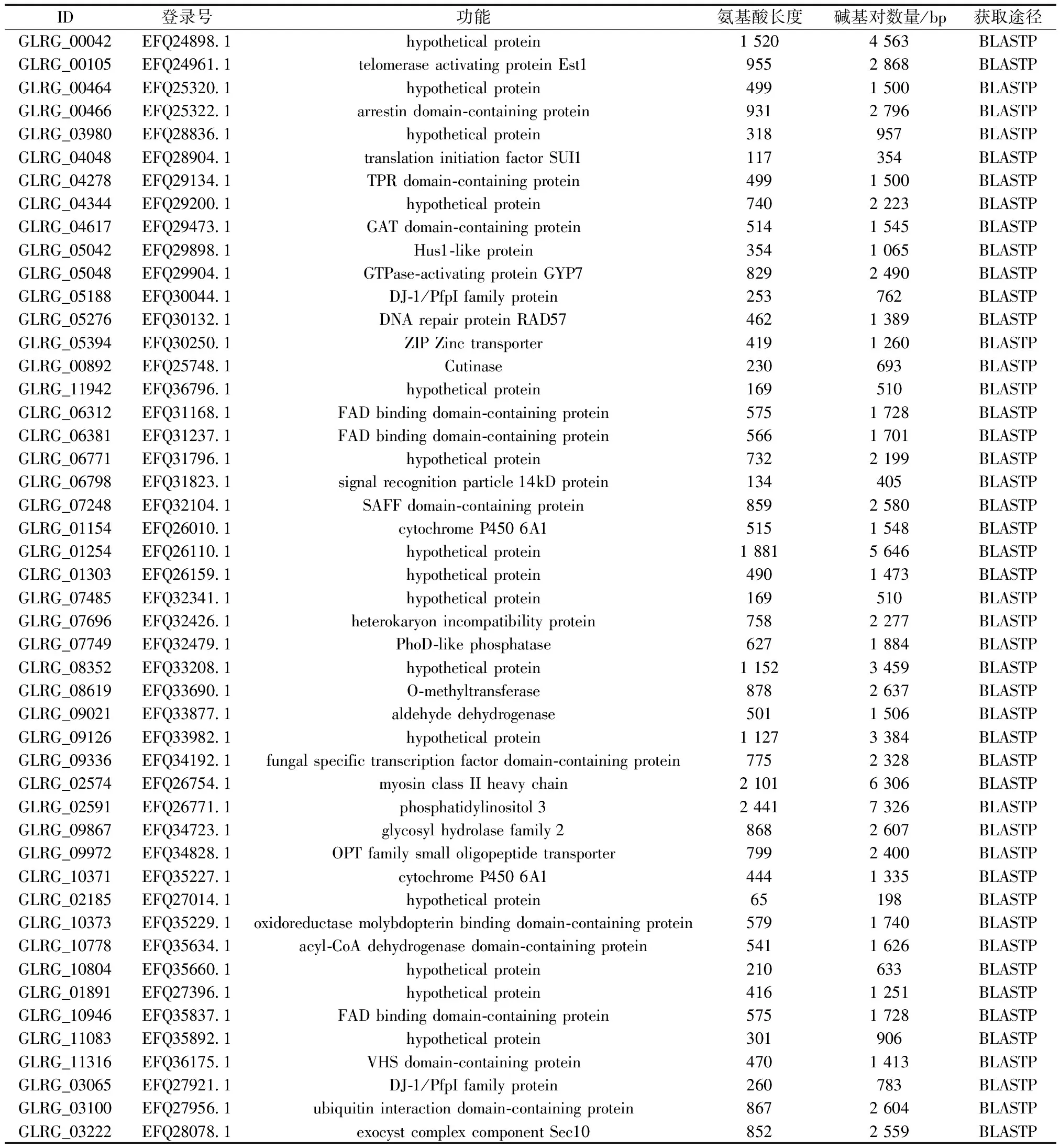
表1 禾谷炭疽菌中DPFxD蛋白的基本信息及获取方法
2.2 保守结构域及亚细胞定位分析
基于TMHMM跨膜结构域分析,具有1个及以上跨膜结构域的蛋白共9个,而具有2个及以上跨膜结构域的蛋白共4个,其ID分别为GLRG_05394、GLRG_01154、GLRG_09972、GLRG_01891;进一步利用HMMTOP进行预测发现,具有1个及以上跨膜结构域的蛋白共18个,而具有2个及以上跨膜结构域的蛋白共9个,其中ID为GLRG_09972、GLRG_05394、GLRG_01891的蛋白具有跨膜结构域的数量较多,分别为19、8和7个。
SMART在线分析表明,仅有9个蛋白具有明显的保守结构域,其ID分别为GLRG_00466、GLRG_05048、GLRG_05276、GLRG_00892、GLRG_09336、GLRG_02591、GLRG_10373、GLRG_10778、GLRG_03100(图1)。这些蛋白具有的保守结构域不相同,包括抑制蛋白C末端结构域、Tre-2-BUB2p-Cdc16p结构域、与各种细胞活动相关的ATP酶、水解酶、类GAL4 Zn(II)2Cys6(或C6锌)双核簇DNA结合域、真菌特异性转录因子结构域、UVSB PI-3激酶,MEI-41和ESR-1的结构域、类固醇结合域、泛素相互作用基序等。
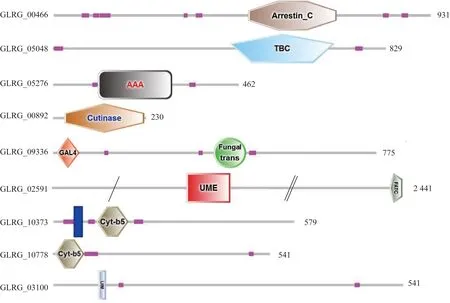
Arrestin_C为抑制蛋白C末端结构域;TBC为Tre-2-BUB2p-Cdc16p结构域;AAA为AAA结构域;Cutinase为角质酶;GAL4为类GAL4 Zn(II)2Cys6(或C6锌)双核簇DNA结合域;Fungal trans为真菌特异性转录因子结构域;UME为UVSB PI-3激酶,MEI-41和ESR-1的结构域;FATC为FRAP、ATM、TRRAP C终端命名的结构域;Cyt-b5为类固醇结合结构域;UIM为泛素相互作用基序图1 DPFxD基序蛋白保守结构域预测Fig.1 Conserved domain prediction of protein with DPFxD motif protein
对蛋白进行亚细胞定位分析(图2),结果表明,ID为GLRG_04048、GLRG_05276、GLRG_09972、GLRG_10373的蛋白亚细胞定位分别在质膜、细胞核、内质网、线粒体,其余44个蛋白均定位在胞外。
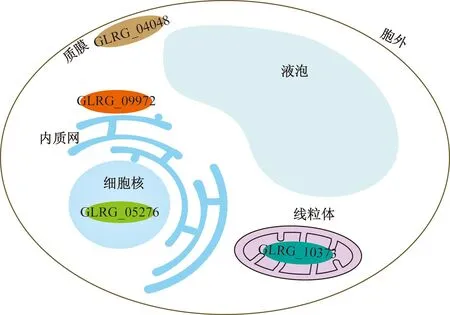
图2 亚细胞定位分析Fig.2 Subcellular localization
2.3 理化性质分析
对48个DPFxD基序蛋白中氨基酸组成情况进行分析,结果显示,A(丙氨酸)含量最高,平均达64个;L(亮氨酸)、S(丝氨酸)含量次之,均为58个;而W(色氨酸)、C(半胱氨酸)含量较低,平均仅为10个和6个(图3)。
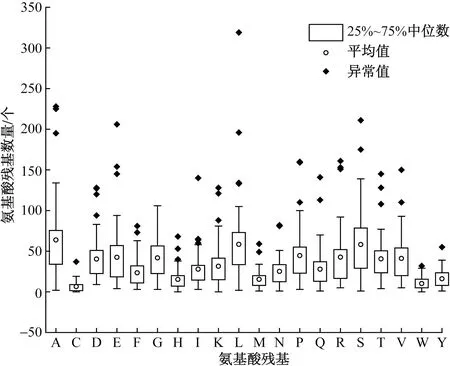
图3 DPFxD基序蛋白氨基酸组成情况Fig.3 Amino acid composition of DPFxD motif protein
理论等电点位于5.51~6.00的蛋白数量最多,达10个,所占比例为20.83%;等电点位于4.51~5.00、5.01~5.50、6.01~6.50和8.50~9.00的蛋白数量次之,均为6个,所占比例为12.50%[图4(a)]。就蛋白稳定性而言,共31个蛋白不稳定系数大于40,所占比例为65.58%亲水性总平均值小于0的蛋白共43个,所占比例为89.58%,且亲水性总平均值总和为-19.69,平均为-0.41[图4(b)],属于亲水性蛋白。就脂肪族氨基酸指数而言,共34个蛋白分布于70~105,所占比例为70.83%,其中脂肪族氨基酸指数在70~80的蛋白数量最多,达16个,所占比例为33%[图4(b)]。
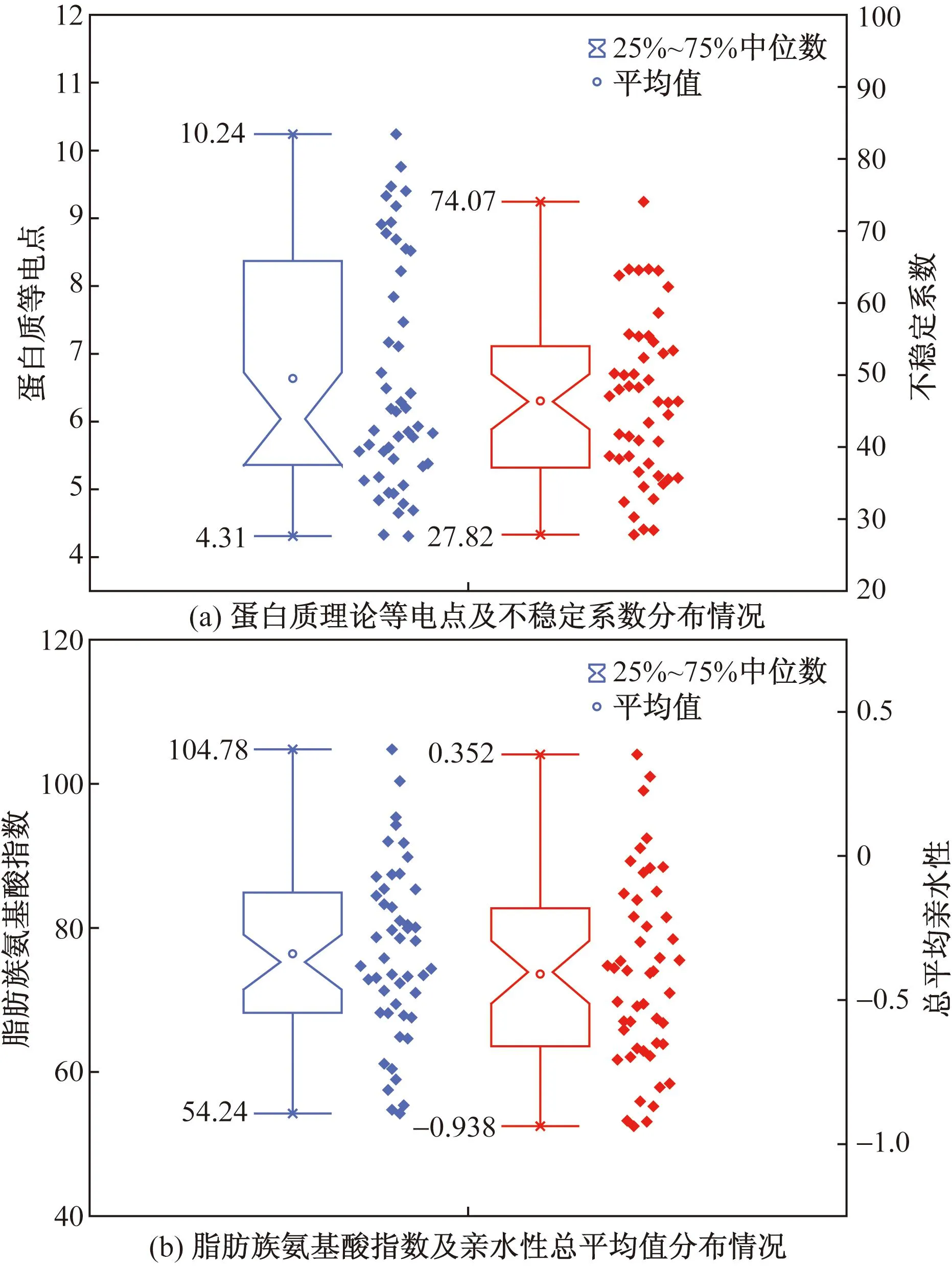
图4 DPFxD基序蛋白基本理化性质Fig.4 Basic physical and chemical properties of DPFxD motif protein
对48个DPFxD基序蛋白的亲(疏)水性进行预测,结果表明,在亲(疏)水性最强氨基酸残基及位置方面也存在较大差异。蛋白ID为GLRG_01254的蛋白中位于1 442位的R亲水性最强,亲水性系数为-4.100;而ID为GLRG_05394的蛋白中位于294位和298位的L和V疏水性最强,疏水性系数为3.744[图5(a)]。进一步对每个蛋白的最强亲(疏)水性氨基酸残基进行统计分析,结果显示:最强亲水性氨基酸残基为D和R的蛋白数量最多,分别为12和11个;而最强疏水性氨基酸残基为L、A和V的蛋白数量最多,分别为12、9、8个[图5(b)]。
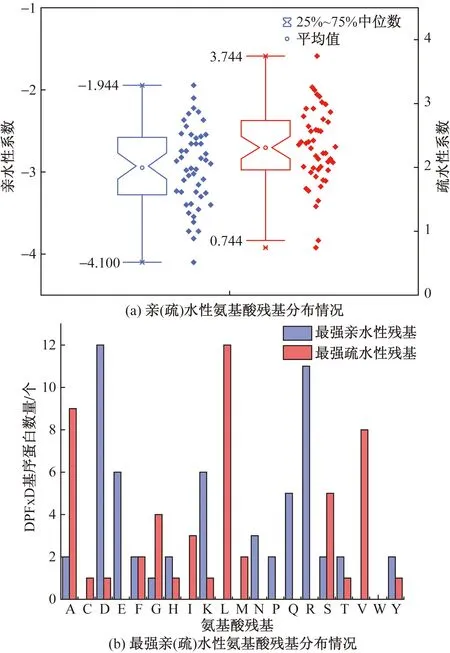
图5 DPFxD基序蛋白的亲(疏)水性氨基酸残基的分布Fig.5 Distribution of hydrophilic (hydrophobic) amino acid residues of DPFxD motif protein
2.4 转运肽及信号肽特征
通过TargetP分析,8个DPFxD基序蛋白定位于信号肽,仅有1个蛋白定位于线粒体,其余蛋白转运肽预测可靠性不高,未得到有效定位情况。其中ID为GLRG_04278的蛋白定位于线粒体,预测可靠性高达90.36%;ID为GLRG_06381的蛋白定位于信号肽,预测可靠性达99.99%。由于TMHMM和HMMTOP程序对于跨膜结构和信号肽的预测存在重叠性,利用SignalP 5.0进一步分析,结果显示,6个DPFxD基序蛋白具有明显的信号肽,其余均无明显信号肽,信号肽切割位点位于15~20的蛋白有4个,所占比例为67%(表2)。
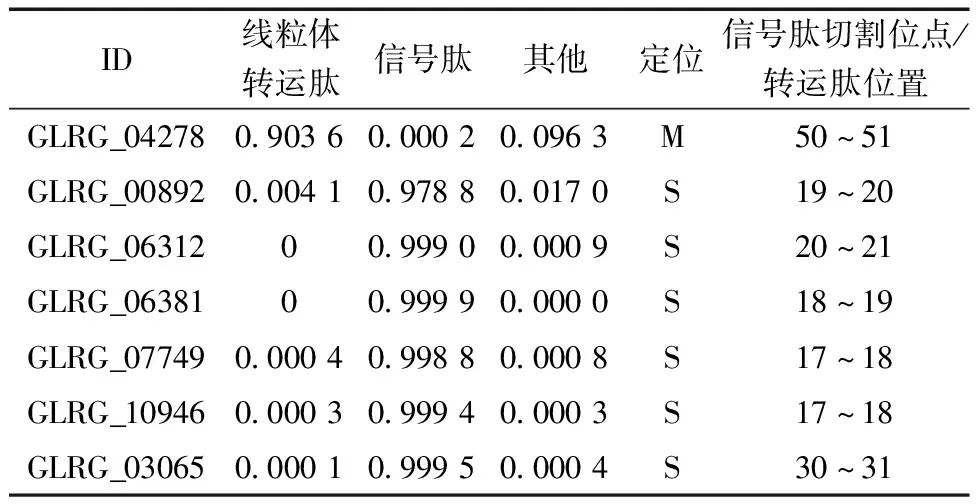
表2 DPFxD基序蛋白的转运肽及信号肽特征
2.5 二级结构分析
蛋白ID为GLRG_07485的蛋白α螺旋比例较高,达84%,但其却不含TM螺旋和β螺旋;ID为GLRG_06771的蛋白无规则卷曲的比例高达96%,而其他3种结构比例较低;总体来看48个蛋白中TM螺旋与β螺旋比例较低,最高仅为43%和37%,如图6所示。
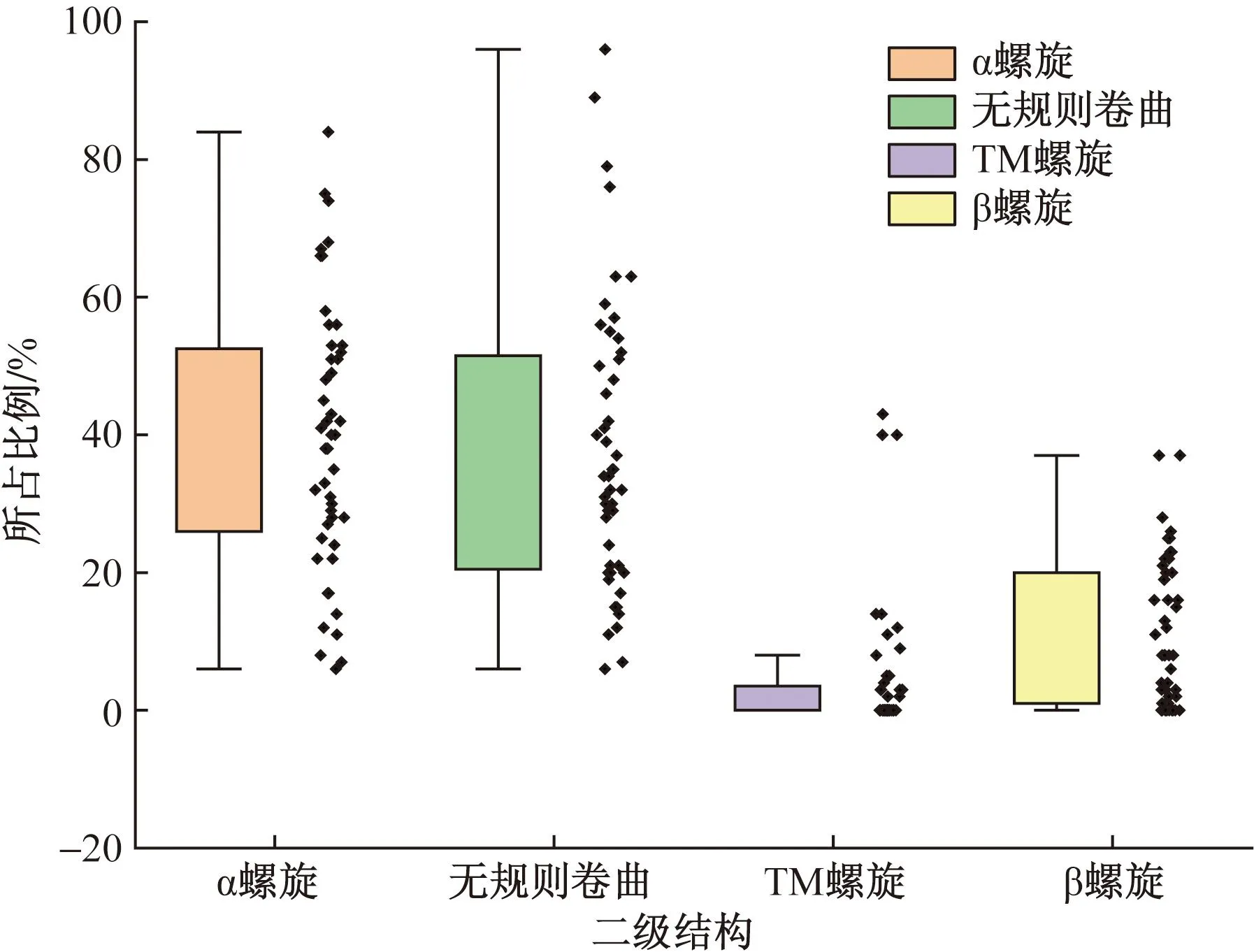
图6 二级结构分析Fig.6 Secondary structure analysis
2.6 遗传关系分析
以C.graminicola中的48个含有DPFxD基序的蛋白序列为基础,在NCBI中进行同源蛋白找寻。结果显示,该菌中的DPFxD蛋白与炭疽菌属中有较高同源性以及较近亲缘关系的病菌有C.sublineola、C.incanum、C.tofieldiae等,分为明显的4大类(图7),表明C.graminicola中大部分DPFxD基序蛋白之间的同源性较高。4个分支中所含DPFxD基序蛋白数量分别为17、17、5和9个,其中ID为GLRG_11942、GLRG_02591、GLRG_09867、GLRG_10778、GLRG_09021的5个蛋白属于同一分支,其亲缘关系较近,但与其他几个分支相比该分支蛋白数量最少;ID为GLRG_06381、GLRG_06312、GLRG_10946、GLRG_05394、GLRG_01891、GLRG_09972、GLRG_07696、GLRG_07485、GLRG_00466的9个蛋白属于同一分支;其余两个分支中蛋白数量最多,均为17个。结果表明,C.graminicola中具有DPFxD基序的蛋白在长期进化过程中总体较为稳定,但部分蛋白在进化过程中产生了较大分化。
3 讨论
植物病原丝状真菌中的分泌蛋白及CAZymes等致病因子在其生长发育及致病过程中发挥着重要作用[30],随着学术界对禾谷炭疽菌致病因子不断的深入研究,一些如GPCR[16]、PI-PLC[31]、Pth11[32]、CFEM[33]等涉及G蛋白信号效应分子逐渐得到进一步确认。然而,对于禾谷炭疽菌DPFxD基序蛋白的研究报道还比较罕见。具有DPFxD基序的蛋白可用于其他正常受体的内化过程,对于缺乏泛素化位点的受体尤为重要[34]。内吞作用可以大量存在丝状真菌菌丝尖端,并且菌丝尖端的快速延伸可能需要配合内吞作用[35]来实现。中外学者对内吞在病菌菌丝生长中的作用已有了相对明确的了解,如稻瘟菌中内吞调控蛋白可以有效抑制寄主免疫反应[36]。根据前人内吞机制调控蛋白研究成果分析,禾谷炭疽菌菌丝成长过程极有可能与内吞机制的调控有关联。当植物通过各种信号途径激活体内的免疫受体,从而减缓病菌等的进一步扩散和传播;同样,当禾谷炭疽菌感应到植物的免疫防御反应时也会采取的应对手段,如分泌毒素、角质酶、果胶酶和纤维素酶等致病因子的方式,破坏寄主植物的免疫受体,建立寄生关系,从而达到侵染的目的。而内吞作用在禾谷炭疽菌寄生过程中的地位和作用,内吞作用与解毒物质分泌之间的联系,内吞作用与植物防御反应情况以及内吞作用对效应分子分泌和植物中营养吸收的影响等问题均有待于对具有DPFxD基序的蛋白功能开展进一步的研究和探讨,从而较好地明确内吞机制,更好地防控植物病原菌。
4 结论
通过关键词搜索、Blastp比对分析、跨膜结构域以及亚细胞定位等进行预测分析,首先明确了C.graminicola中存在48个DPFxD基序蛋白;其次,通过在线分析软件SMART、ProtComp v9.0等,明确其保守结构域、信号肽、蛋白质二级结构、疏水性、遗传关系等情况;最后测定出有多条与DPFxD基序蛋白预测功能相同的蛋白质序列;此外,通过遗传关系分析,明确禾谷炭疽菌DPFxD基序蛋白与炭疽菌属中的C.incanum、C.sublineola、C.tofieldiae等病菌有较高的同源性和较近的亲缘关系等。以上这些成果为深入研究其他炭疽菌属真菌DPFxD基序蛋白提供有益的参考价值。

