Duddingtonia flagrans几丁质酶CTS3/CTS4的分子特征及亲缘关系
韩雨璇,张亚萍,李柠杏,张嘉华,李能秀,焦 健,才学鹏, 乔 军,孟庆玲
(1.石河子大学动物科技学院,新疆 石河子 843002; 2.中国农业科学院兰州兽医研究所,兰州 730046)
【研究意义】几丁质(Chitin)是由N-乙酰-D-氨基葡萄糖(N-Acetyl-D-glucosamine, GlcNAc)以β-1, 4糖苷键连接而成的高分子聚合物[1],该聚合物是大部分真菌细胞壁、昆虫外骨骼、无脊椎动物角质层的重要结构成分[2]。几丁质酶(Chitinase, EC 3.2.1.14)是一种通过切割β-1,4糖苷键将几丁质水解成几丁寡糖和N-乙酰-D-氨基葡萄糖的糖苷水解酶,广泛存在于生物界中[3]。几丁质酶在丝状真菌菌丝的结构形成、线虫卵壳的降解等生理活动中发挥十分重要的作用[4-6],在生物防治中具有潜在的应用价值。【前人研究进展】捕食线虫性真菌(Nematophagous fungi,NTF)作为家畜消化道寄生虫的天敌,是最有潜力的化学药物的替代品。近年来,由于捕食线虫性真菌不存在化学防治的弊端,即使线虫产生耐药性,符合生态可持续性的需求,该菌已成为寄生虫线虫防控的重要研究方向。捕食线虫性真菌分为5个类型:捕食性真菌(Predatory fungi)、杀卵型真菌(Ovicidal fungi)、内寄生型真菌(Endoparasitic fungi)、产毒素型真菌(Toxin-producing fungi)和带有特殊攻击装置的真菌(Producers of special attack devices )[7]。研究证实,NTF经动物消化道后,也不会丧失掠食能力,并能有效控制未成熟阶段线虫(包括虫卵和幼虫)[8],且孢子在粪便中的萌发能力较强。掠食型真菌可以产生诱捕器,是杀死线虫的关键工具。诱捕器由特化菌丝形成,分为黏性诱捕器(黏性菌网、黏性菌丝、黏性菌分枝、黏性菌结)和非黏性诱捕器(非收缩和收缩环)[9],均通过机械力或酶促反应侵染线虫的成虫和幼虫[10-11]。研究表明,胞外水解酶尤其是几丁质酶在捕食性真菌侵染线虫的过程中具有重要作用[12]。研究发现,捕食线虫性真菌产生的几丁质酶可直接用于杀线虫幼虫和虫卵[7,13]。然而,从自然界中分离的几丁质酶酶活性较低,目前对捕食线虫性真菌几丁质酶的结构及功能也了解甚少。【本研究切入点】Duddingtoniaflagrans是一种最具有应用前景的捕食线虫性真菌[14],其产生的厚垣孢子经家畜消化道和其它不良环境仍可萌发形成诱捕器,捕获家畜粪便中的线虫幼虫[15-16]。体外实验研究表明,D.flagrans具有高达96.4%的杀消化道线虫率,杀虫效果优于Monacrosporiumthaumasium和Arthrobotrysrobusta[17]。对D.flagrans基因组分析表明,该菌比其它真菌含有更多与线虫致病性相关的几丁质酶编码基因[18],其生产的粗酶提取物对消化道线虫具有较强的杀灭活性[19-20]。但目前对于D.flagrans几丁质酶的分子特征、功能及其亲缘关系的研究较少。因此,开展D.flagrans几丁质酶的结构、功能及其亲缘关系研究具有重要的现实意义。【拟解决的关键问题】近年来,D.flagrans全基因组的测序成功为研究几丁质酶在捕食线虫性真菌中的作用奠定基础。本研究以D.flagrans为研究对象,克隆CTS3和CTS4几丁质酶基因,利用生物信息学分析其编码蛋白的性质和功能等,并对其与不同真菌来源的几丁质酶氨基酸序列进行进化分析,为D.flagrans几丁质酶在生物防控中的进一步研发利用奠定基础。
1 材料与方法
1.1 菌株、培养基及试剂
捕食线虫性真菌D.flagrans和EscherichiacoliDH5α菌株由石河子大学动物科技学院寄生虫实验室保存,2×TaqPCR Master Mix、2000 bp DNA Marker均购自广州东盛生物科技有限公司,质粒DNA小量制备试剂盒和琼脂糖凝胶电泳回收试剂盒购自诺维森(北京)生物科技有限公司, pMD 19-T载体购自宝生物工程(大连)有限公司。YPSSA (Yeast-phosphate-sulfate-solublestarch-agar)培养基:4 g 酵母提取物,1 g K2HPO4·3H2O, 0.5 g MgSO4,20 g可溶性淀粉,20 g 琼脂,加蒸馏水至1000 mL,经121 ℃、30 min条件下高压灭菌后倾注于灭菌玻璃培养皿,4 ℃保存备用。
1.2 引物设计与合成
根据GenBank发布的D.flagrans全基因组序列(GenBank登录号: SAEB01000001.1),采用Primer 5.0 引物设计软件设计D.flagrans几丁质酶CTS3/CTS4编码序列的特异性引物分别为CTS3-F(5′-TATCGGATCCATGCGGCTGTCTAGTGC-3′)和CTS3-R(5′-GACGGAGCTCTTAACGACGCGGCCTGTA-3′)、CTS4-F(5′-TATCGGATCCATGTTGTTCAAATACATC GC-3′)和CTS4-R(5′-GACGGAGCTCCTAGTTGGACAACAAGCC-3′)。引物由北京六合华大基因有限公司合成。
1.3 D.flagrans 几丁质酶CTS3/CTS4基因的PCR扩增和克隆
将石河子大学动物科技学院寄生虫实验室保存的D.flagrans菌株接种于YPSSA培养基,在28 ℃霉菌培养箱培养1周。取适量菌丝按照真菌基因组DNA提取试剂盒(Solabio)的操作步骤提取D.flagrans全基因组DNA,再以此为模版进行几丁质酶CTS3/CTS4基因扩增。采用20 μL PCR 扩增反应体系:加样量为超纯水9 μL,PCR Mixture 8 μL,上下游引物各0.5 μL, cDNA 模板2 μL,进行 PCR 扩增。反应条件:97 ℃ 5 min;94 ℃ 50 s,54 ℃ 40 s,72 ℃ 120 s,共35个循环;72 ℃ 10 min。PCR扩增产物经琼脂糖凝胶电泳分离,切胶回收。将回收的目的基因片段与pMD 19-T 载体4 ℃过夜连接后转入E.coliDH5α感受态细胞,经氨苄平板筛选后挑取单菌落扩增培养,提取质粒,经琼脂糖凝胶电泳观察结果。将正确的质粒送北京六合华大基因科技股份有限公司进行序列测定。
1.4 D.flagrans 几丁质酶CTS3/CTS4基因及其编码蛋白分子特征分析
据NCBI数据库中其他真菌编码CTS3/CTS4的基因序列推测相应氨基酸序列,在Expasy生物软件利用 Protparam工具预测分析蛋白质的亲水性/疏水性、分子质量、理论等电点等;通过SignalP 5.1服务器预测分析蛋白质信号肽;利用NCBI数据库的CDD功能及SMART数据库联合分析几丁质酶基因编码蛋白催化结构域、氨基酸残基活性位点;使用GOR4分析几丁质酶二级结构,经SWISS-MODEL模拟几丁质酶的空间结构。
1.5 D.flagrans 几丁质酶CTS3/CTS4系统发育树分析
使用MEGA 5.0 软件,对D.flagrans几丁质酶 CTS3/CTS4和其他已报道的真菌几丁质酶序列进行多重比较(ClustalW,默认参数),并采用NJ法(Neighbor-joining, boot-strap 1000)构建系统发育树。
1.6 D.flagrans 几丁质酶CTS3/CTS4序列同源性分析
采用DNAMAN 生物学软件及在线工具(https://www.ebi.ac.uk/Tools/msa/clustalo)进行真菌几丁质酶氨基酸序列比对,获得序列同源性矩阵,比较各序列间的同源性。
1.7 D.flagrans 几丁质酶CTS3/CTS4基于序列比对的保守区和特征位点分析
依据系统发育树和序列相似性分析结果,从已报道的真菌几丁质酶序列中选出适量序列,与D.flagrans几丁质酶CTS3/CTS4进行多重序列比对,并使用GeneDoc软件将比对结果进行展示。结合比对结果,分析D.flagrans几丁质酶CTS3/CTS4的保守区和特征位点。
2 结果与分析
2.1 D.flagrans 几丁质酶CTS3/CTS4基因的克隆
PCR扩增显示,D.flagrans几丁质酶CTS3、CTS4基因扩增目的PCR产物的分子量分别为2896和1469 bp,与预期大小完全相符(图1)。PCR产物成功克隆后,得到阳性克隆。测序结果表明,扩增得到的DNA片段为CTS3和CTS4基因。
2.2 D.flagrans几丁质酶CTS3/CTS4理化特征
CTS3基因CDS全长2565 bp,编码的蛋白质由854个氨基酸残基组成。经预测,该蛋白质分子式为C3883H6008N1022O1386S26,相对分子量为90.02 kDa,理论等电点为4.59。CTS4基因CDS全长1218 bp,编码的蛋白质由405个氨基酸残基组成,预测蛋白分子式为C2009H3100N532O590S13,相对分子量为44.56 kDa,理论等电点为8.69(表1)。
2.3 D.flagrans几丁质酶CTS3/CTS4信号肽分析
SignalIP软件预测分析显示,2个几丁质酶均属于分泌型蛋白,CTS3几丁质酶N端含有信号肽序列,位于1~25氨基酸(MRLSSAFNLLATGLLTLLSSWAVEA);CTS4几丁质酶N端也存在信号肽,位于1~18氨基酸(MLFKYIASLAVLASSAVA)。表明其在蛋白合成后,转运至细胞外参与代谢活动。
2.4 D.flagrans几丁质酶CTS3/CTS4高级结构预测分析
几丁质酶CTS3和CTS4均以无规则卷曲(Random coil)、α-螺旋(α-helix)和 β-折叠 (β-turn)为主要结构单元,分别占55.09%、15.09%、6.90%和46.91%、30.62%、6.91%。SWISS-MODEL软件预测结果(图2)显示,几丁质酶CTS3和CTS4与烟曲霉(Aspergillusfumigatus)的相应几丁质酶三级结构(2a3c.1.A)和 (2y8v.1.A)相似,具有(α/β)8的TIM桶形结构, 中心部分是平行的β-折叠组成的内桶,依次为β1~β8,由α-螺旋将其逐个连接起来,外桶由α1~α8组成,内外桶紧贴。在糖苷水解酶18家族中,底物结合区域位于保守序列β3和β4链形成的环状裂隙。

M: DNA分子量标准(DNAmarker DL5000) ; 1~2: CTS3基因PCR扩增产物; 3~4: CTS4基因PCR扩增产物。M: DNA marker; 1-2:PCR products of CTS3 gene; 3-4: PCR products of CTS4 gene.图1 D.flagrans几丁质酶 CTS3和CTS4基因的PCR扩增结果Fig.1 Amplification products of chitinases CTS3 and CTS4 genes of D.flagrans by plasmid PCR

表1 CTS3和CTS4几丁质酶的理化性质
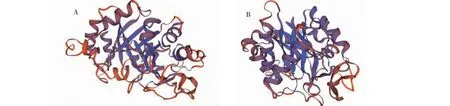
图2 D.flagrans几丁质酶CTS3(A) 和CTS4(B)三级结构预测Fig.2 The predicted tertiary structure of chitinases CTS3 and CTS4 of D.flagrans

图3 CTS3、CTS4与其他几丁质酶的系统发育树Fig.3 Phylogenetic tree between CTS3, CTS4 and other chitinases

图4 D.flagrans CTS3和CTS4与其他几种捕食线虫性真菌的几丁质酶同源性分析Fig.4 The analysis of chitinases homology between CTS3,CTS4 of D.flagrans and other nematode predating fungi
2.5 D.flagrans几丁质酶CTS3/CTS4的系统发育树分析及序列同源性分析
将D.flagrans几丁质酶CTS3和CTS4与18个分别来源于真菌的几丁质酶进行多重序列比对,构建系统发育树并分析其同源性。从图3可看出,CTS3、CTS4均与来源于Arthrobotrysflagrans的几丁质酶亲缘关系更近。从序列同源性分析结果可知,CTS3和CTS4分别与来源于A.flagrans(RVD90058)、A.flagrans(RVD86427)的几丁质酶同源性最高,达到83.8%(图4)。次之是来源于Thermomyceslanuginosus的几丁质酶,同源性为83.2%。
2.6 D.flagrans几丁质酶CTS3/CTS4功能结构域分析
利用SMART软件对CTS3/CTS4几丁质酶序列分析发现,2个几丁质酶均含糖苷水解酶18家族结构域(Glyco-hydro-18),属于糖苷水解酶18家族蛋白。几丁质酶CTS3氨基酸序列的第32~311位为糖苷水解酶18家族结构域;几丁质酶 CTS4氨基酸序列的第34~342位为糖苷水解酶18家族结构域。使用NCBI数据库的CDD功能进行保守功能域分析,几丁质酶CTS3存在1个 LysM受体结合域(图5)。此外,CTS3 第 248~251位的SIGG为底物结合位点,第163~170位的DGFDLDLE为水解酶催化活性位点;CTS4第212~215 位 的SIGG为底物结合位点,第162~169位的 DGFDLDFE为水解酶催化活性位点(图6)。
3 讨 论
D.flagrans被誉为最有前途的捕食线虫性真菌,其在捕食线虫过程中分泌的侵蚀性胞外水解酶,尤其是几丁质酶在杀死线虫幼虫及阻止其卵的孵化方面发挥重要作用,并且该菌产生的厚垣孢子经家畜消化道仍可存活[23],其特性在生防控制家畜胃肠道线虫病害方面具有很大潜力。几丁质是线虫表皮及其卵壳的主要成分,捕食线虫性真菌侵袭线虫要先降解线虫表皮。自1994年从少孢节丛孢菌(Arthrobotrysoligospora)中纯化丝氨酸蛋白酶PII并证明其具有降解线虫角质层作用[24]以来,已经陆续从包括D.flagrans在内的其他捕食线虫性真菌中成功纯化、鉴定出同样具有杀线虫活性的胞外水解酶[25-28]。目前尚未对D.flagrans中发现的几丁质酶进行深入研究。本研究对D.flagrans中2个重要的几丁质酶基因进行分子特征与亲缘关系分析,将为研发新型家畜消化道线虫病生防制剂提供理论依据。
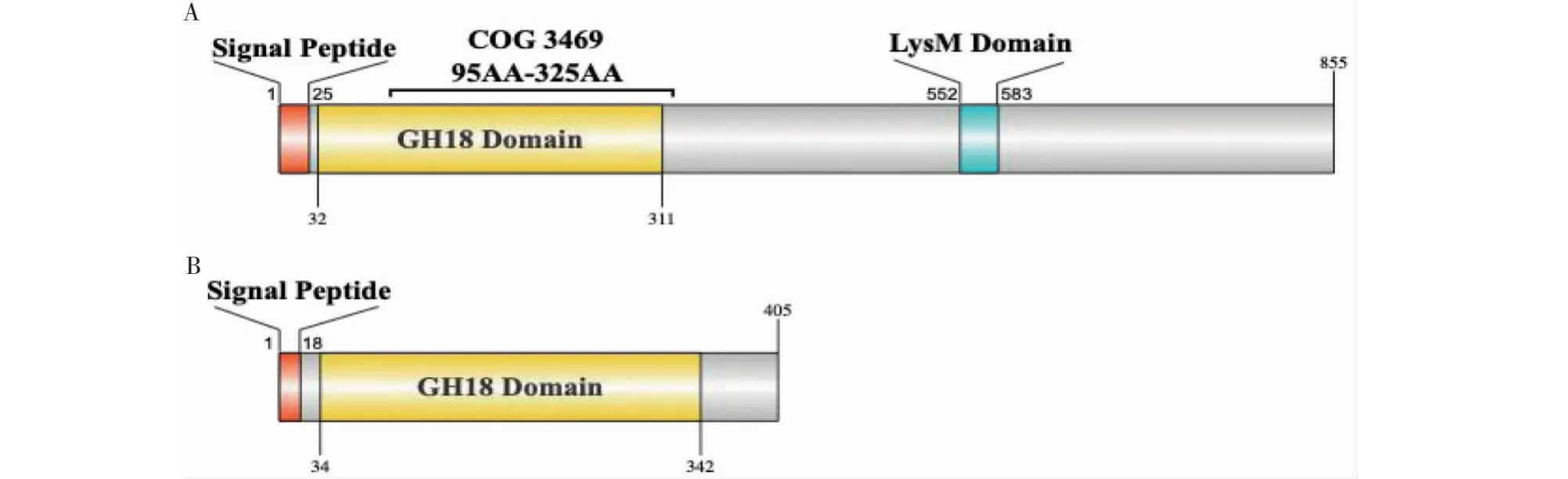
图5 D.flagrans 几丁质酶CTS3(A)和CTS4(B)的功能结构域分析Fig.5 Analysis of the conserved domains in chitinases CTS3(A) and CTS4(B) of D.flagrans

黑色部分为高度同源(100%)的区域,中间空白部分为几丁质酶间的可变区域,***表示糖苷水解酶18家族保守的催化结构区域和几丁质结合结构域。Areas shaded in black are high degree homology (100%) and unshaded areas are regions of variability between the chitinases.*** indicates the conserved catalytic domains of glycosyl hydrolase, family 18 and chitin-binding domains.图6 D.flagrans与其他不同真菌来源几丁质酶氨基酸序列比对Fig.6 Alignment of amino acid sequences of D.flagrans and other chitinases from different fungal sources
本研究对D.flagrans几丁质酶CTS3和CTS4基因序列分析显示,D.flagrans几丁质酶CTS3/CTS4基因CDS分别编码由854、405个氨基酸残基组成的蛋白质。D.flagrans几丁质酶CTS3和CTS4的氨基酸序列从N端起始依次含有信号肽(Signal peptide)、底物结合位点(CBD)和几丁质结合域(ChBD)[29]。2个几丁质酶均为含有信号肽的分泌型蛋白,信号肽氨基酸序列分别在其N端的第1~25和第1~18位。分子特征分析显示,几丁质酶CTS3和CTS4含有几丁质酶催化结构域的2个高度保守序列SIGG和DGFDLDXE及1个典型的(α/β)8桶状结构域,属于糖苷水解酶18家族(GH18)。分子结构分析表明,几丁质酶CTS3和CTS4三级结构具有(α/β)8的TIM桶形结构, 中心部分为平行的β-折叠组成的内桶,外桶由α1~α8组成;底物结合区域位于保守序列β3和β4链形成的环状裂隙。系统发育分析结果显示,D.flagrans几丁质酶CTS3和CTS4与昆虫致病菌几丁质酶的遗传距离较近,说明它们的进化关系更接近,推测它们在殖入宿主的过程中发挥着类似功能[30-31]。D.flagrans产生的几丁质酶CTS3则与亲缘关系较近的来源于Arthrobotrysflagrans(RVD90058)的几丁质酶单独形成一个小分支,提示CTS3几丁质酶与CTS4在遗传进化上存在一定的差异。
4 结 论
Duddingtoniaflagrans几丁质酶CTS3和CTS4均含有信号肽,均含有催化域保守基序 SIGG和DGXDXDWE。分子结构分析表明,几丁质酶CTS3和CTS4三级结构具有(α/β)8的TIM桶形结构, 中心部分为平行的β-折叠组成的内桶,外桶由α1~α8组成;底物结合区域位于保守序列β3和β4链形成的环状裂隙。
作为一种在家畜寄生虫病生物防治方面具有广阔应用前景的捕食线虫性真菌,D.flagrans几丁质酶在侵染线虫过程中发挥了重要作用,是一种极具应用潜力的生物防控制剂研发对象,然而有关捕食线虫性真菌几丁质酶的进化及生物学功能研究相对较少。因此开展捕食线虫性真菌几丁质酶基因的克隆、表达及生物学功能等相关基础研究,对于开发新型、高效的家畜消化道线虫病生物防控制剂具有重要的意义。

