甘松属资源遗传多样性及其ITS 二级结构系统发育信息分析
崔 琪 ,俸明康 ,丰日落 ,王 涛 ,张绍山 ,李 莹 ,李文兵 ,陈 晨 *,刘 圆
1.西南民族大学药学院,四川 成都 610041
2.西南民族大学青藏高原研究院,四川 成都 610225
3.四川省羌彝药用资源保护与利用技术工程实验室,四川 成都 610225
4.青藏高原民族药用资源保护与利用国家民委重点实验室,四川 成都 610225
甘松NardostachyschinensisBat,又名甘松香、香松,作为药用始载于《本草拾遗》[1]。甘松以干燥根及根茎入药,因富含甘松新酮、甘松新酮二醇、甘松香酮A、甘松香酮B、当归香素、蒙花苷[2-3]等成分,具有理气解郁、祛湿消肿[4]的功效,具有重要的药用价值。另外,甘松作为藏香、香水[5]、化妆品[6]等产品的重要成分,亦具有重要的工业价值。甘松的市场需求增大,但其分布范围小,生长周期长,对生长环境要求严苛,无差别和非系统性采集使其野生资源遭到严重破坏,野生资源日益减少,以致于被纳入《濒危动植物国际贸易公约》(CTES)附录Ⅱ[7]。因此,为满足甘松市场需求,可持续利用该资源,展开种质资源收集、评价、开发、利用及新品种选育等工作至关重要。
早期研究认为,败酱科(Valerianaceae)甘松属NardostachysL.植物共有3种,分别为甘松N.chinensisBet.、匙叶甘松N.jatamansiDC.和大花甘松N.grandifloraDC.,三者均分布于喜马拉雅山区[8]。在我国主要分布的为甘松和匙叶甘松2 种,位于青海、四川、云南、西藏和甘肃等省份地区[1];大花甘松N.grandifloraDC.,多分布于印度、尼泊尔等地[9]。三者均为青藏高原生态系统的组成部分。目前大多数研究认为,甘松、匙叶甘松、大花甘松是同种植物,统称为“甘松”。由此可见,甘松属植物如何分类,目前并未有确切依据。
真核生物核糖体 DNA 内转录间隔区序列(internal transcribed spacer,下简称ITS)作为国际公认的植物DNA 条形码候选序列[10],序列十分保守,但在近缘种之间存在明显差异[11],广泛运用于中药材真伪[12]、近缘种亲缘性鉴定[13]、基原鉴定[14]、遗传多样性[15]等方面,相比其他标记手段具有进化速率快、扩增效率高、灵敏准确等有点,已成为主流的核基因分子标记之一[16-17]。本研究通过调查国内甘松属资源的分布,从四川、青海、甘肃、西藏的野生甘松资源分布区收集甘松属资源20份,利用ITS DNA 条形码序列,对青藏高原甘松属植物资源展开遗传多样性研究,为甘松属分类、资源生态适应机制及种质筛选等的研究提供理论指导,同时为野生甘松属植物资源的保护选育提供科学依据。
1 材料与方法
1.1 试验材料
2020 年6 月于四川省阿坝藏族羌族自治州、四川省甘孜藏族自治州、青海省黄南藏族自治州、青海省果洛藏族自治州、甘肃省甘南藏族自治州、西藏自治区日喀则市采集甘松属资源样本20 份,移栽至西南民族大学青藏高原基地。经西南民族大学刘圆教授鉴定,四川甘孜藏族自治州的4 份样本为匙叶甘松N.jatamansiDC.,其余地区样本均为甘松N.chinensisBat,鉴定结果[8]及样本来源见表1。

表1 甘松属样本采集信息Table 1 Samples information of Nardostachys
1.2 方法
1.2.1 试验样地概况 于西南民族大学青藏高原基地甘松资源圃内(102°35′00″E,32°50′07″N)展开试验,试验地海拔3 490 m。2020 年年平均温度2.9 ℃,年降水量860.8 mm,年日照时数2 212.3 h,气候干燥寒冷。
1.2.2 样品DNA 的提取、PCR 扩增及测序 待植株长出新叶,分别取长势一致的叶片,首先用无菌水洗净,然后滤纸吸干后剪碎加液氮研磨,采用CTAB 法提取样品DNA。提取后对样品的DNA 进行纯度与浓度的检测。ITS1、ITS2 序列扩增所用引物及PCR 反应条件见表2。所用PCR 反应试剂为北京擎科生物科技有限公司所生产的I-5TM2×High-Fidelity Master Mix,将1.3%琼脂糖凝胶电泳单一、清晰、明亮的扩增产物送至北京擎科生物科技有限公司进行测序。
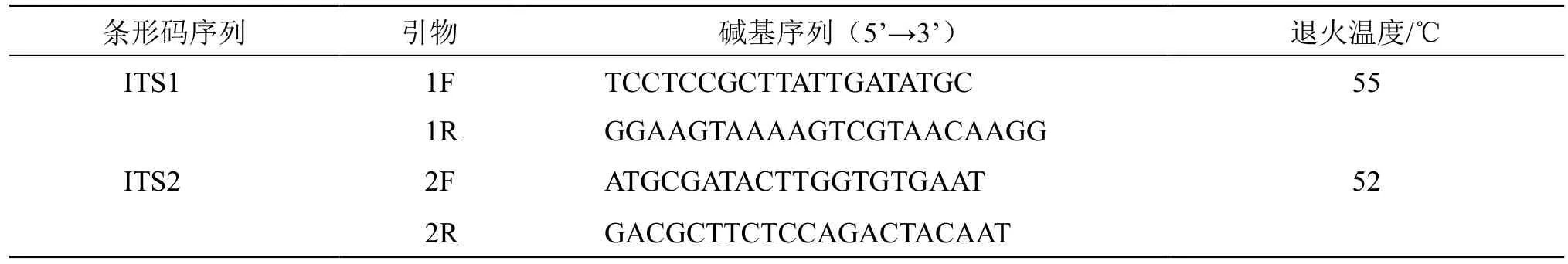
表2 PCR 扩增与扩增程序Table 2 PCR amplification and amplification procedures
经检测,所有样本提取的DNA 在260、280 nm 的吸光度比值A260/A280在1.8~2.0,A260/A230在1.8~2.3,所有样本DNA 纯度达到检测要求。提取的DNA 质量浓度在34.9~90 ng/μL,且纯度均达到要求,见图1。

图1 ITS1 (A) 和ITS2 (B) 扩增产物的琼脂糖凝胶检测电泳图Fig.1 Agarose gel electrophoresis of amplified products in ITS1 (A) and ITS2 (B) sequences
2 数据处理
测序结果用DNA EditSeq 软件进行剪切、拼接和统计等分析,并用MEGA7.0 软件进行多重序列比对并用邻接法(neighbor-joining,NJ)构建系统发育树,并计算遗传距离,利用DNASP ver.5.0 软件对 各居群的遗传多样性参数进行计算,并估算群体间的遗传分化系数(genetic differentiation coefficient,Gst),并将不同单倍型序列用网络在线版Mfold 进行二级结构分析。
3 结果与分析
3.1 ITS 序列G/C 含量与长度多态性分析
分析所有材料的ITS1 和ITS2 序列G+C 含量发现,ITS1 G+C 含量为67.92%~68.46%,ITS2 G+C 含量为66.88%~67.09%,G+C 含量普遍较高,都在66%以上,最高达68.46%(S18)。比较各ITS1和ITS2 序列,可见其长度变化不大,ITS1 序列长度范围为687~689 bp,仅2 bp 差异,ITS2 序列长度比较一致为468 bp。因此,从序列G+C 含量和长度多态性来看,甘松属资源ITS1 的变异较ITS2的序列变异大,但二者种内变异均较小,见表3。

表3 20 个甘松属样本ITS1 和ITS2 序列长度及G+C 值Table 3 ITS1 and ITS2 sequences length and G + C content in 20 samples of Nardostachys
3.2 序列特征及遗传距离分析
利用MEGA 软件对20 个甘松属样本ITS1 和ITS2 序列进行对比分析,结果见表4。ITS1 序列比对长度为689 bp,变异位点为15 个,占该区位点的21.8%,简约信息位点11 个,其中1 个位点发生G/C 或A/T 碱基的颠换、10 个位点发生GC/AT 碱基的转换,简约信息位点为变异较大的位点。如表5 所示,ITS2 序列比对长度为468 bp,变异位点为7 个,占15.0%,简约信息位点6 个,其中1 个位点发生G/C或A/T碱基的颠换、5个位点发生GC/AT碱基的转换。

表4 ITS1 简约信息位点Table 4 Parsimony informative site of ITS1

表5 ITS2 简约信息位点Table 5 Parsimony informative site of ITS2
序列变异结果分析表明,ITS1 序列的变异位点较ITS2 序列变异位点多,甘松与匙叶甘松的ITS1和ITS2 序列差异较大,差异的碱基及位点的变化呈现出一定的规律性,即在特定的位点变异,且变异的碱基大多相同;不同产地的甘松之间、不同产地的匙叶甘松之间变异均较小。
基于K2P 模型对实验材料进行遗传距离分析,结果(表6)表明,甘松种内平均遗传距离为0.001 7,匙叶甘松种内平均遗传距离为0.005 2,种间平均遗传距离为0.005 3,种内平均遗传距离小于种间平均遗传距离。

表6 甘松属2 种药用植物的遗传距离Table 6 Genetic distances among two medicinal plants in Nardostachys
3.3 聚类与同源性分析
构建的系统发育树(图2)可看出,20 个甘松属样本可分为2 大类,其中各个地区的甘松(G1~G16)聚为一组,匙叶甘松(S17~S20)聚为另一组。上述结果与形态学分类中归为2 个不同的种的分类结果一致,说明ITS 序列可用于鉴别甘松与匙叶甘松。此外,本研究所用甘松样品可分为2 个亚类,其中第一个亚类包括14 个种质资源,3 个来自四川、9 个来自青海、2 个来自甘肃;第2 个亚类包括2 个种质资源均来自西藏。ITS 聚类结果与种质的纬度空间分布具有一定的相关性。可见,ITS 分子标记在一定程度上反映了甘松属种质的地理分布特点。

图2 基于ITS 序列构建甘松属样本NJ 聚类树Fig.2 NJ Phylogenetic trees of Nardostachys based on ITS sequences
本研究进一步利用DNASTAR 软件对甘松属20 个样品的ITS 序列间的同源性和差异性进行分析,结果如图3 所示,物种水平上看,甘松(G1~G16)ITS 序列的同源性在99.0%~100%,差异性在0~0.8%,其中G1~G14 的ITS 序列完全一致,说明若尔盖县、河南蒙古自治县、久治县、碌曲县和玛曲县几个地区的甘松没有因为地理变化而发生碱基上的突变,而G15、G16 与G1~G14 的同源性在99.0%~99.3%,差异性在0.7%~0.8%,说明东亚县、吉隆县几个地区与其余地区的甘松相比,碱基序列有较小变化,可能是由于地理变化引起的。匙叶甘松(S17~S20)同源性在99.0%~99.8%,差异性在0.2%~1.0%,说明不同地区的匙叶甘松碱基序列有一定差异。从属的水平看,甘松与匙叶甘松的同源性在98.5%~99.8%,差异性在0.7%~1.3%,甘松与匙叶甘松ITS 碱基序列有较小差异,表明二者亲缘关系较近。

图3 20 个甘松属样本ITS 序列的同源性及差异性Fig.3 Homology and difference of ITS sequences among 20 samples of Nardostachys
3.4 ITS 序列二级结构分析
在甘松属的2 个品种的ITS 基因中,分别选择遗传距离最远的2 个序列,ITS1 序列选择甘松G01和G16、匙叶甘松S17 和S20,ITS2 序列选择甘松G01和G16、匙叶甘松S17和S18,采用RNAfold web server 在线版绘制其二级结构,见图4。甘松与匙叶甘松ITS1 序列二级结构构象、茎环及内部环的结构均有明显差异,而ITS2 序列二级结构差异较小,这与ITS1 和ITS2 序列的碱基变异程度不同有关,结果与序列特征分析一致。

图4 基于ITS 序列构建的rRNA 二级结构Fig.4 rRNA secondary structure based on ITS sequences
3.5 甘松与匙叶甘松遗传多样性比较
利用DNAsp 软件对甘松属资源进行遗传多样性进行分析,分离点位数(number of segregating sites,S)、单倍型数量(number of haplotypes,Hh)、单倍型多样性(haplotypes diversity,Hd)、平均核苷酸差异(average number of nucleotide differences,k)和核苷酸多样性(nucleotide diversity,π)分别进行计算,结果如表7 所示,甘松种群分离位点数为9,共3 个单倍型,单倍型多样性为0.242,平均核苷酸差异为1.992 和核苷酸多样性为0.001 72;匙叶甘松分离位点数为11,共4 个单倍型,单倍型多样性为1.000,平均核苷酸差异为6.000 和核苷酸多样性为0.005 19。综合遗传多样性水平指标也显示,甘松的遗传多样性小于匙叶甘松(图5)。
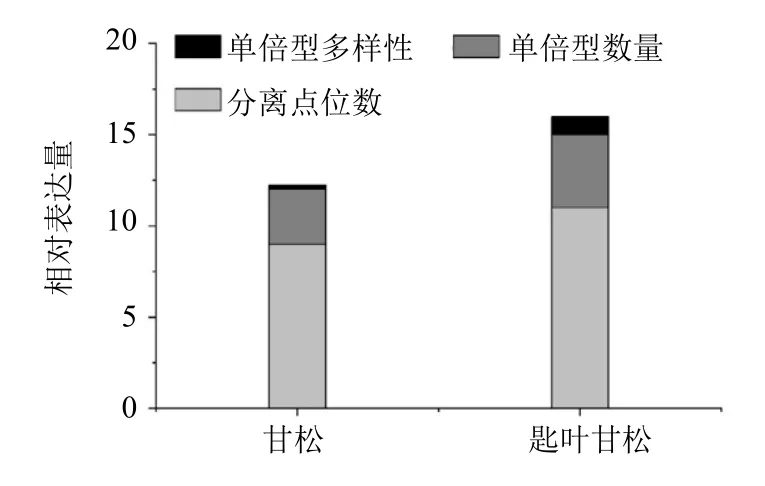
图5 甘松与匙叶甘松的遗传多样性比较Fig.5 Comparison of genetic diversity between N.chinensis and N.jatamansi

表7 甘松与匙叶甘松的遗传多样性参数统计Table 7 Statistics of genetic diversity parameters of N.chinensis and N.jatamansi
4 讨论
4.1 形态学
甘松为重要的中药材及民族药材,基原准确是保证药材质量的基础及关键。早期研究认为世界甘松属资源分为甘松、匙叶甘松和大花甘松3 种,其中,我国产甘松和匙叶甘松2种,但它们现在被Flora of China(《中国植物志》英文版)认为是同一种[18]。因此,对我国甘松属资源进行鉴定,认为我国甘松属从形态学上应为甘松和匙叶甘松2 种,二者有以下显著差异:(1)甘松叶鞘残基呈片状,而匙叶甘松呈纤维状,(2)甘松叶钝形,呈倒披针状,而匙叶甘松则叶尖尖形,呈长匙状,(3)甘松果实光秃,匙叶甘松果实被毛,(4)甘松花后主轴和侧轴明显伸长,而匙叶甘松并无明显伸长,(5)甘松花冠内光滑无毛,匙叶甘松则密布白毛。以上结论与耿晓萍等[19]的研究结果一致。
4.2 ITS 分子标记鉴定甘松属资源的适用性
ITS 序列因受到的选择压力小,具有较高的遗传变异,被视为中草药及其近缘种的鉴定、药材真伪的鉴定、物种遗传多样性最常用的DNA 条形码技术[20]。缘毛紫菀Astersouliei及其近缘物种[21]、鸡血藤Spatholbicaulis及其混伪品的鉴定及雷公藤属Tripterygium的遗传多样性[22-23]均成功利用ITS 序列进行鉴定。在甘松属中,金乾等[24]利用rbcL 序列和ITS2 序列均无法鉴定甘松资源。本研究将ITS2 和ITS1 结合,利用MEGA 软件对ITS序列进行系统发育分析,结果表明,甘松与匙叶甘松各自聚为一类,且与形态学鉴定结果一致,表明ITS 可用于甘松属资源的划分与基原鉴定,且我国甘松属应分为甘松和匙叶甘松2 种。
4.3 甘松属资源的遗传多样性与地理分布
种质资源多样性的核心是遗传多样性,资源多样性为新品种选育提供重要基础[25]。甘松种质资源的多样性分析研究目前主要集中在形态遗传标记及生物化学遗传标记,偶见分子遗传标记研究。耿晓萍[19]对甘松的形态学进行观测与分析,研究发现甘松与匙叶甘松表型差异明显,同时利用HPLC 指纹图谱分析甘松属资源的化学成分,从生物化学方面对甘松资源鉴定提供依据。上文也提及金乾[24]利用rbcL 序列对甘松属资源进行了鉴定。本研究发现形态学鉴定与ITS 序列系统发育分析,均能对甘松属资源进行有效区分。
植物的遗传多样性与其地理分布范围存在着密切联系,自然分布广泛的物种往往具有更高的遗传多样性。甘松与匙叶甘松分布位置分析发现,二者分布在喜马拉雅山脉至横断山脉区域⎯青藏高原东南边缘地带。该地区是我国特有植物种类最丰富的地区,同时也是现代北温带和高山植物区系的重要分化和起源中心,具有植物种类多样化的特点[26]。地理谱系研究的两种高原植物分布演化模式均表明,青藏高原植物群体在冰期后向边缘演化。甘松属植物分布演化可能遵循此规律从而多分布在青藏高原的东南边缘地区,分布面狭窄。本研究发现甘松属资源遗传多样性较其他物种低,这可能是造成其分布面狭窄的原因之一。同时,本研究发现,甘松遗传多样性低于匙叶甘松,但走访调查发现甘松自然分布范围却大于匙叶甘松。甘松在四川、青海、甘肃、西藏多个地区均有分布,而匙叶甘松仅在四川省甘孜藏族自治州、云南省有分布。分析原因,可能是由于气候类型不同引起的。在长期的演化过程中,基因和气候之间形成了稳定的相互作用关系。气候变化必然影响植物的遗传物质,从而导致植物的遗传多样性发生改变。甘松分布区主要为高原高山气候,其遗传物质相对稳定。而匙叶甘松分布区既有高原高山气候还受亚热带季风气候影响,使得其为适应环境发生遗传变异[27]。马丽娟等[28]研究发现,东北野生杏遗传多样性受气候环境影响发生遗传变异。李明等[29]研究发现不同气候环境显著影响油松PinustabulaeformisCarr.居群的遗传多样性。在后续工作中,本课题组将继续走访调查甘松属资源分布区,进一步研究验证该推论。
本研究结合形态学特征分析,利用ITS 条形码技术,研究了中国主要野生甘松分布区甘松属资源的遗传多样性,并对其分类进行分析,结果表明青藏高原分布区采集的甘松属植物资源分为甘松和匙叶甘松2 种,为甘松属资源的分类提供分子层面的支撑。
利益冲突所有作者均声明不存在利益冲突

