小麦转录因子基因TaPHR1参与调控每穗小穗数
张逸宁 张艳菲 汪 敏 王景一 李 龙 李超男 杨德龙 毛新国,* 景蕊莲
小麦转录因子基因参与调控每穗小穗数
张逸宁1,2张艳菲2汪 敏2王景一2李 龙2李超男2杨德龙1,*毛新国1,2,*景蕊莲2
1省部共建干旱生境作物学国家重点实验室 / 甘肃农业大学生命科学技术学院, 甘肃兰州 730070;2中国农业科学院作物科学研究所 / 农作物基因资源与基因改良国家重大科学工程 / 农业农村部农作物种质资源创新与利用重点开放实验室, 北京 100081
利用水分高效基因资源创制新型小麦品种是应对气候变化和人口高速增长的有效途径。MYB (v-myb avian myeloblastosis viral oncogene homolog)是植物中最大的转录因子家族之一, 参与调控植物生长发育, 生物和非生物胁迫。本研究在和中分别鉴定出19个和15个SNP, 基于这些多态性位点开发了分子标记。关联分析表明,-4B-I是小穗数多的优异单倍型。通过创制两个回交导入系群体, 进一步证实-4B-I有利于改善小麦穗部性状。的转录表达分析发现-4B-I单倍型幼穗中的表达水平均高于-4B-II单倍型。此外,在水稻中的异源表达导致穗分支变多, 也证实参与调控每穗小穗数。小麦育成品种的时空分布分析发现尽管-4B-II在我国现代育成品种中占比最多, 但随小麦育种时间的推进,-4B-I的占比在逐渐增多。总之,是小麦每穗小穗数的正调节因子。因此, 本研究开发的分子标记可作为小麦标记辅助选择和遗传改良的重要来源。
小麦; MYB转录因子; 农艺性状; KASP标记; 关联分析
小麦()是世界上最重要的谷类作物之一, 占世界粮食总产量的三分之一[1]。在气候变化和人口快速增长的条件下, 小麦的生产面临巨大挑战, 在小麦育种中探索和利用资源高效基因则是解决这一问题的有效途径[2]。小麦的产量由单位面积穗数、每穗粒数和粒重这三要素构成。因此, 小麦的穗部结构是一个关键性状, 并直接决定了其产量, 而且每穗小穗数与每穗粒数密切相关[3-6]。
MYB (v-myb avian myeloblastosis viral oncogene homolog)转录因子是植物中最大的转录因子家族之一。MYB转录因子具有多种生物学功能, 参与调控植物生长发育、初生代谢及次生代谢[7-11]。目前, 已在水稻()[12]、拟南芥()[12]、小麦[13]中分别鉴定了155、197、393个MYB家族的转录因子。研究发现可以调节拟南芥细胞周期及根的生长[14]; MYB转录因子、、、和等多个成员共同调控拟南芥花药的发育和功能[15-16];和过表达促进木质素、纤维素和半纤维素的合成[17-18];不仅参与植物外种皮细胞的分化, 还具有毛状体调控的作用[19]; 拟南芥MYB-relate类转录因子中的()、(C)和()是重要的光周期调节蛋白, 参与调节拟南芥开花[20-21]; 玉米中MYB-relate类转录因子绒毛白蜡smh1端粒结合蛋白, 对维持染色体结构的完整性和调节基因转录有重要的作用[22]。MYB转录因子同样响应干旱、高盐、极端温度、营养缺乏等多种非生物胁迫和多种生物胁迫[23-30]。例如,是一个受干旱、ABA及盐胁迫诱导表达的基因, 它可能通过参与调控表皮的合成及细胞增殖来响应各种逆境胁迫[31-32]。拟南芥中的受到病原菌侵染时可以对细胞的死亡进行调控[33]。
MYB家族MYB-CC亚族PHR (Phosphate Starvation Response)类蛋白是磷信号通路中重要的中心调控因子, 其C端含有MYB结构域和CC结构域(Coiled-Coil domain)。其中, MYB结构域为DNA结合域, CC结构域有助于MYB结构域与顺式作用元件的准确结合。PHR1的N端含有DNA激活结构域。研究发现PHR1以二聚体的形态结合在靶基因的P1BS (PHR1 Binding Sequence)顺式作用元件上, 从而调控下游基因的表达[34-36]。在小麦中的研究发现, 过表达使小麦穗粒数增加从而增加产量[37]; 此外,在水稻中异位表达导致生物量、穗粒数和穗分支的增加[38]。
本研究通过发掘小麦、、基因上的多态性位点, 开发功能分子标记, 通过关联分析解释多态性位点与农艺性状的关系, 发掘优异单倍型, 为小麦分子育种提供分子标记和优异基因资源。
1 材料与方法
1.1 试验材料及培养条件
试验中使用了3个小麦自然群体(NPs)分析农艺性状, 采用主要分布在我国北方冬麦区和黄淮冬麦区[39]的323份材料组成的NP1进行关联分析。NP2主要来自中国小麦迷你核心种质[40], 由157份农家品种组成; NP3主要来源于中国小麦核心种质[40], 由348份现代育成品种组成, 用于鉴定的单倍型及地理分布。
2015—2017年在北京顺义(40°230′N, 116°560′E)和北京昌平(40°130′N, 116°130′E)中国农业科学院作物科学研究所田间试验站种植了NP1。田间水分管理分为雨水灌溉(干旱胁迫, DS)和正常灌溉(WW)两种情况。DS是指在小麦的整个生长季节不给予任何人工灌溉, 完全由雨水浇灌。WW是指在越冬前、拔节期、开花期和灌浆期各阶段浇水750 m3hm–2(75 mm)。此外, 还通过覆盖塑料膜创造高温胁迫环境对开花后1周的小麦行热处理(HS)。2015、2016和2017年生长季的总降水量分别为161、173和127 mm。在所有不同条件下, 每份材料均匀点播在长2.0 m间距0.3 m的4行小区内, 每行播种40粒。收获时在每个小区中随机选取5株测定株高(PH)、穗长(SL)、花序梗长(PLE)、倒二节长(LPN)、每穗小穗数(NCS)、每穗粒数(NGS)和千粒重(TGW)等农艺性状, 获得了16个环境下的小麦表型数据。
利用有显著耐旱性的小麦品种旱选10号进行基因序列的克隆和基因组序列分离。使用水稻品种Kitaake作为转基因实验的受体。选择具有不同表达水平的的3个T3纯合转基因水稻系进行表型分析。
1.2 实时定量PCR检测TaPHR1的表达
为了确定小麦不同单倍型中的表达水平, 将小麦均匀点播在长1 m间距0.15 m的4行小区内, 每行播种20粒。待小麦度过越冬期生长到叶鞘直立期时, 取出发育完整的幼穗, 检测在小麦幼穗中的表达水平。取转基因水稻的叶片检测在转基因水稻中的表达量。
使用RNA Easy Fast Plant Tissue Kit (ThermoFisher Scientific, 中国香港)试剂盒提取穗和叶中的总RNA; 并使用试剂盒中的DNase I消除基因组DNA的潜在污染。根据制造商的说明书, 使用TIANScript cDNA First-Strand Synthesis Kit (Tiangen,中国北京)将RNA (2 μg)合成cDNA的第一链。使用ABI PRISM 7900分析仪(Roche, 瑞士), SYBR Green PCR Master Mix (TaKaRa Biotechnology, 日本)进行实时定量PCR (qRT-PCR)。利用小麦微管蛋白(TaActin)和水稻肌动蛋白(OsTubulin)作内参基因, 标定小麦和水稻中的相对表达水平(表1)。
PCR反应条件包括95℃反应2 min, 然后95℃ 10 s, 60℃ 30 s循环45次, 最后72℃时延长10 min。为了保证试验结果的准确性, 每组试验进行3次重复。使用2–ΔCT计算基因的相对表达量[41]。
1.3 TaPHR1序列多态性分析
使用的cDNA序列进行BLAST (https:// urgi.versailles.inra.fr/blast/blast.php)查询, 获得小麦中国春的基因组序列作为参考。利用小麦基因组变异联合数据库(http://wheat.cau.edu.cn/WheatUnion/)对进行序列多态性分析, 寻找(Gene ID: TraesCS4A02G131700)、(Gene ID: TraesCS4B02G172900)、(Gene ID: TraesCS4D02G175000)的基因组序列及其启动子上的SNP位点。利用聚类分析筛选、、的单倍型。其中本研究中使用的所有基因组DNA均使用CTAB法从小麦中提取[42]。
1.4 标记开发和关联分析
对于高通量基因分型, 根据标准KASP指南(http://www.lgcgenomics.com/)开发KASP引物。对于TaPHR1-SNPA1和TaPHR1-SNPB1, 设计了2个正向引物(带有特异尾部序列)和一个普通反向引物。然后使用KASP标记应用于NP1、NP2、NP3和回交导入系群体, PCR反应体系所用引物由2个18 μL的正向引物(100 mmol L–1)、45 μL的反向引物(100 μmol L–1)和49 μL ddH2O组成。使用384PCR微孔板(LGC, 英国)进行分析, 在3 μL的混合体系中进行反应, 该体系包括20 ng μL–1的模板DNA 1.5 μL, 1.5 μL KASP Mix (LGC Genomic, 384,Standard content ROX, 英国), 0.042 μL引物混合物和1.5 μL ddH2O。PCR反应条件包括3步: 第1步: 94℃ 15 min, 94℃ 20 s, 第2步: 61~55℃10个循环, 每个循环递减0.6℃, 第3步: 94℃ 20 s, 55℃ 60 s, 26个循环, 于高通量水浴锅PCR仪(Hydrocycle 64, LGC Genomic)进行扩增反应。利用Pherastar荧光扫描仪采集原始荧光信号。利用Kraken (V163.16.16288)软件进行基因分型, 使用SNPviwer2软件分析分型结果。
运用TASSEL (version 5.2.51)软件的一般线性模型GLM (General Liner Model), 把群体结构(Q)作为协变量, 将、的基因型和表型性状进行关联分析, 当<0.05时认为该性状具有统计学意义。用SPSS (version 16.0)软件通过独立T检验检测不同基因型/单倍型性状差异的显著性。
1.5 转基因水稻的表型
使用旱选10的cDNA和pfu DNA聚合酶扩增的全长ORF序列, 在引物上游和下游分别添加I和I两个限制性内切酶切割(表1)位点,并在I位点和的起始密码子之间插入30 bp的MYC标签序列, 然后将其克隆到双元表达载体pCAMBIA1391中。将测序正确的质粒转入农杆菌菌株EHA105, 再使用农杆菌介导转化水稻(Kitaake)。通过PCR检测T0代转基因水稻, 并通过测序确定阳性植株。
为了筛选单拷贝转基因株系, 利用0.1%的潮霉素筛选根分离比为3∶1的T2代转基因系。为了鉴定纯系转基因水稻, 将T3代的种子用潮霉素筛选, 正常发芽没有分离的株系为纯合系。并通过qRT- PCR检测转基因植株中靶基因的表达水平。选择3个不同表达水平的T3纯合转基因水稻株系进行表型分析。将转基因水稻和非转基因水稻野生型(WT)种植在中国农业科学院作物科学研究所实验站(北京)的水稻田中(39°48′N, 116°28′E)。将水稻种子于5月下旬或6月初播种在塑料箱中。发芽15 d后, 将幼苗移植到水稻田中。在成熟时测定株高、分蘖数、主穗长、穗分枝数和每穗粒数等农艺性状。使用自动万深考种仪(SC-G, http://www.wseen.com/)测量了包括千粒重、粒长和粒宽在内的籽粒性状。使用SPSS (version 16.0)软件利用Duncan’s multiple range test进行多组样本间差异显著性分析。
1.6 顺式作用元件预测
启动子从起始密码子上游开始, 长度为3000 bp。并使用PlantCARE (http://bioinformatics. psb.ugent.be/webtools/plantcare/html/)预测启动子中的顺式作用元件。
2 结果与分析
2.1 TaPHR1的分子表征
在六倍体普通小麦中克隆了的3种基因组序列, 根据它们的染色体位置, 分别命名为、和。的编码区长度为1473 bp, 编码由490个氨基酸组成的蛋白质,的编码区长度为1344 bp, 编码由447个氨基酸组成的蛋白质,的编码区长度为1356 bp, 编码由451个氨基酸组成的蛋白质。3种基因组序列均包含8个外显子和7个内含子(图1), 所编码的3种蛋白质序列均包含了保守的MYB motif和MYB-CC motif (附图1)。其中与的氨基酸序列相似性为98.4%,与的氨基酸序列相似性为97.5%,与的氨基酸序列相似性为97.8% (附图1)。
2.2 TaPHR1s序列多态性分析和分子标记的开发
利用小麦基因组变异联合数据库(http://wheat. cau.edu.cn/WheatUnion/)对基因组序列和上游启动子序列进行序列多态性分析, 在、、上分别发现了19、15、0个SNP位点。聚类分析发现有2种单倍型,有2种单倍型(图1)。
针对上的SNP位点, 在启动子区2490 bp处开发了一个KASP标记TaPHR1-SNPA1 (表1)以区分两种单倍型, KASP分析的散点图显示了在X-(FAM)和Y-(HEX)轴上的种质聚类。强FAM信号聚集在右下角的蓝点, 强烈的HEX信号聚集在左上角的红点。对于TaPHR1-SNPA1, 蓝点的材料具有等位基因T, 红点的材料具有等位基因C (附图3)。
针对的SNP位点, 在启动子区2171 bp处开发了KASP标记TaPHR1-SNPB1区分两种单倍型。对于TaPHR1-SNPB1, 蓝点的材料具有等位基因C, 红点的材料具有等位基因T (图1-D)。

表1 试验中使用的引物及用途
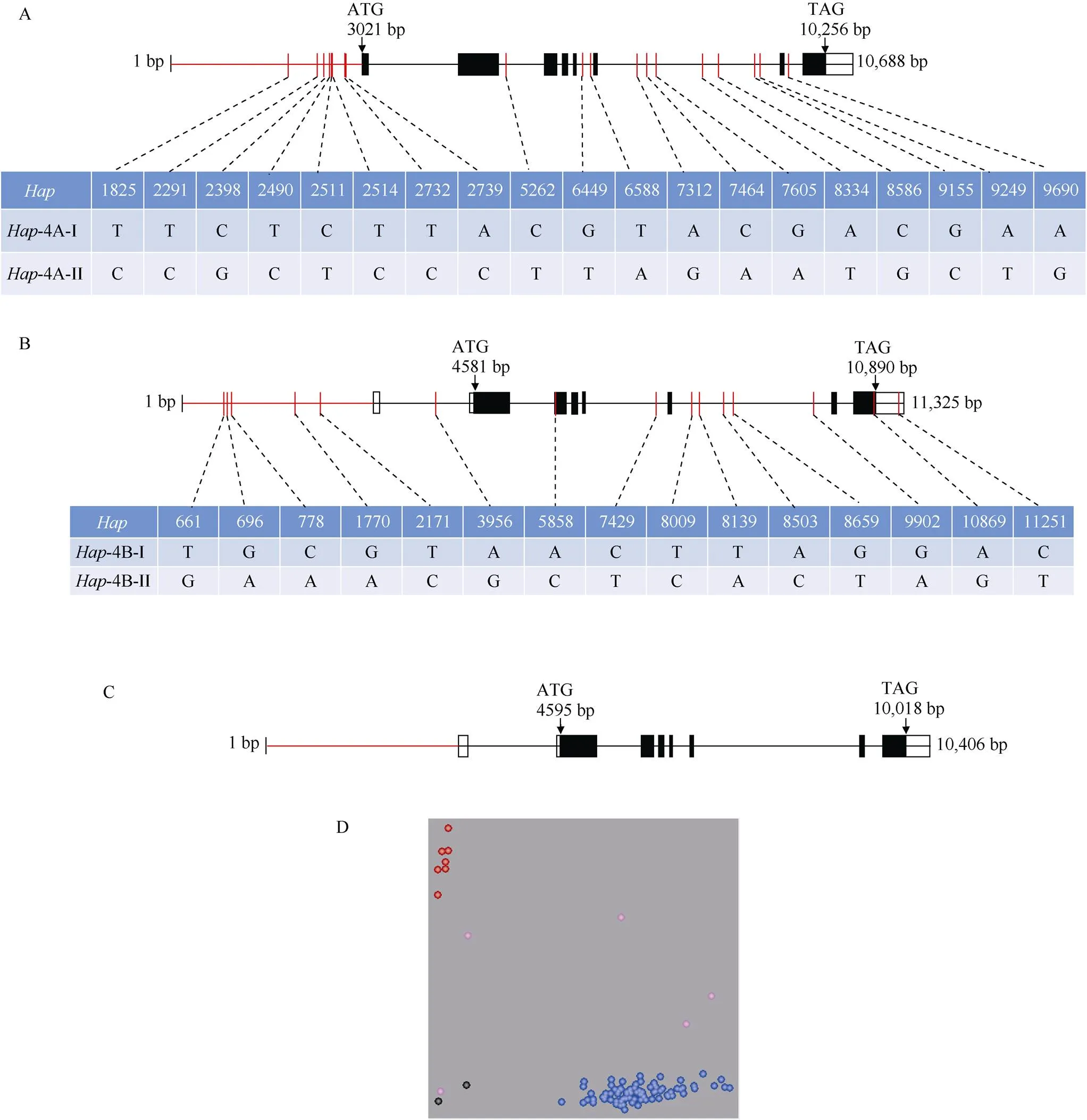
图1 TaPHR1的核苷酸多态性和KASP标记的开发
图A、B、C分别为标注了、和的核苷酸多态性位点; 图D: NP1中SNP-2171 bp的KASP标记(附表1), 其中蓝点代表FAM型等位基因C, 红点代表HEX型等位基因T; 黑点为对照。
Fig. 1-A, B, and C indicate the SNP with,, and, respectively. Fig. 1-D: KASP assays for SNP-2171 bp in the accessions in NP1 (Table S1). The blue circles represent accessions that have the FAM-type allele and the red circles represent the HEX-type allele; the black squares are the non-template control. Allele ‘C’ in FAM and ‘T’ in HEX cluster.
2.3 TaPHR1-4B分子标记与农艺性状的关联分析
在NP1中鉴定了的2种单倍型, 但未与任何农艺性状存在显著关联。在NP1中鉴定了的两种单倍型。其中,-4B-I在NP1中占6%,-4B-II在NP1中占94% (附表1)。表型分析结果显示TaPHR1-SNPB1与每穗小穗数有显著关联(表2)。在16个环境中的13种环境下,-4B-I每穗小穗数(NCS)的平均值显著多于-4B-II (图2), 而在只有旱处理和正常灌溉条件下, 差异依然显著, 说明由多态性位点变异引起的每穗小穗数增加受环境影响较弱。TaPHR1-SNPB1与其他农艺性状, 如粒长、粒宽、粒重、穗粒数等, 均无显著关联。说明主要参与小麦小穗数发育。
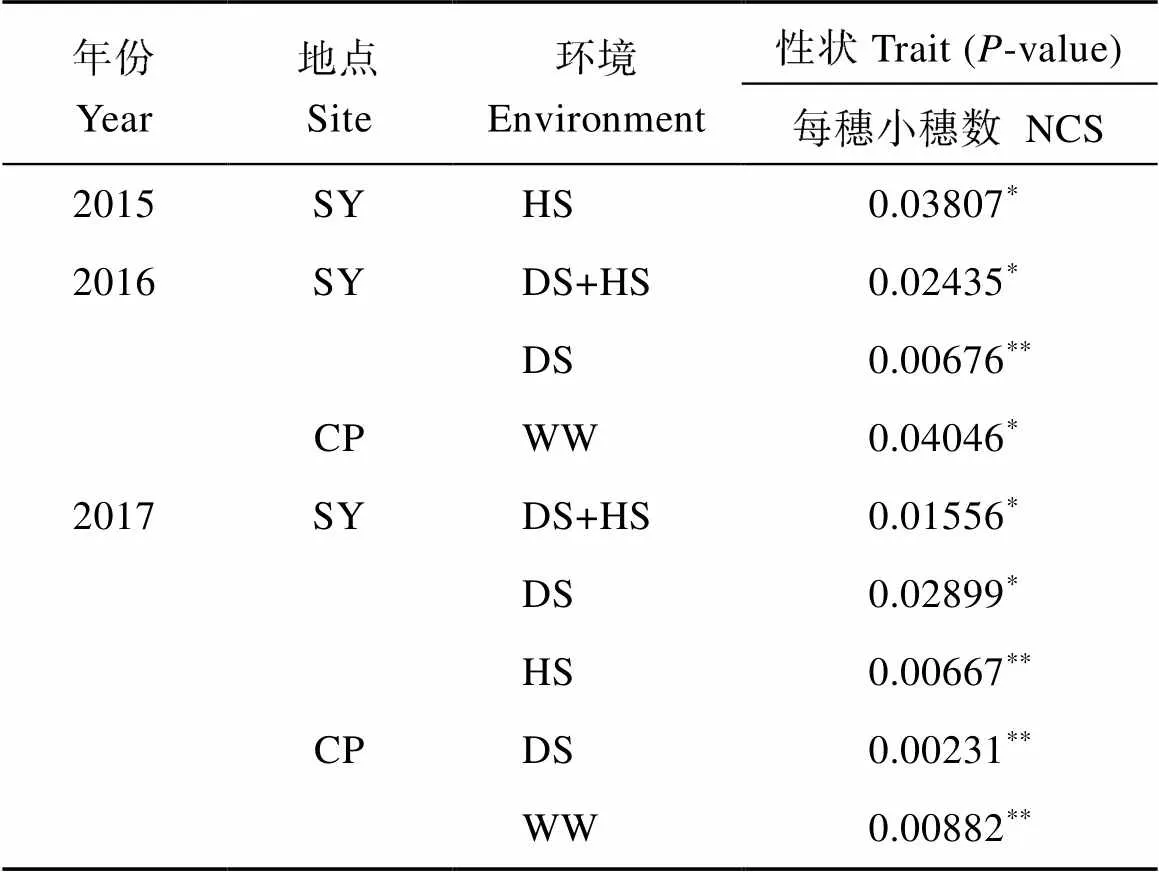
表2 利用一般线性模型分析NP1中TaPHR1-4B单倍型与农艺性状相关性
SY: 顺义; CP: 昌平; DS: 旱处理; HS: 热处理; WW: 正常灌溉; NCS: 每穗小穗数。-value为0.05的概率显著水平;*、**分别表示在0.05、0.01概率水平差异显著。
Environments are Shunyi (SY) and Changping (CP) under well-watered (WW), drought-stressed (DS), heat-stressed (HS) conditions in 2015, 2016, and 2017. NCS: spikelet number per spike. Significant associations were determined using Duncan’s multi-range test in SPSS v.16.0.*:< 0.05;**:< 0.01.
2.4 回交群体材料TaPHR1-4B基因不同单倍型每穗小穗数分析
为进一步验证关联分析的结果, 创制了2个回交导入系群体, 豫麦8-鲁麦14 (BC3F3)和洛阳8628-晋麦47 (BC3F5), 来验证不同单倍型的作用。使用TaPHR1-SNPB1扫描2个回交群体和鉴定亲本单倍型。对于豫麦8-鲁麦14, 其供体亲本豫麦8的基因型为-4B-I, 受体亲本鲁麦14的基因型为-4B-II, 与受体亲本相比, 供体亲本具有更高的NCS (图3-A, B)。在豫麦8-鲁麦14中有4个导入系(L1-L4)被鉴定为-4B-I。对于洛阳8628-晋麦47, 其供体亲本洛阳8628的基因型为-4B-I, 受体亲本晋麦47的基因型为-4B-II, 与受体亲本相比, 供体亲本具有更高的NCS。在洛阳8628-晋麦47中只有一个导入系(L5)被鉴定为-4B-I。亲本和被导入材料的NCS统计结果表明, L1、L2、L3和L4的NCS都显著高于受体亲本鲁麦14, L5的NCS也显著高于受体亲本晋麦47 (图3-C, D)。结果进一步证实参与小麦小穗数发育, 且-4B-I可能为其优异单倍型。

图2 16种环境下TaPHR1-4B两种单倍型每穗小穗数比较
E: 环境, E1: 15-SY-WW; E2: 15-SY-WW-HS; E3: 15-SY-DS; E4: 15-SY-DS-HS; E5: 16-SY-WW; E6: 16-SY-WW-DS; E7: 16-SY-DS; E8: 16-SY-DS-HS; E9: 16-CP-WW; E10: 16-CP-DS; E11: 17-SY-WW; E12: 17-SY-WW-HS; E13: 17-SY-DS; E14: 17-SY-DS-HS; E15: 17-CP-WW; E16: 17-CP-DS。*、**、***分别表示在 0.05、0.01、0.001 概率水平差异显著。误差值: ±SE。
E: environment; E1: 15-SY-WW; E2: 15-SY-WW-HS; E3: 15-SY-DS; E4: 15-SY-DS-HS; E5: 16-SY-WW; E6: 16-SY-WW-DS; E7: 16-SY-DS; E8: 16-SY-DS-HS; E9: 16-CP-WW; E10: 16-CP-DS; E11: 17-SY-WW; E12: 17-SY-WW-HS; E13: 17-SY-DS; E14: 17-SY-DS-HS; E15: 17-CP-WW; E16: 17-CP-DS. *, **, and *** represent significant difference in the 0.05, 0.01, and 0.001 probability levels, respectively. Error bar: ±SE.
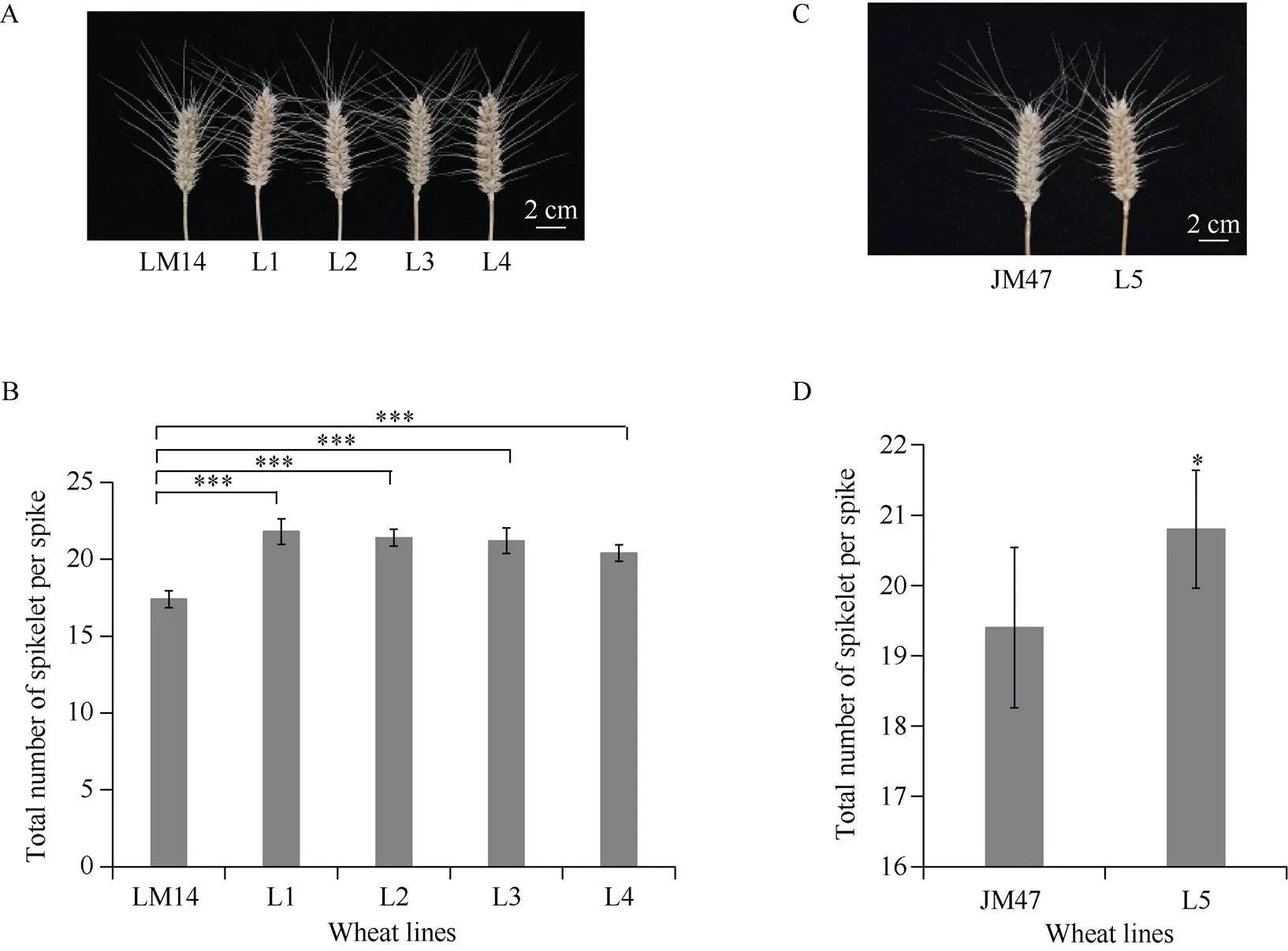
图3 回交导入系群体中不同单倍型小麦每穗小穗数比较
回交导入系群体豫麦8-鲁麦14 (A, B)和洛阳8628-晋麦47 (C, D)中轮回亲本与导入系(L1~L5)的比较。*、**、***分别表示在0.05、0.01、0.001概率水平差异显著。误差值: ±SE。
Comparison of recurrent parents with introgression lines (L1–L5) in donor parent Yumai 8-Lumai 14 (A, B) and Luoyang 8628-Jinmai 47 (C, D). *, **, and *** represent significant difference in the 0.05, 0.01, and 0.001 probability levels, respectively. Error bar: ±SE.
2.5 Hap-4B-I在幼穗中具有较高的转录水平
为研究不同单倍型的SNP与表达水平之间的关系, 在NP1中筛选相应单倍型的材料, 每个单倍型的5份材料(包括2个回交的亲本)进行转录表达分析, qRT-PCR分析发现各-4B-I材料幼穗中的表达量普遍高于各-4B-II材料,-4B-I材料的平均表达量高于-4B-II材料1.57倍(图4)。
2.6 过表达TaPHR1增加水稻的穗分支
为了进一步研究目标基因的功能, 将转入水稻, 观察转基因纯合体水稻的形态和农艺性状。选择表达水平不同的过表达水稻的3个纯合系(附图4)进行性状统计。如图5和图6所示, 转基因植株的穗分支和主穗长都显著高于野生型(图5-A和图6)。进一步说明基因参与植物穂数发育。
2.7 中国小麦群体中TaPHR1-4B单倍型的地理和时间分布
根据地理位置、小麦类型、气候特征以及栽培特点和播种, 中国小麦生产区被划分为10个主要农业生态区, 即北部冬麦区(I)、黄淮冬麦区(II)、长江中下游冬麦区(III)、西南冬麦区(IV)、华南冬麦区(V)、东北春麦区(VI)、北部春麦区(VII)、西北春麦区(VIII)、青藏春-冬麦区(IX)和新疆冬-春麦区(X)[43]。为了确定的优异单倍型是否在小麦育种过程中被正向选择, 研究了它们在两个自然群体中的地理分布。在NP2 (地方品种)中, 除VI外, 所有小麦区域中-4B-II都是显性单倍型(图7-A)。相对于地方品种, 现代育成品种(NP3)中所有麦区-4B-II都是显性单倍型(图7-B)。在现代育成品种中,-4B-I的频率在I区(26.3%~3.6%)、II区(14.3%~5.1%)、IV区(13%~0)、VI (33.3%~18.18%)、VII (33.3%~11.1%)、IX (7.69%~0)降低;-4B-I的频率在III区(0~12.5%)和V区(0~1.96%)增加。为了评估单倍型频率随时间的变化, 将现代育成品种根据种植时间分为6个亚组, 结果可知-4B-I随育种时间占比相对增多(图8)。

图4 TaPHR1在不同单倍型小麦种质中的表达水平
每个单倍型5份材料, 包括2个回交群体的亲本。误差值: ±SE。
Five accessions of each haplotype, including the parents of two backcross populations. Error bar: ±SE.

图5 TaPHR1过表达水稻农艺性状与野生型水稻的比较
转基因水稻穗部特征(A)、成熟植株(B)。
Panicle characteristics (A) and mature plants (B) of transgenic rice.
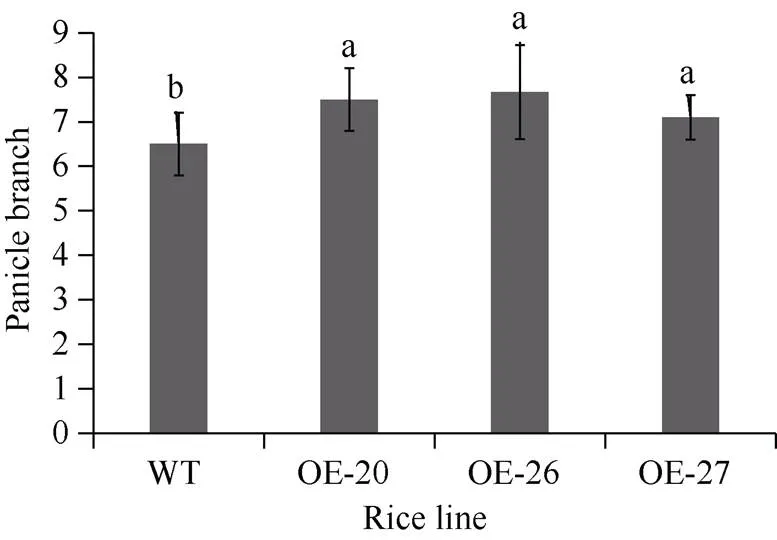
图6 TaPHR1过表达水稻农艺性状与野生型水稻穗分支的比较
转基因水稻(OE-20、OE-26、OE-27)与野生型(WT)穗分枝比较。
Comparison of panicle branch between transgenic rice (OE-20, OE-26, and OE-27) and wild type (WT).
3 讨论
MYB类转录因子是真核生物中分布最广泛的转录因子家族之一, 在植物的生长发育、生物胁迫与非生物胁迫等过程中发挥重要作用[44]。多种MYB转录因子被证明与生长发育相关。对侧芽的发育有一定的控制作用[45]; 拟南芥中参与植物外种皮细胞的分化还和毛状体的调控[19];对根毛的形成具有一定的调控作用[46]。本研究采用关联分析法, 发现参与控制小麦的每穗小穗数, 并通过回交导入系群体和转基因水稻进一步证明上述结果, 说明在控制重要农艺性状发育方面发挥重要作用。
本研究利用小麦基因组变异联合数据库对、、上游启动子区和基因编码区的基因组序列进行序列多态性分析(图1-A, B), 发现TaPHR1-SNPB1在各农艺性状中仅与小麦每穗小穂数存在显著关联(图2和表2)。前人研究发现,在水稻中异位表达导致穗分支增加[38]。本研究中转基因水稻的表型分析发现过表达能增加水稻的穗分枝数, 进一步证实PHR类转录因子参与小穂数发育。但转基因水稻的表型分析发现过表达降低了水稻的千粒重和单株产量(附图5)。前人研究发现在水稻中超量表达PHR类基因可能导致转基因水稻中的磷过量积累, 从而抑制生长导致减产[37,47]。这与本研究结果类似。以上结果也暗示着基因除了影响小穂数外, 可能在其他产量相关性状中起一定的负调控作用。目前, 不少研究发现影响产量的各要素间, 经常存在互相拮抗的作用, 如小穂数增多, 则常伴随着穗粒数, 或者千粒重等农艺性状下降, 从而导致产量降低, 这与本研究的结果类似[48-49]。
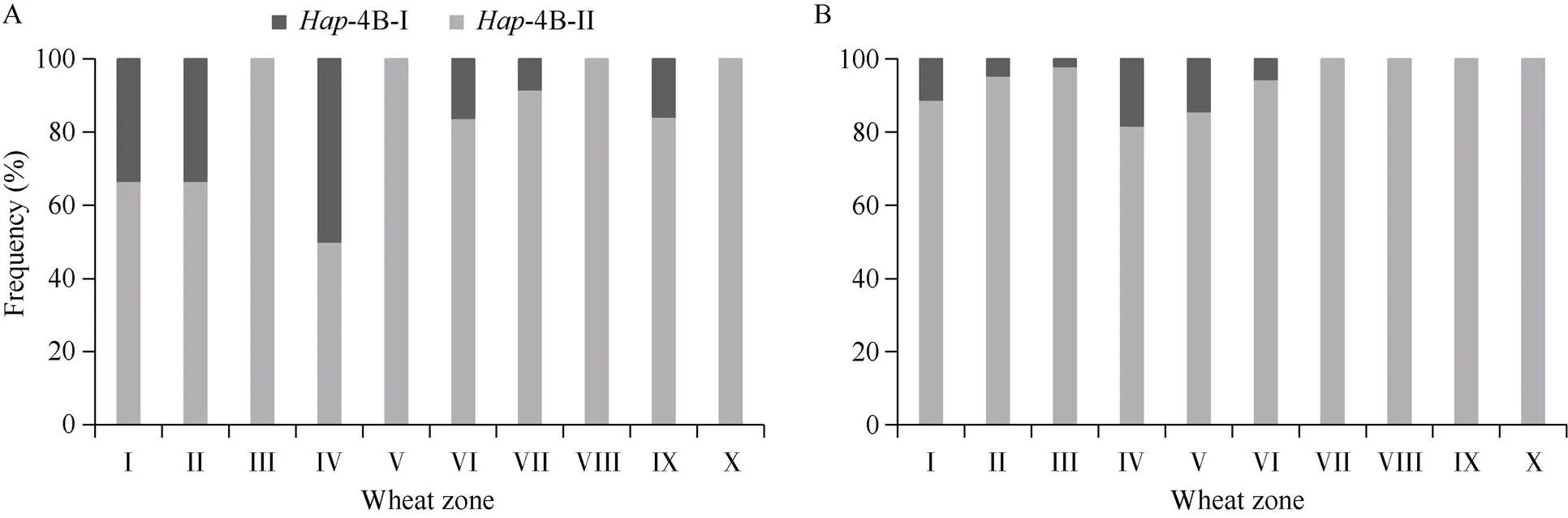
图7 TaPHR1-4B两种单倍型在我国十大麦区的地理分部
两个单倍型在我国十大麦区农家品种(A)和育成品种(B)群体中的分布。I: 北方冬麦区; II: 黄淮冬麦区; III: 长江中下游冬麦区; IV: 西南冬麦区; V: 南方冬麦区; VI: 东北春麦区; VII: 北方春麦区; VIII: 西北春麦区; IX: 青-藏冬春麦区; X: 新疆冬春麦区。饼图的大小与种群中小麦品种的数量成正比。
Haplotype distributions ofin (A) Chinese landraces and (B)modern cultivars. I: Northern Winter Wheat Zone; II: the Yellow and Huai River Valleys Facultative Wheat Zone; III: the Middle and Low Yangtze Valleys Autumn-Sown Spring Wheat Zone; IV: Southwestern Autumn-Sown Spring Wheat Zone; V: Southern Autumn Sown Spring Wheat Zone; VI: Northeastern Spring Wheat Zone; VII: Northern Spring Wheat Zone; VIII: Northwestern Spring Wheat Zone; IX: Qinghai Tibetan Plateau Spring-Winter Wheat Zone; X: Xinjiang Winter-Spring Wheat Zone. The size of pie chart is directly proportional to the number of wheat accessions in the population.

图8 TaPHR1-4B两种单倍型在中国小麦品种中频率随时间的变化
属于MYB-CC亚族成员, 其C端含有经典的MYB结构域和CC结构域。有研究发现, 水稻MYB转录因子()通过调节()的表达控制籽粒的形成, 进而调节粒数和穗结构的发育[50]。MYB蛋白()能够控制水稻籽粒的发育[51]。拟南芥R2R3型转录因子可以调节拟南芥细胞周期及根的生长。进一步研究基因表达, 发现在拟南芥根部大量表达[52]。水稻()编码的MYB蛋白可以调节小穗的分生组织发育, 进一步研究发现在幼穗中表达量最高[53]。本研究发现的优异单倍型-4B-I在幼穗中高表达, 进一步表明可能参与小麦幼穗发育调控, 最终导致产生了更多的小穂。
从两种单倍型频率随时间的变化分析可知-4B-I随育种时间占比相对增多, 暗示着-4B-I作为与小穂数相关联的优异单倍型正在得到正向选择。但从两种单倍型在我国十大麦区的地理分布来看,-4B-II在现代育成品种中普遍占优势; 考虑到转基因水稻除了穗分枝外, 其他与产量相关的农艺性状均呈下降趋势, 因此也可能是每穗小穗数变少的-4B-II单倍型被正向选择, 最终提高了小麦产量。
标记辅助选择(MAS)是精确育种的重要工具之一, 是加速小麦育种进程的有效方法。基于功能基因的等位基因变体开发的, 可以准确区分目标基因的等位基因, 是小麦育种中MAS的理想分子标记[54]。实验结果表明, 在多种环境下,与NCS显著相关, 并在回交导入系群体和水稻转基因系中得到了进一步验证。这些功能标记可有效识别自然群体中的不同等位基因/单倍型, 并可用于小麦育种。
4 结论
小麦基因在小麦自然群体中都存在2种单倍型, 其中-4B-I是每穗小穗数多的优异单倍型。尽管-4B-II在我国现代育成品种中占比最多, 但随小麦育种时间的推进,-4B-I的占比在逐渐增多。TaPHR1-SNPB1可以用于改良农作物的穗部性状, 其功能标记将有利于小麦分子标记辅助选择育种。
附表 请见网络版: 1) 本刊网站http://zwxb. chinacrops.org/; 2) 中国知网http://www.cnki.net/; 3) 万方数据http://c.wanfangdata.com.cn/Periodical- zuowxb.aspx。
[1] Cordell D, Drangert J O, White S. The story of phosphorus: global food security and food for thought.:, 2009, 19: 292–305.
[2] Shiferaw B, Smale M, Braun H J, Duveiller E, Reynolds M, Muricho G. Crops that feed the world 10. Past successes and future challenges to the role played by wheat in global food security., 2013, 5: 291–317.
[3] Slafer G A, Miralles D J. Fruiting efficiency in three bread wheat () cultivars released at different eras. Number of grains per spike and grain weight., 1993, 170. DOI: 10.1111/j.1439-037X.1993.tb01083.x.
[4] Cao S, Xu D, Hanif M, Xia X, He Z. Genetic architecture underpinning yield component traits in wheat., 2020, 133: 1811–1823.
[5] Li Y, Li L, Zhao M, Guo L, Guo X, Zhao D, Batool A, Dong B, Xu H, Cui S, Zhang A, Fu X, Li J, Jing R, Liu X. Wheatactivatesandto regulate spike development in wheat., 2021, 19: 1141–1154.
[6] Ma J, Ding P, Liu J, Li T, Zou Y, Habib A, Mu Y, Tang H, Jiang Q, Liu Y, Chen G, Wang J, Deng M, Qi P, Li W, Pu Z, Zheng Y, Wei Y, Lan X. Correction to: identification and validation of a major and stably expressed QTL for spikelet number per spike in bread wheat., 2020, 133: 367.
[7] Yang C, Li D, Liu X, Ji C, Hao L, Zhao X, Li X, Chen C, Cheng Z, Zhu L. OsMYB103L, an R2R3-MYB transcription factor, influences leaf rolling and mechanical strength in rice (L.)., 2014, 14: 158.
[8] Piao W, Kim S H, Lee B D, An G, Sakuraba Y, Paek N C. Rice transcription factor OsMYB102 delays leaf senescence by down-regulating abscisic acid accumulation and signaling., 2019, 70: 2699–2715.
[9] 徐子寅, 于晓玲, 邹良平, 赵平娟, 李文彬, 耿梦婷, 阮孟斌. 木薯MYB转录因子基因MeMYB60表达特征分析及其互作蛋白筛选, 作物学报, 2023, 49: 955–965. Xu Z Y, Yu X L, Zou L P, Zhao P J, Li W B, Geng M T, Ruan M B. Expression pattern analysis and interaction protein screening of cassava MYB transcription factor., 2023, 49: 955–965 (in Chinese with English abstract).
[10] Koshiba T, Yamamoto N, Tobimatsu Y, Yamamura M, Suzuki S, Hattori T, Mukai M, Noda S, Shibata D, Sakamoto M, Umezawa T. MYB-mediated upregulation of lignin biosynthesis intowards biomass refinery.(Tokyo), 2017, 34: 7–15.
[11] Xiang X J, Sun L P, Yu P, Yang Z F, Zhang P P, Zhang Y X, Wu W X, Chen D B, Zhan X D, Khan R M, Abbas A, Cheng S H, Cao L Y. The MYB transcription factor Baymax1 plays a critical role in rice male fertility., 2021, 134: 453–471.
[12] Katiyar A, Smita S, Lenka S K, Rajwanshi R, Chinnusamy V, Bansal K C. Genome-wide classification and expression analysis oftranscription factor families in rice and., 2012, 13: 544.
[13] Wei Q, Chen R, Wei X, Liu Y, Zhao S, Yin X, Xie T. Genome-wide identification of R2R3-MYB family in wheat and functional characteristics of the abiotic stress responsive gene., 2020, 21: 792.
[14] Xie Z, Lee E, Lucas J R, Morohashi K, Li D, Murray J A, Sack F D, Grotewold E. Regulation of cell proliferation in the stomatal lineage by theMYB FOUR LIPSdirect targeting of core cell cycle genes., 2010, 22: 2306–2321.
[15] Mandaokar A, Browse J. MYB108 acts together with MYB24 to regulate jasmonate-mediated stamen maturation in., 2009, 149: 851–862.
[16] Haga N, Kobayashi K, Suzuki T, Maeo K, Kubo M, Ohtani M, Mitsuda N, Demura T, Nakamura K, Jurgens G, Ito M. Mutations inandcause pleiotropic developmental defects and preferential down-regulation of multiple G2/M-specific genes in., 2011, 157: 706–717.
[17] McCarthy R L, Zhong R, Ye Z H. MYB83 is a direct target of SND1 and acts redundantly with MYB46 in the regulation of secondary cell wall biosynthesis in., 2009, 50: 1950–1964.
[18] Kwon Y, Kim J H, Nguyen H N, Jikumaru Y, Kamiya Y, Hong S W, Lee H. A novelMYB-like transcription factor, MYBH, regulates hypocotyl elongation by enhancing auxin accumulation., 2013, 64: 3911–3922.
[19] Li S F, Milliken O N, Pham H, Seyit R, Napoli R, Preston J, Koltunow A M, Parish R W. TheMYB5 transcription factor regulates mucilage synthesis, seed coat development, and trichome morphogenesis., 2009, 21: 72–89.
[20] Mizoguchi T, Wheatley K, Hanzawa Y, Wright L, Mizoguchi M, Song H R, Carre I A, Coupland G.andare partially redundant genes required to maintain circadian rhythms in., 2002, 2: 629–641.
[21] Park M J, Seo P J, Park C M.alternative splicing as a way of linking the circadian clock to temperature response in., 2012, 7: 1194–1196.
[22] Marian C O, Bordoli S J, Goltz M, Santarella R A, Jackson L P, Danilevskaya O, Beckstette M, Meeley R, Bass H W. The maizegene,, belongs to a novel gene family and encodes a protein that binds telomere DNA repeats., 2003, 133: 1336–1350.
[23] Yu Y T, Wu Z, Lu K, Bi C, Liang S, Wang X F, Zhang D P. Overexpression of the MYB37 transcription factor enhances abscisic acid sensitivity, and improves both drought tolerance and seed productivity in., 2016, 90: 267–279.
[24] Deng M, Wang Y, Kuzma M, Chalifoux M, Tremblay L, Yang S, Ying J, Sample A, Wang H M, Griffiths R, Uchacz T, Tang X, Tian G, Joslin K, Dennis D, McCourt P, Huang Y, Wan J. Activation tagging identifiestranscription factor AtMYB68 for heat and drought tolerance at yield determining reproductive stages., 2020, 104: 1535–1550.
[25] Shingote P R, Kawar P G, Pagariya M C, Muley A B, Babu K H. Isolation and functional validation of stress tolerantgene and its comparative physio-biochemical analysis with transgenic tobacco plants overexpressingand., 2020, 10: 225.
[26] Manna M, Thakur T, Chirom O, Mandlik R, Deshmukh R, Salvi P. Transcription factors as key molecular target to strengthen the drought stress tolerance in plants., 2021, 172: 847–868.
[27] Li X, Jia J, Zhao P, Guo X, Chen S, Qi D, Cheng L, Liu G., an unknown function transcription factor gene from sheepgrass, as a positive regulator of chilling and freezing tolerance in transgenic., 2020, 20: 238.
[28] Tiwari P, Indoliya Y, Chauhan A S, Singh P, Singh P K, Singh P C, Srivastava S, Pande V, Chakrabarty D. Auxin-salicylic acid cross-talk amelioratesmediated defense towards heavy metal, drought and fungal stress., 2020, 399: 122811.
[29] Leng B, Wang X, Yuan F, Zhang H, Lu C, Chen M, Wang B. Heterologous expression of theMYB transcription factor LbTRY inincreases salt sensitivity by modifying root hair development and osmotic homeostasis., 2021, 302: 110704.
[30] Agarwal P, Mitra M, Banerjee S, Roy S. MYB4 transcription factor, a member of R2R3-subfamily of MYB domain protein, regulates cadmium toleranceenhanced protection against oxidative damage and increases expression ofandin., 2020, 297: 110501.
[31] Cominelli E, Sala T, Calvi D, Gusmaroli G, Tonelli C. Over-expression of thegene alters cell expansion and leaf surface permeability., 2008, 53: 53–64.
[32] Hoang M H, Nguyen X C, Lee K, Kwon Y S, Pham H T, Park H C, Yun D J, Lim C O, Chung W S. Phosphorylation by AtMPK6 is required for the biological function of AtMYB41 in., 2012, 422: 181–186.
[33] Raffaele S, Rivas S, Roby D. An essential role for salicylic acid in AtMYB30-mediated control of the hypersensitive cell death program in., 2006, 580: 3498–3504.
[34] Bari R, Pant B D, Stitt M, Scheible W R. PHO2, microRNA399, and PHR1 define a phosphate-signaling pathway in plants., 2006, 141: 988–999.
[35] Bustos R, Castrillo G, Linhares F, Puga M I, Rubio V, Perez-Perez J, Solano R, Leyva A, Paz-Ares J. A central regulatory system largely controls transcriptional activation and repression responses to phosphate starvation in., 2010, 6: e1001102.
[36] Rubio V, Linhares F, Solano R, Martin A C, Iglesias J, Leyva A, Paz-Ares J. A conserved MYB transcription factor involved in phosphate starvation signaling both in vascular plants and in unicellular algae., 2001, 15: 2122–2133.
[37] Wang J, Sun J, Miao J, Guo J, Shi Z, He M, Chen Y, Zhao X, Li B, Han F, Tong Y, Li Z. A phosphate starvation response regulatoris involved in phosphate signalling and increases grain yield in wheat., 2013, 111: 1139–1153.
[38] Zheng X, Liu C, Qiao L, Zhao J, Han R, Wang X, Ge C, Zhang W, Zhang S, Qiao L, Zheng J, Hao C. The MYB transcription factor TaPHR3-A1 is involved in phosphate signaling and governs yield-related traits in bread wheat., 2020, 71: 5808–5822.
[39] Miao L, Mao X, Wang J, Liu Z, Zhang B, Li W, Chang X, Reynolds M, Wang Z, Jing R. Elite haplotypes of a protein kinase geneassociated with important agronomic traits in common wheat., 2017, 8: 368.
[40] Hao C, Wang L, Ge H, Dong Y, Zhang X. Genetic diversity and linkage disequilibrium in Chinese bread wheat (L.) revealed by SSR markers., 2011, 6: e17279.
[41] Livak K J, Schmittgen T D. Analysis of relative gene expression data using real-time quantitative PCR and the 2(–Delta Delta C(T)) method., 2001, 25: 402–408.
[42] Stewart C N, Via L E Jr. A rapid CTAB DNA isolation technique useful for RAPD fingerprinting and other PCR applications., 1993, 14: 748–750.
[43] Liu Y, Lin Y, Gao S, Li Z, Ma J, Deng M, Chen G, Wei Y, Zheng Y. A genome-wide association study of 23 agronomic traits in Chinese wheat landraces., 2017, 91: 861–873.
[44] Jeena G S, Kumar S, Shukla R K. Characterization of MYB35 regulated methyl jasmonate and wound responsive Geraniolgene from., 2021, 253: 89.
[45] Keller T, Abbott J, Moritz T, Doerner P.controls a leaf axil stem cell niche and modulates vegetative development., 2006, 18: 598–611.
[46] Kang Y H, Kirik V, Hulskamp M, Nam K H, Hagely K, Lee M M, Schiefelbein J. Thegene provides a positive feedback loop for cell fate specification in theroot epidermis., 2009, 21: 1080–1094.
[47] Zhou J, Jiao F, Wu Z, Li Y, Wang X, He X, Zhong W, Wu P.is involved in phosphate-starvation signaling and excessive phosphate accumulation in shoots of plants., 2008, 146: 1673–1686.
[48] 张辉. 不同年份小麦产量与主要农艺性状通径分析. 中国农学通报, 2016, 32(27): 24–28. Zhang H. Path analysis of wheat yield and main agronomic traits under different years., 2016, 32(27): 24–28 (in Chinese with English abstract).
[49] 田纪春, 邓志英, 胡瑞波, 王延训. 不同类型超级小麦产量构成因素及籽粒产量的通径分析. 作物学报, 2006, 42: 1699–1705. Tian J C, Deng Z Y, Hu R B, Wang Y X. Yield components of super wheat cultivars with different types and the path coefficient analysis on grain yield., 2006, 42: 1699–1705 (in Chinese with English abstract).
[50] Li G, Xu B, Zhang Y, Xu Y, Khan N U, Xie J, Sun X, Guo H, Wu Z, Wang X, Zhang H, Li J, Xu J, Wang W, Zhang Z, Li Z.controls grain number and shapes panicle architecture in rice., 2022, 20: 158–167.
[51] Ren D, Cui Y, Hu H, Xu Q, Rao Y, Yu X, Zhang Y, Wang Y, Peng Y, Zeng D, Hu J, Zhang G, Gao Z, Zhu L, Chen G, Shen L, Zhang Q, Guo L, Qian Q.encodes a MYB domain protein that determines hull fate and affects grain yield and quality in rice., 2019, 100: 813–824.
[52] Mu R L, Cao Y R, Liu Y F, Lei G, Zou H F, Liao Y, Wang H W, Zhang W K, Ma B, Du J Z, Yuan M, Zhang J S, Chen S Y. An R2R3-type transcription factor generegulates root growth and cell cycle progression in., 2009, 19: 1291–1304.
[53] Li Y F, Zeng X Q, Li Y, Wang L, Zhuang H, Wang Y, Tang J, Wang H L, Xiong M, Yang F Y, Yuan X Z, He G H. MULTI-FLORET SPIKELET 2, a MYB transcription factor, determines spikelet meristem fate and floral organ identity in rice., 2020, 184: 988–1003.
[54] Liu Y, He Z, Appels R, Xia X. Functional markers in wheat: current status and future prospects., 2012, 125: 1–10.
Transcription factor geneinvolved in regulation spikelet number per spike in common wheat
ZHANG Yi-Ning1,2, ZHANG Yan-Fei2, WANG Min2, WANG Jing-Yi2, LI Long2, LI Chao-Nan2, YANG De-Long1,*, MAO Xin-Guo1,2,*, and JING Rui-Lian2
1State Key Laboratory of Arid land Crop Science /College of Life Science and Technology, Gansu Agricultural University, Lanzhou 730070, Gansu, China;2Institute of Crop Sciences, Chinese Academy of Agricultural Sciences / National Key Facility for Crop Gene Resources and Genetic Improvement / Key Laboratory of Crop Germplasm and Utilization, Ministry of Agriculture and Rural Affairs, Beijing 100081, China
The utilization of water elite genetic resources to develop new wheat varieties is an effective approach to deal with the challenges of climate change and rapid population growth. The MYB (v-myb avian myeloblastosis viral oncogene homolog) transcription factors are one of the largest families of transcription factors in plants and are involved in plant growth and development and in the regulation of biotic and abiotic stresses. In this study, 19 SNPs and 15 SNPs were identified inand, respectively, and the molecular markers were developed based on these SNPs. Association analysis showed that-4B-I was the elite haplotype with high Total number of spikelet per spike (NCS).-4B-I was further confirmed to be beneficial for improving wheat spike traits by creating two backcross introgression population lines. Fluorescencequantitative polymerase chain reaction ofrevealed that the relative expression level ofgenes in young spikelets of-4B-I haplotype was higher than that of-4B-II haplotype. In addition, the heterologous expression ofin rice resulted in more panicle branches, which also confirmed the involvement ofin the regulation of NCS. The geographic and temporal distribution of wheat modern varieties revealed that although-4B-II accounted for the largest proportion of modern varieties in China, the proportion of-4B-I was gradually increasing as wheat breeding time progressed. In conclusion,was a positive regulator of NCS in wheat. Therefore, the molecular markers developed in this study could be an important source of marker-assisted selection and genetic improvement in wheat.
wheat; MYB transcription factor; agronomic trait; KASP marker; association analysis
2023-05-24;
2023-07-06.
10.3724/SP.J.1006.2023.31008
通信作者(Corresponding author): 杨德龙, E-mail: yangdl@gsau.edu.cn; 毛新国, E-mail: maoxinguo@caas.cn
E-mail: zyn18845111029@163.com
2023-02-03;
本研究由国家重点研发计划项目(2018YFD0300707), 甘肃省农业重点研发计划项目(21YF5NA089)和甘肃省高等学校产业支撑计划项目(2022CYZC-44)资助。
This study was supported by the National Key Research and Development Program of China (2018YFD0300707), the Key Research and Development Program of Gansu Province, China (21YF5NA089), and Industrial Support Plan of Colleges and Universities in Gansu Province, China (2022CYZC-44).
URL: https://kns.cnki.net/kcms2/detail/11.1809.S.20230705.1720.002.html
This is an open access article under the CC BY-NC-ND license (http://creativecommons.org/licenses/by-nc-nd/4.0/).

