不同Cas9启动子对大豆CRISPR/Cas9系统效率的作用分析
牛志远 秦 超 刘 军 王海泽 李宏宇,*
不同启动子对大豆CRISPR/Cas9系统效率的作用分析
牛志远1,2秦 超1刘 军1王海泽2,*李宏宇1,*
1中国农业科学院作物科学研究所, 北京 100081;2黑龙江八一农垦大学农学院, 黑龙江大庆 163319
CRISPR/Cas9系统作为高效基因编辑系统, 已被广泛应用于动植物中。多种基因启动子, 如、等被报道用于提高CRISPR/Cas9系统的基因编辑效率。但在大豆中, 不同启动子对CRISPR/Cas9基因编辑系统效率的影响还没有被阐明。本研究选择了6个已知功能的高效启动子(、、、、、)和1个大豆内源未知功能的启动子(), 构建CRISPR/Cas9基因敲除载体。通过农杆菌介导的大豆发根系统, 检测这些启动子对大豆内源基因和的编辑效率, 结果显示大豆内源启动子对靶基因的编辑效率最高,、35S、对下游基因的编辑效率高于和。进一步对靶位点测序峰图的分析发现, 使用了和启动子的测序峰图中, 高峰占比较大, 分别为64.0%和58.6%; 而使用的测序峰图中, 低峰占比较高, 为63.3%。这表明和启动子不但编辑效率高, 而且编辑效果好, 更有利于在下一代中分离出纯合突变体植株。这些结果将为构建高效大豆基因编辑载体提供参考, 为提高大豆基因编辑效率提供依据。
基因编辑; CRISPR/Cas9; 发根系统; 启动子; 大豆
基因编辑技术是近年来用于基因功能分析和动植物分子改良的新手段[1]。目前, 主要的基因编辑系统可分为3类: Zinc Finger Nucleases (ZFNs)系统、Transcription activator-like effector nucleases (TALENs)系统和CRISPR/Cas9 (clustered regularly interspaced short palindromic Repeats/CRISPR- associated protein 9)系统[2-3]。CRISPR/Cas9是近年来被广泛应用于植物中的高效基因编辑系统[4-5]。通过CRISPR/Cas9系统可以获得T-DNA free的作物改良新品种, 且与自然基因突变类似具有稳定的遗传能力。避免了传统转基因植株中长期存在T-DNA边界序列和筛选标记等外源基因的困扰。CRISPR/Cas9系统还具有操作简单、周期短、成本低、突变效率高、可多靶点同时突变等优点。
CRISPR/Cas9系统首先在细菌和古细菌中被发现, 是细菌用来抵御外源基因侵略的防御手段[6-8]。应用于植物中的CRISPR/Cas9基因编辑系统, 是经过改良的RNA介导的DNA剪切过程, 该系统包括了Cas9核酸酶和(single guide RNA)两部分[9-11]。在及其5¢端PAM (protospacer-adjacent motif)序列的引导下, Cas9核酸酶到达基因组靶点, 并对靶点DNA进行切割产生DSB (double strand break), 然后通过DNA损伤修复过程在损伤位点附近形成各种类型的突变[12]。虽然CRISPR/Cas9技术在各个领域应用广泛, 但它也存在不足。例如, 不同物种中编辑基因的效率不同, 存在脱靶的风险, 转基因后难以去除转入载体等[13]。因此, 提高CRISPR/Cas9系统基因组编辑的效率, 降低脱靶率一直是科学家们关注的焦点。影响CRISPR/Cas9的编辑效率的因素有很多, 如Cas9核酸酶表达的空间分布、的表达量、的特异性和二级结构、靶DNA的GC含量、Cas9的密码子构成等, 均成为影响CRISPR/ Cas9的编辑效率的关键因素[14]。
Cas9核酸酶的表达模式是影响CRISPR/Cas9载体编辑效率的因素之一。()启动子()和玉米启动子()是双子叶植物和单子叶植物中常用的基因启动子, 两者的表达模式呈非特异的广泛表达[15-19]。在拟南芥中研究发现, 使用作为基因启动子通过花序蘸取农杆菌菌液的转化方法, 在体细胞中对靶基因的编辑效率高于在胚囊细胞中对靶基因的编辑效率, 在单子叶植物中使用驱动基因在胚囊中对下游基因的编辑效率高于使用[20-21]。在拟南芥中, 使用基因启动子驱动基因表达可以提高胚囊中基因的编辑的效率[22-23]。另一个拟南芥中的基因, 在卵细胞和分生组织等发育阶段的细胞中均保持高水平的表达, 将启动子驱动构建CRISPR/Cas9载体对下游靶基因进行编辑发现, 其编辑效率高于启动子[24-25]。这些研究都表明, 对基因启动子的选择, 是影响CRISPR/Cas9载体对下游基因编辑效率的关键因素之一。2015年, 大豆中首次报道了通过发根系统检测CRISPR/Cas9载体的效率[26]。已报道的大豆CRISPR/Cas9基因敲除载体, 大多以为基因启动子[27-28]。本实验室使用拟南芥启动子成功获得了大豆突变体[29]。最近的研究显示, 使用大豆基因驱动基因表达, 也能对大豆基因组靶基因进行有效敲除[30]。但到目前为止在大豆中, 不同启动子对下游靶基因的编辑效率的影响还没有得到比较验证。
本研究选择了4个已知的启动子(、、、)和3个新的大豆内源启动子(、、)驱动基因表达, 利用大豆发状根转化的方法, 在较短的时间内获得转基因发状根, 并检测靶基因编辑情况, 对这7个启动子的编辑效率和编辑效果进行分析, 评价了7个启动子在大豆基因编辑中的效果。
1 材料与方法
1.1 植物材料
本试验中使用的大豆品种为Williams 82和天隆1号。启动子、、、来自于Williams 82的基因组DNA; 大豆发状根转化材料为大豆品种天隆1号, 大豆材料种植在北京市中国农业科学院植物培养室中, 种植条件为16 h光照/8 h黑暗, 温度为27℃。启动子来自于拟南芥()生态型。
1.2 菌株与质粒
本试验所使用的大肠杆菌菌株为, 农杆菌菌株为, 由本实验室保存并制备感受态; 基因编辑载体JRH0645质粒由本实验室根据pCambia3301骨架自行改造并保存, 载体示意图见图1。
1.3 方法
1.3.1 Real-time PCR检测方法及热图绘制方法
除了已见文献报道的高效启动子外, 理论上在大豆中各个组织中表达量均较高基因的启动子, 可作为候选启动子提高大豆中CRISPR/Cas9编辑效率的启动子, 故根据Wang等[31]发表文章中的转录组数据, 从中挑选出11个在根、茎、单叶、复叶中表达量高的基因作为候选基因, 基因编号分别为、、、、、、、、、、。将大豆Williams 82种植在25℃培养室中, 培养条件为16 h光照/8 h黑暗, 当大豆植株长到第3片三重复叶完全展开时, 每5棵植株为1个重复, 分别取根、茎、单叶、第3片三重复叶和顶端分生组织, 3次重复, TRIzol法提取RNA, 参照全式金公司TransScript II One-Step gDNA Removal and cDNA Synthesis SuperMix反转录试剂盒说明书, 进行反转录。在数据库(https://phytozome-next.jgi.doe.gov/)中获得该11个基因的基因组序列, 通过www.oligoarchitect.com网站设计11个基因表达量的检测引物(表1)。以大豆基因作为内参, 使用2-ΔΔCt计算各基因的相对表达量。将计算所得的相对表达量通过GraphPad Prism 7 软件绘制热图, 分析不同植物组织中基因表达水平。

图1 7个CRISPR/Cas9载体构建示意图
绿色箭头代表7个启动子, 红色方框代表终止子。黄色方框代表gRNA序列。
The green arrows, red boxes, and yellow boxes represent the seven promoters, the terminators, and the gRNA sequences, respectively.

表1 PCR引物序列
1.3.2 启动子克隆及载体构建 通过Phytozome数据库网站(https://phytozome-next.jgi.doe.gov/), 获得7个基因的启动子序列。设计启动子扩增引物(表2), 使用高保真聚合酶对7个启动子进行扩增。

表2 启动子序列扩增引物序列
表中小写英文字母为In-fusion enzyme构建载体同源重组边界序。
Different lowercase in the table represents the homologous recombination boundary sequence in the in-fusion enzyme system.
将JRH0645 (本实验室自主改造完成)载体用II单酶切, 对酶切产物进行凝胶回收; 通过Infusion (Clontech)无缝克隆将启动子片段插入到酶切产物JRH0645载体的II位点。构建成:、:、:、:、:、:、:中间载体; 第2步, 将启动子序列和带有基因靶位点的序列(表3)连接到中间载体的I位点与I位点之间, 构建成7个表达载体p:--、:--、:--、:--、:--、:--、:--, 选择编辑效率高的3个启动子, 构建了3个带有靶基因的表达载体:--、:Cas9--、:--。用于验证不同的启动子对靶基因的编辑是否存在偏好性。
1.3.3 大豆发状根诱导及突变位点检测 将上述1.3.2中构建好的7个启动子共10个CRISPR/Cas9载体转入农杆菌菌株中, 侵染大豆子叶节诱导发状根。侵染后在经过10 d左右的发状根培养, 待侵染位点的发状根长到4~5 cm时, 随机挑取单个根提取DNA, 对靶位点和的突变情况进行检测。检测方法为: 根据序列与序列, 进行引物设计(表3), 同时检测到序列与序列的根为阳性转基因根。具体为PCR总反应体系为10 µL, 其中康为MIX buffer (2×) 5 µL, U6-F 0.2 µL, U6-R 0.2 µL, ddH2O 3.6 µL, 模板(单根DNA) 1µL。反应程序为: 95℃ 5 min; 95℃ 30 s, 57℃ 30 s, 72℃ 25 s, 循环35次; 72℃ 5 min。用1%琼脂糖凝胶电泳检测。Cas9检测步骤同上。
通过电泳图观察将同时检测出序列与序列的单根DNA进行突变PCR检测, 设计突变检测PCR引物(表3), 反应体系同上, 将PCR产物测序, 通过测序结果统计靶位点基因的突变类型情况, 对上述实验进行3次技术重复。
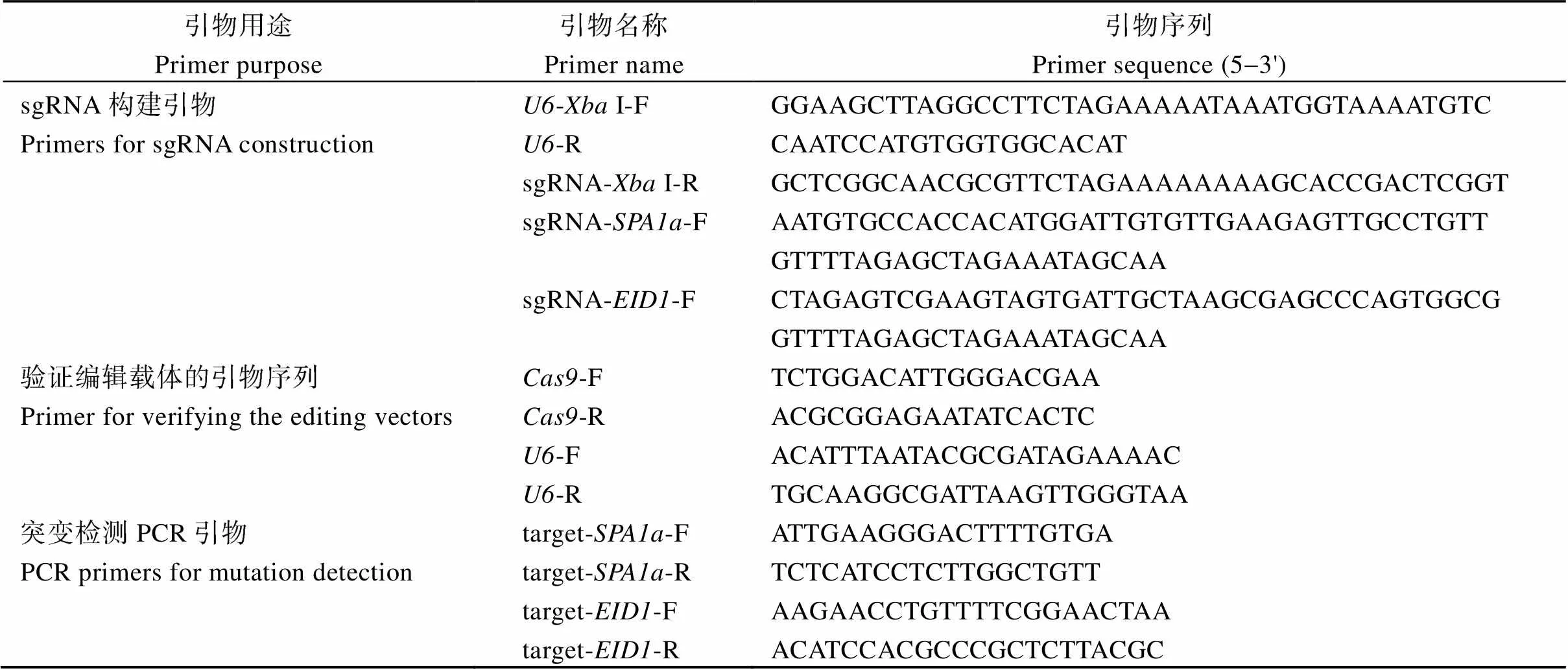
表3 引物序列
2 结果与分析
2.1 Cas9启动子对CRISPR/Cas9编辑效率的比较
为了筛选大豆内源的高效启动子, 将拟南芥基因作为参考基因序列, 在大豆数据库中, 找到4个与拟南芥()同源的基因, 分别命名为()、()、()和()。本研究扩增获得了和的启动子:和, 与拟南启动子一起, 构建了3个敲除大豆的CRISPR/Cas9载体::-:、:-:、:-:(图1)。将拟南芥的基因与大豆基因组序列比对, 发现了1个同源基因, 扩增启动子并构建1个编辑的CRISPR/Cas9载体:-p:(图1)。花椰菜花叶病毒()启动子和玉米()启动子是在双子叶植物和单子叶植物中常用的过表达启动子, 为了比较这2个常用的启动子与其他内源大豆启动子对CRISPR/Cas9编辑系统对基因编辑效率的影响, 本研究同样构建了2个以和为启动子的基因编辑载体:-p:和:- p:, 对下游的进行编辑(图1)。(-)是大豆中光信号转导途径的负调控因子, 本实验室对该基因的研究表明,主要参与到大豆的开花和株高调控过程[32-33]。
通过对同一时期11个基因在不同组织部位的相对表达量比较发现,在根、茎、复叶、单叶和顶端分生组织中的表达量高于其他10个基因(图2), 将该基因命名为(), 并克隆该基因的启动子, 构建了敲除下游的CRISPR/Cas9载体p:- p:(图1)。

图2 热图分析11个基因在根、茎、复叶、单叶和茎端分生组织中的表达量
以大豆基因作为内参, 使用2-ΔΔCt计算各基因的相对表达量。将计算所得的相对表达量通过GraphPad Prism 7软件绘制热图, 相对表达水平以蓝色(高)到白色(低)的色阶表示。
Soybeangene was used as the internal reference, and the relative expression levels of each genes were calculated using 2-ΔΔCtmethod. The relative expression in heat map was made by GraphPad Prism 7 software, and the relative expression level was represented by the color scale from blue (high) to white (low).
2.2 不同启动子对GmSPA1a基因的编辑效率分析
为比较这7个启动子对CRISPR/Cas9载体基因编辑效率的影响, 本研究采用农杆菌介导的发根检测法, 检测这7个启动子对下游基因的编辑情况。每个载体进行了3次发根转化, 记录每次转化中挑取的发状根数量、通过PCR检测和序列判定单根中成功转入各CRISPR/Cas9载体的阳性根数量(图3-A和附图1)、最后再测序检测阳性根中位点被编辑的根的数量并计算编辑效率(发生编辑的根的数量/阳性根的数量) (表4)。
通过对上述结果分析发现, 使用大豆内源基因启动子在3次发根检测中对都表现出较高的编辑效率, 被编辑根数平均超过60%; 其次使用、和平均编辑根数接近或超过50%; 但使用大豆内源、、对靶基因的编辑效率较低, 平均被编辑的根数占比20%~30% (图3-B和图4-A)。
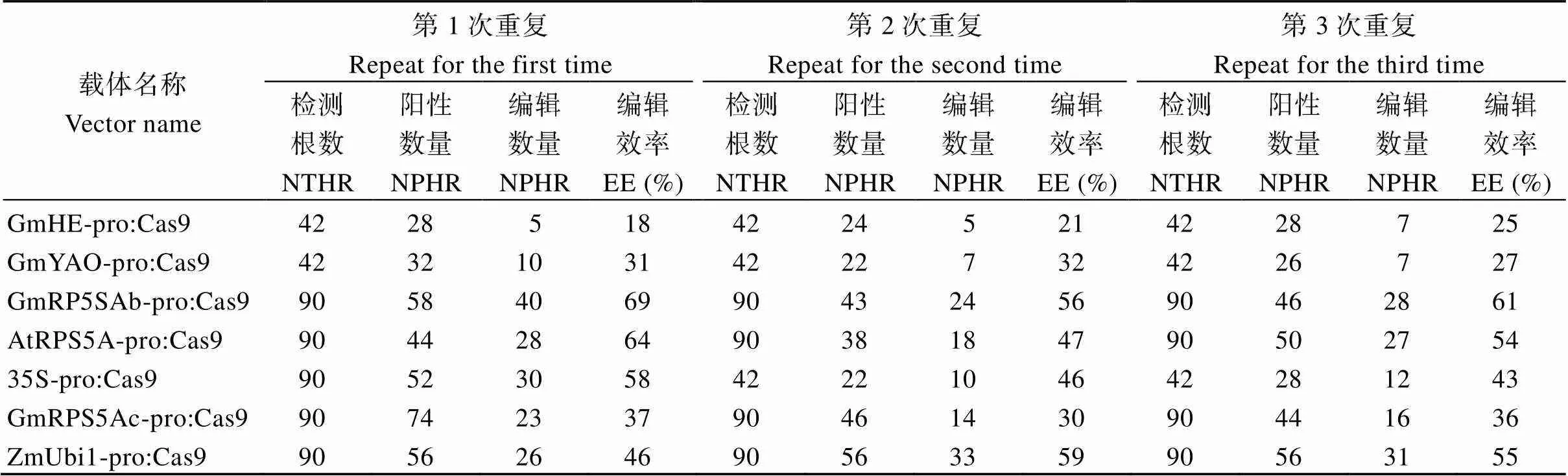
表4 大豆发状根检测中所取发状根数量、阳性数量及编辑效率统计
NTHR: number of total hair roots; NPHR: number of positive hair roots; NPHR: number of editing hair roots; EE: editing efficiency.

A: 使用启动子, 在发状根中PCR检测单根与序列电泳图。M: 分子量marker; 空白对照: 水, 阴性对照: 天隆1号基因组DNA; 阳性对照: 带有、序列质粒。同时检测到序列与序列的阳性根编号用红色数字标识,序列大小为950 bp,序列大小为864 bp。B: 发状根检测所取单根数量、阳性未编辑根数量和被编辑的根数量。3个柱形图为3次重复结果, 其中蓝色柱为检测根总数量, 红色柱为PCR检测出的阳性根数, 绿色柱为位点被编辑的根的数量, 具体取样根的数量见表4。
A:andsequences were detected by PCR in hair roots usingpromoter. M: molecular weight marker; blank control: pure water; Negative control: TL1 genomic DNA; positive control: plasmid withandsequences. The positive root numbers are marked in red, thesequence is 950 bp and that of Cas9 sequence is 864 bp. B: the number of the total roots, positive roots, and edited roots. The three column charts in the figure are the results of three repetitions, among which the blue column is the number of detected roots, the red column is the number of PCR positive roots, and the green column is the number of edited roots at thesite (the specific number of sampled roots in Table 4).
2.3 不同Cas9启动子对GmEID1编辑效率验证
为进一步验证不同的启动子对靶基因的编辑是否存在偏好性, 本研究选择了、和构建了3个编辑大豆内源基因的CRISPR/Cas9载体, 同样使用发根检测法, 验证这3个启动子对的编辑效率(表5)。
分析发现, 使用对的编辑效率最高, 被编辑的发状根数占比超过50%; 使用的编辑效率也较高, 被编辑的发状根数接近50%, 与使用接近; 但使用对的编辑效率较低, 仅接近30% (图4-B)。说明,和对下游靶基因的选择性低, 对不同基因的编辑效率较高并保持稳定, 但对下游靶基因的编辑可能存在选择性。
2.4 不同Cas9启动子对靶基因GmSPA1a的编辑效果检测
截至目前, 大豆中通过CRISPR/Cas9基因编辑系统在T0代获得纯合突变体的概率很低, 但通过观察发现, T0代中对靶基因的编辑情况, 是能否在T1代获得纯合突变体的关键。因此, 本研究对编辑位点的测序峰图, 进行了统计分析。首先设置了峰图分类标准, 将峰图分为高峰和低峰2类: 测序峰图中第2高峰的高度超过最高峰高度一半的峰图, 定义为高峰(图5-A), 反之没有超过最高峰一半的峰图, 定义为低峰(图5-B)。从编辑位点开始计数20个碱基的峰图, 出现5个及以上高峰的为高峰峰图; 反之则为低峰峰图。本研究共进行了3次发根转化, 并分别对每次的编辑效果进行了统计(附图2)。对3次结果的统计显示, 使用、和对基因的编辑高峰占比均超过50%; 而、和的高峰占比接近50%;的高峰占比最低,只有20%~30% (图5-C)。表明, 在大豆中使用、和驱动表达的编辑效果好于使用、和, 而在大豆内源基因编辑过程中, 与其他启动子相比效果较差。

表5 大豆发状根检测中所取发状根数量、阳性数量及编辑效率统计
NTHR: number of total hair roots; NPHR: number of positive hair roots; NPHR: number of editing hair roots; EE: editing efficiency.
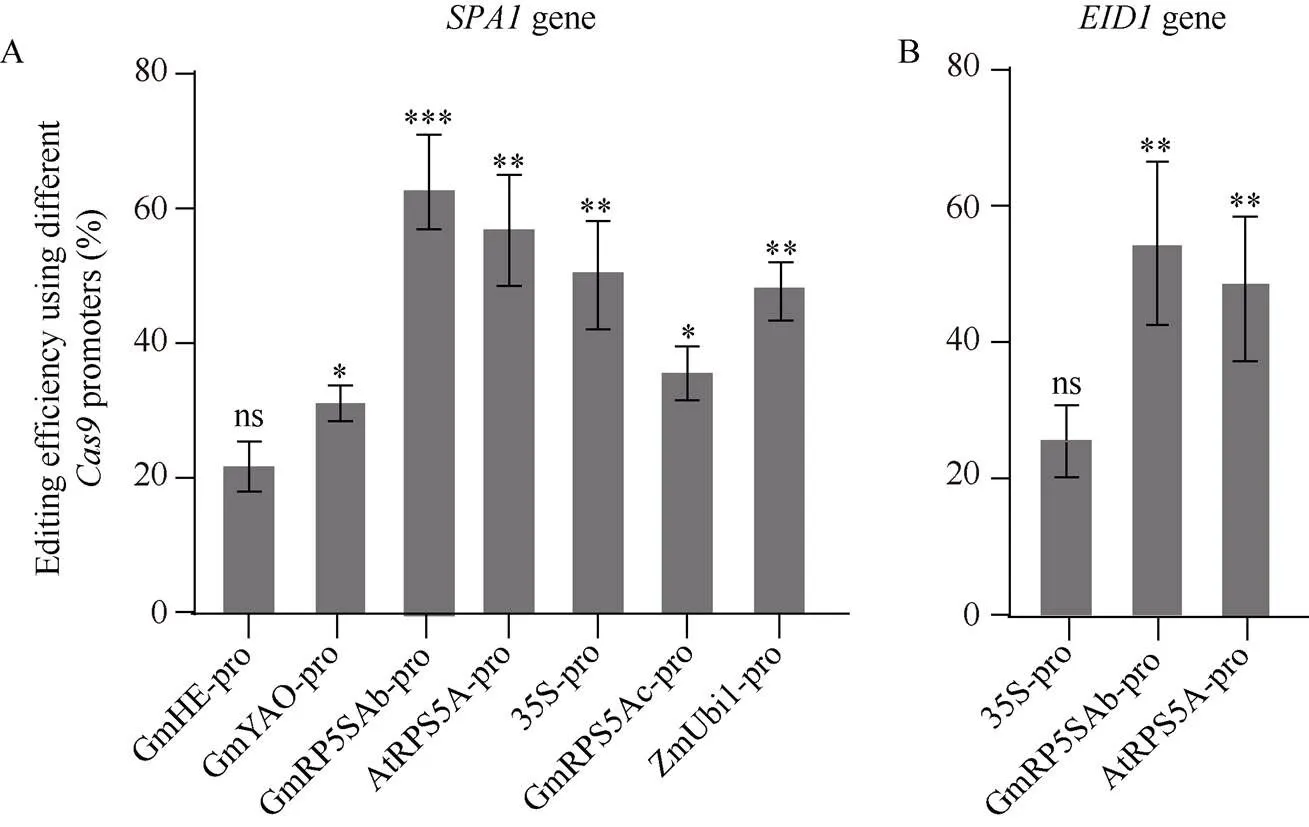
图4 使用不同Cas9启动子的编辑效率
A: 7个启动子对基因的编辑效率。B: 3个启动子对基因的编辑效率。*表示差异显著(-test<0.05), **表示差异极显著(-test< 0.01), ns表示无差异。
A: the editing efficiency of seven promoters ongene. B: the editing efficiency ofgene by three promoters. *:<0.05; **:< 0.01; ns: not significant difference.
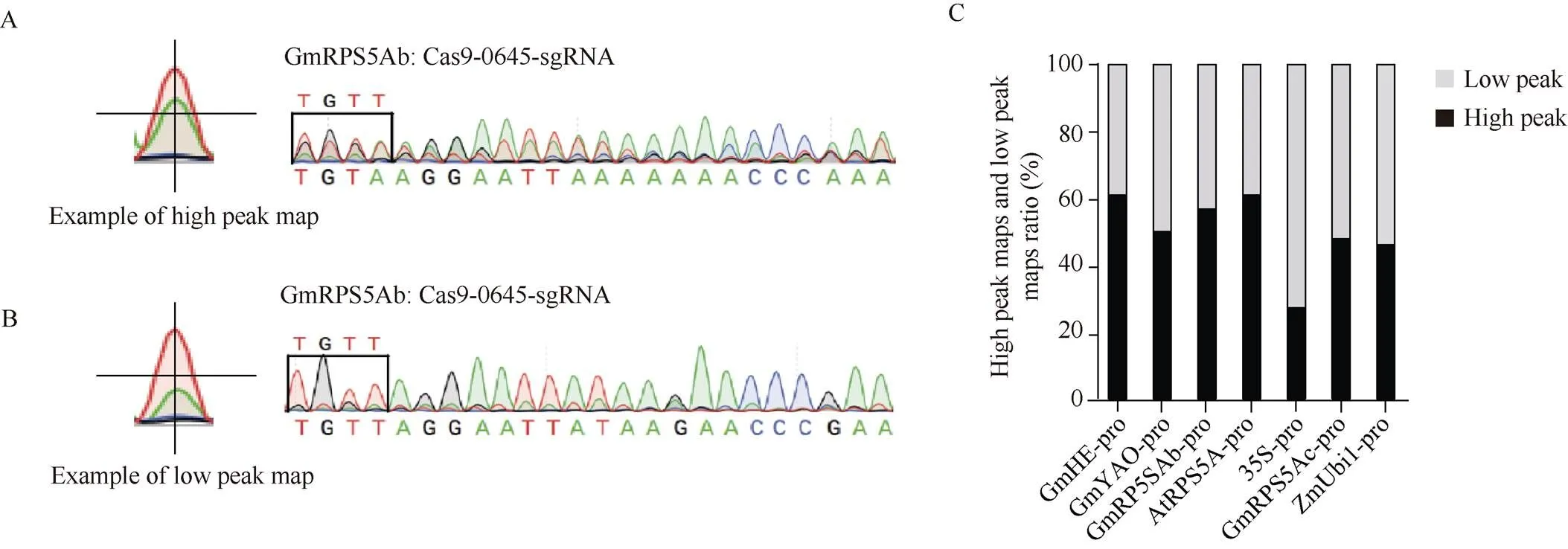
图5 GmSPA1a编辑位点的测序峰图分析
A: 测序峰图高峰示例; B: 测序峰图低峰示例; C: 统计分析3次发根检测中7个启动子对编辑峰图中高峰和低峰比例。柱形图黑色部分代表测序峰图中高峰所占比例, 柱形图灰色部分代测序峰图中低峰所占比例。
A: the example of high peak map; B: the example of low peak map; C: the statistical analysis of the high peak maps and low peak maps ratio of 7 promoters atedited site. Black column represents the high peak maps, and gray represents the low peak maps in sequenced maps.
3 讨论
本研究检测了7个不同的启动子, 在大豆中对下游靶基因的编辑效率和编辑效果。综合各试验结果发现, 使用、和对靶基因的编辑效率和编辑效果好于其他启动子;虽然编辑效率较高, 但其编辑峰中的低峰为主, 编辑效果较差, 不利于T1代植株分离出纯合的突变体植株; 而、和虽然编辑效果较好, 但编辑效率偏低。在拟南芥中研究发现,基因启动子驱动对下游基因的编辑效率比启动子高, 特别是在将变体与碱基脱氨酶融合表达后,启动子显示出对腺苷酸和胞嘧啶位点更高的编辑效率[24]。在烟草中也得到了类似的结果, 与使用启动子相比, 拟南芥启动子在烟草中也提高了CRISPR/Cas9系统对下游基因的编辑效率[34]。本研究的结果与这些研究结果相吻合, 证明拟南芥基因启动子在大豆中也有较高的编辑效率, 同时本研究首次证实大豆内源基因启动子也可作为基因的启动子用于CRISPR/Cas9系统基因编辑。
使用不同的启动子, 会导致基因的表达时期和表达部位不同, 这是影响CRISPR/Cas9系统编辑效率的因素之一。在拟南芥中有研究表明, 在胚胎发育过程中越早期表达的基因启动子, 在子代产生体细胞嵌合体的概率越低[35], 这可能与拟南芥的转化方法相关。在拟南芥中, 转基因主要采取花序组织蘸取农杆菌菌液的转化方法, 在此过程中转基因T-DNA首先作用的是胚囊细胞, 这导致T1代产生的体细胞突变和未转化的种子占比较高, 使用启动子驱动基因在编辑系统中虽然应用广泛, 但使用启动子在花器官中导致更低的编辑效率[20-21,35-36]。本研究中的7个启动子中,、和的表达呈现非特异性广泛表达, 其驱动的基因在各组织器官都有表达; 而基因主要在胚囊、根尖、胚胎、胚乳等细胞分裂活跃的部位表达[23],启动子驱动的基因在胚胎发育早期就已经激活, 从植物卵细胞开始表达, 并表现出组成型表达模式[25,37]。另有研究表明, 特异性的卵细胞表达启动子在拟南芥中能够在T1代获得稳定的纯合植株[38]。在水稻中, 使用启动子驱动进行基因编辑比启动子效果更好, 这可能与启动子能够在愈伤组织中高表达及水稻通过愈伤组织的转化方法相关。根据以上这些研究结果, 不同的启动子会导致的表达模式发生变化,因此推测造成这些启动子对下游靶位点编辑效率的影响很可能与不同启动子的表达时期及植物的转化方法相关。本研究中采用大豆子叶节诱导发根的转化方法, 根据已有的这些研究结果, 推测在农杆菌侵染部位表达量高的启动子可能获得较高的转化效率, 这需要进一步实验证实。
本研究在试验过程中, 对基因使用了不同的启动子, 但并没有对的表达量进行检测。因为本研究通过发根检测的方法检验靶基因和的突变情况, 提取单根发状根的DNA, 无法同时对单根提取蛋白检测的表达量和提取DNA检测编辑情况。已有的研究结果显示, CRISPR/Cas9系统的编辑效率与的表达量相关, 而可能与的表达量关联性较低, 反而与基因的表达时期和表达位置高度相关[20], 因此推测虽然不同启动子可能造成的表达量不同, 但对靶基因的编辑效率影响可能有限。目前, 根据前期的试验结果, 后续将在稳定转化植株中继续检测使用这7个启动子对下游基因的编辑效率的影响, 从而进一步验证本研究的试验结果。
本研究中首次提出了在大豆发状根中判断CRISPR/Cas9系统编辑效果的方法, 即通过观察发状根中杂合突变体测序峰图的状态, 定性判断编辑的效果, 为T1代获得纯合突变体的概率提出参考。到目前为止, 还未见类似报道, 但测序峰图中的高峰和低峰的判定, 与测序产物中突变体DNA分子的数量相关, 即含量越多靶位点DNA, 则测序峰图随之变高, 那么在下一代分离出突变体的概率相对增高, 这为该方法提供了理论支持。在获得稳定的大豆转基因植株后, 将通过计算T0到T1代获得纯合突变体概率, 准确判定这种新方法的准确性。
4 结论
通过本研究, 本研究证明了大豆内源的和外源驱动的表达对下游靶基因的编辑效率最高、编辑效果最优, 是大豆CRISPR/Cas9系统中最适合的启动子; 而大豆内源、子和编辑效率偏低, 不适合作为启动子;在大豆CRISPR/Cas9系统中的编辑效率低于和, 但可作为启动子使用。本研究还提出了一种判定CRISPR/Cas9系统编辑效果的新方法, 通过测量发状根中靶位点的峰图的形态(高峰/低峰)来判断编辑效果, 这将为判定在下一代中获得纯合突变体植株的概率提供参考。
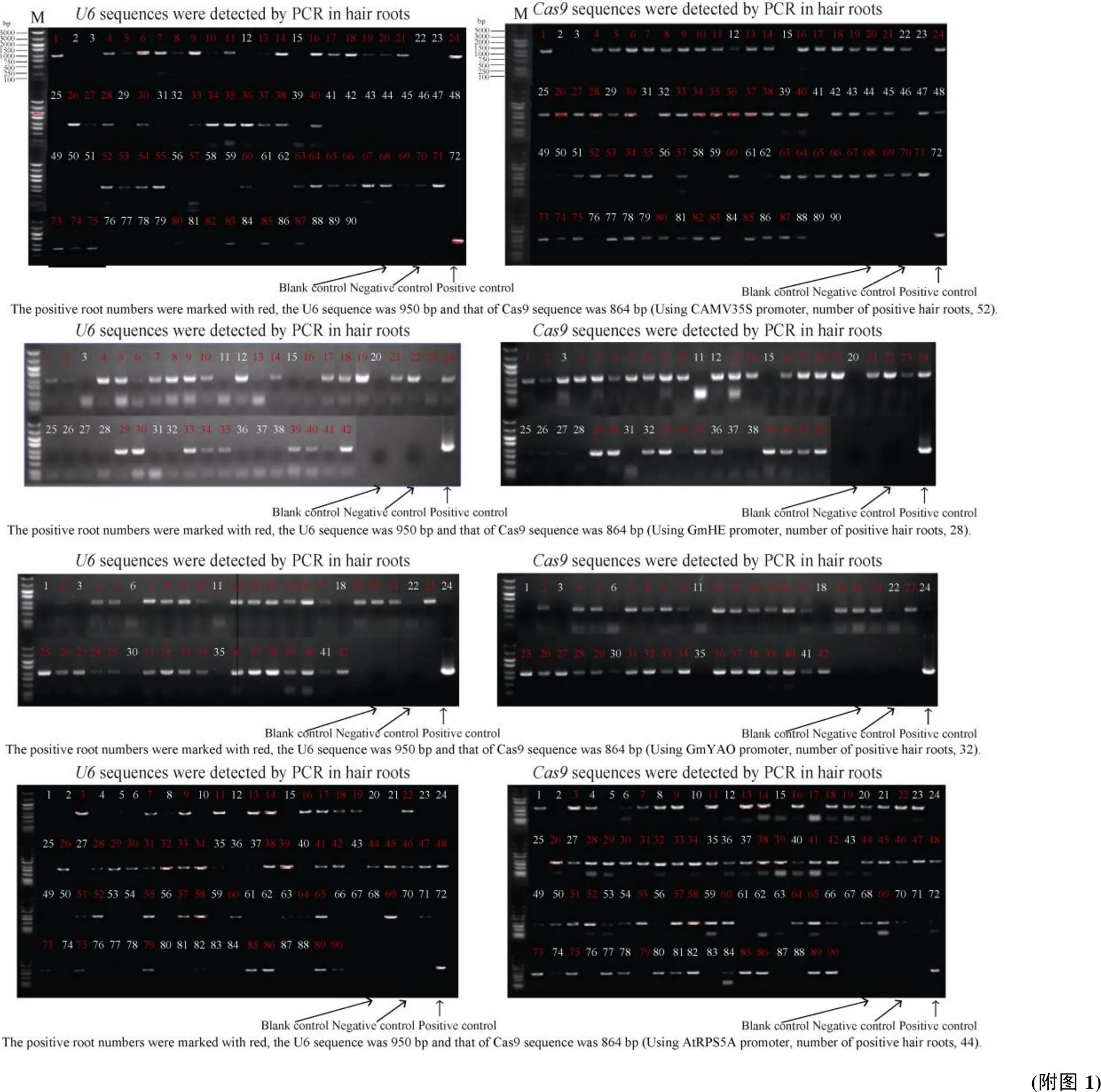
附图1 PCR检测与序列电泳图
Fig. S1 Electrophoresis map of U6 and Cas9 sequence by PCR detection
使用启动子,在发状根中PCR检测单根与序列电泳图。M: 分子量 marker;空白对照:水,阴性对照:天隆1号基因组DNA;阳性对照:带有、序列质粒。同时检测到序列与序列的阳性根编号用红色数字标识,序列大小为950 bp,序列大小为864 bp。
Using,,,,,promoter,andsequences were detected by PCR in hair roots. M: molecular weight marker; Blank control: pure water; Negative control: TL1 genomic DNA; Positive control: plasmid withandsequences. The positive root numbers were marked with red, thesequence was 950 bp and that ofsequence was 864 bp.
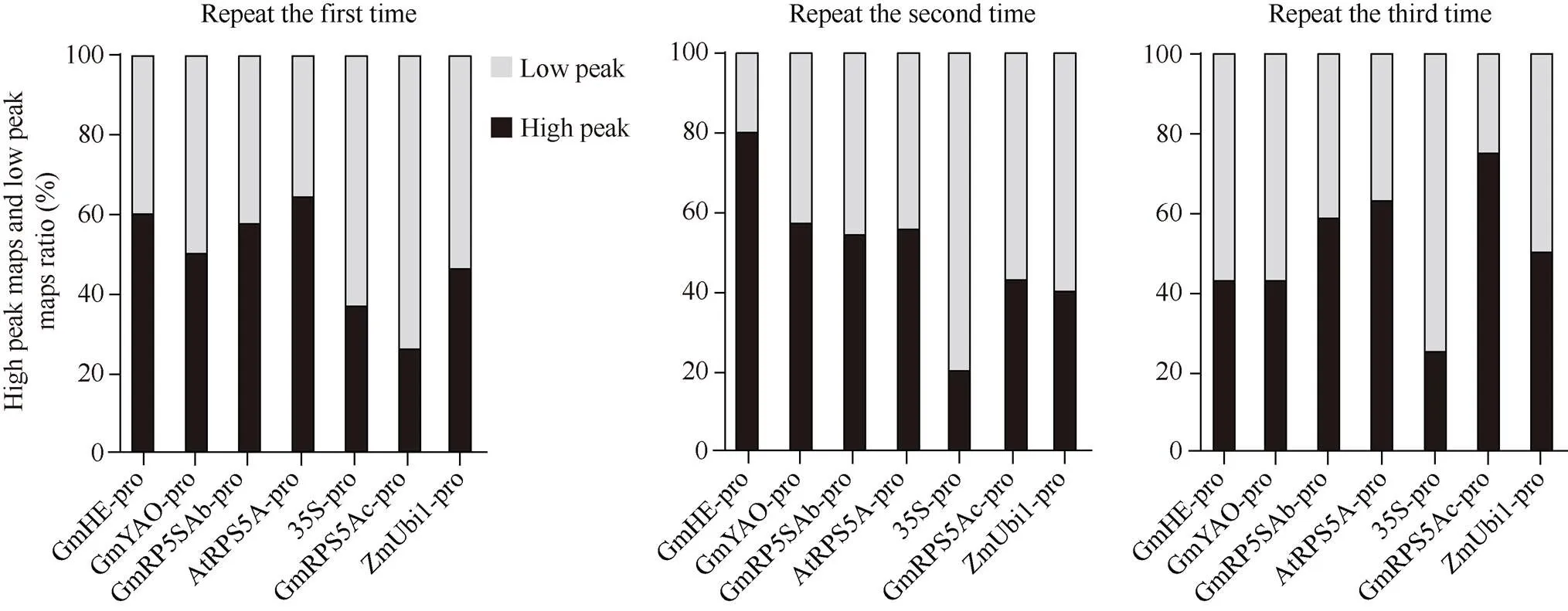
附图2 统计第1次、第2次、第3次发根转化中7个启动子对编辑峰图中高峰和低峰比例
Fig. S2 The high peak maps and low peak maps ratio of 7 promoters inediting sites sequencing maps during the first, second and third root transformation
黑色柱代表测序峰图中高峰比例,灰色代表测序峰图中低峰比例。
Black column represents the high peak maps, and gray represents the low peak maps in sequenced maps.
[1] Manghwar H, Lindsey K, Zhang X, Jin S. CRISPR/Cas system: recent advances and future prospects for genome editing., 2019, 24: 1102–1125.
[2] Christian M, Cermak T, Doyle E L, Schmidt C, Zhang F, Hummel A, Bogdanove A J, Voytas D F. Targeting DNA double-strand breaks with TAL effector nucleases., 2010, 186: 757–761.
[3] Wright D A, Townsend J A, Winfrey R J Jr, Irwin P A, Rajagopal J, Lonosky P M, Hall B D, Jondle M D, Voytas D F. High-frequency homologous recombination in plants mediated by zinc-finger nucleases, 2005, 44: 693–705.
[4] Nekrasov V, Staskawicz B, Weigel D, Jones J D, Kamoun S. Targeted mutagenesis in the model plantusing Cas9 RNA-guided endonuclease., 2013, 31: 691–693.
[5] Li J F, Norville J E, Aach J, McCormack M, Zhang D, Bush J, Church G M, Sheen J. Multiplex and homologous recombination-mediated genome editing inandusing guide RNA and Cas9., 2013, 31: 688–691.
[6] Sorek R, Lawrence C M, Wiedenheft B. CRISPR-mediated adaptive immune systems in bacteria and archaea., 2013, 82: 237–266.
[7] Garneau J E, Dupuis M È, Villion M, Romero D A, Barrangou R, Boyaval P, Fremaux C, Horvath P, Magadán A H, Moineau S. The CRISPR/Cas bacterial immune system cleaves bacteriophage and plasmid DNA., 2010, 468: 67–71.
[8] Jinek M, Chylinski K, Fonfara I, Hauer M, Doudna J A, Charpentier E. A programmable dual-RNA-guided DNA endonuclease in adaptive bacterial immunity., 2012, 337: 816–821.
[9] Mohanraju P, Makarova K S, Zetsche B, Zhang F, Koonin E V, van der Oost J. Diverse evolutionary roots and mechanistic variations of the CRISPR-Cas systems., 2016, 353: aad5147.
[10] Wright A V, Nuñez J K, Doudna J A. Biology and applications of CRISPR systems: harnessing nature’s toolbox for genome engineering., 2016, 164: 29–44.
[11] Komor A C, Badran A H, Liu D R. CRISPR-based technologies for the manipulation of eukaryotic genomes., 2017, 168: 20–36.
[12] Bao A, Burritt D J, Chen H, Zhou X, Cao D, Tran L P. The CRISPR/Cas9 system and its applications in crop genome editing., 2019, 39: 321–336.
[13] Chen K, Wang Y, Zhang R, Zhang H, Gao C. CRISPR/Cas genome editing and precision plant breeding in agriculture., 2019, 70: 667–697.
[14] Yue J J, Hong C Y, Wei P C, Tsai Y C, Lin C S. How to start your monocot CRISPR/Cas project: plasmid design, efficiency detection, and offspring analysis., 2020, 13: 9.
[15] Zheng X, Deng W, Luo K, Duan H, Chen Y, McAvoy R, Song S, Pei Y, Li Y. The cauliflower mosaic virus (CaMV) 35S promoter sequence alters the level and patterns of activity of adjacent tissue- and organ-specific gene promoters., 2007, 26: 1195–1203.
[16] Odell J T, Nagy F, Chua N H. Identification of DNA sequences required for activity of the cauliflower mosaic virus 35S promoter., 1985, 313: 810–812.
[17] Bevan M. Binaryvectors for plant transformation., 1984, 12: 8711–8721.
[18] Cornejo M J, Luth D, Blankenship K M, Anderson O D, Blechl A E. Activity of a maize ubiquitin promoter in transgenic rice., 1993, 23: 567–581.
[19] Christensen A H, Sharrock R A, Quail P H. Maize polyubiquitin genes: structure, thermal perturbation of expression and transcript splicing, and promoter activity following transfer to protoplasts by electroporation., 1992, 18: 675–689.
[20] Ma X L, Zhang Q Y, Zhu Q L, Liu W, Chen Y, Qiu R, Wang B, Yang Z F, Li H Y, Lin Y, Xie Y Y, Shen R X, Chen S F, Wang Z, Chen Y L, Guo J X, Chen L T, Zhao X C, Dong Z C, Liu Y G. A robust CRISPR/Cas9 system for convenient, high-efficiency multiplex genome editing in monocot and dicot plants., 2015, 8: 1274–1284.
[21] Feng Z Y, Zhang B T, Ding W, Liu X D, Yang D L, Wei P L, Cao F Q, Zhu S H, Zhang F, Mao Y F, Zhu J K. Efficient genome editing in plants using a CRISPR/Cas system., 2013, 23: 1229–1232.
[22] Yan L H, Wei S W, Wu Y R, Hu R L, Li H J, Yang W C, Xie Q. High-efficiency genome editing inusing YAO promoter-driven CRISPR/Cas9 system., 2015, 8: 1820–1823.
[23] Li H J, Liu N Y, Shi D Q, Liu J, Yang W C. YAO is a nucleolar WD40-repeat protein critical for embryogenesis and gametogenesis in., 2010, 10: 169.
[24] Choi M, Yun J Y, Kim J H, Kim J S, Kim S T. The efficacy of CRISPR-mediated cytosine base editing with the RPS5a promoter in., 2021, 11: 8087.
[25] Weijers D, Franke-van Dijk M, Vencken R J, Quint A, Hooykaas P, Offringa R. Anminute-like phenotype caused by a semi-dominant mutation in agene., 2001, 128: 4289–4299.
[26] Cai Y, Chen L, Liu X, Sun S, Wu C, Jiang B, Han T, Hou W. CRISPR/Cas9-mediated genome editing in soybean hairy roots., 2015, 10: e0136064.
[27] Cai Y, Chen L, Sun S, Wu C, Yao W, Jiang B, Han T, Hou W. CRISPR/Cas9-mediated deletion of large genomic fragments in soybean.,2018, 19: 3835.
[28] Cai Y, Chen L, Liu X, Guo C, Sun S, Wu C, Jiang B, Han T, Hou W. CRISPR/Cas9-mediated targeted mutagenesis of GmFT2a delays flowering time in soya bean., 2018, 16: 176–185.
[29] Li C, Li Y H, Li Y, Lu H, Hong H, Tian Y, Li H, Zhao T, Zhou X, Liu J, Zhou X, Jackson S A, Liu B, Qiu L J. A domestication-associated gene GmPRR3b regulates the circadian clock and flowering time in soybean., 2020, 13: 745–759.
[30] Carrijo J, Illa-Berenguer E, LaFayette P, Torres N, Aragão F J L, Parrott W, Vianna G R. Two efficient CRISPR/Cas9 systems for gene editing in soybean., 2021, 30: 239–249.
[31] Wang L, Cao C, Ma Q, Zeng Q, Wang H, Cheng Z, Zhu G, Qi J, Ma H, Nian H, Wang Y. RNA-seq analyses of multiple meristems of soybean: novel and alternative transcripts, evolutionary and functional implications., 2014, 14: 169.
[32] 秦超.和基因改良大豆纬度适应性的机制研究. 中国农业科学院博士学位论文, 北京, 2022. Qin C. Mechanism Study of theandGenes in Improving the Latitudinal Adaptability of Soybean. PhD Dissertation of Chinese Academy of Agricultural Sciences, Beijing, China, 2022 (in Chinese with English abstract).
[33] 秦超. 大豆中基因的功能分析. 中国农业科学院硕士学位论文, 北京, 2018. Qin C. Function Analysis ofin Soybean. MS Thesis of Chinese Academy of Agricultural Sciences, Beijing, China, 2018 (in Chinese with English abstract).
[34] Oh Y, Kim S G. RPS5A Promoter-driven Cas9 produces heritable virus-induced genome editing in., 2021, 44: 911–919.
[35] Tsutsui H, Higashiyama T. pKAMA-ITACHI vectors for highly efficient CRISPR/Cas9-mediated gene knockout in., 2017, 58: 46–56.
[36] Ye G N, Stone D, Pang S Z, Creely W, Gonzalez K, Hinchee M.ovule is the target for Agrobacterium in planta vacuum infiltration transformation., 1999, 19: 249–257.
[37] Maruyama D, Hamamura Y, Takeuchi H, Susaki D, Nishimaki M, Kurihara D, Kasahara R D, Higashiyama T. Independent control by each female gamete prevents the attraction of multiple pollen tubes., 2013, 25: 317–323.
[38] Wang Z P, Xing H L, Dong L, Zhang H Y, Han C Y, Wang X C, Chen Q J. Egg cell-specific promoter-controlled CRISPR/Cas9 efficiently generates homozygous mutants for multiple target genes inin a single generation., 2015, 16: 144.
Function analysis of differentpromoters on the efficiency of CRISPR/ Cas9 system in soybean
NIU Zhi-Yuan1,2, QIN Chao1, LIU Jun1, WANG Hai-Ze2,*, and LI Hong-Yu1,*
1Institute of Crop Sciences, Chinese Academy of Agricultural Sciences, Beijing 100081, China;2College of Agriculture, Heilongjiang Bayi Agricultural University, Daqing 163319, Heilongjiang, China
The CRISPR/Cas9 system has been widely used in plants and animals as an efficient gene editing system. Severalpromoters, such asand, have been reported to improve the efficiency of gene editing in CRISPR/Cas9 system. In soybean, the influence of differentpromoters on the efficiency of the CRISPR/Cas9 gene-editing system has not been elucidated. In this study, six efficient promoters with known functions (,,,,, and) and one endogenous soybean promoter with unknown function () were selected to construct the CRISPR/Cas9 knockout vectors. The editing efficiency of thepromoters on the endogenous soybean genesandwas tested bymediated hair roots system, which indicated that soybean endogenous promoterhad the highest editing efficiency. The editing efficiency of,, andwere higher than that ofand. Further analysis of the sequencing maps of target sites showed that the high peak maps accounted for 64.0% and 58.6% in the sequencing maps ofandpromoters, respectively, while in the sequencing maps of-driven hair roots, the low peak accounted for a higher proportion (63.3%). These above results indicated thatandpromoters not only had high editing efficiency, but also had better editing effect, and were more conducive to the isolation of homozygous mutants in the next generation. In conclusion, this study provide the reference for the construction of efficient soybean CRISPR/Cas9 vectors and help to improve the efficiency of soybean gene editing.
gene editing; CRISPR/Cas9; hair roots system; promoter; soybean
2023-05-24;
2023-05-30.
10.3724/SP.J.1006.2023.24285
通信作者(Corresponding author): 李宏宇, E-mail: lihongyu@caas.cn; 王海泽, E-mail: haizewang@163.com
E-mail: niuzhiyuan2022@126.com
2022-12-24;
本研究由中国农业科学院科技创新工程-作物生物信息学及应用项目(2060302-2-20)和大豆藏粮于技项目(CAAS-ZDRW202003)资助。
This study was supported by the Agricultural Science and Technology Innovation Program of Chinese Academy of Agricultural Sciences (2060302-2-20) and the Soybean Grain Storage Technology Program (CAAS-ZDRW202003).
URL: https://kns.cnki.net/kcms2/detail/11.1809.S.20230529.1803.002.html
This is an open access article under the CC BY-NC-ND license (http://creativecommons.org/licenses/by-nc-nd/4.0/).

