基于单细胞测序数据的骨肉瘤预后相关细胞亚群特征分析
王胜涛 朱浩然 徐淑娟 贵鹏 陈明洲 李朝旭


基金項目:广西医疗卫生重点培育学科建设项目(桂卫教科发〔2022〕4号)
摘要:目的 通过分析不同骨肉瘤患者肿瘤组织的细胞组成,探究与骨肉瘤预后相关的细胞亚群及特征。方法 下载不同患者骨肉瘤单细胞测序数据及bulk测序数据,提取细胞样本信息进行降维、注释、细胞功能分析,明确与骨肉瘤预后相关的细胞种类,鉴定骨肉瘤预后相关细胞特征。分析具有预后意义的巨噬细胞发育轨迹,根据分化空间差异基因构建骨肉瘤预后模型。结果 不同骨肉瘤患者的细胞组成存在异质性。骨肉瘤中浸润的单核巨噬细胞具有显著预后意义(P=0.003)。4类巨噬细胞组成关键预后单核巨噬细胞亚群,其特征性转录调控因子包括RUNX3(+)、ETS1(+)、HOXD11(+)、ZNF281(+)、PRRX1(+)。巨噬细胞亚群的预后能力随骨肉瘤的恶性进展而增强,其中Prog_Macro2及Prog_Macro4亚群巨噬细胞位于发育轨迹终端。基于巨噬细胞分化差异基因的预后模型能够有效区分不同风险骨肉瘤患者的生存率(P<0.001)。结论 巨噬细胞亚群与骨肉瘤预后密切相关,可作为骨肉瘤免疫治疗的关键靶细胞。
关键词:骨肉瘤;单细胞测序;单核巨噬细胞系统;巨噬细胞;预后
中图分类号: R738.1 文献标志码: A 文章编号:1000-503X(2023)05-0773-10
DOI:10.3881/j.issn.1000-503X.15573
Characterization of Cell Subsets Associated With Prognosis of Osteosarcoma Based on Single-Cell Sequencing Data
WANG Shengtao1,ZHU Haoran2,XU Shujuan3,GUI Peng4,CHEN Mingzhou1,LI Zhaoxu1
1Department of Joint Surgery and Sports Medicine,Nanxishan Hospital of Guangxi Zhuang Autonomous Region,Guilin,Guangxi 541002,China
2Zonglian College,Xian Jiaotong University,Xian 710061,China
3Department of Hematology,Affiliated Hospital of Guilin Medical University,Guilin,Guangxi 541001,China
4Department of Trauma and Hand Surgery,Nanxishan Hospital of Guangxi Zhuang Autonomous Region,Guilin,Guangxi 541002,China[JZ)]
Corresponding author:LI Zhaoxu Tel:0773-3830680,E-mail:lzxpds@126.com
ABSTRACT:Objective To explore the cell subsets and characteristics related to the prognosis of osteosarcoma by analyzing the cellular composition of tumor tissue samples from different osteosarcoma patients.Methods The single-cell sequencing data and bulk sequencing data of different osteosarcoma patients were downloaded.We extracted the information of cell samples for dimensionality reduction,annotation,and cell function analysis,so as to identify the cell subsets and clarify the cell characteristics related to the prognosis of osteosarcoma.The development trajectory of macrophages with prognostic significance was analyzed,and the prognostic model of osteosarcoma was established based on the differentially expressed genes of macrophage differentiation.Results The cellular composition presented heterogeneity in the patients with osteosarcoma.The infiltration of mononuclear phagocytes in osteosarcoma had prognostic significance(P=0.003).Four macrophage subsets were associated with prognosis,and their signature transcription factors included RUNX3(+),ETS1(+),HOXD11(+),ZNF281(+),and PRRX1(+).Prog_Macro2 and Prog_Macro4 were located at the end of the developmental trajectory,and the prognostic ability of macrophage subsets increased with the progression of osteosarcoma.The prognostic model established based on the differentially expressed genes involved in macrophage differentiation can distinguish the survival rate of osteosarcoma patients with different risks(P<0.001).Conclusion Macrophage subsets are closely related to the prognosis of osteosarcoma and can be used as the key target cells for the immunotherapy of osteosarcoma.
Key words:osteosarcoma;single-cell sequencing;mononuclear phagocyte system;macrophage;prognosis
Acta Acad Med Sin,2023,45(5):773-782
骨肉瘤多起源于四肢长骨,是儿童和青少年群体最常见的原发恶性骨肿瘤[1]。不同病理亚型骨肉瘤的治疗方式及预后完全不同[2],了解骨肉瘤组织细胞组成特征及其特异性功能是临床防治工作开展的重要基础。骨肉瘤组织中的肿瘤细胞、各类免疫细胞以及细胞外基质共同形成骨肉瘤微环境,参与癌变后免疫机制以及骨肉瘤的发生发展,并直接影响患者的诊断治疗及预后[3]。单细胞测序技术能够对癌细胞基因组、转录组进行全面且精准的分析[4],已应用于胃癌[5]、肺癌[6]、乳腺癌[7]等多种肿瘤中。结合R语言等生物信息学分析技术,单细胞测序技术能够捕获不同肿瘤的细胞组成,揭示肿瘤微环境的异质性[8]。然而,目前缺乏单细胞水平的关于骨肉瘤浸润细胞及特征的相关研究。本研究旨在利用单细胞转录测序数据,对骨肉瘤浸润细胞中具有预后意义的细胞群体进行鉴定与分析,以期为临床骨肉瘤防治工作的开展提供理论依据。
资料和方法
资料来源 骨肉瘤单细胞数据集来源于GSE152048[9],共计纳入11例样本。骨肉瘤bulk RNA测序数据集从TARGET数据库(https://ocg.cancer.gov/programs/target)下载,使用R软件TCGAbiolinks包进行分析处理。骨肉瘤样本生存数据从UCSC Xena数据库(http://xena.ucsc.edu/)下载。
数据处理 使用R软件Seurat包[10]筛选出骨肉瘤单细胞数据集中线粒体基因比例<20%的细胞,为避免骨肉瘤中癌变细胞的差异在数据整合中被消除,分析过程并未去除样本的批次效应。
降维聚类 使用RunPCA函数及RunUMAP函数对细胞进行降维聚类,设置dims为1∶50。
细胞注释 结合CellTypist数据库(https://www.celltypist.org/)及R软件SingleR包[11]对筛选后的细胞群进行注释。
R软件工具包 调用R软件Scissor包[12]整合骨肉瘤的bulk测序数据、对应的生存数据以及单细胞数据,检测单细胞样本中对预后具有影响的细胞。调用R软件BayesPrism包[13],将单细胞数据投射到bulk测序数据中以观测每一个bulk测序样本中细胞的组成以及比例差异,同时调用R软件Survival包结合bulk测序样本的细胞比例以及生存数据分析具有预后意义的细胞群。调用R软件pyscenic包[14]分析不同巨噬细胞亚群中转录调控因子的富集差异,从而确定具有显著预后意义的巨噬细胞亚群所具有的转录调控因子富集特征。Monocle3[15]和CytoTRACE包[16]被用来探究骨肉瘤中巨噬细胞的发育轨迹及在细胞发育过程中起关键作用的基因。
预后模型的构建 提取Monocle3计算的在巨噬细胞的UMAP降维图中按照莫兰指数排位前40的基因,采用LASSO回归法构建风险预后模型。将TARGET数据库的85例骨肉瘤组织的bulk测序数据分为训练集(n=43)和测试集(n=42),基于风险得分将患者分为高风险组和低风险组,采用Log-rank检验比较高低风险组患者的生存差异,受试者工作特征(receiver operating characteristic,ROC)曲线可视化风险模型对患者生存的预测效能。
结果
骨肉瘤单细胞样本中细胞的分布与特征 11例骨肉瘤单细胞测序数据经质控、整合、归一化处理后对筛选细胞进行降维聚类,结果显示不同骨肉瘤患者的肿瘤组织中存在多种细胞群,不同细胞群位置離散(图1A);不同骨肉瘤单细胞样本中细胞的组成及比例有着较大差异(图1B)。11例骨肉瘤样本中共鉴定出16种细胞类型(图1C),主要包括成骨细胞、单核巨噬细胞、T细胞、周细胞、成纤维细胞等(图1D)。使用R软件Scissor包对单细胞数据和bulk测序数据联合分析,将具有预后意义的细胞标注在UMAP降维图中,结果显示具有较差预后意义的细胞多为癌变的成骨细胞,而具有较好预后意义的细胞多为单核巨噬细胞(图1E)。
骨肉瘤组织中预后相关细胞群的鉴定 采用R软件BayesPrism包将单细胞的注释结果投射到bulk测序数据中,显示不同bulk测序样本中系细胞比例(图2A)。采用R软件survival包分析不同占比细胞的预后差异,筛选骨肉瘤微环境中具有预后意义的细胞群,生存分析结果显示高浸润比例的单核巨噬细胞(P=0.003)、周细胞(P=0.003)与增殖性成骨细胞(P=0.014)与较好的预后相关(图2B~2D)。
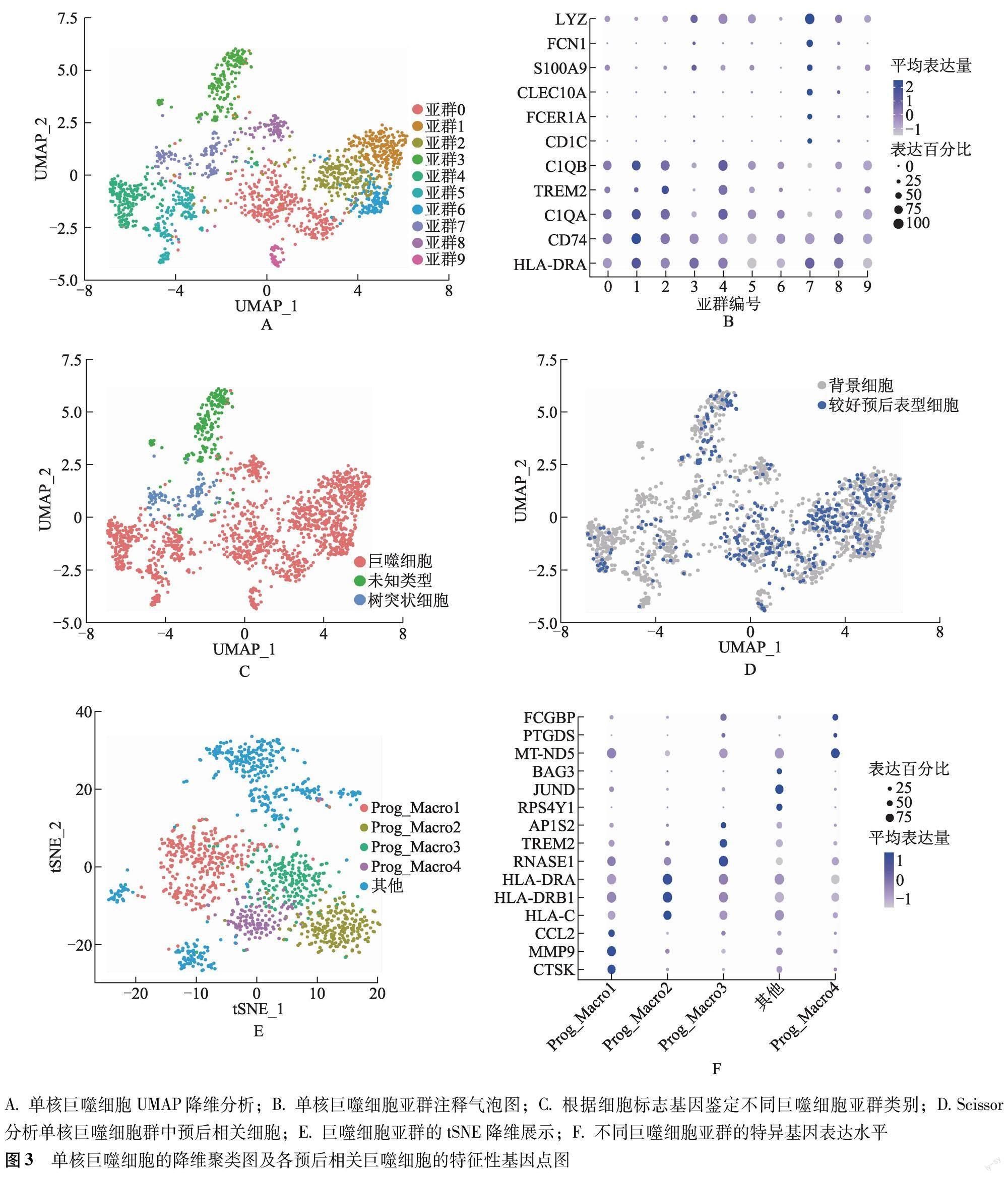
骨肉瘤单核巨噬细胞亚群的组成及鉴定 将单核巨噬细胞降维聚类分为9个亚群(图3A),根据已知单核巨噬细胞marker鉴定细胞亚群类别,气泡图注释浸润单核巨噬细胞亚群标志基因的表达水平(图3B)。调用R软件Scissor包分析单核巨噬细胞群中具有预后意义的细胞,结果显示除背景细胞外单核巨噬细胞均具有较好预后意义(图3C),其中具有较强预后意义的细胞亚群主要为巨噬细胞,以0、1、2、6亚群最为显著(图3D)。对巨噬细胞0、1、2、6亚群重新进行降维聚类,采用Prog_Macro1~4命名(图3E),结果显示Prog_Macro1亚群的特征基因为CTSK、MMP9、CCL2;Prog_Macro2亚群的特征基因为HLA-C、HLA-DRA、HLA-DRB1;Prog_Macro3亚群的特征基因为RNASE1、TREM2、AP1S2;Prog_Macro4的特征基因为MT-ND5、PTGDS、FCGBP(图3F)。
不同巨噬细胞亚群中特征转录调控因子富集分析 采用pyscenic算法对巨噬细胞的转录调控因子进行富集分析(图4A),在每种巨噬细胞亚群中,挑选出差异富集程度排位前5的转录调控因子作为该细胞亚群的特征转录调控因子(图4B)。具有较强预后意义的1、2、6巨噬细胞亚群(Prog_Macro2~4)均差异富集RUNX3(+)和ETS1(+),而0亚群(Prog_Macro1)则差异富集ZNF281(+),同时,可采用PRRX1(+)和HOXD11(+)将0亚群和无预后意义的9亚群区分开(图4C~4E)。
骨肉瘤浸润巨噬细胞亚群的发育轨迹 CytoTRACE预测结果显示,骨肉瘤中无预后意义的巨噬细胞亚群的分化潜能大于有预后意义的巨噬细胞亚群,其中Prog_Macro4分化潜能最低(图5A、5B)。Monocle3计算结果显示,巨噬细胞群可能由无预后意义的细胞群开始分化,并逐渐形成有预后意义的细胞群。同时,代表Prog_Macro2和Prog_Macro4的细胞亚群位于发育轨迹的终端,与CytoTRACE的预测结果一致(图5C、5D)。
基于巨噬细胞分化空间差异基因的预后模型构建 基于最小绝对收缩和选择算子Cox回归算法筛选出的7个特征基因构建风险评估模型,公式为风险评分=PLIN2×(-1.448)+FTL×(-1.274)+HAPLN1×(-0.584)+SLAMF9×0.895+ACKR3×0.516+RPS20×2.777+VEGFA×0.826,生存曲线显示高风险患者的预后更差(P均<0.01)(图6A),模型对1、3和5年患者生存率具有较好的区分度,曲线下面积大于0.75(图6B)。
讨论
骨肉瘤是最常见的原发性恶性骨肿瘤,具有恶性度高、生长迅速、早期转移等特点[17],其预后是患者及临床医生的关注焦点。然而目前缺乏有效指标预测骨肉瘤的预后进展。本研究基于单细胞测序数据,从单细胞水平对骨肉瘤组织浸润细胞的组成与特征展开探究,揭示骨肉瘤预后相关細胞亚群,为临床治疗提供新的思路。
本研究所采用的测序数据来源于TARGET和GEO数据库。11例样本数据降维分析结果显示,不同骨肉瘤患者组织浸润的细胞类型包括成骨细胞、单核巨噬细胞、破骨细胞、T细胞、间充质细胞、内皮细胞、骨髓基质干细胞、周细胞和成纤维细胞等,不同患者肿瘤组织浸润细胞异质性较高。其中,成骨细胞亚群种类较多,包括MT1X/MT1G+、PCP4+、IBSP+、PTGDS/EGFL6+等不同标志亚群,提示成骨细胞在骨肉瘤中的广泛分化与特异性作用。现有文献报道骨肉瘤中成骨细胞的分化与临床进展密切相关[18],研究显示骨钙素是目前成骨细胞较好的特异性标志物,高表达骨钙素的骨肉瘤患者生存率较高[19]。本研究更详尽地描述了骨肉瘤中成骨细胞的分化特征,并确定了MT1X/MT1G、PCP4、IBSP、PTGDS/EGFL6等基因为其主要的差异表达基因。
免疫治疗是目前肿瘤临床治疗的新型手段。研究表明增强人体的免疫功能可能改善骨肉瘤患者的预后[20]。
通常免疫系统能够识别人体癌变的组织和细胞[21],然而肿瘤细胞的免疫逃逸机制使免疫系统失灵,因此,肿瘤免疫治疗旨在通过激活体内失效的免疫系统,进而达到杀伤肿瘤细胞的目的[22]。CD8+和CD4+ T细胞是杀伤肿瘤细胞的主要效应细胞,也是针对肿瘤微环境进行免疫治疗的主要细胞靶点[23]。然而,本研究发现骨肉瘤组织中虽然有大量T细胞浸润,然而其表达水平与预后无相关性。但骨肉瘤组织中浸润的单核巨噬细胞以及周细胞和增殖性成骨细胞与患者预后呈显著正相关。为进一步探究单核巨噬细胞在骨肉瘤中的重要作用,本研究根据细胞标志基因表达差异将单核巨噬细胞分为9个不同亚群,其中巨噬细胞与预后关联最显著。研究表明肿瘤浸润的单核巨噬细胞在肿瘤微环境中既承担主要抗原提呈细胞的功能,也是肿瘤发生发展的关键调节因子[24]。虽然多项靶向单核巨噬细胞的治疗策略已经进入临床前试验阶段[25-26],然而由于缺乏对肿瘤微环境复杂性的了解,单核巨噬细胞在肿瘤中的关键作用机制尚不清楚。本研究发现巨噬细胞在骨肉瘤预后中的关键作用,并对其特征进行了进一步标注。9种单核巨噬细胞亚群中,3、7亚群细胞经鉴定为树突状细胞和未知细胞,0、1、2、6亚群细胞为不同特征的巨噬细胞(命名为Prog_Macro1~4),且均能提示较好的骨肉瘤预后。本文通过分析上述细胞亚群的转录调控因子对细胞进行鉴别标志。结果显示,1、2、6亚群为RUNX3(+)+、ETS1(+)+、HOXD11(+)-巨噬细胞群,0亚群为ZNF281(+)+、PRRX1(+)-巨噬细胞群。这些巨噬细胞亚群对骨肉瘤均具有关键的预后价值。此外,巨噬细胞发育分析轨迹表明,巨噬细胞并非从肿瘤初始阶段即存在预后效应,而是随着骨肉瘤的不断进展,持续分化的巨噬细胞才具有预测预后的价值。在具有预后意义的巨噬细胞群体中,Prog_Macro2和Prog_Macro4细胞亚群位于发育终端,且空间差异表达基因的表达下调。使用这些差异基因构建模型能够对骨肉瘤预后及风险进行有效预测。而无预后意义的巨噬细胞的具体作用及特征有待进一步研究。
近年来有多项研究通过单细胞测序技术揭示了骨肉瘤微环境的异质性以及浸润的免疫细胞对患者预后的影响。Liu等[27]研究表明骨肉瘤组织中以CD83+、CCR7+、LAMP3+为标志物的成熟调节性树突状细胞可通过募集Treg细胞促进肿瘤免疫耐受,进而影响患者的预后。Tu等[28]以及He等[29]研究结果提示骨肉瘤微环境中浸润的C1Q+和LPAR5+巨噬细胞具有较好的预后意义。这些研究主要根据CIBERSORTx算法推断bulk测序样本中免疫细胞富集程度,并以此为依据将收集样本划分为高低浸润组,跟据生存情况计算高低浸润组生存结局及差异。然而,这种方法仅对推断异质性较强的细胞亚型有效。骨肉瘤中浸润的巨噬细胞各亚型间基因表达谱相似,CIBERSORTx算法难以获得足够的特征基因标志,进而丢失部分细胞对于患者预后的影响。本研究所采用的Scissor算法可以避免细胞亚型间的基因表达谱过于相似对推断结果造成的干扰,进而对骨肉瘤组织中影响患者预后的巨噬细胞亚群进行更准确推断。同时,结合拟时序算法monocle3以及细胞干性算法CytoTrace对具有不同预后意义的巨噬细胞亚群出现的先后进行推测,分析了骨肉瘤浸润巨噬细胞在不同的分化阶段对患者预后影响的差异。Zhou等[9]虽然也对骨肉瘤微环境中浸润的巨噬细胞进行了系统分析,但只做了传统的M1、M2以及脂肪酸结合蛋白4+肿瘤相关巨噬细胞分型,同时,也缺乏骨肉瘤浸润巨噬细胞对于患者预后影响的分析。而本研究进一步完善了骨肉瘤中巨噬细胞群的不同分型及其对于患者预后的影响。
綜上,本研究结果表明,巨噬细胞亚群与骨肉瘤预后密切相关,可作为骨肉瘤免疫治疗的关键靶细胞。然而本研究未针对不同病理亚型的骨肉瘤患者的细胞组成及预后情况进行分析,未来应扩大样本类型进行更深入的研究;同时,应关注无预后意义的巨噬细胞群体在骨肉瘤中所起到的潜在作用。
参考文献
[1]Czarnecka AM,Synoradzki K,Firlej W,et al.Molecular biology of osteosarcoma[J].Cancers,2020,12(8):2130.DOI:10.3390/cancers12082130.
[2]Gill J,Gorlick R.Advancing therapy for osteosarcoma[J].Nat Rev Clin Oncol,2021,18(10):609-624.DOI:10.1038/s41571-021-00519-8.
[3]Yang C,Tian Y,Zhao F,et al.Bone microenvironment and osteosarcoma metastasis[J].Int J Mol Sci,2020,21(19):6985.DOI:10.3390/ijms21196985.
[4]Tang X,Huang Y,Lei J,et al.The single-cell sequencing:new developments and medical applications[J].Cell Biosci,2019,9(1):1-9.DOI:10.1186/s13578-019-0314-y.
[5]Sathe A,Grimes SM,Lau BT,et al.Single-cell genomic characterization reveals the cellular reprogramming of the gastric tumor microenvironment[J].Clin Cancer Res,2020,26(11):2640-2653.DOI:10.1158/1078-0432.CCR-19-3231.
[6]Maynard A,McCoach CE,Rotow JK,et al.Therapy-induced evolution of human lung cancer revealed by single-cell RNA sequencing[J].Cell,2020,182(5):1232-1251.e22.DOI:10.1016/j.cell.2020.07.017.
[7]Grosselin K,Durand A,Marsolier J,et al.High-throughput single-cell ChIP-seq identifies heterogeneity of chromatin states in breast cancer[J].Nat Genet,2019,51(6):1060-1066.DOI:10.1016/j.cell.2020.07.017.
[8]Levitin HM,Yuan J,Sims PA.Single-cell transcriptomic analysis of tumor heterogeneity[J].Trends Cancer,2018,4(4):264-268.DOI:10.1016/j.trecan.2018.02.003.
[9]Zhou Y,Yang D,Yang Q,et al.Single-cell RNA landscape of intratumoral heterogeneity and immunosuppressive microenvironment in advanced osteosarcoma[J].Nat Commun,2020,11(1):6322.DOI:10.1038/s41467-020-20059-6.
[10]Stuart T,Butler A,Hoffman P,et al.Comprehensive integration of single-cell data[J].Cell,2019,177(7):1888-1902.e21.DOI:10.1016/j.cell.2019.05.031.
[11]Aran D,Looney AP,Liu L,et al.Reference-based analysis of lung single-cell sequencing reveals a transitional profibrotic macrophage[J].Nat Immunol,2019,20(2):163-172.DOI:10.1038/s41590-018-0276-y.
[12]Sun D,Guan X,Moran AE,et al.Identifying phenotype-associated subpopulations by integrating bulk and single-cell sequencing data[J].Nat Biotechnol,2022,40(4):527-538.DOI:10.1038/s41587-021-01091-3.
[13]Chu T,Wang Z,Peer D,et al.Cell type and gene expression deconvolution with BayesPrism enables Bayesian integrative analysis across bulk and single-cell RNA sequencing in oncology[J].Nat Cancer,2022,3(4):505-517.DOI:10.1038/s43018-022-00356-3.
[14]Aibar S,González-Blas CB,Moerman T,et al.SCENIC:single-cell regulatory network inference and clustering[J].Nat Methods,2017,14(11):1083-1086.DOI:10.1038/nmeth.4463.
[15]Qiu X,Mao Q,Tang Y,et al.Reversed graph embedding resolves complex single-cell trajectories[J].Nat Methods,2017,14(10):979-982.DOI:10.1038/nmeth.4402.
[16]Gulati GS,Sikandar SS,Wesche DJ,et al.Single-cell transcriptional diversity is a hallmark of developmental potential[J].Science,2020,367(6476):405-411.DOI:10.1126/science.aax0249.
[17]Sadykova LR,Ntekim AI,Muyangwa-Semenova M,et al.Epidemiology and risk factors of osteosarcoma[J].Cancer Invest,2020,38(5):259-269.DOI:10.1080/07357907.2020.1768401.
[18]Araki Y,Yamamoto N,Hayashi K,et al.The number of osteoclasts in a biopsy specimen can predict the efficacy of neoadjuvant chemotherapy for primary osteosarcoma[J].Sci Rep,2021,11(1):1-9.DOI:10.1038/s41598-020-80504-w.
[19]Cascini C,Chiodoni C.The immune landscape of osteosarcoma:implications for prognosis and treatment response[J].Cells,2021,10(7):1668.DOI:10.3390/cells10071668.
[20]Wang Z,Wang Z,Li B,et al.Innate immune cells:a potential and promising cell population for treating osteosarcoma[J].Front Immunol,2019,10:1114.DOI:10.3389/fimmu.2019.01114.
[21]Gonzalez H,Hagerling C,Werb Z.Roles of the immune system in cancer:from tumor initiation to metastatic progression[J].Gene Dev,2018,32(19-20):1267-1284.DOI:10.1101/gad.314617.118.
[22]Binnewies M,Roberts EW,Kersten K,et al.Understanding the tumor immune microenvironment(TIME)for effective therapy[J].Nat Med,2018,24(5):541-550.DOI:10.1038/s41591-018-0014-x.
[23]Waldman AD,Fritz JM,Lenardo MJ.A guide to cancer immunotherapy:from T cell basic science to clinical practice[J].Nat Rev Immunol,2020,20(11):651-668.DOI:10.1038/s41577-020-0306-5.
[24]Luo ZW,Liu PP,Wang ZX,et al.Macrophages in osteosarcoma immune microenvironment:implications for immunotherapy[J].Front Oncol,2020,10:586580.DOI:10.3389/fonc.2020.586580.
[25]Goswami S,Anandhan S,Raychaudhuri D,et al.Myeloid cell-targeted therapies for solid tumours[J].Nat Rev Immunol,2022:1-15.DOI:10.1038/s41577-022-00737-w.
[26]Veglia F,Sanseviero E,Gabrilovich DI.Myeloid-derived suppressor cells in the era of increasing myeloid cell diversity[J].Nat Rev Immunol,2021,21(8):485-498.DOI:10.1038/s41577-020-00490-y.
[27]Liu W,Hu H,Shao Z,et al.Characterizing the tumor microenvironment at the single-cell level reveals a novel immune evasion mechanism in osteosarcoma[J].Bone Res,2023,11(1):4.DOI:10.1038/s41413-022-00237-6.
[28]Tu J,Wang D,Zheng XT,et al.Single-cell RNA datasets and bulk RNA datasets analysis demonstrated C1Q+ tumor-associated macrophage as a major and antitumor immune cell population in osteosarcoma[J].Front Immunol,2023,14:438.DOI:10.3389/fimmu.2023.911368.
[29]He Y,Zhou H,Huang X,et al.Infiltration of LPAR5+ macrophages in osteosarcoma tumor microenvironment predicts better outcomes[J].Front Immunol,2022,13:909932.DOI:10.3389/fimmu.2022.909932.
(收稿日期:2023-03-14)

