产黑普氏菌刺激原代人口腔角质细胞的时间序列基因表达谱研究
邵如如, 郑章龙, 徐 盼, 郭艺婷, 何 园
(同济大学口腔医学院·同济大学附属口腔医院口腔黏膜病教研室,上海牙组织修复与再生工程技术研究中心,上海 200072)
口腔扁平苔藓(oral lichen planus, OLP)是一种常见的口腔黏膜慢性炎症性疾病。据估计,该疾病影响了大约1%的普通人群,平均发病年龄约为60岁,女性较为多见。该疾病被WHO归类于口腔潜在恶性疾病,恶性转化率为0.44%-1.4%。OLP病理学表现为固有层存在具有带状外观的炎性浸润以及上皮基底细胞层液化变性,浸润的淋巴细胞主要CD4+和CD8+T细胞,其发病机制目前尚不清楚[1]。
微生物在维持口腔健康方面起着重要的作用。特定情况下,微生物与人体之间的动态平衡被打破,导致菌群失衡,一些共生菌群转化成致病菌,诱发多种疾病[2]。近年来,微生物感染在OLP发病机制中的作用引起人们的广泛关注,最近多个研究证明了OLP患者的唾液和组织样本中微生物群落多样性和组成与健康受试者相比存在显著差异[3-4]。本课题组前期研究发现,OLP患者颊黏膜表面微生物群落结构与健康人群相比明显不同。进一步通过高通量测序分析发现,产黑普氏菌(Prevotellamelaninogenica,Pm)在OLP颊黏膜表面构成比显著增加,并且Pm侵入OLP组织的上皮层和固有层[5-6],这均提示我们Pm可能在OLP的发生发展中起到重要作用。
本研究对Pm刺激原代人口腔角质形成细胞(primary human oral keratinocytes, pHOKs)的转录组测序数据,通过Mfuzz包进行时间序列分析,以探讨相关生物学分子的动态模式以及Pm与宿主细胞相互作用的潜在机制。
1 材料与方法
1.1 Pm-pHOKs细胞共培养模型及转录组数据的获取
Pm-pHOKs细胞共培养模型及转录组数据来自课题组前期郭等人的研究[7]。
1.2 数据预处理和差异表达基因的筛选
用HTSeq分别对共培养组和对照组样本进行reads计数。使用DESeq2进行差异表达基因(differentially expressed genes, DEGs)筛选,Ashr算法对logFC进行校正。取差异倍数绝对值≥1且P≤0.01的基因筛选为差异具有统计学意义的DEGs。
1.3 DEGs的聚类分析
用于软聚类微阵列数据的Mfuzz[8]克服了传统聚类的弱点。在这项研究中,Mfuzz用于进行DEGs的聚类分析。其中C值设为3,m值为3.71。分别在Pm处理不同时间下筛选具有一致表达趋势的基因。根据聚类结果鉴定上调基因和下调基因,簇中上调基因表现为持续上升趋势,下调基因表现为持续下降趋势。
1.4 功能富集分析
利用R语言富集常用包ClusterProfiler分别对3个簇中包含的基因进行功能富集分析,包括基因本体论“生物学过程”注释类别和重要的京都基因和基因组百科全书(Kyoto Encyclopedia of Genes and Genomes, KEGG)途径。校正后P<0.05表示DEGs的显著富集的GO和KEGG途径。
1.5 蛋白质-蛋白质相互作用网络和模块分析
用于检索相互作用基因的搜索工具STRING是一种探索潜在蛋白质-蛋白质相互作用的在线工具,它由已知的和预测的PPI组成。分别提取3个簇中的DEGs,利用STRING数据库(https:∥cn.string-db.org/)进行蛋白-蛋白互作分析。使用Cytoscape软件(版本3.9.1)对蛋白互作结果进行可视化,进一步利用Cytohubba插件对DEGs按EPC、Degree、Closeness、MNC算法计算评分位于前10的基因,再取交集,得到枢纽基因(Hub基因)。
1.6 HaCaT细胞培养
将HaCaT细胞(中国科学院细胞库)接种于含有10%的胎牛血清的DMEM培养基(Hyclone,美国)中,并在37 ℃,5%CO2环境下培养。培养基2-3 d更换1次。常规传代,取对数生长期细胞进行后续实验。
1.7 细菌培养
将P.melaninogenica(ATCC®25845TM)涂布于厌氧基础血平板,厌氧环境下培养,挑取单个菌落接种于厌氧基础肉汤中,置于37 ℃摇床过夜培养,用新鲜肉汤将菌液稀释至OD600为0.1,培养至对数期收集菌液,离心(离心半径10 cm,3 000 r/min,5 min)弃上清液,PBS清洗3次后,用新鲜培养基调整OD600=1.0,用于实验。
1.8 实时荧光定量PCR验证Hub基因
将HaCaT细胞以2×105个/mL的细胞密度均匀种植于12孔板,24 h后去除培养基,PBS清洗3次后加入不含胎牛血清的DMEM培养基,感染组和对照组分别吸取1 μL菌液和1 μL的DMEM培养基,放置于含5%CO2的37 ℃孵箱内培养4、24 h后去除培养基,PBS清洗3次,加入TRIzol试剂,冰上孵育5 min后将RNA收集至离心管内,氯仿法提取RNA。根据反转录试剂盒(TaKaRa,日本)说明书将每个样本的总RNA转化为cDNA。根据实时荧光定量试剂盒(TaKaRa,日本)说明,确定相应的反应条件: 95 ℃ 30 s,随后进行40个95 ℃ 5 s和60 ℃ 31 s的循环。所有样本一式3份。GAPDH作为内部对照,所用引物信息见表1。

表1 关键基因的引物序列
1.9 统计学处理
目的基因相对表达量采用CT对比法确定水平(2-ΔΔCt)。采用单因素方差分析对多组数据间进行比较,P<0.05为差异有统计学意义。
2 结 果
2.1 DEGs筛选
通过分析Pm刺激pHOKs的高通量测序数据发现,在本次测序所获取的全部基因中,相较于对照组,4 h组与24 h组分别有1 455个、1 385个基因的表达存在统计学差异,见图1。
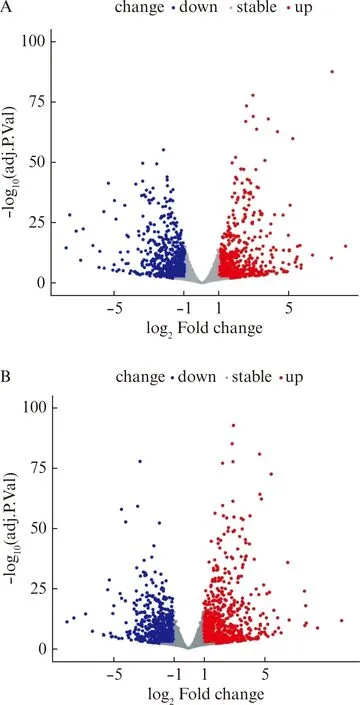
图1 实验组和对照组的基因表达
2.2 DEGs的聚类分析
通过软Mfuzz聚类算法,将4 h和24 h组的DEGs按照其随时间变化表达的相似性分为3个簇,分别获得簇1(包括561个基因),簇2(包括262个基因),和簇3(包括303个基因)。簇1中的基因随时间延长其表达呈现下降的趋势,相反,簇2中的基因表达值呈现上升的趋势,簇3的基因表达值在刺激前期(4 h)显著上调,后期(24 h)显著下调,见图2。

图2 3个聚类的基因表达变化
2.3 DEGs的GO功能富集分析
图3显示了3个簇包含的DEGs生物学过程功能富集结果。其中,横轴代表各个簇,纵轴代表GeneOntology数据库中对应GO的条目名称。与簇1中的DEGs相关的生物学过程为上皮细胞分化的调控、上皮细胞向间充质细胞转化的负调控、间充质细胞生长。簇2中的DEGs主要富集的生物学过程为细菌来源的分子的反应、对脂多糖的反应、烯烃化合物代谢过程、二萜代谢过程、视黄醇代谢过程、细胞对生物刺激的反应。簇3中的DEGs主要富集的生物学过程为对脂多糖的反应、细胞因子介导的信号通路、白细胞迁移、细胞趋化作用、对IL-1的反应。
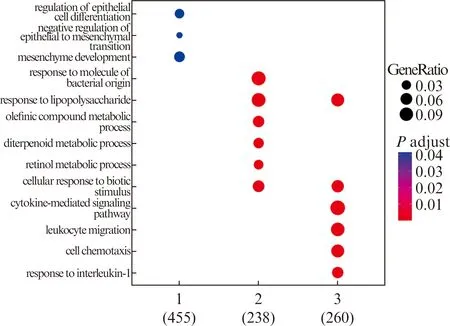
图3 差异表达基因GO功能富集分析结果示意图
2.4 DEGs的信号通路分析
对3个簇中的DEGs进行了KEGG信号通路分析,如图4所示,簇1中的基因没有富集到相关通路,簇2中的基因主要参与金黄色葡萄球菌感染、类风湿性关节炎、TNF信号通路、IL-17信号通路、甾类激素生物合成、细胞因子与细胞因子相互作用等通路。簇3中的基因主要富集在类风湿性关节炎、TNF信号通路、IL-17信号通路、细胞因子与细胞因子相互作用、病毒蛋白与细胞因子及细胞因子受体的相互作用、癌症中的转录失调、脂质和动脉粥样硬化、Toll样受体信号通路等通路。
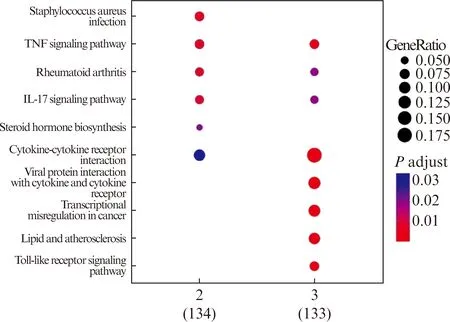
图4 差异表达基因KEGG富集分析
2.5 PPI网络和Hub基因
通过利用STRING在线工具,构建3个簇中DEGs的PPI网络,见图5。

图5 3个聚类中差异基因的PPI网络
在PPI网络中筛选出簇1中的2个Hub基因为VEGFA、GDNF;簇2中的8个Hub基因分别为CSF2、CCL5、CXCL1、PTGS2、ICAM1、CD86、MYD88、TNFAIP3;簇3中的8个Hub基因分别为TNF、IL-6、CXCL8、CXCL10、CCL2、IRF1、MX1、IFIT1,见图6。
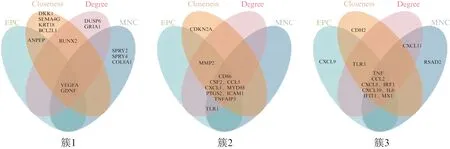
图6 用Cytohubba插件中4种不同算法所确定的Hub基因
2.6 实时荧光定量PCR验证结果
随机选取3个簇中部分Hub基因进行实时荧光定量PCR检测,结果表明,簇1中的GDNF在4 h时与对照组相比显著下调(P<0.05),24 h时下调趋势更为显著(P<0.01)。而簇2中的基因PTGS2则随着时间延长其表达量逐渐上调。簇3中的基因IRF1及IFIT1在4 h时均显著上调(P<0.01),而24 h时与对照组相比差异无统计学意义,实时荧光定量PCR结果与转录组测序结果一致,见图7。
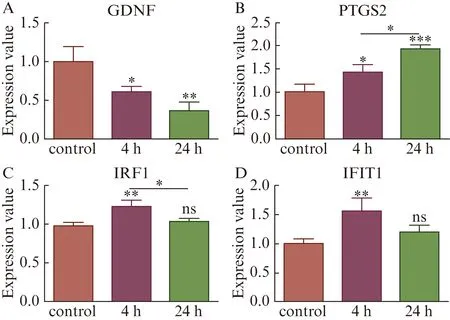
图7 实时荧光定量PCR验证RNA-Seq结果
3 讨 论
宿主和微生物之间的相互作用在时间和空间上是高度动态的,特别是在感染的情况下,病原体数量、微生物表型和宿主反应的性质通常会随着时间的推移而发生巨大变化[9]。对时间序列中动态变化的基因的探究有助于更好地理解宿主与微生物相互作用的机制,为疾病预防与临床治疗提供信息。故本研究基于时间序列分析,对Pm刺激pHOKs的RNA-seq数据进行数据挖掘,根据数据的特点及需要采用Mfuzz算法将DEGs划分为3个聚类,并对每个聚类中的基因进行GO、KEGG以及STRING网络分析,并筛选出Hub基因,利用实时荧光定量PCR对部分基因进行验证。
Mfuzz算法可以对来自不同时间点的DEGs进行聚类,以显示它们的渐进式表达式动态[10]。分析微阵列数据时常用的方法是聚类技术,但通常是基于硬聚类方法,即其中一个基因(或样本)被精确地分配到一个聚类。然而,硬聚类存在信息丢失等缺点。相比之下,软聚类方法可以将一个基因分配到多个聚类,它克服了传统硬聚类技术的不足,并具有进一步的优势[11]。除了Mfuzz算法之外,还有一些类似的基于模糊聚类的算法,例如: Fuzzy C-means(FCM)算法[12]、Possibilistic C-means(PCM)算法等[13]。
上皮-间充质转化(epithelial-mesenchymal transition, EMT)是指上皮细胞通过特定程序转化为具有间质表型细胞的生物学过程,已被证明为一种可逆过程[14]。当负调控上皮细胞向间充质细胞转化过程相关基因下调时可促进EMT,上皮细胞失去细胞-细胞连接和基底-顶端极性。此前有研究报道称EMT相关标志物claudin-1,claudin-4和E-钙黏蛋白在OLP中的表达与对照组相比显著降低。提示OLP的上皮层存在细胞黏附和极性失调[15]。本研究结果显示,簇1中的基因与上皮细胞向间充质细胞转化的负调控相关,提示EMT可能是OLP的早期病理过程,当Pm与口腔角质形成细胞相作用,可激活EMT,使上皮去极化并破坏其紧密和黏附,导致细菌进一步地侵入上皮与固有层。
此外,簇2的差异表达基因随时间延长其表达持续上调,通过GO分析提示与视黄醇代谢过程相关。进一步分析数据发现,在Pm刺激原代人口腔角质形成细胞模型中与视黄醇代谢途径相关的基因AKR1B10的表达显著上调。AKR1B10是视黄酸代谢中的关键酶,即醛固酮还原酶家族1成员B10。目前研究表明,AKR1B10在乳腺癌[16],口腔鳞状细胞癌[17],肺癌[18],肝癌[19],喉癌[20]等肿瘤中过表达,此外AKR1B10的上调还见于银屑病、瘢痕疙瘩、特应性皮炎等患者的皮肤慢性炎症中。OLP相关转录组数据集GSE52130[21]中,AKR1B10的表达水平较健康人群的上皮样本高,提示该基因可能在OLP病理过程中起到重要作用。
簇3中的基因主要富集于与对脂多糖的反应、细胞因子介导的信号通路、白细胞迁移、细胞趋化作用等生物学过程相关,其表达在刺激前期上调,后期下调。有研究发现一些微生物已经进化出许多适应性机制逃避免疫监视,如牙龈卟啉单胞菌通过利用宿主自噬机制在牙龈上皮细胞中成功存活[22],此外牙龈卟啉单胞菌能够在细胞间传播,有学者研究发现牙龈卟啉单胞菌的细胞间易位是通过基于肌动蛋白的膜突起介导,绕过了将细菌释放到细胞外基质中的需要,从而避开免疫系统的清除[23]。综合上述,我们推测早期Pm黏附于上皮细胞表面,细胞分泌细胞因子、趋化因子和防御素等免疫效应物抵抗其入侵。后期Pm可能通过类似机制入侵上皮细胞,逃避免疫监视,从而导致炎症的慢性迁延与复发。对Pm可能产生的适应性机制进行研究有助于深入理解OLP的病理过程。
本研究进一步筛选了3个簇的Hub基因,簇1中的GDNF负责编码神经营养因子,该因子的下调与炎症性肠病患者的屏障功能降低密切相关[24-25]。在本研究中GDNF的转录在Pm刺激的pHOKs中显著下调,这表明GDNF可能在介导Pm刺激后pHOKs屏障功能障碍中发挥重要作用。簇2中PTGS2又称为COX2,是产生疼痛介质PGE2的重要酶,PGE2的水平升高可能使周围感觉神经敏感,导致疼痛信号更频繁地传递到大脑[26]。有学者发现与OLP网状型相比,糜烂型病变中COX-2的表达显著增加[27]。临床上,OLP患者通常会表现为口腔的烧灼感或疼痛症状,这可能与Pm刺激后pHOKs中COX-2表达升高有关。此外簇2中还包括基因GM-CSF,有文献报道,口腔鳞状细胞癌患者存在唾液微生物组生态失调,产黑普氏菌的丰度较高,且患者唾液中IL-8、IL-6、TNF-α、GM-CSF和IFN-γ显著升高,表明产黑普氏菌有可能增加口腔鳞癌患者口腔唾液中GM-CSF的表达,促进鳞癌的发展[28]。提示OLP中GM-CSF的表达上调可能与产黑普氏菌的毒力相关。我们还注意到簇3的Hub基因包括IL-6,研究表明铜绿假单胞菌(Pseudomonasaeruginosa,PA)分泌的胞外毒力蛋白LasB是炎症反应的触发因素,在PA感染急性期,LasB激发宿主产生大量促炎细胞因子,如IL-6[29]。同时LasB可降解宿主细胞表面受体及细胞因子,直接破坏宿主组织细胞和免疫防御系统。如在囊性纤维化患者中,LasB下调IL-6/STAT3通路,抑制上皮细胞修复,导致慢性炎症的建立和维持[30]。故推测早期Pm刺激pHOKs诱导免疫反应,导致促炎因子IL-6上调,后期Pm可能产生某种毒力因子通过直接降解免疫介质IL-6或间接下调IL-6的产生来逃避免疫调节。为检验转录组数据是否准确,随机选取4个Hub基因进行实时荧光定量PCR检测,结果表明测定结果与转录组测序结果得到的差异基因表达变化趋势一致,表明本研究中转录组测序得到的数据是可信的。
本研究旨在通过时间序列分析,全面了解人口腔角质形成细胞对Pm感染反应的动态变化。通过研究我们发现与视黄醇代谢相关的基因以及负调控EMT的基因在OLP中异常表达可能与Pm入侵上皮细胞有关,并且Pm入侵上皮细胞后可能通过一些适应性机制逃避免疫监视,从而导致慢性炎症持续存在。这些发现将有助于更好地理解Pm在OLP的发生发展过程中与宿主细胞相互作用的潜在机制,同时为Pm感染所致疾病的发病机理研究提供一定的参考,为Pm感染相关疾病的预防和临床治疗提供理论基础。但本研究仍存在一些局限性: 在本次实验中,只选择了3个时间点收集样本,对于分析的结果有待进一步通过扩大不同时间点的样本量来验证。此外我们只分析了部分基因,还有更多生物分子信息有待深入研究。针对已挖掘的关键基因,后续将在实验样本中验证其在Pm与细胞相互作用中发挥的功能,以期为Pm感染相关疾病的临床治疗提供理论基础和实验证据。

