假单胞菌属中新型IncpGRT1质粒的遗传特性及其潜在传播风险
李昕悦,王 鹏,陈方舟,穆小飞,卢秀慧,贺家琪,郑亚丽,周冬生,殷 喆
假单胞菌属(Pseudomonas)属于假单胞菌科(Pseudomonadaceae)假单胞菌目(Pseudomonadales),包含200多个亚种[1]。假单胞菌属菌多为条件致病菌,对人和动物均有致病性,并且有很强的环境适应性。细菌感染及耐药率的上升,很大程度上与质粒的传播相关。质粒是细菌染色体外能自主复制的环状DNA分子,可通过接合在细菌之间进行水平转移[2]。目前根据质粒不相容性(plasmid incompatibility)对质粒进行分类的方法已经非常普遍[3]。已报道的假单胞菌中的质粒Inc群包括IncP-1至IncP-14、IncpRBL16等[4]。如今可以获得的假单胞菌中的质粒全序不断增多,其中很多质粒尚不能归于现有的Inc群,给假单胞菌中质粒的遗传特性研究造成很大的困难。该研究采用生物信息学方法,为新的IncpGRT1型别的质粒的保守性及多态性,以及该型别质粒之间的演化关系提供了更深入的见解。
1 材料与方法
1.1 菌株来源与鉴定菌株15420352分离自湘雅医院呼吸内科的50岁肺部感染女性患者的尿液样本。原始菌种记录为恶臭假单胞菌。本研究对其16S rDNA基因扩增测序进一步确定菌种。
1.2 药敏试验使用VITEK 2 Compact全自动细菌鉴定及药敏分析仪对菌株15420352进行药敏MIC检测,依据美国临床与实验室标准协会(Clinical and Laboratory Standards Institute, CLSI)2020标准判读结果。
1.3 质粒测序及组装对菌株15420352进行三代测序以获得质粒的完整序列,利用德国Qiagen公司的Ultra Clean® Microbial DNA Isolation kit(CAT.12224-250)试剂盒,提取恶臭假单胞菌15420352的基因组DNA。构建均值约15 kb(片段大小10~20 kb)的DNA文库,通过PacBio RSII测序仪进行三代测序。同时,构建均值约400 bp左右(片段大小150~600 bp)的双端测序(paired-end)文库,通过HiSeq测序仪进行二代测序。利用Proovread软件通过短的片段校正PacBio长的片段,校正过的PacBio片段通过HGAPv3.0(基因组覆盖率100×)进行拼接组装,最终获得恶臭假单胞菌15420352的完整染色体及质粒序列。
1.4 质粒的生物信息学和比较基因组学分析获得质粒全序列以后,首先通过RAST2.0(https://rast.nmpdr.org/)初步预测质粒的开放阅读框(ppen reading frame, ORF),利用Plasmidfinder2.1(https://cge.food.dtu.dk/services/PlasmidFinder/)确定质粒的复制子基因,在此基础上,利用Blast(https://blast.ncbi.nlm.nih.gov/Blast.cgi)、ConservedDomains(https://www.ncbi.nlm.nih.gov/Structure/cdd/wrpsb.cgi)等进行每个基因的功能注释。利用在线数据库ResFinder(https://cge.food.dtu.dk/services/ResFinder/)和CARD(https://card.mcmaster.ca)对耐药基因进行筛查,ISfinder(https://www-is.biotoul.fr)、INTEGRALL(http://integrall.bio.ua.pt/)和TheTransposonRegistry(https://transposon.lstmed.ac.uk/tn-registry)对质粒的外源插入区结构进行精细注释。注释列表完成后,利用Inkscape1.0绘制质粒整体结构圈图、质粒线性结构比较图和外源插入区线性结构比较图。
1.5 序列登录号进行全基因组测序后,得到质粒p420352-strA序列,提交至GenBank,登录号为MT074087。
2 结果
2.1 菌种鉴定及药敏结果测定菌株15420352经16S rDNA扩增初步确定菌种,后将全基因组DNA序列与恶臭假单胞菌参考菌株KT2440(登录号:NC_002947.4)进行平均核苷酸一致性(average nucleotide identity,ANI)比对分析(http://www.ezbiocloud.net/tools/ani),一般以95%做为判定是否为同一菌种的阈值,ANI值计算结果为96.99%,最终核定该株菌为恶臭假单胞菌。野生株对β-内酰胺类、头孢类、青霉烯类、喹诺酮类抗生素耐药,对阿米卡星及四环素类抗生素敏感,见表1。
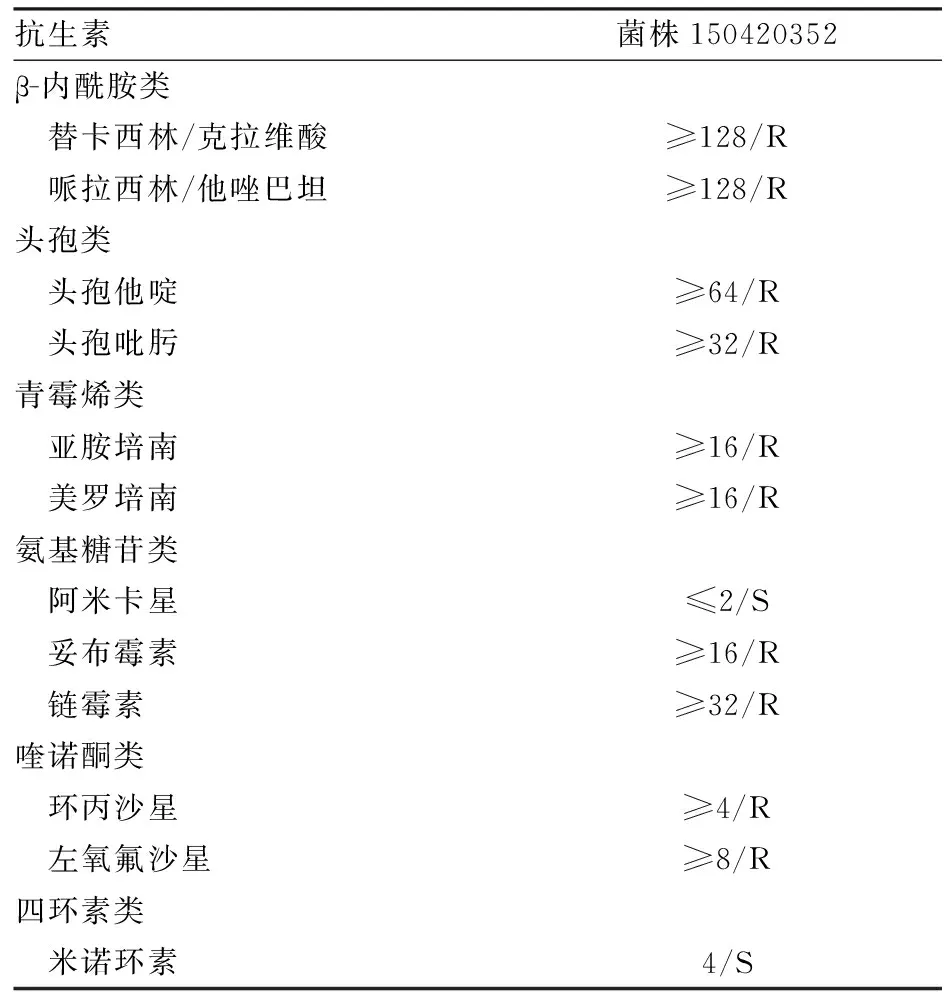
表1 15420352 MIC测定结果
2.2 IncpGRT1型质粒遗传特性
2.2.1测序质粒p420352-strA及纳入分析的质粒概述 质粒p420352-strA大小为153.6 kb,平均GC含量为56.67%,共预测得到174个开放读码框,见图1。本研究纳入分析的质粒共5个,见表2,除上述本研究测序质粒,另有截至2022年9月1日从GenBank中检索到的4个质粒,包括pGRT1、pZDHY414、pPp1290、pBYT5-2。这5个质粒的复制子(replication)基因与repAIncpGRT1的同源性≥95%。且均包含IncpGRT1型质粒的保守骨架结构。由于不能归于现有的Inc群,均属于新的Inc型别IncpGRT1。pGRT1在恶臭假单胞菌DOT-T1E中被发现,因为该质粒为最早测序的该型别质粒,本研究将其作为IncpGRT1型质粒的参考质粒。每个质粒都可分为骨架区和多个独立的外源插入区,外源插入区分别插入在不同的骨架区位点。
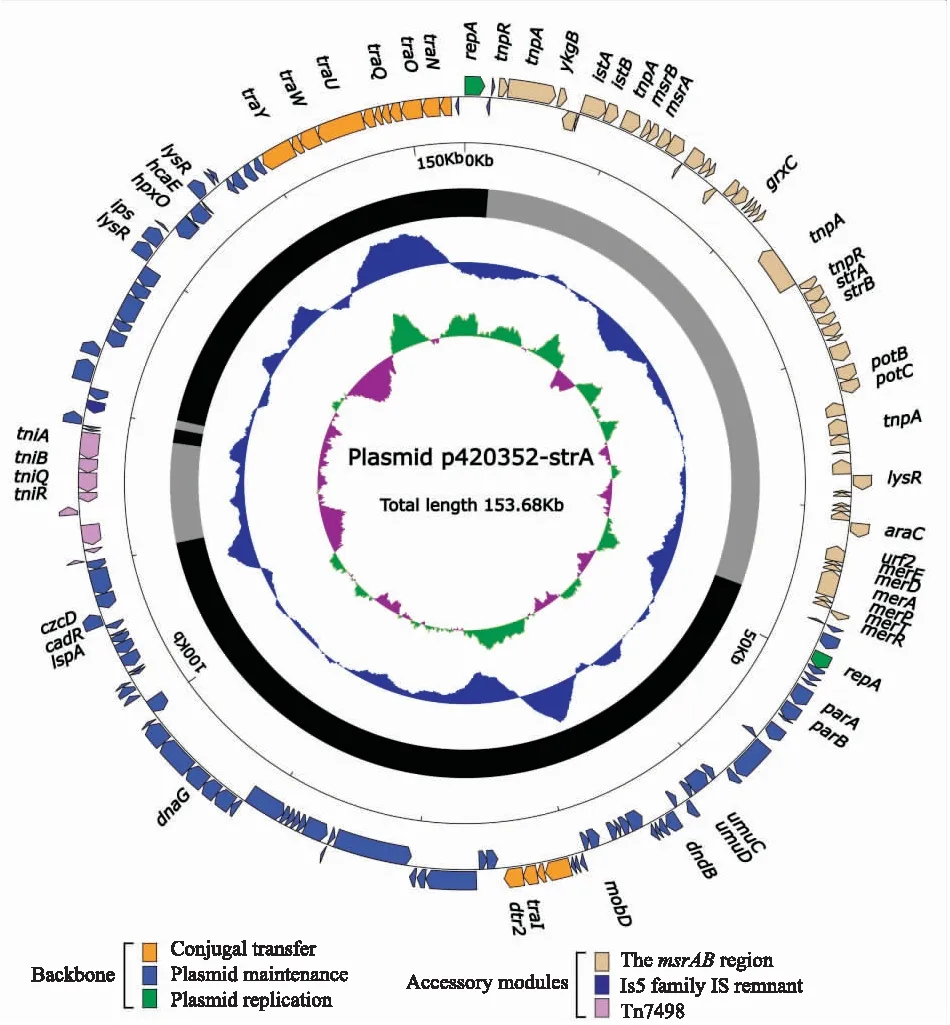
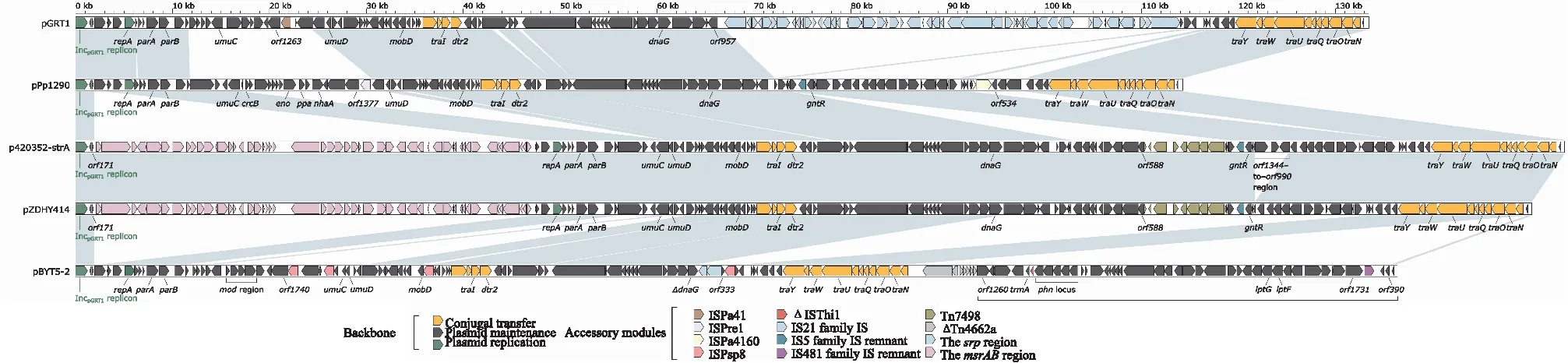
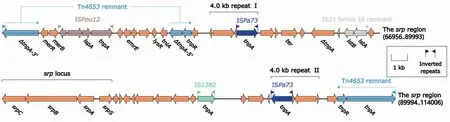
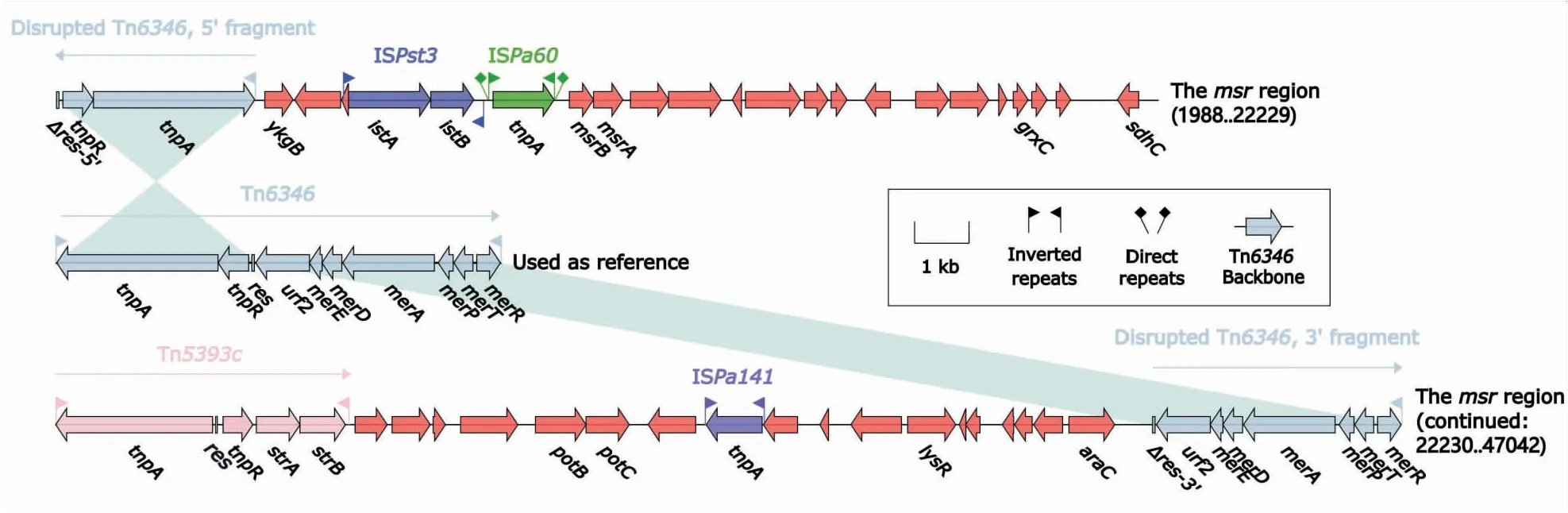
图1 p420352-strA全序列示意图最内环表示GC偏移,向外和向内分别表示G和C的相对含量G>C和G 表2 IncpGRT1型别质粒的基本特征 2.2.2IncpGRT1型质粒的骨架区比较分析 IncpGRT1型的5个质粒的骨架区可进一步分为与质粒复制起始相关的区域,维持质粒稳定性相关的区域和接合转移区,见图2。这5个质粒>52%的骨架区的同源性>80%。此外,这些质粒的的复制起始区各含有两个编码新型复制子的基因,除了上述主复制子repAIncpGRT1,均还含有一个辅复制子,为编码Rep_3家族复制起始蛋白RepA的repA。IncpGRT1型质粒的保守骨架结构包括:repAIncpGRT1及其iterons(复制区)、parAB(质粒分割区)、dtr2和几个tra基因(接合转移区),以及其他调控质粒稳定性相关的基因umuCD、mobD、dnaG等。其中,pBYT5-2与其他四个质粒的骨架区差异最大,orf1260-to-orf390区是其独有的骨架区结构,长4.2 kb,该区域包括磷酸盐转运系统(phosphonate transport system,phnCDE)基因座位和脂多糖转运酶(LPS export ABC transporter permease,lptGF)基因座位,是菌体内重要的代谢相关基因。此外,pBYT5-2骨架区还发现了mod区,该区有钼酸盐转运蛋白(molybdate ABC transporter,modABE)基因,这些基因均参与调控宿主关键的生理功能。p420352-strA和pZDHY414的同源性最强,这两个质粒的差异只在于p420352-strA的骨架区存在一个orf1344-to-orf990区。 图2 IncpGRT1质粒序列线性比较图箭头表示基因,不同颜色代表不同的基因功能分类;阴影部分表示同源区域(核苷酸相似性>90%) 2.2.3质粒外源插入区 pGRT1的外源插入区包括一个4.7 kb长的srp区和一个ISPa41,见图3。srp区的两端发现了Tn4653的残余,Tn4653为Tn3家族转座子。该区域内的溶剂外排RND转运体外膜蛋白(solvent efflux RND transporter outer membrane subunit,srpABCS)基因,可以使菌体在高浓度有机溶剂如甲苯、苯乙烯和二甲苯的环境中具有高耐受能力,甚至生长能力。推测这些与细菌环境适应性相关的外排泵基因由移动元件携带并进入菌体中,导致该菌对甲苯的耐性增强。这一区域内还发现有汞转运(mercuric transcriptional regulator,mer)基因,以及多药转运蛋白(escherichia coli multidrug transporter,emrE)基因。因此,质粒携带的srp区在提高细菌耐药性方面也发挥了很大作用。此外该区域另有插入序列ISPpu12、ISPa73、IS1382以及一个IS21家族IS残余的插入。 图3 pGRT1 srp区域箭头指示基因,不同颜色标示不同的基因功能分类 p420352-strA和pZDHY414的外源插入区主要为msr区(图4)和一个单元转座子Tn7498(图5)。 图4 p420352-strA和pZDHY414的msr区域箭头表示基因,不同颜色代表不同的基因功能分类;阴影部分表示同源区域(核苷酸同源性>90%);Tn5393c的登录号为AF262622 在这两个质粒的msr区中,发现了不完整的Tn6346成分,Tn6346为Tn3家族单元转座子,由Tn5051的核心转位模块tnpA(转座酶基因)-tnpR(解离酶基因)-res(重组位点)和Tn501的mer区组成,该转座子在其res位点被打断为两部分并发生了颠转,分别为该区域两端的Tn6434的5′端片段和3′端片段。Tn5393c被发现插入到该区域中,Tn5393c也是Tn3家族的单元转座子,其包含两个链霉素类耐药基因strA和strB。该区域中还发现了肽甲硫氨酸亚砜还原酶(peptide methionine sulfoxide reductase,msrAB)基因,比对结果注释为毒力因子。其他的移动元件ISPst3、ISPa60和ISPa141也在该区域中被发现。 Tn7498是本研究新发现的一个Tn5053亚家族的单元转座子,Tn5053为Tn7家族转座子。最初在黄单胞菌属W17中被发现,其骨架区核心转座结构为tniABQR。Tn7498插入到质粒骨架区基因orf588上游的87 bp处,并产生了5 bp的正向重复序列(direct repeats, DRs)。Tn7498未发现其参考元件Tn5053中的mer区。 假单胞菌广泛分布在世界各地的各种环境中,并且质粒在假单胞菌中普遍存在。质粒介导的基因转移不仅在抗生素耐药基因的传播中发挥重要作用,还在微生物降解途径和毒力因子传播中发挥重要作用。为探究假单胞菌属中新型IncpGRT1质粒的遗传特性,本研究对5个IncpGRT1质粒进行了全面的比较基因组学分析。揭示了该型别质粒介导假单胞菌产生耐药性和致病性的潜在风险。 目前该类质粒仅被发现在假单胞菌中传播(搜索截止日期为2022年9月1日),并且这些宿主菌在临床和环境中均有分布。本研究中的5个质粒有3个来自恶臭假单胞菌,1个来自黄褐假单胞菌,1个来假单胞属菌。其中,菌株1290分离时间最早,1985年分离自美国的环境样本[5],据此推测该类质粒最初来自于环境。 通过对IncpGRT1型质粒的比较基因组学分析,更深层次地了解了该型别质粒的保守性以及多样性。该类质粒基因组均发现了两个复制子,其中repAIncpGRT1上游发现了8个重复的正向序列iterons,该序列在调控质粒DNA复制中起重要作用。5个质粒包含至少3种主要的外源插入区:srp区域、msr区域以及Tn7498。外源插入区增加了这一类质粒序列的多样性。复杂的转座机制是这些外源插入区形成的主要原因。 IncpGRT1型质粒已经成为一些耐药基因以及毒力基因在假单胞菌属中积累和传播的重要载体。在这些质粒的外源插入区中,已经发现了2类已知的抗生素耐药和汞金属抗性基因:strAB和mer。在p420352-strA和pZDHY414两个质粒中,发现了同源性很高的毒力相关的msr区,均编码两个毒力基因msrAB,这两个基因在细菌黏附及侵袭作用中发挥了重要作用[6-7]。一些关键的转运蛋白也在该型别质粒中被发现,pGRT1中包括可以抵抗有机溶剂的外排泵基因srpABCS,根据相关报道携带该质粒的恶臭假单胞菌DOT-T1E由于获得了这类相关基因,其可以在高浓度的甲苯溶液中生存,且耐有机溶剂[8]。该质粒中还发现了emrE基因,许多研究[9-10]已发现在不同携带该基因的的菌种中都产生了多种药物的外排现象。在pBYT5-2的骨架区发现了编码转运蛋白的mod、phn、lpt,可以维持宿主的基本生理活动[11- 12]。总之,质粒中这些关键的抗性基因和调控基因可以提高宿主在环境压力下的生存能力。 该研究表明IncpGRT1型质粒在假单胞菌中宿主的广泛性以及其传播耐药及毒力基因的潜力,假单胞菌可以通过传播这类质粒获得相关基因。该类型质粒已经在临床中分离出,推测其在院内环境中有广泛传播的潜力,临床防治过程中需要给予重视。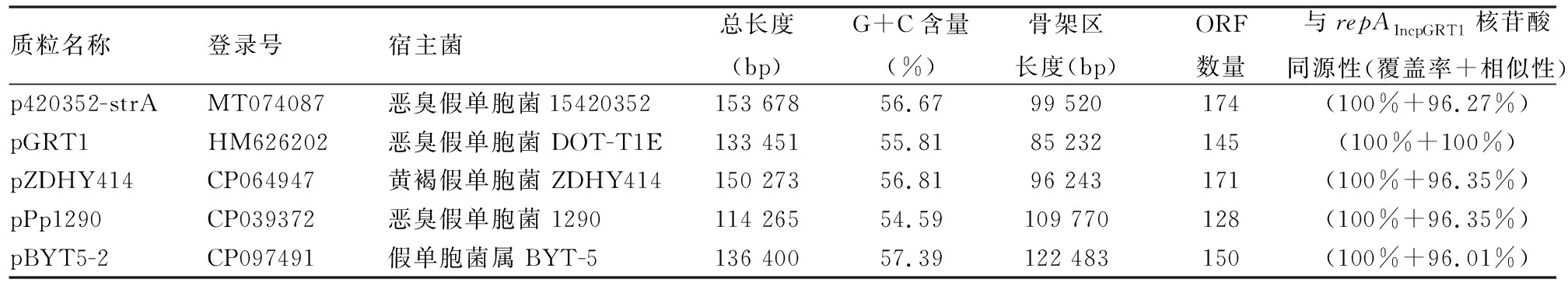
3 讨论

