深度学习在皮肤OCT医学图像中的应用
王常科 马琼 蔡清裕 孙海洋 汤晓安 康宏向

摘 要:深度学习作为机器学习领域的重要研究方向,其强大的特征学习能力以及优秀的可移植性使得深度学习技术渗入到科学研究的各个领域,尤其是深度学习中前沿领域的成果对医学图像的识别、分类、分割、量化等方面起到了积极作用。光学相干断层扫描成像(OCT)技术具有非侵入、分辨率高的特点,能够无创获取生物组织内部微米级的高分辨率三维切面图像,在生物医学的眼科、血管、皮肤科等方面有着广泛应用。本文首先介绍了OCT及深度学习的基本原理以及二者相结合在皮肤领域的应用优势,然后详细阐述了深度学习在皮肤OCT医学图像领域的应用,主要包括在皮肤癌的分析分类、美容领域、指尖图像的分割以及在损伤皮肤的定性定量评估中的应用。
关键词:光学相干断层扫描成像;医学图像;深度学习;皮肤疾病;卷积神经网络
中图分类号:Q-334 文献标志码:ADOI:10.3969/j.issn.1007-7146.2023.03.001
Application of Deep Learning in OCT Medical Images of Skin
WANG Changke1, 2, MA Qiong1, CAI Qingyu2, 3, SUN Haiyang2, 3, TANG Xiaoan2, KANG Hongxiang1*
(1. Institute of Radiation Medicine, Academy of Military Medicine, Academy of Military Sciences, Beijing 100850, China;
2. College of Information Engineering, Henan University of Science and Technology, Luoyang 471023, China; 3. Hunan SANY Industrial Vocational Technical College, Changsha 410129, China)
Abstract: As an important research direction in the field of machine learning, deep learning technology has been infiltrated into various fields of scientific research due to its powerful feature of learning ability and excellent portability. In particular, the achievements in the frontier fields of deep learning have played a positive role in the recognition, classification, segmentation and quantification of medical images. Optical coherence tomography (OCT) technology has the characteristics of non-invasive, high resolution, non-invasive acquisition of micron-level high resolution 3D sectional image of biological tissue, and has been widely used in ophthalmology, vascular, dermatology and other biomedical fields. This paper first introduces the basic principles of OCT and deep learning and the advantages of their combination in the field of skin, and then elaborates the application of deep learning in the field of skin OCT medical images, including the analysis and classification of skin cancer, beauty, fingertip image segmentation, and the qualitative and quantitative evaluation of damaged skin.
Key words: optical coherence tomography; medical imaging; deep learning; skin disease; convolutional neural network
(Acta Laser Biology Sinica, 2023, 32(3): 193-199)
光学相干断层扫描成像(optical coherence tomography,OCT)是一种非侵入性光学成像技术,自1991年发现以来发展迅速[1]。OCT能够无创获取生物组织微米级的高分辨率三维切面图像[2],在皮肤科有着很好的应用前景。近二十年来,临床上不断尝试使用OCT评估皮肤结构,用于表皮、真皮、毛囊和汗腺等[3]皮肤組织的结构成像。使用OCT辅助临床皮肤疾病诊断能够无创获取活体皮下组织的病灶信息,是皮肤镜和皮肤电子计算机断层扫描(computed tomography,CT)等常用辅助诊断成像技术无法实现的。
作为一种非侵入性光学成像方法,OCT在非侵入性诊断和疾病监测方面具有相当大的应用场景。尤其在皮肤科领域,OCT已经在正常皮肤[4]、角化细胞癌[5]、黑色素瘤[6]、炎症皮肤[7]的评估或识别中得到了应用。Boone等[8]基于3D OCT影像中的某些特殊特征设计了一种决策树算法,用于从正常皮肤和鳞状细胞癌中区分光化性角化病;Cobb等[9]使用中值滤波对皮肤损伤小鼠的OCT图像去噪,在此基础上使用Canny边缘检测算法分割OCT图像,用以监测皮肤伤口的愈合特征;Gao等[10]基于OCT图像中强度、纹理和分形特征对黑色素瘤、基底细胞癌和色素痣进行分类,并使用广义估计方程检验皮肤肿瘤之间的差异。上述研究证实了OCT在皮肤领域应用的可行性以及科学性,但临床OCT图像的分析通常需要经过培训的专家来完成,高度依赖医生的临床经验,且医学影像的复杂性、皮肤病灶边界模糊等会对诊断造成巨大的干扰。对于大量需要处理的皮肤OCT图像,传统处理方式的速度及精度无法满足临床应用的需要。
深度学习(deep learning,DL)作为机器学习(machine learning,ML)领域中的一个重要研究方向,解决了很多复杂的模式识别难题,在语音和图像识别领域的效果要远超之前的相关技术[11]。目前,深度学习在医学图像处理中的应用尤为广泛,特别是深度学习中前沿领域的成果对医学图像的识别、分类、分割、量化等方面起到了积极作用[12]。将深度学习技术与皮肤OCT图像处理相结合,利用深度学习的优势,建立自动化的皮肤疾病智能诊断系统,可以提升皮肤病变图像的检测效率,帮助皮肤科医生快速准确地制定诊疗决策,能够大大降低医疗人力成本[13],对智能医疗的发展有着重要的意义,同时深度学习可推动OCT在临床皮肤疾病诊断领域应用的进一步发展。
1 深度学习与OCT
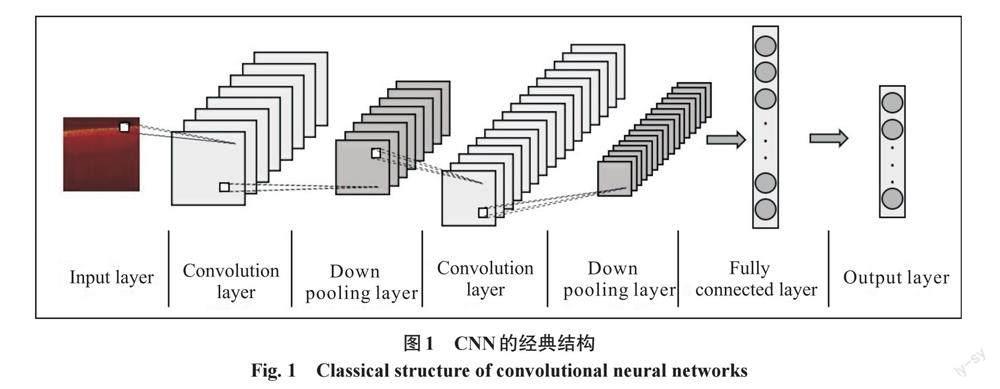
1.1 深度学习在皮肤科的应用优势
深度学习作为机器学习的重要分支,在计算机视觉、语音识别、自然语言处理方面有着广泛的应用[14]。深度学习在皮肤疾病诊断中具有两大优势,一是有深度学习的辅助可以大幅降低皮肤疾病诊断成本、提高效率以及节约医疗资源,原本需要人工分割、识别、分析、诊断的皮肤疾病影像处理任务可全部交给计算机高质量、高效率地完成[15];二是基于深度学习的辅助诊断技术具有客观、标准化的特点,并且可以发现人眼无法发现的高阶特征信息,提升疾病的诊断能力并提高诊断的正确率,在早期防治皮肤疾病中十分重要[20]。卷积神经网络(convolutional neural networks,CNN)作为主流深度学习模型之一,由多层感知机演化而来,具有局部感知和权值共享、降采样等特点[15],在图像处理领域表现出色。CNN是一个拥有输入层、卷积层、池化层、全连接层以及输出层的多层结构(图1)。经典的CNN包括AlexNet[17]、VGGNet[18]、GoogleNet[19]等。
1.2 OCT在皮肤科的应用优势
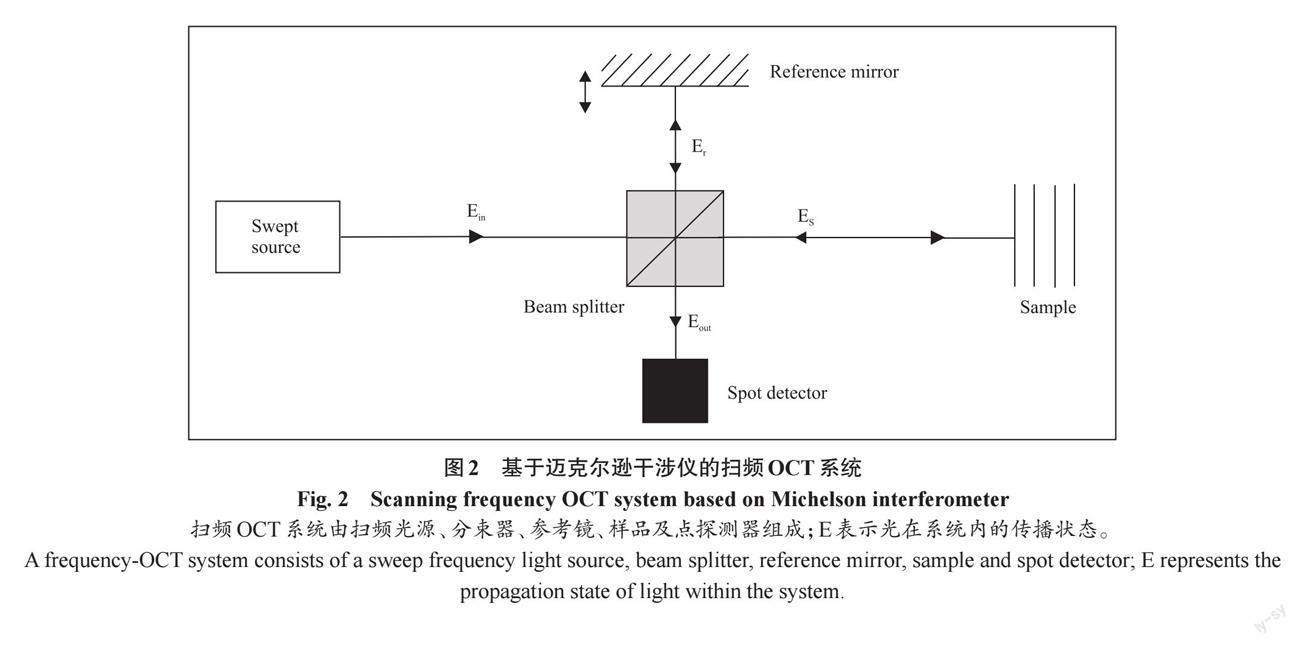
OCT基于光学相干原理,利用光的低相干原理获取组织深度信息,通过扫描获得生物组织的实时二维或三维成像,分辨率可达到微米量级,成像深度为毫米量级[21]。OCT以超发光二极管作为光源,迈克尔逊干涉仪为核心光学结构,通过样品组织中射回来的光信号发生干涉效应,提取组织信息,形成组织图像。图2展示了基于迈克尔逊干涉仪的激光扫频OCT系统结构[22]。OCT技术在皮肤疾病诊断中具有三大优势:一是OCT不局限于皮肤病灶表面,能够无创采集皮下断层图像,通过二维断层图像进行三维重建,为皮肤疾病诊断提供重要的病灶信息[23];二是OCT技术在保证高分辨率的同时,拥有更大的视场和更深的扫描深度,横向、纵向分辨率为1~10 μm,扫描深度约2 mm;三是OCT可以对皮肤病变组织进行无创、原位、动态、实时的成像,可以动态监测病变组织发展过程,已被证明在诊断和监测皮肤疾病(包括恶性肿瘤和炎症)中具有良好的实用性和精确度[24]。
2 深度学习在皮肤OCT图像分类中的应用
2.1 在皮肤癌诊断中的应用
皮肤癌是人类最常见的恶性肿瘤,以非黑色素瘤为主,其中80%为基底细胞癌(basal cell carcinoma,BBC),其余大部分为鳞状细胞癌(squamous cell carcinoma,SCC)[25]。近年来OCT逐步被应用于皮肤癌成像,Mandache等[26]将OCT和深度学习相结合,建立了一种计算机辅助诊断技术,對从莫氏手术、活检以及常规切除组织中获取的OCT图像进行专家级的手工分割和标注,并在整张的OCT图像上提取了大小为256×256的小块图像进行训练。该研究提出了一个专用的网络模型,使用了VGG的思想,并在Dropout层去除部分神经元以防止过拟合。研究结果表明,该模型对SCC病变区域的分类准确率可达到95.93%,对应的敏感性为95.20%,特异性为96.54%。该研究可实现对皮肤癌区域的自动诊断,从而加快手术速度,具有在临床上应用的潜力。
Ho等[27]将全场OCT技术和深度学习相结合对SCC进行分类,从小鼠背部采集了正常、发育不良以及SCC 3类皮肤OCT图像(图3),在使用三维平均滤波对其进行降噪的基础上用减少了参数的ResNet18进行训练,以提升模型的训练效果以及减少计算量。结果表明,模型的整体分类准确度超过了80.00%,具有在临床上应用的潜力。
2.2 在皮肤美容领域中的应用
根据组织学分析,人体皮肤的老化主要是由真皮层特定结构的组织紊乱造成的。真皮基质质量可在一定程度上反应皮肤的老化状态,因此,观察真皮在衰老过程中的结构变化在皮肤美容方面具有重要价值[28]。Josselin等[29]对人体皮肤老化相关的真皮基质质量进行自动评估,使用OCT设备从57名不同年龄段(22岁到79岁之间,平均年龄为56岁)的志愿者面部采集图像,并由经过培训的专家根据4个等级对图像进行打分;在此基础上,学者使用深度学习模型MobileNet-V3在ImageNet数据集上进行预训练,然后在15 993张图像上进行训练和测试,并按4个等级进行分类。研究结果表明,该模型对皮肤基质状态的评分表现出良好性能,分类准确度达到了83.00%,召回率达到了82.00%,可辅助医师评估老化相关的皮肤基质质量变化。
脱发是皮肤美容领域的常见问题,寻找准确可靠的方法监测治疗脱发进展是研发有效脱发治疗手段的挑战之一。Urban等[30]提出了一种有效监测头皮毛发以及毛囊数量的自动评估方法,使用OCT技术结合深度学习对毛发、毛囊数量自动计数。该方法可准确有效监测头发生长治疗的过程。在该研究中,在14名患者的不同头皮位置采集70张OCT图像并进行人工标记(其中20张由2名专家进行冗余标记),使用一种独特设计的CNN进行评估。研究结果表明:深度学习系统预测毛囊数量的平均错误率为11.80%,而预测毛发数量的平均错误率为18.70%;OCT系统可以在3 s内捕获一个头皮位置图像,经过训练后的模型能够在处理OCT扫描图像后1 s内做出有效预测。该方法较人工评估方法节省了大量的时间和精力,可以作为一种监测脱发治疗进展的有效手段。
3 深度学习在皮肤OCT图像分割中的应用
3.1深度学习对指尖皮肤OCT图像分割
指纹识别作为生物特征识别的重要方式,已广泛应用于我们的日常生活中。现有的指纹采集技术测量的都是皮肤的表皮特征,而皮肤的表皮特征可以被扭曲、伪造等方式篡改,因此,提取指尖内部特征成为指纹识别领域的一个重要趋势[31]。OCT作为一种非侵入性、高分辨率的活体成像技术,已广泛应用在指尖皮肤生物特征采集中,可测量皮肤表面、内部的信息,如内部指纹、汗腺等[32](图4)。
Ding等[33]使用定制的光谱域OCT系统(中心波长为1 310 nm,带宽为85.6 nm的宽带光源)采集人体指尖皮肤图像,在此基础上对采集到的OCT图像使用BM3D方法进行降噪,并提出一种改进的U-Net模型即BCL-U-Net。该模型结合了混合空洞卷积、双向长短期记忆网络和残差学习机制,对指尖OCT图像进行分割。研究结果表明:与U-Net、3D U-Net、V-Net等比较,该模型在区域预测精度、轮廓提取精度上都表现更好,且其可以更好地识别汗腺;在内表面混合指纹匹配试验中,等错误率为3.67%,验证了内外部指纹的一致性;在此基础上对皮肤条件较差的手指进行匹配试验,结果表明,内部指纹的等错误率为2.47%,低于表面指纹,表明了内部指纹具有补充或替代表面指纹的潜力。刘凤等[34]提出了一种基于深度学习的指尖OCT图像亚表面指纹重构方法,使用HDCRes-34网络将OCT采集到的B-scan图像分割成角质层、脊部以及谷部3层,然后通过滑窗函数和权值函数重构算法,实现指纹重构(重构效果见图5)。在该研究中,学者提出的网络分割模型的平均像素准确率为0.956,平均交并比为0.873,证明了亚表面指纹在指纹识别领域的潜力和科学意义。
3.2 深度学习对损伤皮肤OCT分割
表皮和结痂检测在研究皮肤伤口的愈合过程以及再上皮化过程中具有重要意义,其中,表皮和结痂厚度是判断再上皮化是否正常的重要指標。为了解皮肤伤口愈合过程以及阐明治疗对伤口愈合的加速作用,Ji等[35]对小鼠皮肤OCT图像中的表皮和结痂区域进行人工标记,在伤口愈合的过程中(对照组及伤后第3天、第7天、第10天、第14天)使用多种深度学习模型对图像进行分割(分割效果见图6),并对模型的分割性能进行了定性和定量比较,用以对啮齿动物皮肤损伤模型的愈合过程中表皮和结痂组织厚度的自动定量进行评估。研究表明,U-Net模型在分割目标结构的同时,能最大限度减少其他低散射干扰组织和噪声的影响,与其他模型(Res-U-Net、DeepLab-V3、PSP-Net和Seg-Net)相比,U-Net具有更高的分割精度,且在各项评价指标上均优于其他模型,尤其是在模型运行速度方面明显快于其他CNN结构,每张图像的处理速度仅为6.7 ms。
Gao等[36]在小鼠皮肤分层的基础上构建了激光诱导的小鼠皮肤激光损伤模型,提出了一种基于深度神经网络模型的激光皮肤损伤定量分析方法。该研究采用U-Net模型及多种加入注意力机制的U-Net模型针对小鼠正常皮肤的多层结构和损伤区域分别进行了精确分割,并对分割结果进行了比较分析。研究表明,基于双注意机制的U-Net模型性能最佳,分割小鼠真皮层和损伤区域的Dice系数达到了0.90以上,分割脂肪层和肌肉层的Dice系数达到了0.80以上。该研究对未来皮肤损伤疾病的早期无创检测和术后恢复检测具有潜在的应用价值。
4 总结与展望
在皮肤科领域,相较于皮肤镜等手段,OCT可以直观地呈现出皮肤内部的组织结构形态,能够无创采集皮下断层图像,可以针对皮肤病变组织进行无创、原位、动态、实时的成像,可以动态监测病变组织的发展过程,已被证明在皮肤疾病的诊断中能够较好地发挥实际效用。随着深度学习逐渐应用到医学图像处理领域,以深度学习为基础的医学图像处理系统能极大地降低人力成本、提升图像处理速度。将深度学习与皮肤OCT技术相结合,在OCT能够采集更丰富病灶信息的同时,利用深度学习实现皮肤疾病OCT图像的自动分析,可为皮肤疾病以及其他疾病的辅助诊断与治疗提供重要参考。
本文综述了深度学习在皮肤OCT图像处理领域的应用。目前二者相结合的应用还处于起步阶段,主要用于皮肤组织结构的分割与识别分类,如皮下指纹结构分割、皮肤损伤评估、皮肤癌的识别分类等。但将深度学习应用于皮肤OCT图像处理还存在一些局限性:如当前的研究都是基于自行采集的数据集,并无公开的、标准的数据集供研究人员使用,数据标准、格式不一,同一类型的研究缺乏横向对比;且由于OCT系统采用了干涉技术,受低相干光源和成像样本自身结构的共同影响,成像过程中的散斑噪声会影响成像质量,对后续深度学习模型的学习效果造成影响。同时,对比OCT在其他生物医学领域的应用,如视网膜分割、血管识别评估等,已有使用深度学习方法对OCT图像预处理的大量研究,如利用生成对抗网络对图像进行去噪等,类似研究在皮肤OCT领域尚未见报道。
未来,OCT技术将在成像分辨率、成像速度上进一步提高,会在皮肤领域得到进一步的应用,对于皮肤OCT图像的处理、评估等需求也会大大提高。深度学习作为图像处理领域的利器,势必会越来越广泛地应用到皮肤OCT图像的处理中。结合深度学习领域的成果,愈发成熟的皮肤疾病自动分析技术将辅助医师快速、准确地进行临床诊断治疗,推动智慧医疗的进一步发展。
參考文献(References):
[1] HUANG D, SWANSON E A, LIN C P, et al. Optical coherence tomography[J]. Science, 1991, 254(5035): 1178-1181.
[2] 倪波, 范应威, 马琼, 等. 光学相干断层成像在临床医学中的应用[J]. 激光生物学报, 2018, 27(6): 481-497.
NI Bo, FAN Yingwei, MA Qiong, et al. Application of optical coherence tomography in clinical medicine[J]. Laser Biology Journal, 2018, 27(6): 481-497.
[3] PATHANIA Y S, SALERNI G, APALLA Z, et al. Non‐invasive diagnostic techniques in pigmentary skin disorders and skin cancer[J]. Journal of Cosmetic Dermatology, 2022, 21(2): 444-450.
[4] KIM K H, PIERCE M C, MAGULURI G, et al. In vivo imaging of human burn injuries with polarization-sensitive optical coherence tomography[J]. Journal of Biomedical Optics, 2012, 17(6): 066012.
[5] COLEMAN A J, PENNEY G P, RICHARDSSON T J, et al. Automated registration of optical coherence tomography and dermoscopy in the assessment of sub-clinical spread in basal cell carcinoma[J]. Computer Aided Surgery, 2014, 19(10): 1-12.
[6] RAJABI A, BITTAR J M, NASCIMENTO V, et al. Optical coherence tomography imaging of melanoma skin cancer[J]. Lasers in Medical Science, 2019, 34(2): 411-420.
[7] ALDAHAN A S, CHEN L, FERTIG R M, et al. Vascular features of nail psoriasis using dynamic optical coherence tomography[J]. Skin Appendage Disord, 2017, 33(2): 102-118.
[8] BOONE M, MARNEFFE A, SUPPA M, et al. High-definition optical coherence tomography algorithm for the discrimination of actinic keratosis from normal skin and from squamous cell carcinoma[J]. Journal of the European Academy of Dermatology & Venereology, 2015, 29(8): 1606-1615.
[9] COBB M J, CHEN Y, UNDERWOOD R A, et al. Noninvasive assessment of cutaneous wound healing using ultrahigh-resolution optical coherence tomography[J]. Journal of Biomedical Optics, 2006, 11(6): 064002.
[10] GAO W, ZAKHAROV V P, MYAKININ O, et al. Medical images classification for skin cancer using quantitative image features with optical coherence tomography[J]. Journal of Innovative Optical Health Sciences, 2016, 9(2): 150628195857004.
[11] LECUN Y, BENGIO Y, HINTON G, et al. Deep learning[J]. Nature, 2015, 521(7553): 436-444.
[12] MAMOSHINA P, VIEIRA A, PUTIN E, et al. Applications of deep learning in biomedicine (Review)[J]. Molecular Pharmaceutics, 2016, 13(5): 1445-1454.
[13] HAN S, PARK I, CHANG S E, et al. Augment intelligence dermatology: deep neural networks empower medical professionals in diagnosing skin cancer and predicting treatment options for 134 skin disorders[J]. Journal of Investigative Dermatology, 2020, 140(9): 1753-1761.
[14] CHEN X, WANG X, ZHANG K, et al. Recent advances and clinical applications of deep learning in medical image analysis[J]. Medical Image Analysis, 2021, 79: 102444.
[15] HEMAVATHI S, VELMURUGAN K J. Skin disease prediction and provision of medical advice using deep learning[J]. Journal of Physics Conference Series, 2021, 1724(1): 012048.
[16] ALJABRI M, ALGHAMDI M. Deep learning in neural networks: an overview[J]. Neural Networks, 2015, 61: 85-117.
[17] KRIZHEVSKY A, SUTSKEVER I, HINTON G. ImageNet classification with deep convolutional neural networks[J]. Advances in Neural Information Processing Systems, 2012, 25(2): 89-90.
[18] SIMONYAN K, ZISSERMAN A. Very deep convolutional networks for large-scale image recognition[C]//3rd International Conference on Learning Representations (ICLR), 2015: 1-14.
[19] SZEGEDY C, LIU W, JIA Y, et al. Going deeper with convolutions[C]//2015 IEEE Conference on Computer Vision and Pattern Recognition(CVPR), 2015: 1-9.
[20] LI L F, WANG X, HU W J, et al. Deep learning in skin disease image recognition: a review[J]. IEEE Access, 2020, 8: 208264-208280.
[21] TADROUS P J. Methods for imaging the structure and function of living tissues and cells: optical coherence tomography[J]. The Journal of Pathology, 2000, 191(2): 115-119.
[22] TOMLINS P H, WANG R K. Theory, developments and applications of optical coherence tomography[J]. Journal of Physics D-applied Physics, 2005, 38(15): 2519-2535.
[23] BREZINSKI M E, FUJIMOTO J G. Optical coherence tomography: high-resolution imaging in nontransparent tissue[J]. IEEE Journal of Selected Topics in Quantum Electronics, 2002, 5(4): 1185-1192.
[24] ZIOLKOWSKA M, PHILIPP C M, LIEBSCHER J, et al. OCT of healthy skin, actinic skin and NMSC lesions[J]. Medical Laser Application, 2009, 24(4): 256-264.
[25] HO C, CALDERON-DELGADO M, LIN M, et al. Classification of squamous cell carcinoma from FF-OCT images: data selection and progressive model construction[J]. Computerized Medical Imaging and Graphics, 2021, 93: 101992.
[26] MANDACHE D, DALIMIER E, DURKIN J R, et al. Basal cell carcinoma detection in full field OCT images using convolutional neural networks[C]//2018 IEEE 15th International Symposium on Biomedical Imaging, 2018: 784-787.
[27] HO C, CALDERON M, CHAN C, et al. Detecting mouse squamous cell carcinoma from submicron full-field optical coherence tomography images by deep learning[J]. Journal of Biophotonics, 2021, 14(1): e202000271.
[28] HAYDONT V, BERNARD B A, FORTUNEL N O. Age-related evolutions of the dermis: clinical signs, fibroblast and extracellular matrix dynamics[J]. Mechanisms of Ageing and Development, 2019, 177(2): 150-156
[29] JOSSELIN B, PAULINE R, SOPHIE G, et al. Utilizing deep learning for dermal matrix quality assessment on in vivo line-field confocal optical coherence tomography images[J]. Skin Research and Technology, 2022, 29(1): 1-8.
[30] URBAN G, FEIL N, CSUKA E, et al. Combining deep learning with optical coherence tomography imaging to determine scalp hair and follicle counts[J]. Lasers in Surgery and Medicine, 2020, 53(1): 171-178.
[31] CHENG Y, LARIN K V. Artificial fingerprint recognition by using optical coherence tomography with autocorrelation analysis[J]. Applied Optics, 2006, 45(36): 9238-9245.
[32] YU Y, WANG H, SUN H, et al. Optical coherence tomography in fingertip biometrics[J]. Optics and Lasers in Engineering, 2022, 151: 106868.
[33] DING B, WANG H, CHEN P, et al. Surface and internal fingerprint reconstruction from optical coherence tomography through convolutional neural network[J]. IEEE Transactions on Information Forensics and Security, 2020, 16: 685-700.
[34] 劉凤, 张文天, 刘浩哲, 等. 基于深度学习的亚表面指纹重构[J]. 计算机学报, 2021, 44(10): 2033-2046.
LIU Feng, ZHANG Wentian, LIU Haozhe, et al. Subsurface fingerprint reconstruction based on deep learning[J]. Journal of Computer Science, 2021, 44(10): 2033-2046.
[35] JI Y, YANG S, ZHOU K, et al. Deep-learning approach for automated thickness measurement of epithelial tissue and scab using optical coherence tomography[J]. Journal of Biomedical Optics, 2022, 27(1): 015002.
[36] GAO T X, LIU S, FAN Y W, et al. Automatic segmentation of laser-induced injury OCT images based on a deep neural network model[J]. International Journal of Molecular Sciences, 2022, 23(19): 11079-11091.

