洋金花与木本曼陀罗叶绿体基因组特征与系统进化分析
毕光耀,丁怡宁,王 丽,胡赛文,李贺敏,雷 明,张占江,夏 至*
洋金花与木本曼陀罗叶绿体基因组特征与系统进化分析
毕光耀1,丁怡宁1,王 丽2,胡赛文1,李贺敏1,雷 明3,张占江3,夏 至1*
1. 河南农业大学农学院,河南 郑州 450046 2. 方城县柳河农业技术推广区域站,河南 方城 473200 3. 广西壮族自治区药用植物园,广西 南宁 530023
以洋金花和木本曼陀罗为材料,分析其叶绿体基因组结构和特征,基于叶绿体组数据探讨洋金花和木本曼陀罗及茄科其他物种的系统发育关系。利用华大MGISEQ-2000PE150测序平台,双末端测序策略对基因组DNA建库测序,用NOVOPlasty组装叶绿体基因组,采用最大似然法(maximum likelihood,ML)构建系统进化树。洋金花和木本曼陀罗的叶绿体基因组全长为155 934 bp和155 939 bp,分别包含131和130个基因,GC值32.3%,具有典型的四分区域结构,包括1个大单拷贝区(large single copy,LSC)、1对反向重复区(inverted repeats,IR)和1个小单拷贝区(small single copy,SSC),各区域序列长度分别为86 354、86 278、25 609、25 720、18 362、18 221 bp。系统发育分析表明,洋金花与曼陀罗属的曼陀罗构成1单系分支,具有100%支持率,而木本曼陀罗属与曼陀罗属构成单系分支,具有100%支持率。结果支持洋金花隶属于曼陀罗属,与曼陀罗亲缘关系较近,木本的木曼陀罗属从曼陀罗属分出,独立为一个属。洋金花和木本曼陀罗叶绿体基因组信息为后期分子鉴定和群体遗传研究奠定基础。
洋金花;木本曼陀罗;叶绿体全基因组;组装;系统发育
药用植物洋金花L.来源于茄科(Solanaceae)曼陀罗属L.,为一年生草本[1],在《中国药典》2020年版中以干燥花入药,中药材名为洋金花[2]。洋金花有效成分为东莨菪碱,临床具有平喘止咳、解痉定痛的功效。用于哮喘咳嗽、皖腹冷痛、风湿痹痛、小儿慢惊及外科麻醉[2]。木本曼陀罗(L.) Lagerh.隶属于木曼陀罗属L.,原产于南美洲,我国引入栽培。《中国植物志》将木曼陀罗属作为一个组放在曼陀罗属[1],2者系统发育关系一直存在争议。近年来,基于分子条形码序列的系统发育结果支持木曼陀罗属为一个独立的属[3]。曼陀罗属植物在我国分布范围广,生态适应幅度大,导致该属植物在形态上产生很多变异,种内的遗传变异十分显著,且该属植物花部特征相似,不易区分[4],形态鉴别困难。中药材洋金花常见的伪品来源于其近缘种曼陀罗L.和毛曼陀罗Miller的花[5]。此外木本曼陀罗(L.) Lagerh.的花东莨菪碱含量较高,也常被误用作洋金花使用[4],严重影响洋金花中医临床用药安全。近年来,有关洋金花的研究主要集中在化学成分分析和药理学研究等方面[6-7],药用植物洋金花和木本曼陀罗的叶绿体基因组组装、特征和系统进化分析未见报道。
叶绿体是植物最重要的细胞器之一,具有一整套用于光合作用、能量代谢、蛋白质合成及氮、硫同化相关的基因,分布在大小为120~180 kb的环状基因组上,具有结构保守、母性遗传等特点[8-9]。叶绿体基因组一般为闭环双链DNA结构,陆生植物的叶绿体基因组结构通常由1个大单拷贝区域(large single copy,LSC)、1个短单拷贝区域(small single copy,SSC)和2个反向重复区域(inverted repeat,IR)组成[10]。叶绿体基因组拥有相对独立的基因组和遗传序列,并且不像核基因组一般有着复杂的重复序列,其基因序列保守,间隔区变异位点丰富,适宜的进化速率能够为植物不同等级的亲缘关系,系统进化关系及遗传多样性研究提供较为可靠的信息[10]。
随着测序技术的不断改进,测序平台的不断升级,一系列组装和注释软件如plasmid SPAdes[11]、NOVOPlasty[12]、GetOrganelle[13]等的开发与更新,多种重要的药用植物叶绿体基因组已完成测序和分析,如人参C. A. Meyer[14]、红豆杉var.(Pilger) Florin[15]、三七(Burkill) F. H. Chen ex C. Chow & W. G. Huang.[16]、铁皮石斛Kimura et Migo[17]、地黄(Gaert.) Libosch. ex Fisch. et Mey.[18]、射干(L.) Redouté[19]、连翘(Thunb.) Vahl[20]等的叶绿体全基因组序列的分析已有相关报道。本研究以洋金花和木本曼陀罗为材料,利用高通量测序方法测定其基因组DNA序列,并对其叶绿体基因组进行组装和注释。分析药用植物洋金花和木本曼陀罗叶绿体基因组序列特征,IR边界特征,间隔区信息位点变异的特征,并对洋金花和木本曼陀罗及其近缘物种共22种(25个样品)植物的叶绿体基因组序列进行系统发育分析,验证其系统的位置,探讨曼陀罗属与木曼陀罗属的系统关系。为药用植物洋金花及其近缘物种的种质资源的鉴定、群体遗传学和遗传多样性研究奠定基础。
1 材料与方法
1.1 材料
洋金花和木本曼陀罗新鲜叶片釆集于广西壮族自治区药用植物园(22°50′47.14′′N, 108°19′30.06′′E),凭证标本号为XZ-2020-17和XZ-2020-16。叶片用硅胶干燥后,装入取样袋带回实验室,置于−80 ℃冰箱备用。样品由河南农业大学农学院中药材系高致明教授和夏至教授鉴定为茄科植物洋金花L.和木本曼陀罗(L.) Lagerh.,凭证标本保存于河南农业大学标本馆,NCBI的GenBank登录号为OK040953和OK040952。其近缘物种叶绿体基因组序列来源于NCBI数据库,实验材料详细信息见表1。
1.2 DNA的提取和高通量测序
采用植物DNA提取试剂盒(天根生化科技有限公司)提取样品干燥新鲜叶片的总DNA,利用1%的琼脂糖凝胶电泳检测DNA完整性。样品送至华大生物科技公司后,进行NanoDrop 2000微量分光光度计(美国Thermo Scientific公司)检测总DNA的纯度和浓度。MGISEQ-2000 PE150测序平台测序,测序完成后,利用华大自主开发的过滤软件SOAPnuke过滤参数。(1)过滤接头:测序read匹配上adapter序列的25%或者以上则删除整条read;(2)过滤低质量数据:如果测序read中质量值低于20的碱基占整条read的30%或者以上则删除整条read;(3)去N:如果测序read中N含量占整条read的1%或者以上,则删除整条read;(4)获得clean reads。数据以FASTQ格式储存,用于后续的拼接和注释。
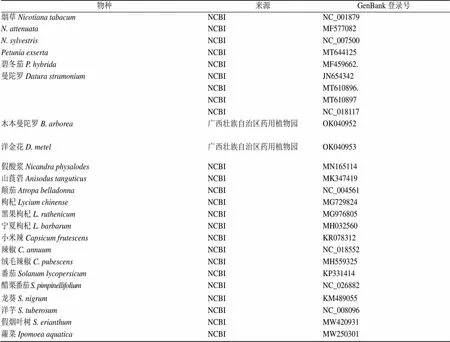
表1 植物样品来源
1.3 叶绿体基因组的组装和注释
叶绿体基因组的拼接釆用NOVOPlasty-master[12]程序,插入片段大小设为150 bp。过滤后的reads用Geneious 11.0.3[21]拼接软件组装成重叠群,并对组装中的简并碱基,进行人工修正。利用Geneq-Annotation of Organellar(https:// chlorobox.mpimp golm.mpg.de/geseq.html),结合NCBI上已报道的曼陀罗L.(GenBank登录号NC_018117)注释结果对洋金花和木本曼陀罗叶绿体全基因组进行基因注释,参数为默认值,最后进行手动调整。tRNA用ANAGORNV1.2.38(https://chlorobox.mpimp golm. mpg.de/geseq.html)预测。注释完成后,提交到NCBI数据库(https://www.ncbi.nlm.nih.gov/ genbank/),GenBank登录号为OK040953和OK040952。利用在线工具OGDRAW-DRAW Organelle Genome Maps(https://chlorobox.mpimp-golm.mpg.de/ OGDraw. html)绘制叶绿体结构图。
1.4 叶绿体基因组IR边界的收缩和扩张分析
IR区域在叶绿体基因中具有高度保守性,IR边界的膨胀和收缩被认为是被子植物叶绿体全基因组大小变化的主要机制[20]。其中被子植物叶绿体基因组长度为120~180 kb。在植物进化过程中,IR/SC边界不同程度的扩张和收缩是导致边界和基因组长度多样性的原因[22]。本研究使用Geneious 11.0.3[21]软件获得茄科10个属11种药用植物叶绿体基因组的IRa/IRb、LSC和SSC和边界基因的序列长度,进行比较分析,探讨茄科植物叶绿体基因组IR边界的收缩和扩张特征。并使用Adobe illustrator软件绘制11种茄科植物叶绿体基因组IR边界对比图。
1.5 叶绿体基因组基因间隔区信息位点分析
在近缘物种中叶绿体基因间隔区往往比叶绿体基因编码区具有更高的变异位点,常被用来分析属间、属内种间物种亲缘关系和系统发育关系。本研究基于茄科10个属22种植物的叶绿体基因组序列特征,利用Geneious 11.0.3[21]的MAFFT[23]功能进行多重比较,导出Fasta格式文件,后用Phylosuite vl.2.1[24]提取22个物种叶绿体基因组32个共有的间隔区,统计这些间隔区的信息位点百分率,为下一步构建茄科物种系统进化关系提供分子标记。
1.6 系统发育分析
基于本研究新报道的洋金花和木本曼陀罗的叶绿体基因组,同时选取NCBI已报道的茄科物种叶绿体基因组共22个(25个样品),以近缘的旋花科(Convolvulaceae)植物蕹菜Forsk为外类群(表1)。利用Phylosuite vl.2.1[24]软件基于MAFFT[23]参数设置进行多重比对。系统发育分析采用最大似然法(maximum likelihood,ML),利用CIPRES Science Gateway服务器(http://www. phylo.org/)中RAxML(version 8.2)软件[25]构建系统发育树。利用Bootstrap(BS,1000次重复)检验各分支的支持率。系统发育树导出后利用FigTree version 1.4.2进行查看。
2 结果与分析
2.1 洋金花和木本曼陀罗叶绿体基因组的结构特征
测序结果去除接头和低质量的数据后,组装和注释后得到洋金花和木本曼陀罗的完整叶绿体基因组。结果表明,洋金花和木本曼陀罗叶绿体基因组均为共价闭合的双链环状分子(图1)。洋金花和木本曼陀罗的叶绿体全基因组长度分别为155 934 bp(表2)和155 939 bp(表2),其中LSC长度分别为86 354 bp和86 278 bp,SSC分别为18 362 bp和18 221 bp,二者的2个IR长度分别为25 609 bp和25 720 bp。洋金花和木本曼陀罗的叶绿体全基因组中GC含量分别为37.9%、37.8%。在LSC区域中GC含量皆为35.9%,SSC区域中GC含量分别为32.3%和32.1%,IRs区域中GC含量相同,皆为43.1%。洋金花和木本曼陀罗的叶绿体全基因组分别注释131和130个基因,洋金花包括85个蛋白编码基因(protein coding genes,PCGs),38个tRNA以及8个rRNA,其中LSC区完全包含58个PCGs和23个tRNA,SSC区包含11个PCGs和1个tRNA,IRs区域内包含11个PCGs和14个tRNA以及全部(8个)的rRNA,另外、、基因分别横跨LSC/IRb、LSC/IRb、IRb/SSC和SSC/IRa边界。而木本曼陀罗包括85个PCGs,37个tRNA以及8个rRNA,其中LSC区完全包含58个PCGs和22个tRNA,SSC区包含10个PCGs和1个tRNA,IRs区域内包含12个PCGs和14个tRNA以及全部(8个)的rRNA,另外、、、基因分别横跨LSC/IRb、LSC/IRb、IRb/SSC和SSC/IRa、IRb/SSC边界。详细信息见表2。
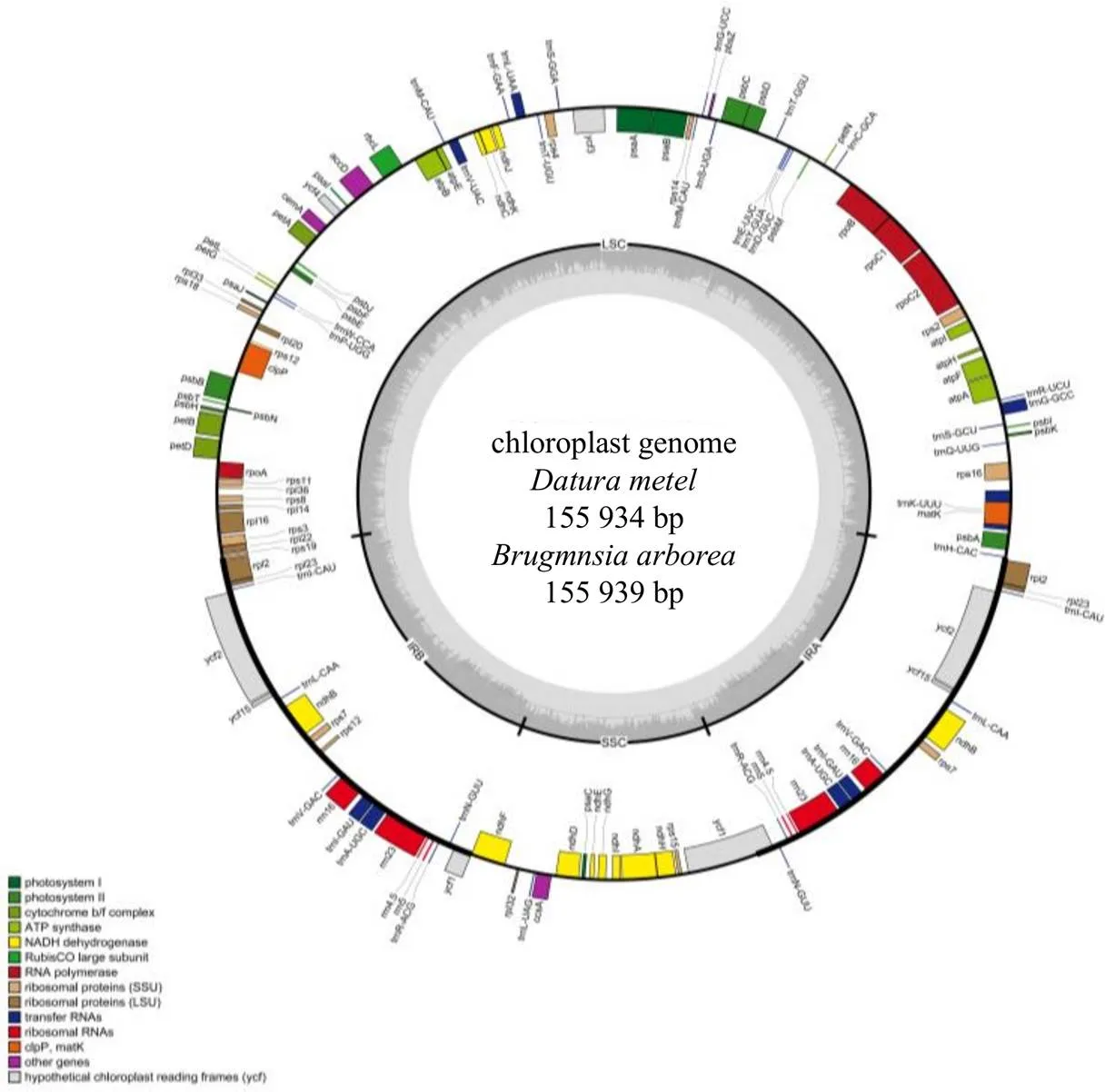
图1 洋金花和木本曼陀罗的叶绿体基因组
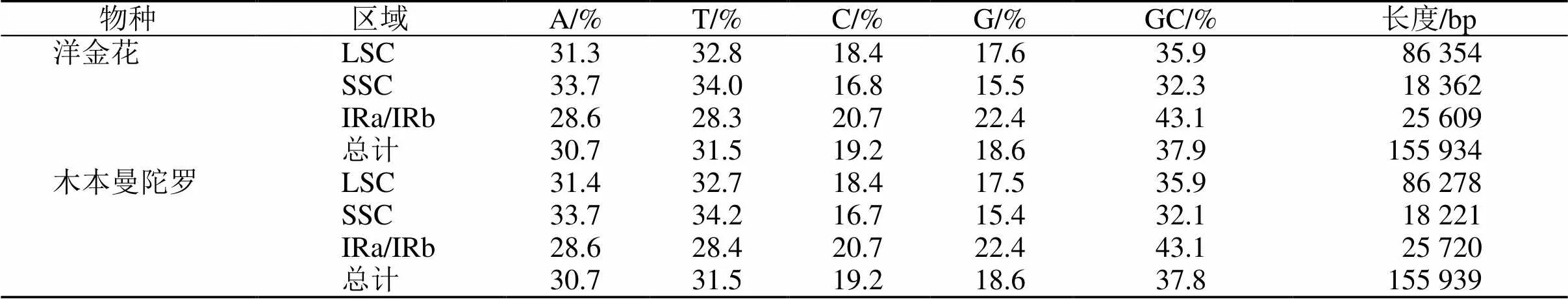
表2 洋金花和木本曼陀罗叶绿体基因组碱基组成及特征
2.2 洋金花和木本曼陀罗叶绿体基因组的组成和特征分析
洋金花叶绿体基因组共包括131个基因,非重复基因113个,其中85个PCGs、8个rRNA基因与38个tRNA基因。其中,PCGs中与自我复制相关基因除rRNA基因和tRNA基因外(表3),还包括13个核糖体小亚基基因、11个核糖体大亚基基因和4个RNA聚合酶亚基基因;光合作用相关的基因有45个,包括12个NADH脱氢酶基因、5个光系统I基因、14个光系统II基因、6个细胞色素复合物编码基因、6个ATP合酶基因、1个二磷酸核酮糖羧化酶大亚基基因和1个依赖ATP的蛋白酶单元p基因;此外还有4个其他功能基因及8个未知功能基因。在tRNA中、、、、、、各有2个拷贝;4个rRNA均有2个拷贝,分别位于反向重复区IRa和IRb。核糖体蛋白大小亚基编码的基因中,、、和这3个基因均有2个拷贝,其余为1个拷贝。NADH脱氢酶亚基中的基因及未知功能蛋白基因和的拷贝数均为2。内含子在基因表达调控中发挥重要作用,洋金花叶绿体基因组中有15个基因有内含子。其中,、、、、、、、、、、、、、、各有1个内含子,而、具2个内含子。基因位于基因内,整个编码区为内含子的一部分,存在序列共用现象;基因的3′端与基因的5′端,基因的3′端与基因的5′端、的3′端与的5′端重叠。木本曼陀罗和洋金花相比在tRNA上缺少了,其他基因组成与洋金花一致。

表3 洋金花和木本曼陀罗叶绿体基因组编码的基因
a和b分别表示含有1个和2个内含子;c表示含有2个拷贝基因;d表示木本曼陀罗缺少该基因
a and b represent one and two introns, respectively; c indicates that it contains two copies of genes; d indicates thatlacks this gene
2.3 茄科部分物种叶绿体全基因组特征比较分析
茄科植物叶绿体基因组特征的比较分析见表4,洋金花和木本曼陀罗及其近缘物种(共22个物种)叶绿体基因组的序列长度范围介于155 004~157 390 bp,其中,黑果枸杞的叶绿体全基因组长度最短(155 004 bp),绒毛辣椒的叶绿体基因组最长(157 390 bp)。洋金花的叶绿体基因组长度为155 934 bp,木本曼陀罗的叶绿体基因组长度为155 939 bp,位于茄科其他20个物种的叶绿体基因组长度范围之内。洋金花和木本曼陀罗及其近缘物种(共22个物种)叶绿体基因组GC含量的范围为37.6%~37.9%,山莨菪的GC含量最低(37.6%),洋金花的GC值最高为37.9%,而木本曼陀罗GC含量为37.8%。利用Geneious 11.0.3软件获取洋金花和木本曼陀罗及其近缘物种(共22个物种)叶绿体基因组的IR、LSC和SSC区序列。结果表明,IR区的序列长度范围介于25 342~25 904 bp,其中的IR区最短(25 342 bp),颠茄的IR区最长(25 904 bp),洋金花的IR区的长度为25 609 bp,木本曼陀罗的IR区的长度为25 720 bp,2者均位于茄科其他20个物种IR区长度范围之内。LSC区序列长度范围介于85 737~87 688 bp,其中洋芋的LSC区长度最短(85 737 bp),绒毛辣椒的LSC区长度最长(87 688 bp),洋金花的LSC区的长度为86 354 bp,木本曼陀罗的LSC长度为86 278 bp,都介于茄科其他20个物种LSC区长度范围之内。SSC区序列长度范围介于17 487~18 642 bp,其中山莨菪的SSC区最短(17 487 bp),的SSC区最长(18 642 bp),洋金花SSC区长度为18 362 bp,木本曼陀罗SSC区长度为18 221 bp,二者介于茄科其他20个物种SSC区长度范围之内。
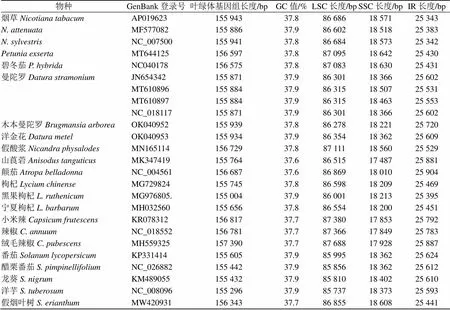
表4 茄科22种植物叶绿体基因组的特征
2.4 茄科10个属11个物种叶绿体全基因组边界的特征
选取茄科10个属11个物种的叶绿体全基因组,比较分析洋金花和木本曼陀罗叶绿体基因组与茄科其他物种在进化过程中IR/SC边界的扩张和收缩情况(图2)。茄科10个属11个物种叶绿体全基因组的IR-LSC和IR-SSC边界比较显示,IR/SC边界具有高度的保守性,其LSC/IRb边界(JLB),SSC/IRb边界(JSB)、SSC/IRa边界(JSA)和LSC/IRa边界(JLA)的侧翼基因完全相同,但边界基因的扩张程度存在一定的差异。茄科11个物种中,9个物种JLB边界全部位于基因内,其中洋金花和木本曼陀罗的JLB边界扩张范围,位于9个物种之内。但烟草和碧冬茄的基因却全部位于LSC区域,距JLB边界有5 bp。JSB边界扩张范围显示,茄科物种的侧翼基因完全相同,均为和基因,洋金花和木本曼陀罗的SSC/IRb边界(JSB)与洋芋,辣椒和曼陀罗一致,存在基因于基因重叠现象。JSA边界的扩张范围显示,SSC和IRa区域基因完全相同,均为基因,基因在IRa区长度范围为995~1440 bp,在SSC区长度范围为4205~4726 bp。其中洋金花和木本曼托罗的基因在SSC区域为4527 bp和4422 bp,在IRa区域为1101 bp和1049 bp,位于茄科11个物种JSA边界扩张范围之内。JLA边界扩张范围显示,其侧翼基因完全相同,均为(位于IRa区)和(位于LSC区)2个基因。其中11个物种的和2个基因距JLA边界碱基长度范围变异不大,分别为56~166 bp和4~30 bp。洋金花和木本曼陀罗的和2个基因距JLA边界的序列长度为136、124 bp和8、7 bp,位于11个物种JLA边界扩张范围之内。
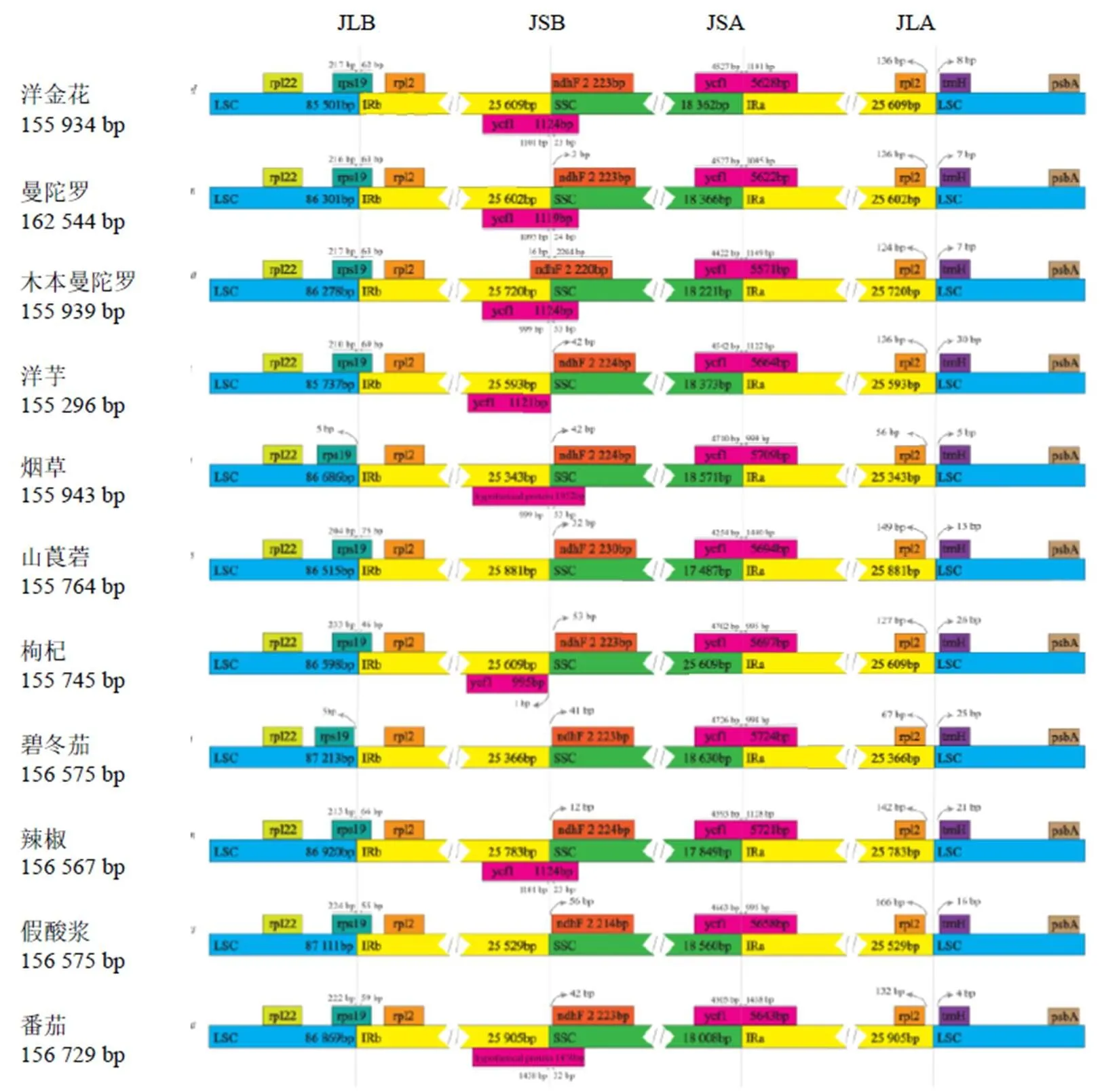
图2 洋金花和木本曼陀罗及其近缘种的叶绿体全基因组边界
2.5 茄科叶绿体基因组基因间隔区信息位点分析
根据茄科10个属22种植物的32个叶绿体基因组间隔区信息序列特征统计结果(表5),在32个共同的叶绿体基因间隔区中,变异位点百分率变化范围为3.49%~18.70%,最高的为基因间隔区,其变异位点百分率为18.70%。信息位点超过10%的基因间隔区有11个,分别为、、、、、、、、、、。这些变异位点百分率较高的叶绿体基因间隔区,能提供足够多的信息位点,为茄科属间和种间物种进化关系及分子鉴定提供较高的分辨率。
2.6 系统发育分析
选取洋金花和木本曼陀罗及其近缘物种共22种(25个样品)叶绿体全基因组进行系统发育树的构建,以旋花科植物蕹菜Forsk作为外类群,利用ML法构建系统发育树(图3)。结果显示取样茄属、辣椒属、枸杞属、曼陀罗属、烟草属、碧冬茄属的单系性都得到100%支持。24个分枝节点中有22个的支持率都为100%,另外2个为98%和99%,聚类结果较为可靠。洋金花和曼陀罗以100%支持率聚在一起,它们所在的曼陀罗属与木曼陀罗属聚在1支,且有100%的支持率。

表5 茄科22个物种的32个叶绿体基因间隔区矩阵位点信息

图3 基于叶绿体全基因组构建的系统发育树
3 讨论
中药材洋金花作为我国药典收载的药材之一,与其近缘物种木本曼陀罗均富含东莨菪碱,具有重要的药用和经济价值。本研究完成了药用植物洋金花和木本曼陀罗的叶绿体基因组的测序,组装与注释。结果表明,药用植物洋金花和木本曼陀罗的叶绿体基因组,均具有典型的4分区域结构(图1),包括1个LSC区、1对IR区和1个SSC区,长度分别为155 934 bp和155 939 bp。其叶绿体基因组长度位于茄科其他20个物种叶绿体基因组长度范围之内。洋金花和木本曼陀罗的叶绿体全基因组分别包含131和130个基因,洋金花包括85个PCGs,38个tRNA以及8个rRNA,木本曼陀罗和洋金花相比,在tRNA上缺少了基因,其余基因组成一样。
基于茄科10个属11个物种的叶绿体全基因组IR边界比较分析(图2),洋金花和木本曼陀罗与茄科其他8个属植物的叶绿体基因组IR边界均具有高度的保守性。其LSC/IRb边界(JLB),SSC/IRa边界(JSA)和LSC/IRa边界(JLA)的侧翼基因完全相同,但边界侧翼基因的扩张序列长度存在差异,洋金花和木本曼陀罗的上述边界侧翼基因扩张范围,均为于茄科物种叶绿体基因组IR边界的扩张范围之内。洋金花和木本曼陀罗的SSC/IRb边界(JSB)与洋芋、辣椒和曼陀罗一致,存在基因于基因重叠现象。基因与基因重叠现象同样也出现在龙胆科药用植物叶绿体基因组[26]。IR边界比较分析结果表明洋金花和木本曼陀罗与茄科其他物种的叶绿体全基因组具有较高保守性,叶绿体基因组适合用来解决茄科属级以上分类等级的系统发育关系。
基于茄科10个属22种植物的32个叶绿体基因组间隔区信息序列特征统计表明(表5),茄科的叶绿体基因组编码区较为保守,在32个共同的叶绿体基因间隔区中,变异位点百分率变化范围为3.49%~18.70%,变异位点百分率最高的为基因间隔区,其变异位点百分率为18.70%。本研究结果支持韩建萍等[3]结果,支持基因间隔区作为药用植物洋金花与其伪品的DNA条形码序列。此外,信息位点超过10%的叶绿体基因间隔区有11个,分别为、、、、、、、、、、。这些基因间隔区在茄科的属间和种间提供了丰富的信息位点。由于叶绿体基因组在大多数被子植物中为母系遗传,重组率低,核苷酸置换率适中[27],进一步结合双亲遗传的核基因片段联合分析,为茄科植物属下物种杂交起源,多倍体物种的形成和系统进化分析提供可靠的分子标记片段。
为进一步界定药用植物洋金花和木本曼陀罗在茄科的系统位置,探讨曼陀罗属与木曼陀罗属的系统关系,基于茄科22个物种(25个样品)叶绿体基因组构建系统发育树(图3)显示,茄科各属植物均聚成有较高支持率的分枝。其中洋金花与曼陀罗构成1单系分支(支持率100%),表明药用植物洋金花隶属于曼陀罗属。木曼陀罗属(木本曼托罗为代表)与曼陀罗属(包括洋金花与曼陀罗为代表)组成1单系分支(支持率100%),二者互为姐妹群。木曼陀罗属与曼陀罗属的系统关系一直存在争议,一种认为木曼陀罗属应独立成一属,有的认为应作为曼陀罗属的一个组[1]。Persoon把曼陀罗属内具有木本特性,光滑而不规则开裂蒴果的木本曼陀罗分出,成立木曼陀罗属L.[28]。《中国植物志》认为Persoon所提出的特征:光滑而不规则开裂的蒴果,在曼陀罗属的另一个组中同样是存在的,因此将它作为曼陀罗属的一个组[1]。本研究的叶绿体基因组数据结果支持近年来分子系统学研究结果[3,29-30],草本的曼陀罗属和木本的木曼陀罗属是2个独立的属。木曼陀罗属具有一系列独特形态特征(近裔共性)也支持分子数据的结果,如木本的习性、俯垂的花、俯垂的浆果且表面平滑等这些形态特征与曼陀罗属有着显著的区别。
近年来,随着新一代测序技术的成本降低,基于叶绿体基因组序列作为超级条形码对物种的分子鉴定应用越来越广泛[31]。洋金花作为常用中药材之一,其商品中常混有同属有毒植物曼陀罗的花,影响临床用药安全[3-4]。本研究首次报道药用植物洋金花和木本曼陀罗的叶绿体基因组,丰富了曼陀罗属及其近缘属叶绿体基因组数据,比较分析这些近缘物种叶绿体基因组的特征和差异,为中药材洋金花的精准鉴定提供理论依据。此外,茄科植物是许多大宗蔬菜和药用植物的来源,本研究结果不仅为曼陀罗属药用植物的提供分子鉴别,也为茄科植物的分子鉴定,遗传多样性及种质资源保护研究奠定基础。
利益冲突 所有作者均声明不存在利益冲突
[1] 中国植物志编辑委员会. 中国植物志(第67(1) 卷) [M]. 北京: 科学出版社, 1978: 147.
[2] 中国药典 [S] 一部. 2020: 279.
[3] 韩建萍, 李美妮, 罗焜, 等. DNA条形码鉴定洋金花及其伪品 [J]. 药学学报, 2011, 46(11): 1408-1412.
[4] 周太炎, 徐国钧, 裴鑑, 等. 五种曼陀罗的植物分类及生药鉴定研究 [J]. 药学学报, 1955(2): 149-178.
[5] 黄淑贤, 李书渊, 房志坚. 木曼陀罗花与洋金花的鉴别研究 [J]. 广东药学院学报, 2005(1): 17-18.
[6] 井佳楠, 吕邵娃, 王秋红, 等. 洋金花化学成分和药理作用及临床应用研究进展 [J]. 中草药, 2016, 47(19): 3513-3521.
[7] 杨炳友, 杨春丽, 刘艳, 等. 洋金花根化学成分研究 [J]. 中国中药杂志, 2018, 43(8): 1654-1661.
[8] Twyford A D, Ness R W. Strategies for complete plastid genome sequencing [J]., 2017, 17(5): 858-868.
[9] Yu X Y, Zuo L H, Lu D D,. Comparative analysis of chloroplast genomes of fivespecies: Genome comparative and evolution analysis [J]., 2019, 689: 141-151.
[10] 邢少辰, Clarke J L. 叶绿体基因组研究进展 [J]. 生物化学与生物物理进展, 2008, 35(1): 21-28.
[11] Antipov D, Hartwick N, Shen M,. plasmidSPAdes: assembling plasmids from whole genome sequencing data [J]., 2016, 32(22): 3380-3387.
[12] Dierckxsens N, Mardulyn P, Smits G. NOVOPlasty:assembly of organelle genomes from whole genome data [J]., 2017, 45(4): e18.
[13] Jin J J, Yu W B, Yang J B,. GetOrganelle: A fast and versatile toolkit for accurate de novo assembly of organelle genomes [J]., 2020, 21(1): 241.
[14] Kim K J, Lee H L. Complete chloroplast genome sequences from Korean ginseng (Nees) and comparative analysis of sequence evolution among 17 vascular plants [J]., 2004, 11(4): 247-261.
[15] Zhang Y Z, Ma J, Yang B X,. The complete chloroplast genome sequence ofvar.(Taxaceae): Loss of an inverted repeat region and comparative analysis with related species [J]., 2014, 540(2): 201-209.
[16] 宋菊, 龙月红, 林丽梅, 等. 五加科植物叶绿体基因组结构与进化分析 [J]. 中草药, 2017, 48(24): 5070-5075.
[17] Konhar R, Biswal D K, Debnath M,. Complete chloroplast genome sequence offrom northeastern India [J]., 2016, 4(5): e01088-16.
[18] Xia Z, Li C C, Hu S W,. The complete chloroplast genome of Chinese medicine cultivar species of(Orobanchaceae) [J]., 2021, 6(1): 290-292.
[19] Li C C, Hu S W, Ding Y N,. The complete chloroplast genome of Chinese medicinal herb(L.) Redouté (Iridaceae) [J]., 2021, 6(2): 331-332.
[20] Wang W B, Yu H, Wang J H,. The complete chloroplast genome sequences of the medicinal plant(Oleaceae) [J]., 2017, 18(11): 2288.
[21] Kearse M, Moir R, Wilson A,. Geneious Basic: An integrated and extendable desktop software platform for the organization and analysis of sequence data [J]., 2012, 28(12): 1647-1649.
[22] Dong W L, Wang R N, Zhang N Y,. Molecular evolution of chloroplast genomes of orchid species: Insights into phylogenetic relationship and adaptive evolution [J]., 2018, 19(3): 716.
[23] Katoh K, Standley D M. MAFFT multiple sequence alignment software version 7: Improvements in performance and usability [J]., 2013, 30(4): 772-780.
[24] Zhang D, Gao F L, Jakovlić I,. PhyloSuite: An integrated and scalable desktop platform for streamlined molecular sequence data management and evolutionary phylogenetics studies [J]., 2020, 20(1): 348-355.
[25] Stamatakis A. RAxML version 8: A tool for phylogenetic analysis and post-analysis of large phylogenies [J]., 2014, 30(9): 1312-1313.
[26] 董博然, 赵志礼, 倪梁红, 等. 龙胆科叶绿体基因组结构特征及其药用植物鉴定意义 [J]. 中草药, 2020, 51(6): 1641-1649.
[27] Drouin G, Daoud H, Xia J N. Relative rates of synonymous substitutions in the mitochondrial, chloroplast and nuclear genomes of seed plants [J]., 2008, 49(3): 827-831.
[28] Persoon C H.[M]. Parisiis Lutetiorum: C.F. Cramerum, 1805: 216-217.
[29] Olmstead R G, Bohs L, Migid H A,. A molecular phylogeny of the Solanaceae [J]., 2008, 57(4): 1159-1181.
[30] Bye R, Sosa V. Molecular phylogeny of the jimsonweed genus(Solanaceae) [J]., 2013, 38(3): 818-829.
[31] 姜汶君, 郭梦月, 庞晓慧. 叶绿体基因组在药用植物鉴定及系统进化研究中的应用 [J]. 世界中医药, 2020, 15(5): 702-708.
Characterization and phylogenetic analysis of complete chloroplast genome ofand
BI Guang-yao1, DING Yi-ning1, WANG Li2, HU Sai-wen1, LI He-min1, LEI Ming3, ZHANG Zhan-jiang3, XIA Zhi1
1. College of Agronomy, Henan Agricultural University, Zhengzhou 450046, China 2. Liuhe Agricultural Technology Extension Regional Station of Fangcheng County, Fangcheng 473200, China 3. Guangxi Botanical Garden of Medicinal Plants, Nanning 530023, China
To analyze the structure and feature of complete chloroplast genome of medicinal plantsand, so as to construct the phylogenetic relationship between the two species and their relatives.MGISEQ-2000PE150 was used to sequence the geomic DNA ofwith the paired-end strategyin Beijing Genomics Institute (China). The complete chloroplast genome was assembled using NOVO Plasty software, and sequence analysis was performed based on gene annotation results. Phylogenetic analyses were performed using Maximum-Likelihood (ML) methods.The complete chloroplast genome ofandwere 155 934 bp and 155 939 bp in length including 131 and 130 genes respectively, with a GC content of 32.3%. The chloroplast genome exhibited a typical quadripartite structure, including a large single copy region (LSC), a pair of inverted repeats (IR), and a small single copy (SSC), and the each region lengths of which were 86 354, 86 278, 25 609, 25720, 18 362, 18 221 bp, respectively. Phylogenetic analyses result indicated thatwas sister towith bootstrap 100%. The generaandformed one monophyletic group with bootstrap 100%.This study verified thatbelonged to the, and it was related to. The genusshould be separated from genus. The information of chloroplast genome provided foundation for subsequent studies on molecular identification and genetic diversity.
L.;(L.) Lagerh.; chloroplast genome; assembly; phylogenetic analysis
R282.12
A
0253 - 2670(2022)22 - 7191 - 10
10.7501/j.issn.0253-2670.2022.22.023
2022-04-07
河南省科技攻关项目(222102110137);河南省高等学校重点科研项目计划(22A360010);国家自然科学基金-河南联合基金项目(U1404302)
毕光耀(1998—),男,硕士研究生,研究方向为中药资源的分子鉴定。E-mail: 903592548@qq.com
夏 至,教授,主要从事中药资源的分子鉴定及分子生药学研究。E-mail: xiazhiemail@126.com
[责任编辑 时圣明]

