SPU测试及其在全基因组多元统计分析中的应用*
郑州大学公共卫生学院计算机与卫生统计学教研室(450001)
罗子潇 杨永利 贾晓灿 王玉平 施学忠△
全基因组关联分析(genome-wide association study,GWAS)在识别疾病的常见变异方面取得了巨大进展,目前已经报道上万个单核苷酸多态性(single nucleotide polymorphism,SNP)位点与数百种复杂疾病存在关联,这为表型变异的遗传基础提供了前所未有的视角[1-2]。但GWAS是基于个体水平基因型和表型数据的分析,因此需要更有效的方法基于汇总统计数据来识别复杂疾病中的罕见变异[3-4]。MGAS(multivariate gene-based association test by extended Simes procedure)和metaCCA(summary statistics-based multivariate meta-analysis of genome-wide association studies using canonical correlation analysis)是目前已知分析多元表型和基因型相关关系的有效方法,有不涉及候选基因等优点,但同时在确定样本量和识别罕见变异等方面也存在局限性[5-7]。对此,Wei Pan等[8]提出了动力分数测试(sum of powered score tests,SPU),即利用不同的参数值来构造SNP数据驱动和变化的权重,从而适应多个SNP之间未知的关联强弱和关联方向,该方法可以在基因和通路两水平上进行表型与基因型相关分析。本文将重点介绍不同关联分析下SPU测试的方法、原理和实现,尤其是在基因和通路水平的多元表型与基因型相关分析,并探讨其应用前景。
不同关联性分析下的SPU测试
1.多SNP-单性状关联性分析
GWAS是逐个检测每个SNP,然后进行多次检测调整,选出符合要求的SNP。然而,由于罕见变异内包含弱相关信号以及极小的等位基因频率(minor allele frequency,MAF)导致GWAS效能较低[8]。Wei Pan等[9]提出了一类适用于汇总统计数据自适应权值及其相应的加权检验法(adaptive sum of powered score tests,aSPUs)。该测试考虑了SNP之间的关联性强弱及方向,适用于多SNP单性状关联性研究,其对多个GWAS的大规模meta分析以及单个GWAS或对单个SNP的GWAS汇总统计具有实际可用性。基于路径的GWAS方法是利用基因功能方面的先验生物学知识来促进对GWAS数据集的分析,在此基础上aSPUs测试也扩展到通路分析(aSPUspath)[10]。
2.单SNP-多性状关联性分析
aSPU测试的主要思想是在广义估计模型(generalized estimation equation,GEE)的框架下构建不同权重的测试,从这类加权测试中选择最强大的一种,使其能够在多种情况下保持高效能。aSPU测试旨在分析在个体水平或汇总统计下的单性状多SNP相关性。因此,Wei Pan等[11]又将aSPU扩展到适应于汇总统计数据的多性状-单SNP关联性研究(the SPU and aSPU tests for multiple traits-single SNP association with GWAS summary statistics,MTaSPUs)。
3.多SNP-多性状关联性分析
基因与多种性状的遗传关联研究已经变得越来越重要,不仅因为它有潜力提高统计效能,也因为其考虑了复杂疾病不同表型之间的相关性。2016年II-Youp Kwak等[12]提出了基于基因的自适应的测试(MTaSPUsSet),用GWAS汇总统计对多个性状进行关联分析并将其扩展到通路水平(MTaSPUsSetPath)。与传统多基因关联分析相比,该方法在SNP和性状层面上都是自适应的,并考虑了SNP之间和不同性状之间可能存在的各种关联模式,使该测试在大部分情况下保持高功率。该方法也适用于从单个GWAS或多个GWAS的meta分析中获得的Z统计量或P值的汇总数据[12-13]。
基因和通路水平SPU模型的建立
1.aSPU模型的建立
(1)构建GEE模型:
假设对于每个目标有i=1,…,n,有k个性状Yi=(yi1,yi2,…,yik)′,xi=0,1或2为相关SNP的基因型得分,zi=(zi1,zi2,…,ziq)是协变量q的行向量。SNP和协变量通过边际广义线性模型(GLM)建模:
g(μi)=ηi=Ziφ+Xiβ=Hiθ
通过求解GEE,得到了两个相容的渐近正态估计:
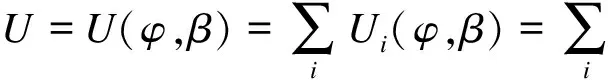
(2)建立零假设H0:β=(β1,…,βk)′=0;H1:β≠0;
为了构建具有协变量Zi的基于分数的测试,在假设特征具有独立工作相关结构的零假设下的得分向量为:
(3)构造SPU测试:
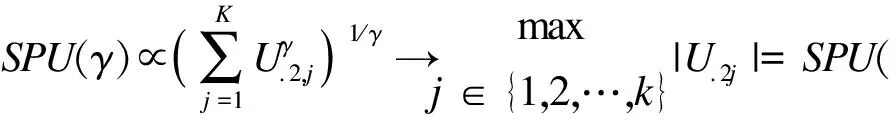
由于SPU测试的幂函数曲线很难描述,所以我们用SPU测试的P值来估计它的幂函数。自适应地选择一个SPU测试,形成aSPU测试:
2.多SNP-单性状关联性分析的模型建立
(1)基因水平的关联性分析——aSPUs的模型建立
首先假设个体的基因型和表型数据是可用的,采用如下广义线性模型:
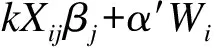
U的协方差矩阵可以估计为:
在H0成立的条件下,其均值为:
通过模拟SPU和aSPU测试的个体基因型和表型数据,仅利用汇总数据Z值就可以定义相应的测试:
(2)通路水平的关联性分析——aSPUsPath的模型建立
用Z分数代替P值构建模型的想法也可以扩展到适应性通路测试,定义基于基因和通路水平的SPU检验为:
PathSPU(γ,γG:S)=∑g∈SSPU(γ:g)Γg
其中两个整数γ>0和γG>0分别用于SNP和基因水平上的自适应加权。例如,当仅有较少的基因(或SNP)与性状相关时,要想效率更高则需要更大的γG(或γ)。但由于(γ,γG)的最佳值是未知的,为了自适应的选择(γ,γG),则提出:

3.多SNP-多性状关联性分析的模型建立
(1)基因水平的关联性分析——MTaSPUsSet的模型建立
假设有d个SNP(例如在基因检测中)其加性基因型分数g=(g1,…,gd)′通过应用广义线性模型我们首先考虑表型Yh:
对于给定的数据集{(Yih,gi,ci):i=1,…,n}有n个对象,βh的得分向量Uh=(Uh1,…,Uhd)′如下:
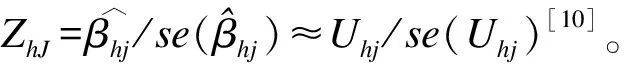
由于(γ1,γ2)的最优值是未知的,从而提出了一个自适应选择(γ1,γ2)的方法:
(2)通路水平的关联性分析——MTaSPUsSetPath的模型建立
对仅有GWAS汇总统计的案例进行基于路径的多性状关联测试,给出一个含有|S|基因的途径S,在SNP水平上,对于基因g的第dg个SNP其Z值为:
Z(ig)=(Z(ig)1,Z(ig)2,…,Z(ig)dg)
将基于基因和路径的测试定义为一个性状,则多个性状为:
SPUsPath(γ1,γ2;Z(i),S)=(∑g∈SSPU(γ1;Z(ig)γ2)/|S|)1/γ2
MTSPUsSetPath(γ1,γ2,γ3;Z,S)=
其中γ1≥1,γ2≥1,γ3≥1分别对SNP、基因和性状进行加权以自适应的选择(γ1,γ2,γ3),从而提出:
SPU模型的软件实现
通过R软件的aSPU软件包实现(https://cran.r-project.org/web/packages/aSPU/),本研究中以MTaSPUsSet和MTaSPUsSetPath为例,介绍模型的实现。
1.MTaSPUsSet的模型实现
将原始GWAS汇总统计结果整理后得到包含在LCORL基因上的单个SNP对应不同性状的P值(Ps)或Z值(Zs)的数据集。MTaSPUsSet的软件实现过程如下:
(1)利用Plink计算SNP之间的相关性(corSNP),以欧洲后裔人群为例,输人文件为SNP_id,代码为:
plink2—file hapmap3—extract SNP_id—keepCEU_ hapmap—r2 inter-chr with-freqs—ld-window-r20-make-bed-out uppro
输出结果为uppro,即为corSNP。
(2)下载aSPU安装包,利用estcov函数计算表型之间的相关性(corPhe),代码为:
corPhe=estcov(Ps,Ps=True)
(3)使用以下命令执行MTaSPUsSet:
library(aSPU)
(outFP<-MTaSPUsSet(PsF,corSNP=corSNPF,corPhe=corPheF,pow=c(1,2,4,8),pow2=c(1,2,4,8),n.perm=100,Ps=TRUE))
即可得出女性在不同SNP权重和不同性状权重下用MTaSPUsSet计算的LCORL基因与身高、体重、BMI、腰围、臀围和腰臀围这六个性状相关性的P值,结果如表1。MTaSPUsSet=0.821782,即在不同权重取值下均适用的该基因-多性状关联测试的P值为0.821782。
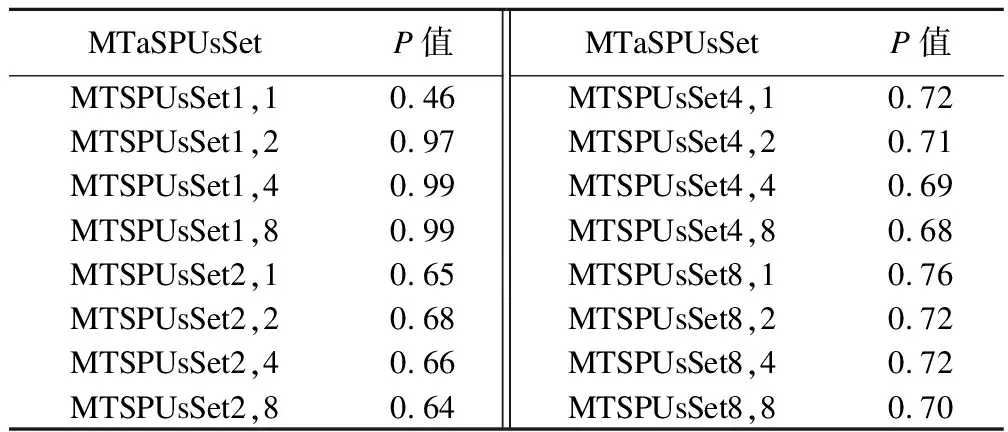
表1 MTaSPUsSet的模型实现结果
(4)在LCORL基因上绘制SNP图谱
plotG(someGs$LCORL[[1]],main=“LCORL(P-values)”,zlim=c(0,18))
结果如图1所示,呈现了基因LCORL上的SNP分别与身高、体重、BMI、腰围、臀围和腰臀围相关关系的P值,与身高相关的SNP更多。

图1 基因LCORL相关单核苷酸与六种性状的对数转换P值
2.MTaSPUsSetPath的模型实现
MTaSPUsPath的软件实现过程如下:
(1)生成待测SNP的相关矩阵(corSNP)和待测性状的相关矩阵(corPhe),代码为:
corPhe=estcov(Zs,Zs=True)
plink2-file hapmap3-extract SNP_ id-keep CEU_ hapmap-r2 inter-chr with-freqs-ld-window-r20-make-bed-out corPhe
(2)生成一个SNP信息矩阵和基因信息矩阵,代码为:
snp.info<-as.data.frame(snp.info)
gene.info<-as.data.frame(gene.info)
(3)下载aSPU安装包,执行MTaSPUsSetPath命令,代码为:
library(aSPU)
out<-MTaSPUsSetPath(Zs,corPhe=corPhe,corSNP=corSNP,n.perm=100,snp.info=snp.info,gene.info=gene.info)
out
即可得出不同SNP权重、不同基因权重和不同性状权重下该通路与多个性状间关联测试的P值,结果中MTaSPUsSetPath即为该测试在不同权重取值下均适用的P值。
SPU测试的应用
II-Youp Kwak等[12]将MTaSPUsSet测试应用于“人体特征基因调查联盟”(genetic investigation of ANthropometric traits,GIANT)的汇总统计数据,分别对男性和女性的身高、体重、BMI、腰围、臀围和腰臀围这六个人体测量学特征进行了基于基因水平的关联测试。通过MTaSPUsSet测试,共有2722976个SNPs被定位到17562个基因(每个基因加上2kb上游和2kb下游区域),共鉴定出137个对男性或女性人体测量特征具有全基因组意义的基因:男性为81个,女性为125个,两者共有为69个。而在相同的参考面板下采用MGAS方法,使用“kgg”软件仅能识别出19个显著基因。同时使用MTaSPUsSet和MGAS两种方法,MTaSPUsSet识别出27个对男性显著的基因和39个对女性显著的基因,而MGAS分别识别出7个和14个基因,结果显示基因RPGRIP1L和RPS10-NUDT3等仅能通过MTaSPUsSet检测得到,这表明MTaSPUsSet有更高的效能。另外,由于metaCCA需要所有单核苷酸多态性-性状对的样本量相同,而一些单核苷酸多态性的样本量在性状间从大约200到大约70000不等,因此metaCCA不适用于不同疾病之间含有重复研究对象的数据,如GIANT等[12]。目前还有其他进行全基因组关联性研究的相关应用的研究[8,14],结果表明相比于一些新的适应性测试,如KBAC(kernel-based adaptive clustering test),PWST(P-value weighted sum test)和aSSU(adaptive sum ofsquared score test),SPU测试具有更高的适应性和效能并在模拟实验中效果更好[8]。
SPU测试的优缺点
首先,与传统的GWAS相比,SPU测试在任何参考面板下性能都较优,其估计膨胀因子k接近于1[8]。其次,不同情况下可选择不同的SPU测试,由于SPU测试都是基于一般回归模型的得分向量,当同一SNP具有多个特征且这些特征的影响范围很小时,aSPU测试效能相比其他方法更强[8,15]。最后,MTaSPUsSet与传统多基因关联分析相比,在SNP和性状层面上都是自适应的,该方法考虑了SNP和性状之间可能存在的不同关联模式,例如关联强度和方向,从而在多数情况下保持高效能。另外,MTaSPUsSet可应用于混合类型的性状,也适用于从单个GWAS或多个GWAS的meta分析中获得的Z统计量或P值的汇总数据。同时,仿真和实际数据的数值研究表明,此法具有良好的应用前景[12]。此外,除本文介绍的几种典型SPU测试外还有许多适用于不同情况的自适应关联测试[11,16]。
然而,SPU测试是基于汇总数据统计分析得到,而没有进行生物验证,因此我们无法推测任何确定位点的因果影响[17]。可在此基础上进行转录组广泛关联研究,以推断基因表达状态并进一步探索候选基因与结果之间的因果关系[18-19]。
小 结
SPU测试是一种新的基于基因和路径的自适应关联测试,该测试可使用GWAS汇总统计数据,其I类错误率得到了很好控制,克服了目前已知方法不能应用于大规模数据以及弱相关或多重相关时不敏感等缺点[20-21]。该方法在基因组学、蛋白质组学等方面有较好的应用前景,为人类了解复杂性疾病的发病机制提供更多的线索,但其理论和方法仍需在应用中进一步完善。

