染色体核型分析联合CNV-seq在NT增厚胎儿产前诊断中的应用
胡艳平,袁静,李琴,周培,程龙凤
胎儿颈项透明层(nuchal translucency,NT)指胎 儿颈后部皮下组织内液体集聚的厚度;孕11~13+6周NT增厚是早期筛查胎儿染色体非整倍体的重要指标。传统染色体核型分析无法检出5~10 Mb以下的染色体微缺失/微重复,随着拷贝数变异测序(copy number variation sequencing,CNV-seq)在产前诊断中的应用,我国专家共识建议对有介入性产前诊断指征或需求的孕妇,在其充分知情的前提下,可将CNV-seq作为一线的产前诊断方法供其选择[1]。安徽医科大学第一附属医院(我院)产前诊断中心以NT≥2.5 mm为界值,采用染色体核型分析联合CNV-seq技术,探讨其在NT增厚胎儿中的应用。
1 对象与方法
1.1 对象及分组选择2020年1月—2021年10月就诊于我院孕11~13+6周NT≥2.5 mm的单胎孕妇189例。根据是否合并其他介入产前诊断指征(高龄、血清学异常、超声异常等)分为单纯NT增厚组(119例)和非单纯NT增厚组(70例)。根据NT厚度分为2.5 mm≤NT<3.5 mm组(123例)和NT≥3.5mm组(66例)。孕妇及家属充分知情选择并签署知情同意书。
1.2 实验室方法孕16~24周在超声引导下行羊膜腔穿刺术,抽取羊水21~23 mL,其中16~18 mL羊水进行G显带染色体核型分析,5 mL羊水用于CNV-seq检测。
1.2.1 染色体核型分析采用原位细胞培养法,常规进行细胞培养、收获、制片和G显带、全自动扫描仪扫描、拍照,按照人类细胞遗传学国际命名体制参照ISCN(2020)标准进行染色体核型分析。
1.2.2 CNV-seq方法患者羊水标本进行离心沉淀,取羊水中胎儿脱落细胞提取DNA,构建测序文库并进行质控,通过HiSeq 2000测序平台进行CNV-seq检测。将染色体全基因组测序检测到的基因组CNV,参照国际基因组CNV多态性数据库Decipher、UCSC Genome Browser、Online Mendelian Inheritance in Man(OMIM)、International Standards for Cytogenomic Arrays(ISCA)等以及相关文献评估CNV-seq结果是否有致病性,并附上相关文献。检测报告严格按照2019年中国低深度全基因组测序技术在产前诊断中的应用专家共识[1]进行CNV-seq分级,CNV-seq结果有5个等级:致病性、可能致病性、临床意义不明、可能良性及良性。
1.3 统计学方法应用SPSS 26.0统计学软件进行数据分析。定性资料用率表示,组间比较用χ2检验或Fisher确切概率法。P<0.05为差异有统计学意义。
2 结果
189例NT增厚的胎儿中染色体核型检出异常31例(检出率16.40%),包括非整倍体24例(12.70%)和结构异常7例(3.70%);CNV-seq检出致病性拷贝数变异(pathogenic copy number variations,pCNVs)38例(20.11%),其中非整倍体24例(12.70%),其他pCNVs 14例(7.41%),包括微缺失/微重复综合征10例(5.29%)和染色体片段缺失/重复4例(2.12%)。CNVseq在158例染色体核型正常者中额外检出pCNVs 9例(5.70%)。
2.1 染色体核型分析24例非整倍体中21-三体16例、18-三体5例、Turner综合征3例,分别占染色体核型异常的51.61%(16/31)、16.13%(5/31)和9.68%(3/31)。7例染色体结构异常中46,XX,t(11:15)(q14:q26)dup(11)(q14q25)、46,XY,der(8)、46,XX,?der(15),del(18)、46,XY,del(13)(q14,q34)、46,XY,dup(4)(q27q35)del(4)(p16)各1例,2例46,XX,inv(9)(p12q13)。染色体多态性8例。
2.2 CNV-seq结果分析CNV-seq检出非致病性变异者19例(10.05%),其中临床意义不明者13例(6.88%)、可能良性、良性者各3例。非单纯NT增厚组pCNVs、染色体非整倍体检出率均高于单纯NT增厚组,差异有统计学意义(P<0.05),2组其他pCNVs检出率差异无统计学意义(P>0.05)。NT≥3.5 mm组pCNVs、染色体非整倍体检出率均高于2.5 mm≤NT<3.5 mm组,差异有统计学意义(P<0.05),2组其他pCNVs检出率差异无统计学意义(P>0.05),见表1。14例其他pCNVs包括10例(5.29%)微缺失/微重复综合征,4例(2.12%)非综合征致病性片段缺失/重复,见表2。
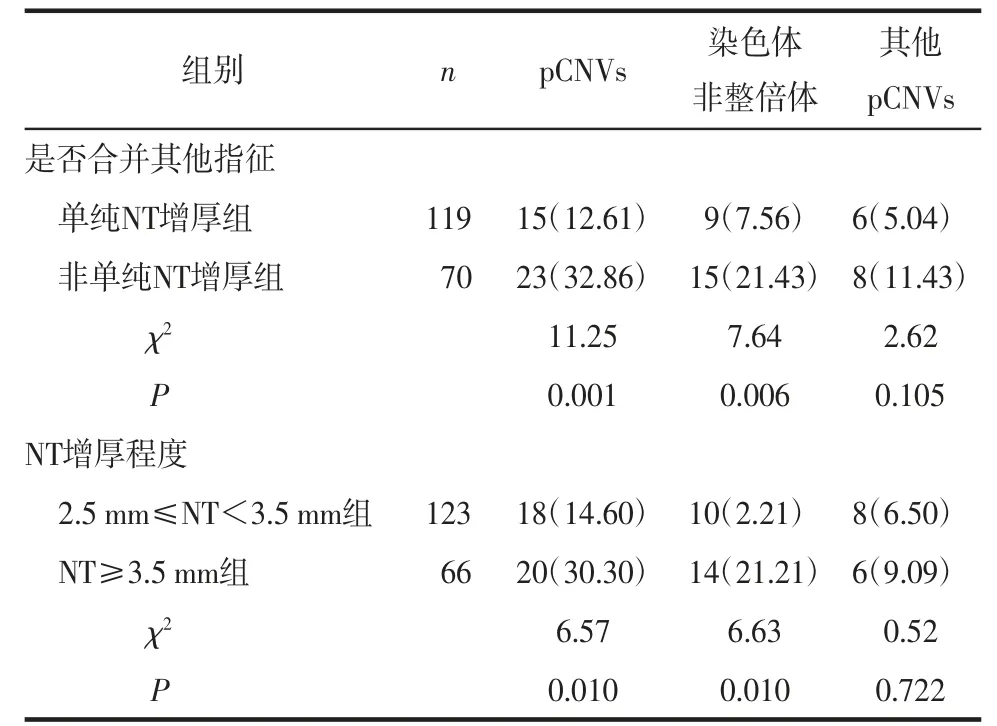
表1 不同NT增厚类型及程度分组pCNVs分析[例(%)]

表2 14例其他pCNVs及染色体核型分析
3 讨论
3.1 NT增厚核型分析研究表明,染色体异常发生率随NT厚度的增加而增加;当NT增厚合并其他超声异常时胎儿染色体异常的发生率明显增高[2]。NT增厚中染色体异常包括21、18和13三体综合征,以及Turner综合征、三倍体,微缺失/微重复综合征和某些单基因病(如Noonan综合征)[3]。在NT增厚的染色体异常胎儿中,50%为21三体综合征,25%为18或13三体综合征,10%为Turner综合征,5%为三倍体,10%为其他染色体异常[4]。有文献报道NT值位于2.4~3.4 mm、3.5~4.4 mm、4.5~5.4 mm、5.5~6.4 mm及≥6.5 mm时,胎儿染色体核型异常的发生率分别为10%、12%、33%、46%和58%[5]。研究表明NT≥2.5 mm染色体核型异常发生率为14.63%,其中染色体核型非整倍体发生率13.41%[6],本研究与文献报道相似。非单纯NT增厚组及NT≥3.5 mm组染色体非整倍体风险(约2 1%)分别明显高于单纯N T增厚组和2.5 m m≤N T<3.5 m m组。N T增厚合并其他指征或N T厚度≥3.5 m m时,对于焦虑的孕妇可选择绒毛穿刺术(孕1 1~1 3+6周),早期诊断可降低孕妇终止妊娠带来的伤害。由于绒毛穿刺对技术要求及风险高于羊膜腔穿刺术,本中心尚未开展绒毛穿刺术。本中心一般选择1 8周后进行羊膜腔穿刺术,但针对N T≥3.5 m m或N T增厚合并超声结构异常的孕妇知情选择可在孕1 6周后进行羊膜腔穿刺术。本研究染色体核型检出7例染色体结构异常的病例中3例染色体易位导致染色体重排时发生染色体片段缺失/重复,2例9号染色体臂间倒位,C N V-s e q可检出染色体缺失或重复,但无法检出染色体易位或倒位等。在我国欠发达地区的产前机构,针对N T增厚的胎儿染色体核型分析仍应作为产前诊断的首要检查方法。
3.2 致病性CNV分析随着产前诊断检测技术日益成熟,在NT增厚胎儿中pCNVs逐渐被发现。有研究发现NT≥3.0 mm胎儿pCNVs风险随NT厚度增加而增加[7],pCNVs检出率21.8%~25.9%,其中染色体非整倍体、其他pCNVs检出率分别为16.93%~22.0%和3.9~4.91%[7-8]。文献报道对于NT≥3.5 mm染色体核型正常的胎儿,pCNVs检出率为5%~6.8%[9-10]。本研究发现当NT≥2.5 mm时,在染色体核型正常胎儿中额外检出5.63%的pCNVs,与选择NT≥3.0 mm胎儿或NT≥3.5 mm的胎儿研究相比基本相似。目前对NT界值作为介入性产前诊断的标准不一致,部分机构以NT≥3.0 mm为界值[7-8],部分以NT≥2.5 mm为界值[2,4,6,10]。本研究9例染色体核型正常者中3例NT厚度在2.5~3.0 mm者检出pCNVs,且本研究发现其他pCNVs与NT增厚程度无明显相关性,这与其他研究一致[8,11],因此笔者推荐当NT≥2.5 mm时,排除胎儿染色体非整倍体外,也应关注是否具有微缺失/微重复。此外,本研究其他pCNVs与NT增厚合并其他指征亦无相关性,但非单纯NT增厚组检出率(11.43%)高于单纯NT增厚组(5.04%),故认为胎儿NT增厚合并其他指征者是CNV-seq检查的重点对象,以避免漏诊其他的pCNVs,但此结论仍需大样本进一步研究。
3.3 微缺失/微重复综合征在NT增厚中最常见的pCNVs微缺失/微重复为22q11.2缺失、22q11.2重复、10q26.12q26.3缺失和12q21q22缺失[9]。22q11.2微缺失/微重复综合征表型均存在外显不全且高度可变,且存在相同或相似的临床表型[12-13]。22q11.2微缺失综合征主要与先天性心脏病、腭部异常等有关[13],Tbox1转录因子(TBX1)基因缺失是心脏异常主要因素,本研究1例22q11.2微缺失综合征合并胎儿心脏异常的表型,因此,产前发现NT增厚合并心脏病的胎儿在排除非整倍体时,首先考虑是否合并22q11.2微缺失综合征。本研究发现3例合并1q21.1微缺失综合征,该综合征患者的表型存在外显不全且高度可变[14],常见表型为面部畸形、小头畸形和发育迟缓等。16p13.11微缺失/重复区域为神经认知障碍易感区,存在外显不全,部分携带者无明显临床症状[15-16]。综上,NT增厚中微缺失/微重复综合征表型多数为外显不全或高度可变,即使胎儿与父母携带相同的致病性微缺失/微重复,也无法通过父母的表型准确判断胎儿出生后是否发病[1]。因此,在NT增厚检出外显不全的微缺失/微重复综合征的胎儿,在超声结构正常且染色体核型正常的情况下,主要的难点是产前咨询对其表型的预判,需充分告知孕妇及家属胎儿出生后的可能表型,使其知情并选择是否继续妊娠。
综上,随着NT增厚或合并其他介入产前诊断指征,胎儿染色体异常风险增加,主要为染色体非整体异常风险增加,但与其他pCNVs无明显相关,由于本研究样本量及其他pCNVs检出数量少,结果可能存在偏倚,仍需大样本进一步研究。染色体核型分析联合CNV-seq可避免染色体结构异常及染色体微缺失/微重复的漏诊,建议临床采用并积累经验提高诊断准确率。

