基于通用引物的多重PCR对小圆胸小蠹的分子鉴定
黄卫东,梁建锋,彭 锋,桑 文,陈晓胜,王兴民*
(1.广东省生物农药创制与应用重点实验室,生物防治教育部工程技术研究中心,广州 510640;2.华南农业大学林学与风景园林学院,广州 510640)
小圆胸小蠹EuwallaceafornicatusEichhoff,1868隶属于鞘翅目Coleoptera象甲科Curculionidae小蠹亚科Scolytinae方胸小蠹属Euwallacea(Smithetal.,2019)。该虫在我国分布广泛,主要分布于福建、海南、广东、广西、台湾、四川、云南、贵州等地,国外则分布于东南亚、澳洲、美洲的23个国家和地区(Rabagliaetal.,2006;Hulcretal.,2007;何华和伍苏然,2016)。小圆胸小蠹是一种国际性的重大林木害虫,也是近年国内外爆发成灾的重大蛀干害虫之一,其寄主植物十分广泛,在全球范围内包括63科342种(Cooperbandetal.,2016;李巧等,2018)。小圆胸小蠹主要通过虫、菌结合来危害寄主植物,成虫在寄主植物体内羽化后,即开始钻蛀到新寄主植物枝干内进行产卵。在成虫钻蛀的过程中,其体表携带的真菌孢子散落在蛀道内,并开始生长。菌丝的过度生长会阻塞寄主植物的维管束,影响树体水分传输,与该虫共同危害树木(Walgama,2012;何华和伍苏然,2016;李巧等,2018)。
对害虫进行快速、准确地鉴定是实施害虫防治的基础和关键。目前,对于农业害虫的鉴定大都采用传统的形态学方法,但昆虫形态学鉴定主要是以成虫为研究对象,而昆虫的卵、幼虫和蛹的形态特征很难辨认。因此,利用以分子生物学手段为基础的DNA条形码技术对害虫进行分子鉴定,能够在很大程度上弥补形态学鉴定中的不足。Hebert等(2003)首次提出DNA条形码的概念,即利用线粒体细胞色素氧化酶亚基I(mitochondrial cytochrome c oxidase subunit I,COI)约658 bp的分子片段对物种进行快速鉴定。目前,该技术已广泛应用于象甲科昆虫的新种描述和物种鉴定等领域,并展现出了极大的应用潜力(Skuhrovecetal.,2018;Cognatoetal.,2020)。除序列COI外,在鞘翅目昆虫DNA条形码研究中涉及的分子片段还包括核基因的28S rDNA(28S)、18S rDNA(18S)、延长因子EF-1α等,线粒体基因的16S rDNA(16S)、12S rDNA(12S)、COII等(张媛等,2011)。
多重PCR(multiplex PCR)是指在一次PCR反应体系中同时加入多对引物,从而产生多条目的片段的技术,最早由Chamberlain等(1988)提出。目前,多重PCR技术主要应用于病原微生物的快速检测:如Potrykus等(2014)利用多重PCR成功检测了欧洲马铃薯SolanumtuberosumL.上侵染的Pectobacterium病原菌;Kim等(2015)则利用多重PCR成功检测了感染人类的5种Vibrio病原菌。该技术目前也被应用于节肢动物物种的鉴定中,如王彦坤等(2020)利用COI和16S的通用引物结合双重PCR技术对3纲8目14科的14种节肢动物物种进行了鉴定,结果显示双重PCR技术不仅可以保证物种鉴定的高准确率,还可以明显减少时间与DNA样本量的消耗。然而,关于使用多重PCR技术(即在一个反应体系内同时扩增3个或3个以上的分子标记)对昆虫进行快速鉴定仍未见报道,其可行性需要进一步探究。
本研究选择在鞘翅目分子鉴定中常用的COI、16S和28S三个分子片段,利用多重PCR的方法开展小圆胸小蠹的分子鉴定。目的在于:一方面探究多重PCR在昆虫分子鉴定中的可行性,另一方面建立小圆胸小蠹的分子鉴定方法,以期为开展小圆胸小蠹的有效、准确鉴定及综合防治等提供重要依据。
1 材料与方法
1.1 标本采集
小圆胸小蠹成虫样本采自于广东省潮州市潮安区凤凰镇(纬度:23.851108;经度:116.632647;海拔:539.32 m)。田间采集后,至于无水乙醇中保存,带回实验室至于-20℃低温贮存备用。成虫的形态鉴定主要依据Gomez等(2018)和Smith等(2019)的文献描述。
1.2 基因组DNA的提取
血液/组织/细胞基因组织提取试剂盒(TIANamp Genomic DNA Kit)用于目标昆虫DNA的提取。选择小圆胸小蠹后足组织用于DNA的提取,详细的提取步骤严格按照试剂盒的说明书进行。提取后的DNA至于-20℃保存备用。
1.3 PCR扩增及测序
试验设计了4组多重PCR体系,(1)COI+28S+16S;(2)COI+16S;(3)COI+28S;(4)16S+28S;并且分别扩增了28S、16S和COI的单个片段。引物序列信息和参考文献见表1。

表1 本研究使用的引物Table 1 Primers used in this study
PCR扩增体系为50 μL:PCR Mix 24 μL(TransGen Biotech,Beijing),上下游引物各1 μL,DNA模板1 μL,然后使用无菌水补充至50 μL。反应程序为:94℃预变性3 min;94℃变性30 s,50℃退火30 s,72℃延伸1 min,共35个循环;72℃最终延伸5 min。最后将PCR产物在1.5%琼脂糖凝胶中电泳,经EB染色后,在紫外灯下照射,对符合目的片段长度的产物进行回收,并送至上海生工生物工程有限公司进行双向测序。
1.4 序列分析
在软件Geneious 7.1.4(Kearseetal.,2012)中将测序获得的基因序列进行拼接和人工校准,去除两端引物。然后将拼接完成的序列输出文件格式为FASTA,在NCBI(https://www.ncbi.nlm.nih.gov/)网站上进行BLAST序列同源性分析,以确认目的片段被扩增。使用软件MEGA 7(Kumaretal.,2016)分别统计了小圆胸小蠹COI、16S和28S的碱基组成。为了计算种间的遗传距离,本研究从数据库中下载了部分小圆胸小蠹和其它方胸小蠹属的分子序列,并利用MUSLE(Edgar,2004)对序列进行比对,然后使用MEGA 7分析各种间的遗传距离(表2)。由于本研究扩增的小圆胸小蠹的28S片段与数据库中小圆胸小蠹的28S片段不属于同一区域,因此本研究中28S的扩增仅用于探究多重PCR的可行性而不用于后续的遗传距离计算和系统发育树的构建。

表2 本研究所用到的序列信息Table 2 Information of sequences used in this study
1.5 系统发育树构建
本研究分别基于单个分子片段使用邻接法(Neighbor-Joining,NJ)和最大似然法(Maximum likelihood,ML)用于系统发育树的构建。NJ树的构建实施于MEGA 7中,基于K2P(Kimura-2-parameter)双参数模型。ML树的构建实施于RAxML 8.2.8(Stamatakis,2014)中,同时使用自举值重复1 000次以检验各节点的可信度。在ML树构建之前,使用Modeltest(Posada and Crandall,1998)分别推算COI和16S数据集的最佳替换模型。ML分析在CIPRES Science Gateway上运行(Milleretal.,2010)。
2 结果与分析
2.1 通用引物的多重PCR检测
基于通用引物的小圆胸小蠹的多重PCR检测结果显示:在包含COI、16S和28S的反应体系1中产生3条目的条带,但COI条带较弱。在包含COI和16S的体系2中,COI条带也较弱,16S则产生明亮条带。对比包含COI和28S的体系3中,COI和28S均产生明亮条带。在包含16S和28S的反应体系4中,也产生了2条明亮条带。此外,基于单个PCR反应的COI、16S和28S均产生了符合目的片段大小的明亮条带(图1)。

图1 小圆胸小蠹成虫生态照Fig.1 Ecological photograph of the adults of Euwallacea fornicatus
2.2 基因序列分析
将序列拼接完成、去除两端引物后,扩增得到小圆胸小蠹的COI序列长度为658 bp,16S序列长度为302 bp,28S序列长度为908 bp。在GenBank中进行BLAST检索显示所获得序列为目的序列。在COI的所有位点中,碱基A、T、G和C的含量分别为33.0%、33.1%、14.9%和19.0%,且A+T含量高于G+C含量。在16S的所有位点中,碱基A、T、G和C的含量分别为37.4%、39.0%、15.1%和8.5%,且A+T含量高于G+C含量。在28S的所有位点中,碱基A、T、G和C的含量分别为24.3%、21.8%、29.9%和24.0%,而A+T含量稍低于G+C含量。本研究新测序的分子序列均已上传至GenBank中;COI登录号为MT946291,16S登录号为MT946906,28S登录号为MT940909。
2.3 遗传距离分析
基于COI的遗传距离分析显示本研究扩增获取的序列与数据库中小圆胸小蠹的COI序列的遗传距离为0.009~0.014;而与其它方胸小蠹属的遗传距离为0.104~0.162(表3)。基于16S的遗传距离分析显示本研究扩增获取的序列与数据库中小圆胸小蠹的16S序列的遗传距离为0.000~0.007;而与其它方胸小蠹属的遗传距离为0.081~0.114(表4)。

表3 基于COI基因的小圆胸小蠹与其它小蠹的遗传距离分析Table 3 Genetic distance between Euwallacea fornicatus and other species of Euwallacea based on COI gene

表4 基于16S基因的小圆胸小蠹与其它小蠹的遗传距离分析Table 4 Genetic distance between Euwallacea fornicatus and other species of Euwallacea based on 16S gene
2.4 系统发育树构建
系统发育分析结果表明,无论是基于何种建树方法还是不同的分子片段,本研究扩增的小圆胸小蠹的分子序列与数据库中小圆胸小蠹的分子序列均聚为一支,且得到较高的支持率(图2,图3)。此外,小圆胸小蠹与其它方胸小蠹属的物种区分明显,表明COI和16S分子序列可作为小圆胸小蠹分子鉴定的依据。

图2 小圆胸小蠹的多重PCR扩增结果Fig.2 Amplification result of Euwallacea fornicatus with multiplex PCR注:1,Marker 2000;2,COI+16S+28S;3,COI+16S;4,COI+28S;5,16S+28S;6,COI;7,16S;8,28S。
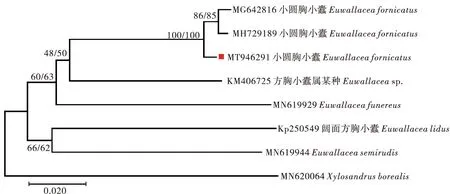
图3 基于COI基因的小圆胸小蠹与其它小蠹构建的系统发育树Fig.3 Phylogenetic tree of Euwallacea fornicatus and other species of Euwallacea based on COI gene注:图中节点旁边为自举值(左:NJ;右:ML),红色方形表示本研究中生成的序列。Note:Numbers at nodes represented bootstrap value for NJ (left) and ML (right).Red rectangle represented this sequence was new generated in this study.
3 结论与讨论
DNA条形码鉴定的前提是DNA条形码间隙(barcode gap)的存在,即种间遗传距离大于种内遗传距离(Hebertetal.,2003;2004;Puillandreetal.,2012;Taylor and Harris,2012)。Hebert等(2004)指出种间遗传距离分化至少应大于种内遗传分化的10倍;然而,后续的研究表明遗传距离阈值对于不同类群来说是不一致的(Havermansetal.,2011;Songetal.,2018)。例如,2%~3%被表明适用于膜翅目、蜉蝣目、襀翅目和毛翅目部分类群的种类鉴定(Monaghanetal.,2005;Zhouetal.,2010;Webbetal.,2012;Schmidtetal.,2015);3%~5%适用于一些双翅目和鞘翅目物种的种类鉴定(Linetal.,2015;Nzeluetal.,2015;Huangetal.,2020);而6%~8%则适用于毛翅目纹石蛾科Hydropsychidae的物种鉴定(Paulsetal.,2010)。在本研究中,基于COI基因的遗传距离分析表明本研究扩增的小圆胸小蠹的DNA条形码序列与数据库中的序列差异小于2%,而和其它方胸小蠹属的序列差异则为10.4%~16.2%。同样地,在基于16S基因的遗传距离分析中也揭示了本研究扩增的小圆胸小蠹的16S序列与数据库中小圆胸小蠹的16S序列具有极高的相似性。目前,一些研究表明基于COI单个分子的物种鉴定结果经常被一些偏差所影响,包括假基因的存在、不完全谱系选择和基因渗透(Funk and Omland,2003;Dupuisetal.,2012;Talaveraetal.,2013;Huangetal.,2020)。因此,为了增加分子鉴定的可信度,其它的基因片段,如12S、16S、18S和28S被提出作为互补的分子片段用于物种鉴定(Lerayetal.,2013;Elbrechtetal.,2016)。本研究中,利用多个分子片段同时用于小圆胸小蠹的分子鉴定,遗传距离和系统发育分析的结果表明COI和16S分子片段可用于小圆胸小蠹的快速、准确鉴定,同时也强调了使用多个分子片段在准确鉴定物种方面的重要性。

图4 基于16S基因的小圆胸小蠹与其它小蠹构建的系统发育树Fig.4 Phylogenetic tree of Euwallacea fornicatus and other species of Euwallacea based on 16S gene注:图中节点旁边为自举值(左:NJ;右:ML),红色方形表示本研究中生成的序列。Note:Numbers at nodes represented bootstrap value for NJ (left) and ML (right).Red rectangle represented this sequence was new generated in this study.
在本研究中,首次使用3对通用引物来开展小圆胸小蠹的分子鉴定,并探究多重PCR在昆虫中应用的可行性。结果表明,在设置的4个多重PCR反应体系中均可以产生符合目的片段大小的单一条带。但在体系1和体系2中,COI产生的条带则较暗,通过比较体系3和体系4中的引物配置,推测由于体系1和体系2中包含了16S的扩增引物,从而影响了COI的扩增效果。引物的选择与优化被认为是影响多重PCR效果的最主要因素(Elnifroetal.,2000)。由于COI和16S同属于线粒体基因,且大多数昆虫线粒体基因组排列与昆虫线粒体基因组原始排列顺序一致(Cameron,2014),因此这两个片段的引物可能会发生相互干扰。由于本研究中的多重PCR反应体系为50 μL,使得仍可以通过胶回收获取足量的COI扩增产物以满足测序的要求。为解决在多重PCR反应中部分条带较弱的问题,一些研究提出在反应体系中增加对应引物的量,可以有效的增加引物与模板结合的概率(Elnifroetal.,2000;郝少东等,2015)。
本研究证实了多重PCR应用于昆虫分子鉴定的可行性。在一个PCR反应体系中产生的多条分子序列,不仅提高了物种鉴定的准确率,而且节省了PCR反应所需时间和模板的消耗;在样品材料十分珍贵的情况下,这两点显得尤为重要。此外,通过COI、16S和28S提供的序列信息和多重PCR方法,建立了小圆胸小蠹的分子鉴定方法,为开展小圆胸小蠹的准确鉴定及综合防治等奠定了基础。考虑到当前数据库丰富的分子序列和引物设计的便捷,建议在研究不同类群时挑选合适的基因和不同的引物组合,采用多重PCR开展物种的鉴定工作,以增加物种鉴定的准确性。

