基于生物信息学分析探讨类风湿关节炎与骨关节炎之间的关系①
章晓云 李华南 陈 锋 柴 源 甘 斌 陈丁鹏 (江西中医药大学,南昌 330004)
类风湿关节炎(rheumatoid arthritis,RA)是一种自身免疫性疾病,可引起慢性炎症表现、肥胖、心血管疾病等,影响全球0.3%~1.0%的人口,在中国每年至少500 万人被诊断出RA[1-2]。骨关节炎(osteoarthritis,OA)是一系列能引起软骨损伤和变性的退行性关节炎的总称,影响了全球约2.4亿人口,无论是从生活质量还是疾病负担方面,都已成为全球范围内严重的公共卫生问题[3-5]。两者是目前最常见的关节炎类型,病因均未明确,暂无有效的根治方法,给个人和社会带来了巨大的经济压力。
尽管两种疾病的病因学不同,但均以滑膜炎症反应、关节软骨破坏、骨质流失等为特征,最终导致关节进行性退变,临床上许多患者同时患有这两种疾病。研究表明RA 是OA 发病的重要危险因素之一,RA 患者罹患OA 的风险比正常人高2.75 倍[4]。随着基础研究的不断深入,已经发现两种疾病之间存在诸多共同发病机制和受调控因子,在蛋白质水平和组织炎症评分水平上具有很强的连续性[6-8]。因此,本研究通过GEO数据库查找RA和OA血清基因芯片表达谱进行生物信息学分析,筛选出差异miRNA 并进行靶基因预测,最后交集筛选出核心mRNA,为确定RA 和OA 之间的共同发病机理创造条件,并为RA 和OA 治疗药物的研发提供有效的作用靶点。
1 资料与方法
1.1 数据收集 以“Rheumatoid arthritis”“Osteoar⁃thritis”“miRNA”为关键词,在GEO 公共数据库(https://www.ncbi.nlm.nih.gov/geo/)[9]检索与RA 及OA 相关的芯片,获得编号为GSE115885 的RA 血清基因表达芯片,共包含15 例RA 患者和5 例健康对照,其所处平台为GPL25134;获得编号为GSE105027 的OA 血清基因表达芯片,共含12 例OA患者和12例健康对照,其所处平台为GPL21575。
1.2 差异miRNA 的数据分析 利用Perl(V5.30.0.1)对数据进行重注释,并利用R 语言校正、分类,limma 包对miRNA 进行差异分析,以P<0.05 和|logFC|≥0.5 作为过滤条件,筛选差异miRNA(differentially expressed miRNAs,DEmiRNAs)。最后运用Venny 平台(http://bioinfogp. cnb. csic. es/tools/venny/)将所得RA 和OA 的DEmiRNAs 进行映射取交集,得到交集DEmiRNAs。
1.3 miRNA 转录因子预测及功能富集分析 Fun⁃Rich(V3.1.3)[10]是一个用于基因和蛋白质功能富集和相互作用网络分析的软件。利用FunRich 软件将筛选出的交集miRNA 进行转录因子预测及功能富集分析,并根据P值显示前10位进行可视化。
1.4 靶基因预测 通过miRTarBase 数据库(http://mirtarbase. mbc. nctu. edu. tw/php/index. php)、Target Scan 数据库(http://www. targetscan. org/vert_72/)和miRDB数据库(http://mirdb.org/)分别预测DEmiRNA的靶基因,使用Venny 平台取3 个数据库均能预测到的靶基因,生成交集基因,并与步骤1.2所得的交集DEmiRNAs 构成关系网络。整合得到miRNAmRNA 调控网络,并导入Cytoscape(V3.7.2)绘制网络图。
1.5 蛋白互作网络构建 为进一步探究miRNA 所调控靶基因的作用机制,将靶基因导入String 数据库(http://string-db. org/cgi/input. pl)[11],限定研究物种为“Homo Sapiens”获得蛋白互作关系,设置连接评分(Combined score)>0.95,再导出蛋白互作关系的数据文件,选取文件中的node1、node2 和Com⁃bined score 三列导入Cytoscape 中,最后利用Cyto⁃scape 软件的“NetworkAnalyzer”工具进行可视化处理,构建蛋白互作(PPI)网络图。
1.6 基因富集分析 利用DAVID 数据库(https://david. ncifcrf. gov/)[12]对mRNA 进行GO 功能富集分析,研究靶基因的主要生物功能;对潜在靶点进行KEGG 信号通路富集分析,研究靶基因的主要信号通路,P<0.05 代表富集结果显著。最后利用R 语言绘制GO富集分析图及KEGG富集分析气泡图。
2 结果
2.1 DEmiRNA 的鉴定 利用R语言对芯片进行重注释和数据校正后,再对其进行差异分析。结果显示,与健康对照相比,RA 患者的血清中共存在41个明显改变的miRNAs,其中上调31个,下调10个;OA患者的血清中共存在265个明显改变的miRNAs,其中上调124 个,下调141 个;对二者取交集后共得到4个交集DEmiRNAs,详见表1。
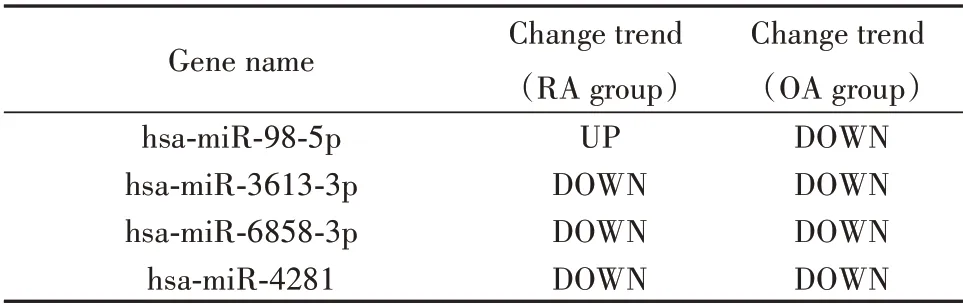
表1 交集DEmiRNAsTab.1 Intersection DEmiRNAs
2.2 miRNA 调控的转录因子及富集分析结果 通过FunRich 软件对4 个交集DEmiRNA 进行转录因子预测,共得到197个转录因子,根据其所调控的基因数与P值,选取富集程度显著的10 个转录因子进行展示,见图1。差异最显著的10 个转录因子分别为LHX3、SP4、NFIC、VSX2、HOXA7、TCF3、MYC、HOXB4、ETS1、SP1,其基本信息见表2;对4 个交集DEmiRNAs 进行功能富集分析,其生物过程主要涉及肽代谢、转录、翻译等;细胞成分主要涉及受体复合体、Ⅰ型胶原、Ⅸ型胶原等;分子功能主要涉及细胞外基质结构成分、蛋白质酪氨酸/丝氨酸/苏氨酸磷酸酶活性、辅助转运蛋白活性等,见图2。
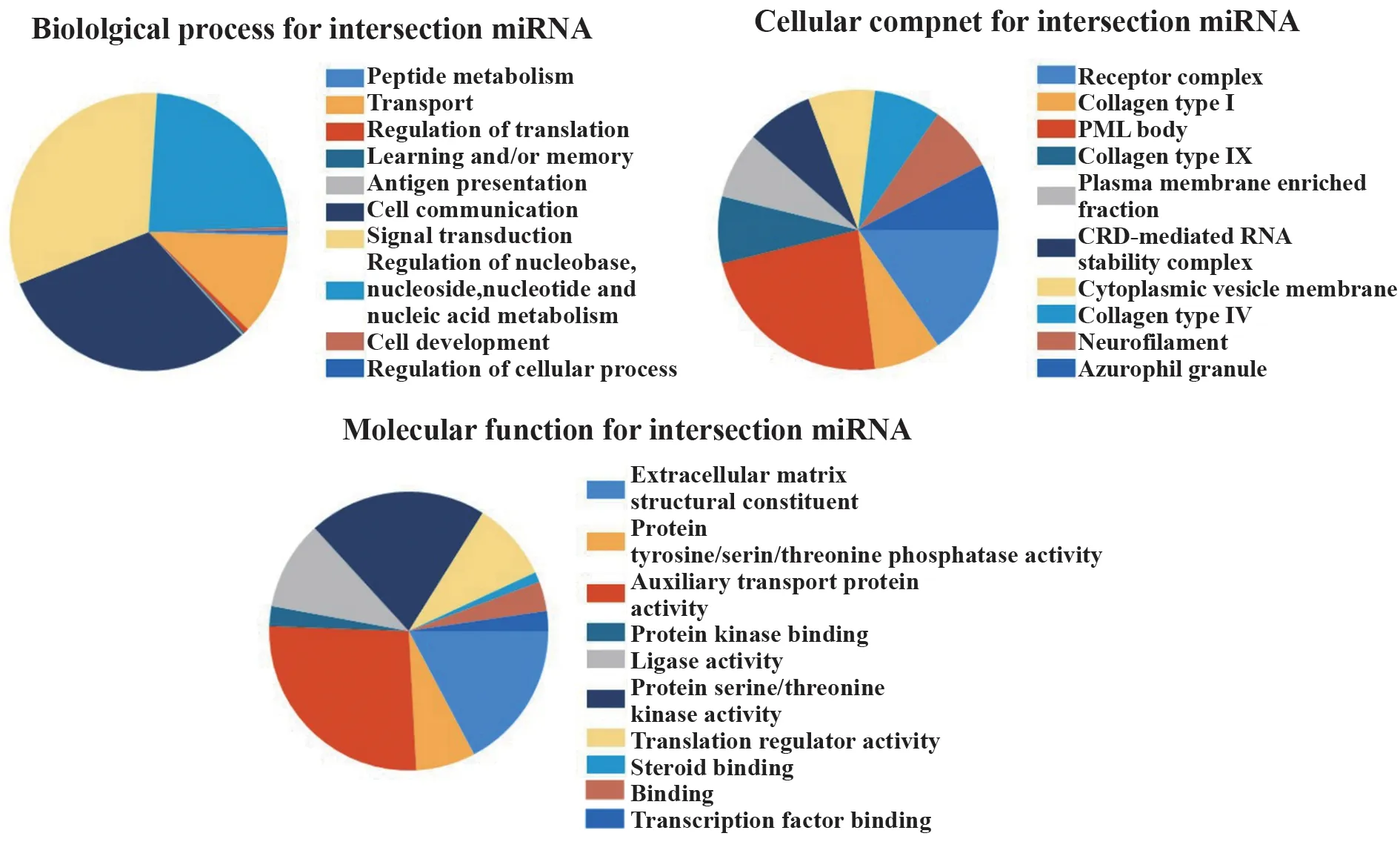
图2 交集DEmiRNAs的功能富集分析Fig.2 Functional enrichment analysis of intersection DEmiRNAs
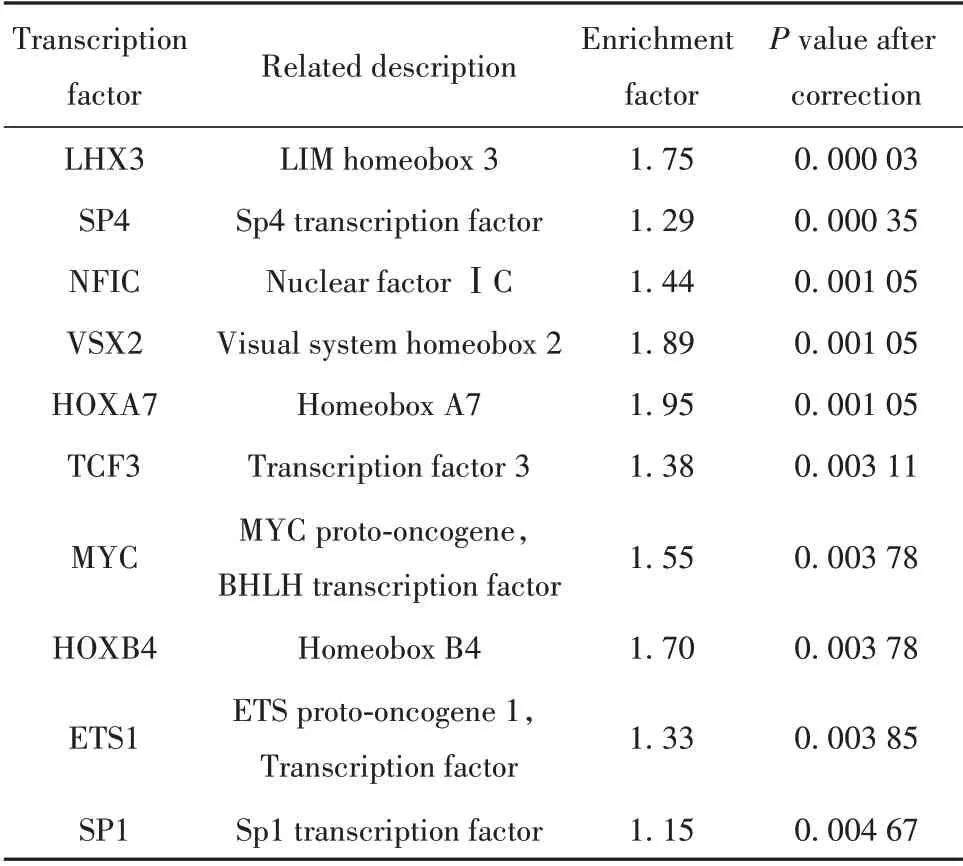
表2 关键转录因子的基本信息Tab.2 Basic information on key transcription factors
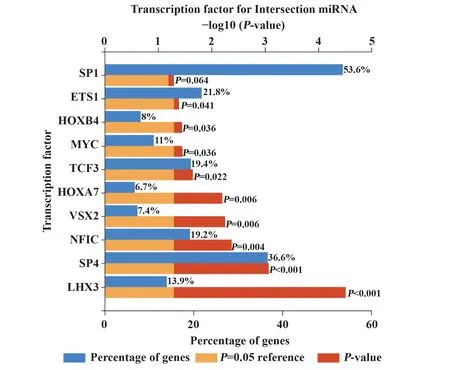
图1 交集DEmiRNAs的转录因子预测Fig.1 Transcription factor prediction of intersection DEmiRNAs
2.3 “miRNA-基因”调控网络预测 本研究运用miRTarBase、TargetScan、miRDB数据库预测上述4个DEmiRNAs 的靶基因,通过Venny 平台筛选靶基因在3 个数据库的交集,共得到433 个靶基因,见图3。最后将得到的靶基因与交集DEmiRNAs构成关系网络导入Cytoscape 软件构建miRNA-mRNA 调控网络图,见图4。
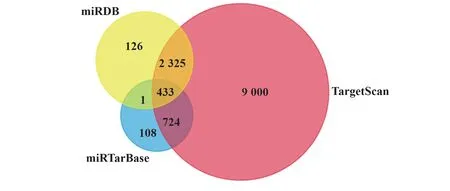
图3 交集DEmiRNAs的靶基因韦恩图Fig.3 Target gene Venn diagram of intersection DEmiRNAs

图4 交集miRNA-mRNA调控网络Fig.4 Intersection miRNA-mRNA regulatory network
2.4 PPI网络 借助String数据库和Cytoscape软件构建PPI网络,见图5。图中共涉及40 个节点、43 条边,其中节点代表蛋白,边则代表蛋白间的互作关系,节点越大、颜色越深则度值越大;边越粗,则蛋白间的关系越紧密。度值排名前5 的蛋白质为CDC5L、HNRNPU、GATAD2A、CHD4、ACTB。这些度值较大的蛋白质在整个网络中发挥关键作用,可能是RA 跟OA 发生发展的关键基因,其基本信息见表3。

表3 关键靶点的基本信息Tab.3 Basic information on key targets
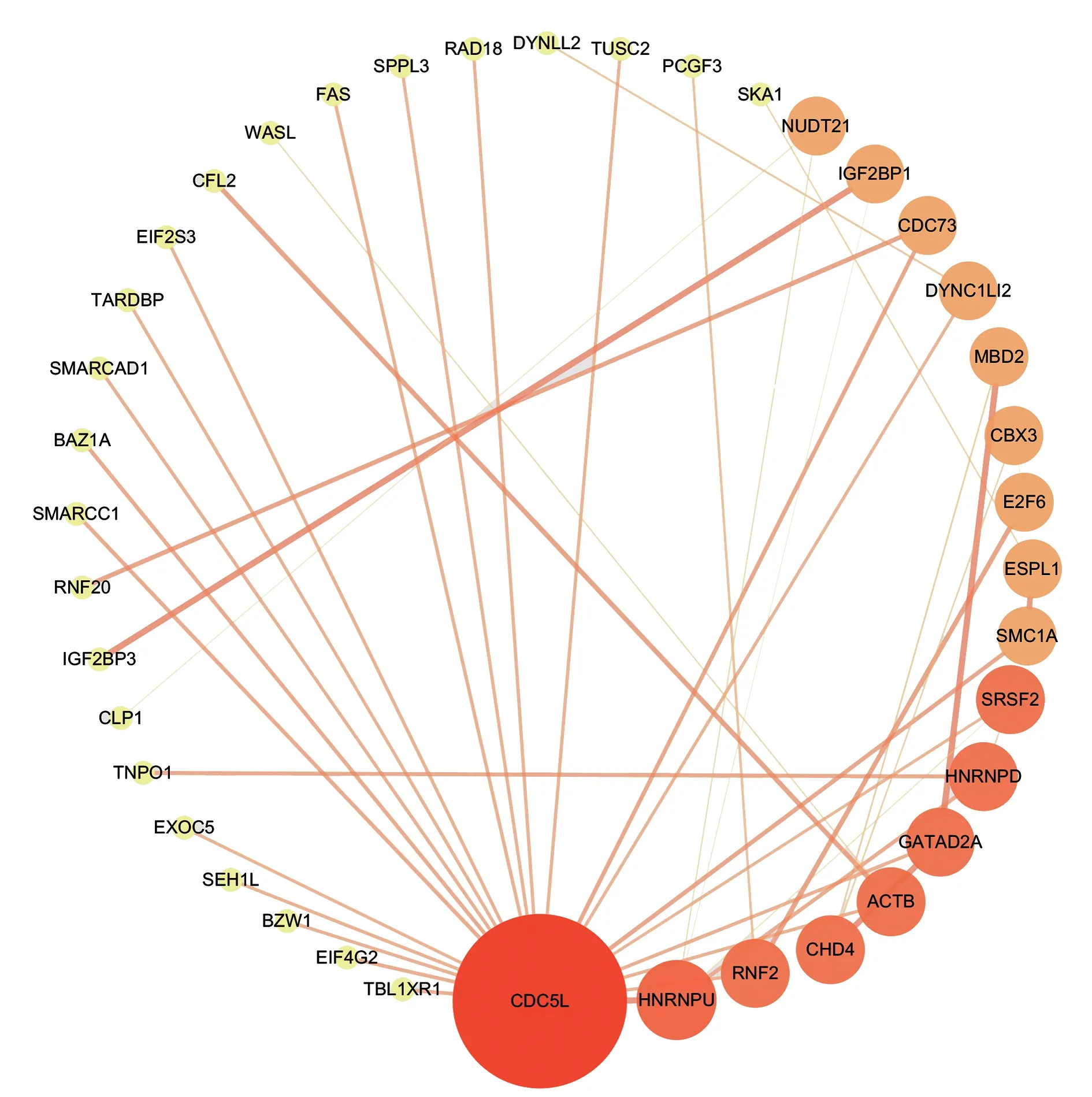
图5 蛋白互作网络Fig.5 Protein interaction network
2.5 GO 功能与KEGG 信号通路富集分析 将靶基因导入DAVID 数据库,分析获得疾病基因和蛋白信息所富集的通路,并将基因信息进行可视化处理,见图6。结果显示,靶基因的生物过程主要涉及mRNA代谢过程的调控、细胞周期过程的负调控、有丝分裂细胞周期等;细胞成分主要涉及转录因子复合物、转移酶复合物、蛋白激酶复合物等;分子功能主要涉及泛素蛋白转移酶活性、蛋白激酶调节剂活性、DNA 解旋酶活性等;信号通路主要涉及调控干细胞多能性的信号通路,Hippo信号通路、FoxO 信号通路、Apelin信号通路、p53信号通路等。
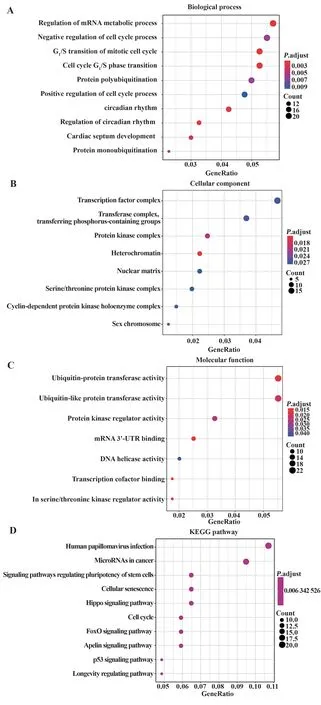
图6 GO和KEGG信号通路可视化结果Fig.6 Visualization results of GO and KEGG signaling pathway
3 讨论
近年来,miRNA 的研究引起了人们的极大兴趣,已经发现了2 000 多种miRNAs,它们在真核基因表达调控中具有广泛作用,参与细胞分化,增殖及凋亡等多种生物过程,人类基因组中约有1/3 的基因处于miRNA的控制之下[13-14]。研究表明miRNA通过调控自身抗体与炎症细胞因子释放、软骨细胞自噬凋亡、调节免疫等机制在RA 与OA 疾病的发病过程中发挥至关重要的作用[13-15]。随着以基因芯片为代表的高通量测序技术的发展,目前可在短时间获取疾病的相关信息,进而从基因层面进行更全面的生物标志物筛选。本研究通过生物信息学分析了解RA 与OA 之间的相关性,了解两病的相关发病机理,为药物研发提供一定的参考资料。
本研究通过GEO 数据库查找获取RA 血清基因表达芯片GSE115885 和OA 血清基因表达芯片GSE105027,对两种芯片进行生物学信息分析共获得4 个DEmiRNAs,分别为hsa-miR-98-5p、hsa-miR-3613-3p、hsa-miR-6858-3p、hsa-miR-4281。研究结果显示除hsa-miR-98-5p 对两种疾病的作用变化趋势相反,其他三种miRNA 的作用趋势一致。相关研究表明RA 与OA 疾病发生和发展过程中涉及多种转录因子,主要涉及滑膜炎症反应,软骨与骨组织的破坏[16]。本研究对4 个交集DEmiRNA 转录因子预测结果显示与其相关的转录因子共有197 个,差异最显著的为LHX3、SP4、NFIC、VSX2、HOXA7、TCF3、MYC、HOXB4、ETS1、SP1。研究表明miRNAs参与了几乎所有已知细胞过程的转录后调控机制,包括发育、分化、凋亡以及对病原体感染的先天性和适应性免疫反应,而转录因子与RNA 聚合酶Ⅱ形成的转录起始复合体在转录起始过程中具有重要的作用[17-18]。本研究结果对交集DEmiRNAs 进行功能富集分析显示其生物过程主要涉及肽代谢、转录、翻译等,与既往研究完全符合[17-18]。因此,笔者认为这4种交集DEmiRNAs与RA 和OA 的发病过程中有着紧密的关系,可以为阐明RA 与OA 之间的关系以及后续研究提供依据。
对自身靶基因的调控是miRNAs 在疾病发病过程中发挥作用的研究重点。本研究结果显示4 个DEmiRNAs 共调控433个靶基因,其中有359个靶基因为共同调控,其中CDC5L、HNRNPU、GATAD2A、CHD4、ACTB 排名较靠前,在整个网络中起着关键的作用。CDC5L 参与细胞周期调控,影响炎症及细胞凋亡过程,因此其可通过调节炎症细胞及软骨细胞的凋亡从而影响RA 与OA 疾病的发生与发展[19-20]。研究表明HNRNPU 参与免疫基因转录调节过程,对RA 发病具有关键作用[21-22]。HNRNPU 可与功能性基因间重复RNA 元件相互作用,增强mRNA的稳定性,可促进炎症基因的表达水平,从而可减轻RA 与OA 患者的炎症反应[23]。CHD4是低氧诱导因子(hypoxia inducible factor,HIF)的共激活因子,CHD4 可增强HIF1α 的募集,从而促进HIF 目标基因转录[24]。研究表明HIF1α 介导了免疫炎症细胞的激活,在RA 患者滑膜中表现出缺氧性并呈现HIF1α 上调[25],HIF1α 也被认为是OA 的关键调节因子,因此笔者认为CHD4 可通过与HIF1 相互作用从而影响两种疾病的发生。ACTB 参与软骨细胞自噬与凋亡过程,从而影响RA 与OA 患者软骨退变情况[26]。目前对于GATAD2A 的研究主要集中在癌症疾病与精神分裂疾病,其可影响患者精神状态[27],尚未有关于其在RA 与OA 疾病中的研究,但RA 与OA 患者因为疾病的难治性可影响患者的心理及精神情绪,因此未来对于GATAD2A 与RA 与OA 疾病之间的关系有待进一步研究。
为充分了解受DEmiRNAs 调控的mRNA 在RA与OA 中涉及的共同通路及功能,本研究进行了GO及KEGG 分析。GO 结果显示靶基因主要涉及mRNA代谢过程的调控、细胞周期过程的负调控、有丝分裂细胞周期等生物过程,主要涉及泛素蛋白转移酶活性、蛋白激酶调节剂活性、DNA 解旋酶活性等分子功能,这些生物过程及分子功能均与炎症细胞与软骨细胞的增殖与分化,自噬与凋亡有关,从而影响RA 与OA 的炎症变化与关节退变程度。KEGG 分析结果显示信号通路主要涉及调控干细胞多能性的信号通路,Hippo 信号通路、FoxO 信号通路、Apelin 信号通路、p53 信号通路等。研究发现通过对诱导性多能干细胞进行编程,可使其具有对炎症刺激作出反应的能力,并产生强大的自主调节的抗炎细胞因子,因此调控干细胞多能性的信号通路是未来研究RA 与OA 发病机制的主要途径[28]。研究表明Hippo 信号通路及p53 信号通路均可影响细胞增殖和凋亡,抑制其表达可减少软骨细胞凋亡以维持软骨形态,FoxO 信号通路可调节软骨细胞自噬和防御氧化应激,从而有效改善RA 与OA 患者的病情,减缓关节退变[29-31]。Apelin 可通过自分泌/旁分泌和内分泌途径有效地调节炎症,调节软骨、滑膜、骨骼等组织和各种免疫细胞的变化,对于RA 和OA的发病机制至关重要[32]。
总而言之,RA 和OA 均存在庞大的疾病基因网络,但这些网络并非各自独立,而是存在密不可分的联系。本研究获得RA 和OA 各自疾病基因网络的交叉部分正是二者相关联的部分,也是研究RA和OA 相互关系的突破点,同时也可为药物同时干预两种相互关联的疾病提供较为可靠的路径和作用靶点。且从侧面反映出不同疾病可通过应用同一药物治疗的特色,与中医辨证论治中的“异病同治”思想不谋而合,以此促进中药现代化发展。此外本研究尚存在一些缺点:首先,由于筛选条件的限制,只能对主要转录因子、作用靶点、信号通路进行分析,在一定程度上使得研究结果具有局限性;其次,对差异基因信息进行整合处理依赖于生物信息学技术的发展,疾病数据库的完整性、准确性直接决定着整合后信息的可靠性。故本文虽然能够筛选出大量靶点和通路,但仍需后续结合体内外实验进一步验证与支持,使理论更加可靠。

