基于分子动力学模拟的TRPM8通道门控特性分析
高志伟, 李军委, 史赛, 付强, 贾钧儒, 安海龙
基于分子动力学模拟的TRPM8通道门控特性分析
高志伟1, 李军委2, 史赛1, 付强1, 贾钧儒1, 安海龙2
(1. 河北工业大学理学院, 2. 生命科学与健康工程学院, 生物物理研究所, 河北省分子生物物理重点实验室, 天津 300401)
TRPM8通道的温度感知等生理功能依赖于正常的门控, 但现有晶体结构中S6跨膜螺旋C末端形成的门控结构存在氨基酸缺失, 所以其门控特性未能揭晓. 本文基于已有的晶体结构和AlphaFold算法构建了 11个完整不同构象的TRPM8通道, 发现其S6跨膜螺旋C末端构成的门控存在回环和螺旋2种构象. 在回环构象中, 多个氨基酸参与形成阻碍离子通透的孔道区; 而在螺旋构象中, 仅有关键氨基酸V956发挥门控作用. 由于回环构象的柔性大于螺旋构象, 导致回环构象参与阻碍离子通透的关键氨基酸构象和数量变化多样. 二级结构预测与模建结果表明, S6跨膜螺旋C末端存在回环构象向螺旋构象的转变, 此过程中柔性的回环构象结构域向胞外侧上移, 关键氨基酸向孔道衬外扭转, 增强了与相邻跨膜螺旋S5的相互作用以及S5与TRP螺旋之间的相互作用, 进而形成刚性、 稳定且有序的螺旋构象. 这增加了TRPM8通道各结构域间的协同性, 使能量信息更高效地传递到门控结构域, 为TRPM8通道开启蓄势.
TRPM8通道; 门控; 同源模建; 分子动力学模拟
TRPM(Transient receptor potential melastatin)家族是TRP超家族中最大和最多样化的亚家族[1]. 该亚家族成员与温度、 味觉等环境信号感知密切相关[2]. TRPM亚家族在激活模式、 阳离子选择性和组织分布等方面存在差异, 但在结构上TRPM通道跨膜螺旋S6的C末端区域有一段高度保守的氨基酸序列, 称为TRP螺旋[3].
瞬时受体电位M8型离子通道(TRPM8)是TRPM家族的成员之一[4]. 该通道主要表达于外周感觉神经元, 但也存在于前列腺、 支气管肺组织、 膀胱和泌尿生殖道中[5]. 目前, 已知TRPM8参与各种病理过程, 如与炎症或神经损伤后的冷痛症密切相关, 以及在炎症后冷性痛觉超敏等疼痛条件中起到关键作用[6].
TRPM8通道是一种多模态、 非选择性阳离子通道, 对Ca2+等阳离子敏感. TRPM通道的多模态[7]则表现在受低温(<28 ℃)、 膜去极化及冷相关化合物(薄荷醇、 Icilin和桉树脑等)等多种物理化学刺激的调控[4]. TRPM8通道是冷刺激感受离子通道, 是感受温度和疼痛的受体单元[8], 还可以通过激活该通道增加眼泪缓解干眼症[9]. 因此, TRPM8通道可作为药物开发的作用靶点. 结构与功能关系研究可为靶向调控药物或小分子探针的设计提供研究基础[10~12].
目前, 关于TRPM8通道已经报道了8种晶体结构(表S1, 见文本支持信息), 其中包括3种激动剂复合结构(PDBID: 6NR2, 6NR3, 6NR4), 2种拮抗剂复合结构(PDBID: 6O72, 6O6R), 1种高浓度Ca2+结合结构(PDBID: 6O77)和2种无配体蛋白结构(PDBID: 6BPQ, 6O6A)[13~15]. TRPM8通道是一种同源四聚体, 每个单体包含跨膜结构域(TMD)和胞质结构域(CD). 胞质结构域由N端结构域(NTD)和C端结构域(CTD)组成. N端结构域由4个MHR(Melastatin homology regions, MHRs)组成. C端结构域由位于TRP结构域C端的3段螺旋(CTDH1/2和CC)组成. 在跨膜结构域中, 跨膜螺旋S1~S4组成电压感受域(VSLD), 跨膜螺旋S5和S6及1个孔螺旋(PH)形成孔域(PD). 在孔域中, 4个同源单体的S6螺旋 C末端相互交叉形成阻碍离子通透的空间位阻[16]. S6螺旋向C端方向延伸是TRP螺旋, 其将跨膜结构域连接组成一个整体, 是跨膜结构域与胞内结构域重要的链接结构[14].
离子通道功能是通过其构象变化实现的, 而构象变化的产生则依赖于调控因子与通道间的相互作用[10,17,18]. 由此可知, 作用位点对通道的变构至关重要. TRPM8通道的电压感受结构域(S1~S4螺旋)形成腔室是激动剂(WS-12, Icilin和薄荷醇等), 拮抗剂(TC-Ⅰ 2014和AMTB等), 以及Ca2+的重要结合位点[16]. S1, S4和TRP螺旋形成的狭缝是小分子磷脂酰基醇4,5-2磷酸(PIP2)的结合位点. S4-S5连接子运动是薄荷醇激活TRPM8通道的重要表现[7]; 磷脂分子PIP2在TRPM8通道激活过程中可能影响S4-S5链接子和TRP螺旋的空间结构, 达到调控通道功能的目的[14,15]. 生物学湿实验表明, 薄荷醇对TRPM8通道的激活机制与S4-S5连接子运动相关[16]; 对比冷冻电镜三维结构发现, 磷脂分子PIP2在TRPM8通道激活过程中可能影响S4-S5链接子和TRP螺旋的空间结构, 达到调控通道功能的目的[14]; 晶体结构分析表明, 在门控发生大尺度构象变化时, S6螺旋发生弯折, 弯折点处的螺旋转变为螺旋,是门控开启的重要结构变化[13].
迄今, 结构生物学家虽然提供了多个TRPM8通道的晶体结构, 但关键区域氨基酸或者氨基酸侧链存在缺失. 静态的晶体结构和生物学实验均不能明确通道的门控微观结构, 需进一步挖掘TRPM8通道的门控特性, 揭示其门控动力学行为, 从而为基于TRPM8通道靶给药物研发设计提供理论支持. 本文基于现有TRPM8通道晶体结构和AlphaFold计算结构, 通过“嫁接”优化同源模建模板, 重构了完整的通道结构, 并对模建结果进行了分析评估. 研究结果表明, TRPM8通道S6跨膜螺旋C末端(门控)存在柔性、 不确定、 无序的回环构象和刚性、 稳定、 有序的螺旋构象2种不同构象. 推测回环向螺旋变构过程中胞内结构域向胞外侧上移, 门控氨基酸向孔道衬外扭转, 增强了跨膜螺旋S5, S6和TRP螺旋间的相互作用, 提升了各结构域间的协同性, 有利于TRPM8通道门控的开启.
1 实验方法
1.1 同源模建
根据白领姬鹟(PDBID: 6NR2)的氨基酸序列, 以PDB数据库(https://www.pdbus.org/)和AlphaFold (https://www.alphafold.ebi.ac.uk/)云计算鼠源(UniprotKB: Q8R455, Q8R54D5)、 人源(UniprotKB: Q7Z2W7)结构为基础模板[19], 通过模板的构象互补, 大规模缺失的区域嫁接补充, 对基础模板进行了优化重构, 使用SWISS-MODEL模建服务平台(https://swissmodel.expasy.org/)模建了11个完整的TRPM8通道结构[20].
通过比较现有的晶体结构发现, 6NR2缺失了胞外环区结构(Ⅰ区域), 但是在Ⅲ区域缺失最少 [图1(A)]; 6O77的Ⅰ区域是TRPM8唯一完整的晶体结构, 但是在Ⅲ区域缺失大量氨基酸[图1(C)]. 6NR2和6O77大部分的缺失区域存在互补性, 所以进行相互“嫁接”, 优化模建的模板. 使用SWISS-MODEL平台同源模建, 得到2种完整的TRPM8通道结构6NR2-2[图1(B)]和6O77-2[图1(D)]. 依据此方法模建了6O6R, 6NR2, 6NR4, 6O72, 6BPQ, 6O6A, 6NR3和6O77的结构模型, 并以AlphaFold云计算结构为模板同源模建得到3个TRPM8通道蛋白结构.
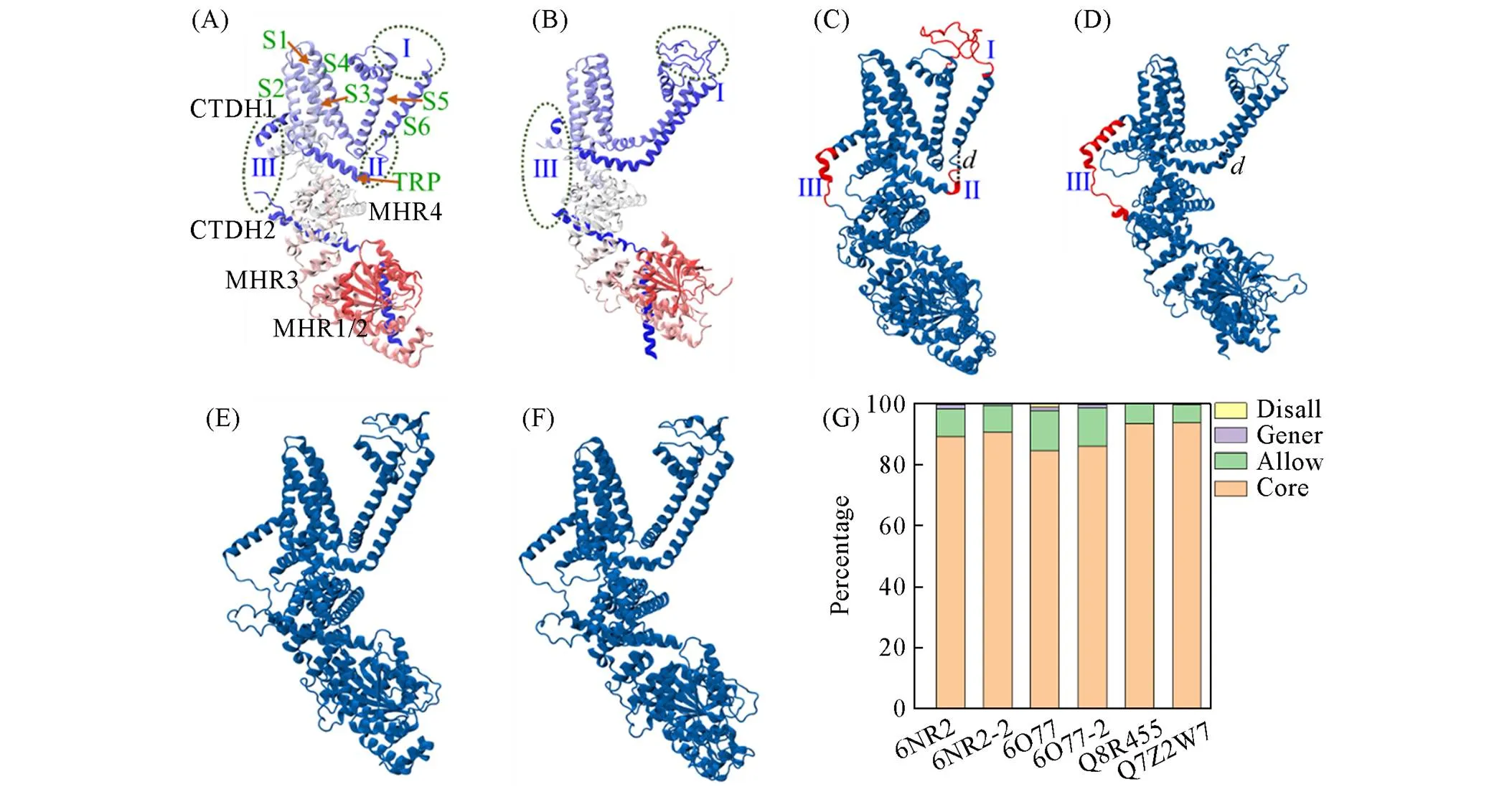
Fig.1 Structural models and the Ramachandran Plot statistics
The crystal structures of 6NR2(A) and 6O77(B); the homology modeling structures of 6NR2(C), 6O77(D) and AlphaFold(E, F); the statistical results of the Ramachandran Plot(G).
1.2 分子动力学模拟参数设置
首先, 用同源模建结果的最优结构构建同源四聚体, 并将TRPM8的四聚体结构嵌入1-棕榈酰-2-油酰-卵磷脂(POPC)的磷脂膜中. 将蛋白质双层系统浸入大小为15.6 nm×15.7 nm×14.9 nm的水分子盒中, 添加0.4 mol/L的CaCl2离子, 使模拟系统净电荷为0, 构建成约363000个原子的动力学模拟体系.
使用NAMD进行分子动力学模拟, 整个体系使用CHARMM36全原子力场[21,22], 溶剂水分子使用TIP3P模型, 在 310 K的恒定温度下进行, 并在所有方向应用周期性边界条件[23]. 采用Particle Mesh Ewald(PME)方法计算长程静电相互作用, 以距离1.2 nm截断半径计算范德华相互作用. 在动力学模拟运行前先进行500000步的能量最小化以消除不良接触, 逐步释放膜磷脂分子、 水分子、 离子和蛋白后, 在NPT系综下进行100 ns分子动力学模拟, 并2 ps记录一次轨迹[11].
1.3 氨基酸相互作用计算
氨基酸间的相互作用使用VMD插件NAMD Energy(V1.4)计算完成[24,25]. NAMD Energy使用标准VMD原子选择语言标识2处氨基酸, 则计算2处氨基酸之间的相互作用能. 指定计算能量类型为非键合能量包含静电相互作用和范德华相互作用; 孔道图使用Hole程序计算绘制得出[26]; 氨基酸二级结构预测使用JPred4计算完成[27]; 结构可视化、 数据分析和图形绘制使用VMD软件[28].
2 结果与讨论
2.1 模型可靠性分析
虽然TRPM8通道的晶体结构描述了整体构象, 但也存在部分氨基酸的缺失(表S1)或侧链不完整. 根据晶体结构相似互补, 采用相互“嫁接”的方法, 进行同源模建. 模建结果中, 不仅6NR2和6O77的“嫁接”部分(Ⅰ区域)与AlphaFold计算的相应结构很相似(RMSD值相差0.19 nm), 而且6O77的S6螺旋 C末端、 链接TRP螺旋的Ⅱ区域也与AlphaFold计算结构相似(RMSD值相差0.11 nm).
在同源模建的结构中, S6螺旋与TRP螺旋链接的Ⅱ区域出现较大差异. 同源模建的6O6R, 6NR2, 6NR4, 6O72和6O6A结构在Ⅱ区域展现为回环构象; 而6NR3和6O77结构中却为螺旋构象. 为了证明 Ⅱ区域存在不同构象, 定义了一个距离(表S2, 见文本支持信息), 即S6螺旋中的氨基酸M958到TRP螺旋N末端的距离[图1(C)]. 假若在距离中填充螺旋, 可根据理论上需要氨基酸个数与实际个数差值分为两类: 一类是差值接近或大于3个氨基酸的模建结果, 包含6O72, 6O6A, 6O6R, 6NR2和6NR4; 另一类是差值小于一个氨基酸的模建结果, 包含6NR3和6O77. 这恰好与模建中形成不同的非螺旋和螺旋构象结果相符. OPM数据计算显示Ⅱ区域在膜内侧, 而非跨膜结构, 所以此结构有可能实现回环构象和螺旋构象间的相互转变[29](图S1, 见文本支持信息). 同时, 研究证实由于Ⅱ区域构象的灵活多变, 导致未解析出此结构域, 这与模建构象具有结构多样性的结果一致[13].
对同源模建的结果模型进行了评估. 模建的氨基酸序列与白领姬鹟序列(6NR2, 6NR4, 6BPQ 和6NR3)的同源性大于96.9%, 与大山雀序列(6O6R, 6O72, 6O6A和6O77)的同源性为97.9%, 与 鼠源和人源(AlphaFold云计算结果UniprotKB: Q8R455, Q8R54D5和Q7Z2W7)的同源性大于78.6%. 拉氏图计算结果表明, 6NR2和6O77模建结果分别在允许区域以上的比值达到99.4%(6NR2-2)和98.4%(6O77-2), 比直接模建模型拉氏结果98.1%(6NR2)和97.4%(6O77)分别上升了1.3%和1% [图1(F)], 说明“嫁接”模建结构的二面角合理性取得进步. TRPM8通道同源模建结构的主链与模板完全重合(图S2, 见文本支持信息), QMEANDisCo Global评分[30]等蛋白三维结构评估均在合理范围内, 充分说明“嫁接”优化模板模建的结果合理可靠.
2.2 基于同源模建的TRPM8通道门控微观结构分析
TRPM8通道4个同源单体的S5和S6跨膜螺旋构成孔道, S6螺旋的C末端区域能够阻碍孔道中阳离子的通透, 形成TRPM8通道的门控结构域. 参与门控的关键氨基酸包含L954~E968. 门控的开启或关闭在孔道图中用不同颜色表示, 蓝色区域表示离子可跨膜通透, 而绿色或者红色区域为阻碍离子通透的路径.
所有模型的孔道图在门控结构域均出现红色或者绿色的狭窄区域, 所以TRPM8通道同源模建结果的所有结构均未出现开放态的构象, 但是门控处的氨基酸构象却表现出很大的差异. 以6NR2结构模建得到的孔道图表明, 氨基酸M958和F959侧链指向孔内, 并形成了绿色或红色狭窄的路径, 与 晶体结构一致[图2(A)]; 同时, 其它氨基酸Y961, Q967和E968也可能参与阻碍离子通透门控过程 [图2(A)~(C)]. 以6NR3结构模建得到的孔道图表明, L954, M958, T962和V963侧链指向孔内, 并形成阻碍离子透过的空间位阻[图2(D)]. 这种构象的关键门控氨基酸数量多, 且构象变化多样.

Fig.2 Conformational changes of the ion permeation pore
The zoom-in view of the bundle crossing gate illustrated that residues known to be critical for ions passing; the solvent accessible pathway as generated by the HOLE software is illustrated as red(radius
以6O77和AlphaFold结构同源模建结果表明, 阻碍离子通透的氨基酸仅有V956, 这与晶体结构一致[13]. 此时, 参与门控的氨基酸L954, M958, F959, T962, V963, Q967和E968都不再指向孔道内侧, 而是指向孔道衬外[图2(E)和2(F)]. 这可能是由于以6NR2模建的门控结构域处于非螺旋态, 而以6O77和AlphaFold模建的门控结构域则处于螺旋态. 在TRPM8通道中跨膜螺旋S6上的氨基酸V956不仅参与影响离子跨膜通透的门控过程, 还具有调控通道离子选择性的作用. 这说明氨基酸V956在控制离子选择通透过程中发挥着关键作用[19,31].
2.3 TRPM8通道门控结构处存在两种不同构象
通过比较多个同源模建结构发现, 门控结构域存在回环和螺旋2种构象[图3(A)和(B)], 其中以6NR2, 6NR4, 6O6A, 6O72, 6O6R为模板构建的完整TRPM8模型门控结构域是回环构象; 以6O77和AlphaFold计算的结构为模板构建的完整模型门控结构域是螺旋构象[图3(B)]. 门控结构域两种构象的差异直观地表现在TRP螺旋N末端最低处到细胞膜中心的距离. 在此, 定义TRPM8通道四聚体S6螺旋上的氨基酸V940主链中心到TRP螺旋N末端最低处的距离(可以表示为孔道的长度)和TRP 螺旋的倾斜角度[图3(A)和(B)], 通过计算得出6O6R, 6NR2, 6NR4, 6O72, 6O6A, 6NR3和6O77同源模建的完整模型的孔道长度分别为3.92, 3.90, 3.86, 3.83, 3.71, 3.26和3.16 nm, 倾斜角度分别为33.61°, 30.49°, 25.65°, 31.33°, 33.86°, 17.11°和18.55°. 可见, 以6O6R, 6NR2, 6NR4, 6O72和6O6A为模板构建的模型孔道长度均大于3.7 nm, TRP倾斜角度大于25°, 此时S6螺旋C末端主链构象为回环; 以6O77为模板构建的模型的孔道长度在3.2 nm左右, TRP倾斜角度约为18°, 此时门控微观结构处于螺旋态[图3(C)]. 以6NR3为模板构建的模型的指标虽与6O77相近, 但门控结构并未完全形成螺旋. 孔道高度和TRP螺旋角度均说明门控微观结构存在回环和螺旋2种构象, 导致孔道构象不同.

Fig.3 Statistical height of the ion permeation pore and angle of TRP helix, secondary structure prediction of gate amino acids
(A) The structure of loop gate; (B) the binding-crossing of helical gate; (C) the distance statistics from S6 helix to N terminal of TRP helix on the left and the angle of TRP helix on the right; (D) prediction of secondary structure of gates amino acids.
对门控区域的氨基酸序列进行了二级结构预测. 结果表明, 门控结构域在JNetPRED (共识预测)、 JNetHMM(基于隐马尔可夫模型的预测)和JNetPSSM(基于位置特定评分矩阵的预测) 3种预测中, 都倾向于形成螺旋, 用红色的长条形表示[图3(D)]. JNetCONF表示预测螺旋的置信度, 用高度值(0~9)或用黑色条形方框的占据来表示. 数值越大, 黑色条形框占据越多, 形成螺旋的可能性越大; 反之形成螺旋的可能性越小. L954~A957的置信度较高, 倾向于形成螺旋, 而M958~T962形成螺旋的置信度较小, 可能形成非螺旋. 以上结果表明, 此段氨基酸既有倾向于形成螺旋构象的趋势, 又存在非螺旋构象的可能性.
二级结构预测与文献报道均表明, TRPM8通道S6螺旋C末端(门控结构域)处在2种不同的结构, 即回环和螺旋态[32]. 由此推测回环构象可以向螺旋构象转变, 此过程中TRPM8通道TRP螺旋倾斜角度降低, 回环构象结构域向胞外侧上移, 门控氨基酸向孔道衬外扭转, 孔道长度变短, S6跨膜螺旋C末端氨基酸形成稳定有序的螺旋构象, 影响门控开启[17]. 该转变现象不仅出现在TRPM8通道中, 在内向整流钾离子Kir2.1通道中也同样存在. Kir2.1通道受PIP2分子调控, 当通道处于关闭态时, PIP2分子与通道的结合位点区域处于回环构象; 当结合PIP2分子时, Kir2.1通道激活, PIP2分子拉动胞内结构域向细胞膜方向移动, 结合位点区域缩短, 回环构象转变为螺旋构象[33].
2.4 门控微观结构处于不同构象的动力学分析
在Hole孔道图中发现处于回环态的TRPM8通道, 使用相同的模板却构建出多个不同的孔道构象; 而处于螺旋时不同的模板则模建出相似的孔道构象, 这可能与其柔性相关. 基于此, 对回环态中以6NR2为模板构建的模型和螺旋态中以6O77为模板构建的模型, 在NPT系综下进行了100 ns自由驰豫的动力学模拟. 2种状态的均方根偏差(RMSD)基本相同, 都稳定在0.32 nm左右[图4(A)], 说明处在2种状态的TRPM8通道均是稳定的结构. 通过分析用于表征结构域柔性的均方根涨落(RMSF), 发现回环态的G960到N970的RMSF值为0.24 nm左右(蓝色曲线), 而螺旋态G960到N970的RMSF值为0.15 nm左右[图4(B)红色曲线]. 以上结果表明, 当门控区域处在回环态时的柔性明显高于螺旋态.

Fig.4 Root⁃mean⁃square deviation(RMSD) and root mean square fluctuation(RMSF) plots of 6NR2 or 6O77 systems under MD simulation
回环构象的柔性大, 形成门控的氨基酸可灵活摆动, 具有不确定性, 是一种无序的构象; 而螺旋构象则刚性强, 门控氨基酸较稳定, 是一种相对有序的构象. 因此, 门控结构域表现出不同的柔性: 回环态的门控结构柔性大, 氨基酸不确定性和无序性导致形成阻碍离子通透的氨基酸数量多且构象多样; 而螺旋态的门控结构刚性大, 氨基酸构象稳定, 且仅有氨基酸V956发挥门控作用.
2.5 处于两种不同构象的关键氨基酸相互作用分析
回环构象的柔性高于螺旋构象, 除与自身相邻氨基酸的相互作用有关外(回环本身柔性大), 还与其周围氨基酸的相互作用密切相关. 因此, 100 ns分子动力学模拟过程中不同构象(回环和螺旋)门控结构域与S5螺旋的相互作用也有差异. 在这2种不同体系中, 虽然回环和螺旋的构象未发生改变, 但S6螺旋C末端氨基酸M958, F959和T962的构象存在较大差异. 在回环态中, 以上3个氨基酸指向孔道域, 形成阻碍离子通透的势垒区[图5(A)]; 而在螺旋态中, 这3个氨基酸由指向孔域转变为偏离孔域, 指向S5跨膜螺旋方向[图5(B)]. 此种现象出现的原因是处在回环构象的氨基酸上升, 扭转形成螺旋构象. 蛋白质构象的转变必定伴随有相互作用的变化.
当TRPM8通道门控结构域为回环构象时, 3个氨基酸M958, F959和T962指向孔道域, 偏离S5跨膜螺旋, 故在动力学过程中这3个氨基酸与相邻跨膜螺旋S5上F849和M843氨基酸存在相互作用, M958与F849相互作用为(-0.21±0.13) kJ/mol[图5(C)], F959与M843相互作用为(-2.80±0.71) kJ/mol[图5(D)], T962与M843相互作用为(-0.08±0.08) kJ/mol[图5(E)]. 当处在螺旋构象时, 3个氨基酸M958, F959和T962偏离孔道域, 指向S5跨膜螺旋, 故在动力学过程中这3个氨基酸分别与相邻跨 膜螺旋S5上F849和M843氨基酸相互作用为(-4.14±1.13) kJ/mol[图5(C)], (-18.70±1.80) kJ/mol [图5(D)]和(-8.66±2.76) kJ/mol[图5(E)]. 除S6螺旋中3个明显偏转的氨基酸与S5螺旋发生相互作用外, 螺旋态的S6门控结构域与S5螺旋总相互作用为(-259.83±47.57) kJ/mol; 回环态的S6门控结构域与S5螺旋总相互作用为(-45.44±19.04) kJ/mol[图5(F)] . 除以上相互作用外, 门控结构域处于不同构象时TRP螺旋与S5螺旋的相互作用也存在不同. 当处于螺旋态时, 位于TRP螺旋上的N970与S5螺旋N末端的R842间的相互作用为(-35.69±5.31)kJ/mol; 当处于回环态时, N970与R842间的相互作用为0~45 ns为(-0.54±0.42) kJ/mol, 45~100 ns为0 kJ/mol[图5(G)]. 氨基酸R842是多个激动剂(薄荷醇、 WS-12和Icilin)的结合位点[7,14], 因此加强了激动剂、 S5螺旋和TRP螺旋彼此间的相互作用, S5螺旋与TRP螺旋间的相互作用对于门控的开启至关重要[34]. 相互作用分析结果表明, 螺旋态的门控结构域与相邻螺旋S5的相互作用均强于回环态, 与TRP螺旋的相互作用也强于回环态.
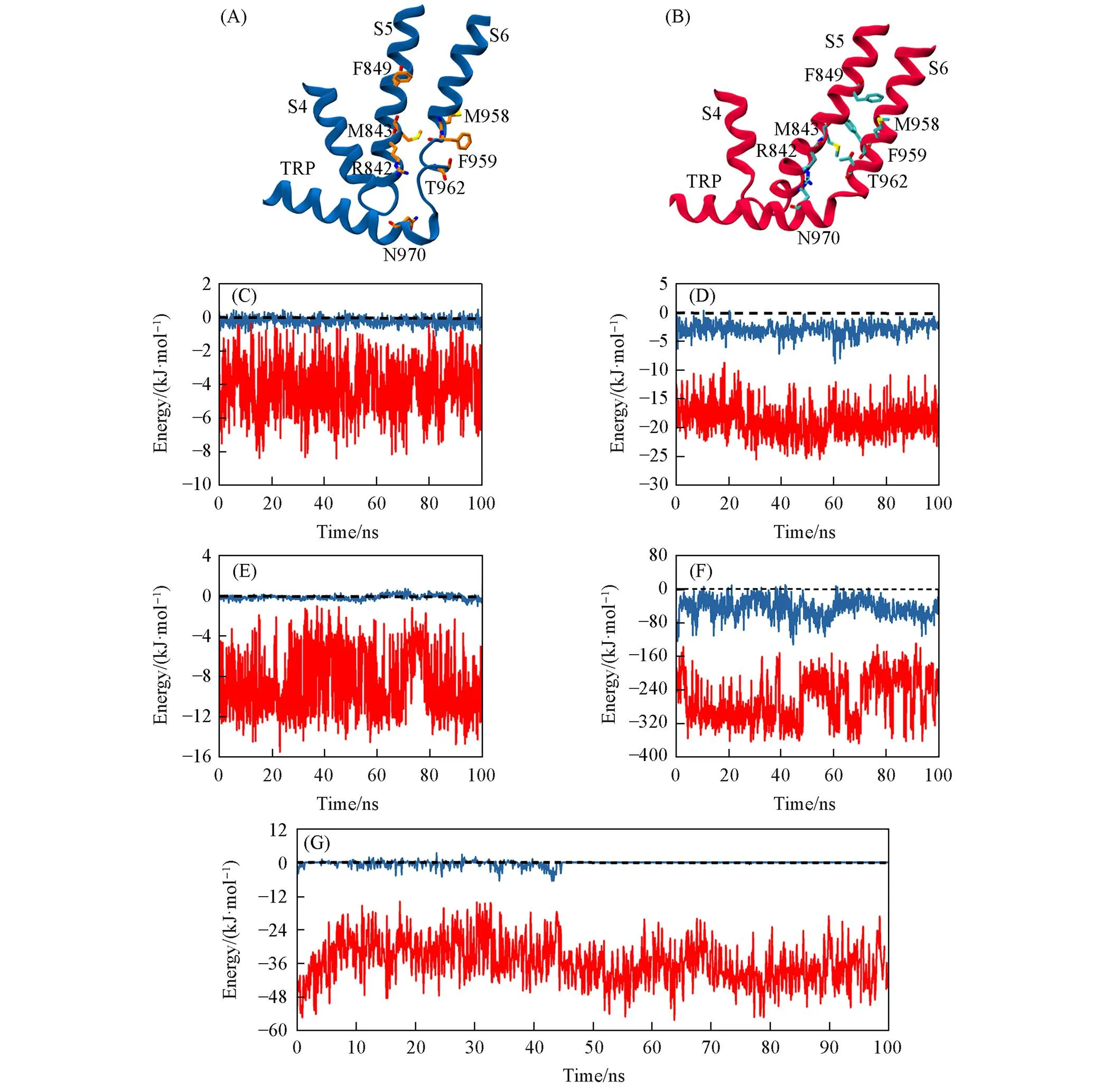
Fig.5 Interaction between the binding⁃crossing domain and S5 helix
The structure of loop gate(A) and helical gate(B); the interaction of M958 with F849(C), F959 with M843(D), T962 with M843(E), the bundle crossing(956 to 970) with S5 N-terminal(840 to 850)(F), N970 with R842(G); (A, B) the main chain is represented by band, and the side chain amino acids are represented by rod; (C—G) blue indicates the interaction of loop gate and red indicates the interaction of amino acids in the bundle crossing of helical gate; theaxis stands for the total energy of electrostatic interactions and Van Der Waals interactions.
当TRPM8通道S6螺旋C末端交叉束结构域, 即门控结构域处于回环态时, 其氨基酸与相邻螺旋S5的相互作用很弱, 柔性强, 导致其门控结构域的构象多变, 形成门控的氨基酸数量不确定, 此时的门控结构域处于一种无序的构象; 当处于螺旋态时, 其氨基酸与相邻螺旋S5的相互作用增强, 柔性减弱, 刚性增强, 形成门控的氨基酸仅有一个, 此时的门控结构域处于一种稳定有序的构象. 以上结果表明, 门控结构域由回环态向螺旋态变构时, 其与相邻跨膜螺旋S5的相互作用增强; TRP螺旋与S5螺旋的相互作用也增强. 以上增强的相互作用不仅能维持S6螺旋C末端处于稳定且有序的螺旋态, 而且加强了TRP螺旋、 跨膜螺旋S5和S6相互作用, 增加了各结构域间的协同性. 因此, TRPM8通道的门控表现出与周围相互作用氨基酸的两种相互作用强度: 当门控结构域处于回环态时, 与周围氨基酸相互作用弱, 不利于电压敏感域的相互作用传递到门控氨基酸; 而处于螺旋态时, 与周围氨基酸相互作用强, 有利于增强各结构域运动的协调性, 为TRPM8通道的开启奠定的结构基础.
TRPM8通道的激活与S5螺旋、 TRP螺旋运动密切相关[1]. 调控因子PIP2磷脂分子能够促进TRPM8通道跨膜螺旋S5向远离孔域方向移动, 且TRP螺旋靠近孔域端向上倾斜[15,17]. 薄荷醇小分子激活TRPM8通道是通过S4-S5连接子运动实现的[7]. TRPM其它亚家族通道调控同样与S4-S5螺旋的连接子和TRP螺旋的运动密切相关. 研究发现, TRPM2通道的TRP螺旋可以整合胞内结构域、 电压感受域和S4-S5连接子的能量和信息, 直接拉拽S6螺旋C末端调控通道开启[35]; TRP螺旋的运动也能够促进激活因子Ca2+与TRPM5通道的结合[35,36], 将TRPM4通道电压感受域S1-S4的信息传递到孔域, 调控通道开闭[37].
3 结 论
TRPM8通道中结合配体的能量信息传递到门控结构域要经过两个途径: 其一是通过S4-S5螺旋连接子作用S5跨膜螺旋; 其二是通过TRP螺旋的运动. 由于有些激活小分子结合在S1-S4电压感受域, 促使电压感受域发生运动, S4螺旋通过S4-S5螺旋连接子与螺旋S5相连接, 进而拉拽S5发生运动; 如果S5螺旋与S6螺旋间存在相互作用, 则有利于门控的开启. 相对S4-S5连接子的间接作用, TRP螺旋直接与S6螺旋C末端(门控结构域)相连接. 当门控结构域处于螺旋构象时, 加强了与S5螺旋、 TRP螺旋间相互作用, 比回环构象更具刚性、 稳定性和有序性. 因此, 门控结构域处于螺旋构象时更具协同性, 有利于TRPM8通道门控的开启.
本文采用同源模建的方法构建了若干个完整的TRPM8通道结构, 采用分子动力学模拟的方法研究了TRPM8通道门控特性. 众所周知, TRPM8通道受多种因素的调控, 包括物理因素和化学因素. 虽然已结晶出部分小分子与通道结合的晶体结构, 呈现出单个和多个小分子与通道的静态结合模式, 然而各因素共同调控通道门控过程中, 各因素间的耦合协同作用机制尚不明确. 同时, 激动剂与拮抗剂结合的位置大致相同, 却能引发不同的门控变构过程, 结合的信息能量如何传递到门控结构域尚不清楚. 在门控变构过程中, 各因素间的耦合协同作用与远程相互作用传递路径有待进一步挖掘.
支持信息见http://www.cjcu.jlu.edu.cn/CN/10.7503/cjcu20220080.
[1] Moran M. M.,, 2018, 58, 309—329
[2] Jimenez I., Prado Y., Marchant F., Otero C., Eltit F., Cabello⁃Verrugio C., Cerda O., Simon F.,, 2020,(12), 2604
[3] Huang Y., Fliegert R., Guse A. H., Lü W., Du J.,, 2020,, 102111
[4] Izquierdo C., Martín⁃Martínez M., Gómez⁃Monterrey I., González⁃Muñiz R.,, 2021,(16), 8502
[5] Señarís R., Ordás P., Reimúndez A., Viana F.,, 2018,(5), 761—777
[6] Moore C., Gupta R., Jordt S. E., Chen Y., Liedtke W. B.,, 2018,(1), 120—142
[7] Xu L. Z., Han Y. L., Chen X. Y., Aierken A., Wen H., Zheng W. J., Wang H. K., Lu X. C., Zhao Z. Y., Ma C., Liang P., Yang W., Yang S. L., Yang F.,, 2020,(1), 3790
[8] González⁃Muñiz R., Bonache M. A., Martín⁃Escura C., Gómez⁃Monterrey I.,, 2019,(11), 2618
[9] Yoon H. J., Kim J., Yang J. M., Wei E. T., Kim S. J., Yoon K. C.,, 2021,(2), 250
[10] Hou S., Gu R. X., Wei D. Q.,, 2017,(11), 2811—2821
[11] Lei X. T., Jin Y. Q., Meng X. Y.,, 2021,(8), 2550—2557 (雷晓彤, 金怡卿, 孟烜宇. 高等学校化学学报, 2021,(8), 2550—2557)
[12] Gu R. X., Liu L. A., Wei D. Q.,, 2013,(10), 571—580
[13] Diver M. M., Cheng Y. F., Julius D.,, 2019,(6460), 1434—1440
[14] Yin Y., Wu M. Y., Zubcevic L., Borschel W. F., Lander G. C., Lee S. Y.,, 2018,(6372), 237—241
[15] Yin Y., Le S. C., Hsu A. L., Borgnia M. J., Yang H. H., Lee S. Y.,, 2019,(6430), eaav9334
[16] Xu L. Z., Han Y. L., Chen X. Y., Aierken A., Wen H., Zheng W. J., Wang H. K., Lu X. C., Zhao Z. Y., Ma C., Liang P., Yang W., Yang S. L., Yang F.,, 2020,(1), 3790
[17] Yin Y., Lee S. Y.,, 2020,(9), 806—819
[18] Gu R. X., Liu L. A., Wei D. Q., Du J. G., Liu L., Liu H.,, 2011,(28), 10817—10825
[19] Jumper J., Evans R., Pritzel A., Green T., Figurnov M., Ronneberger O., Tunyasuvunakool K., Bates R., Žídek A., Potapenko A., Bridgland A., Meyer C., Kohl S. A. A., Ballard A. J., Cowie A., Romera⁃Paredes B., Nikolov S., Jain R., Adler J., Back T., Petersen S., Reiman D., Clancy E., Zielinski M., Steinegger M., Pacholska M., Berghammer T., Bodenstein S., Silver D., Vinyals O., Senior A. W., Kavukcuoglu K., Kohli P., Hassabis D.,, 2021,(7873), 583—589
[20] Waterhouse A., Bertoni M., Bienert S., Studer G., Tauriello G., Gumienny R., Heer F. T., de Beer T. A. P., Rempfer C., Bordoli L., Lepore R., Schwede T.,, 2018,(W1), W296—W303
[21] Vermaas J. V., Hardy D. J., Stone J. E., Tajkhorshid E., Kohlmeyer A.,, 2016,(6), 1112—1116
[22] Phillips J. C., Hardy D. J., Maia J. D. C., Stone J. E., Ribeiro J. V., Bernardi R. C., Buch R., Fiorin G., Hénin J., Jiang W., McGreevy R., Melo M. C. R., Radak B. K., Skeel R. D., Singharoy A., Wang Y., Roux B., Aksimentiev A., Luthey-Schulten Z., Kalé L. V., Schulten K., Chipot C., Tajkhorshid E.,, 2020,(4), 044130
[23] Miao M. Y., Guo Y. C., Shao X. G., Cai W. S.,, 2021,(10), 3116—3124 (妙孟姚, 郭一畅, 邵学广, 蔡文生. 高等学校化学学报, 2021,(10), 3116—3124)
[24] Jurrus E., Engel D., Star K., Monson K., Brandi J., Felberg L. E., Brookes D. H., Wilson L., Chen J. H., Liles K., Chun M. J., Li P., Gohara D. W., Dolinsky T., Konecny R., Koes D. R., Nielsen J. E., Head-Gordon T., Geng W. H., Krasny R., Wei G. W., Holst M. J., McCammon J. A., Baker N. A.,, 2018,(1), 112—128
[25] Acun B., Hardy D. J., Kale L. V., Li K., Phillips J. C., Stone J. E.,, 2018,(6), 1—9
[26] Takematsu K., Pospisil P., Pizl M., Towrie M., Heyda J., Zalis S., Kaiser J. T., Winkler J. R., Gray H. B., Vlcek A.,, 2019,(7), 1578—1591
[27] Drozdetskiy A., Cole C., Procter J., Barton G. J.,, 2015,(W1), W389—394
[28] Humphrey W., Dalke A., Schulten K.,, 1996,(1), 33—38
[29] Shimizu K., Cao W., Saad G., Shoji M., Terada T.,, 2018,(5), 1077—1091
[30] Studer G., Rempfer C., Waterhouse A. M., Gumienny R., Haas J., Schwede T.,, 2020,(6), 1765—1771
[31] Kühn F. J. P., Knop G., Lückhoff A.,, 2007,(38), 27598—27609
[32] Mishra S., Looger L. L., Porter L. L.,, 2021,(10), e23471
[33] Li J. W., Lü S. Q., Liu Y. Z., Pang C. L., Chen Y. F., Zhang S. H., Yu H., Long M., Zhang H. L., Logothetis D. E., Zhan Y., An H. L.,, 2015,, 11289
[34] Ruan Z., Haley E., Orozco I. J., Sabat M., Myers R., Roth R., Du J., Lü W.,, 2021,(7), 604—613
[35] Huang Y. H., Winkler P. A., Sun W. N., Lu W., Du J.,, 2018,(7725), 145—149
[36] Zhang Z., Tóth B., Szollosi A., Chen J., Csanády L.,, 2018,, e36409
[37] Winkler P. A., Huang Y. H., Sun W. N., Du J., Lu W.,, 2017,(7684), 200—204
Analysis of Gating Characteristics of TRPM8 Channel Based on Molecular Dynamics
GAOZhiwei1, LIJunwei2*, SHISai1, FUQiang1, JIAJunru1, ANHailong2*
(,,,,300401,)
Physiological functions of TRPM8 channel, such as temperature sensing, depend on normal gating. Due to the lack and insufficient number of existing crystal structures, the gating characteristics of TRPM8 channels need to be further explored. Therefore, 11 TRPM8 channels with different conformations were constructed, basing on the existing crystal structure and AlphaFold algorithm. It was found that there were two different architectural in the S6 transmembrane helical bundle crossing domain(gating): loop state and helix state. At the loop state, multiple amino acids participated in the formation of pore regions that hinder ion permeability, while in the helical architecture. Only the key amino acid V956 played a gating role. Because the flexibility of the gated loop architecture was greater than that of the helical, and the number of key amino acids involved in gating was different. The secondary structure prediction showed that the loop architecture could change to the helical. In this process, the flexible loop domain moved upward to the outside of the cell, and the gated amino acids twisted to the outside of the pore lining. At the same time, the interaction with the adjacent transmembrane S5 helix was enhanced, and a rigid, stable and orderly helical architecture was formed. This promoted the coordination between the various domains of TRPM8 channel, enabled energy and information to be transmitted to the bundle crossing more efficiently, which was conducive to channel opening.
TRPM8 channel; Gating mechanism; Homology modeling; Molecular dynamics simulation
O641
A
10.7503/cjcu20220080
2022-02-08
2022-04-28.
李军委, 男, 博士, 副教授, 主要从事离子通道结构与功能关系研究. E-mail: junwei_li@hebut.edu.cn
安海龙, 男, 博士, 教授, 主要从事离子通道结构与功能关系、 离子通道与重大疾病及离子通道靶向药物分子设计等方面的研究. E-mail: hailong_an@hebut.edu.cn
国家自然科学基金(批准号: 11647121, 81830061, 11747610)、 河北省自然科学基金(批准号: F2021202001, H20202005, 19JCYBJC28300)和河北省留学引进人员资助项目(批准号: C20210337)资助.
Supported by the National Natural Science Foundation of China(Nos.11647121, 81830061 and 11747610), the Natural Science Foundation of Hebei Province, China(Nos.F2021202001, H20202005 and 19JCYBJC28300), and the Funding Program for Overseas-educated Scholars of Hebei Province, China(No.C20210337).
(Ed.: L, H, W, K)

