柴胡转录组SSR的分布及序列特征分析
刘迪 欧阳艳飞 徐鹏 甄军波 刘琳琳 张利超 迟吉娜


摘要:【目的】分析柴胡轉录组中SSR的分布及序列特征,为开发多态性良好的、功能基因相关的SSR标记提供理论依据。【方法】以不同温度处理的柴胡种子为材料,经高通量转录组测序后,使用Trinity对测序结果进行de novo组装,并利用MISA对组装得到的Unigenes进行SSR位点搜索,最后统计分析SSR的分布及序列特征。【结果】从转录组数据组装获得244194条Unigenes,其N50值为1036 bp,平均长度791 bp,总长度193138105 bp。从Unigenes序列中共检测到50303个SSR位点,去除20405个复合型SSR位点,以29898个单一型SSR位点为后续分析对象。转录组SSR的出现频率为12.24%,平均分布距离6.46 kb,主要重复基元类型为二核苷酸,共17105个,占SSR总数的57.21%,其次为三核苷酸和单核苷酸,分别占SSR总数的20.75%和20.46%,四核苷酸~六核苷酸重复基元数量均较少。转录组SSR中,共有97种重复基元,其中二核苷酸和三核苷酸重复基元分别以AT/AT和ATC/GAT为主,分别占SSR总数的33.33%和3.98%;重复次数5~10次的SSR位点数量最多,共27942个,占SSR总数的93.46%。转录组SSR序列长度存在明显差异(12~76 bp),平均长度15.28 bp。【结论】柴胡转录组的SSR位点出现频率较高,类型较丰富,具有开发出高多态性SSR分子标记的潜力,将其用于柴胡的遗传多样性分析、种质资源评价及分子标记辅助育种等研究。
关键词: 柴胡;转录组;SSR;分布;序列特征
中图分类号: S567.79 文献标志码: A 文章编号:2095-1191(2022)03-0676-08
Analysis of SSR distribution and sequence characteristics of Bupleurum chinense DC. transcriptome
LIU Di OUYANG Yan-fei XU Peng ZHEN Jun-bo LIU Lin-lin ZHANG Li-chao CHI Ji-na
(1Cotton Research Institute,Hebei Academy of Agriculture and Forestry Sciences/Key Laboratory of Cotton Biology and Genetic Breeding in Huanghuaihai Semiarid Area,Ministry of Agriculture and Rural Affairs, Shijiazhuang, Hebei 050051, China; 2Chengde Chinese Herbal Medicine Green Ecological Planting Technology Service Center,
Chengde, Hebei 067000, China)
Abstract:【Objective】The SSR locus information and sequence characteristics in the transcriptome of Bupleurum chinense DC. were analyzed, so as to provide the basis for the development of functional gene-related SSR markers with good polymorphism. 【Method】 The processed seeds of B. chinense were used as materials to perform high-throughput transcriptome sequencing. Trinity was used to assemble the sequencing results for De Novo assembly. The unigenes obtained were searched for the presence of SSR sites by MISA software and then the SSR data was statistically analyzed. 【Result】 244194 unigenes were assembled from B. chinense DC. The transcriptome data had an N50 value of 1036 bp, an average length of 791 bp and a total length of 193138105 bp. A total of 50303 SSR loci were identified from the unigene sequences, among which 20405 were complex SSR loci and 29898 SSR loci were actually analyzed. The frequency of SSR accounted for 12.24% of all unigenes with an average distribution distance of 6.46 kb. The major repeat motifs were dinucleotide (17105), accounting for 57.21% of all SSRs,followed by trinucleotide and mononucleotide (20.75% and 20.46%,respectively). The proportion of tetranucleotide repeat units to hexanucleotide repeat units was low. 97 kinds of repeat motifs were found in the B. chinense DC. transcriptome. The main repeat motif types in dinucleotide were AT/AT and in trinucleotide were ATC/GAT,which accounted for 33.33% and 3.98% of the total SSR, respectively. The types of SSR repeat units with 5 to 10 repeats had the highest proportion, with a total of 27942, accounting for 93.46% of the total SSRs. The sequence length ranged from 12 bp to 76 bp, with an average length of 15.28 bp. 【Conclusion】 The SSR loci in the B. chinense transcriptome have high frequency and diversity, and it is possible to develop SSR primers with high polymorphism, which can be used in the analysis of B. chinense genetic diversity, germplasm resource evaluation and molecular marker-assisted breeding.CBA22FE2-C5D6-49B5-95BB-86A3709C1060
Key words: Bupleurum chinense DC.; transcriptome; SSR; distribution; sequence characteristics
Foundation items:National System of Chinese Medicinal Materials Industry (CARS-21);Hebei Technical Innovation System of Traditional Chinese Medicine Industry(HBCT2018060201);Fundamental Research Funds of Hebei Academy of Agriculture and Forestry Sciences (2018070203)
0 引言
【研究意义】柴胡(Bupleurum chinense DC.)为伞形科柴胡属多年生草本植物,其根为最常见的大宗药材之一,具有疏散退热、疏肝解郁、升阳举气的功效。全世界柴胡属植物种类繁多,约有150种,我国有柴胡属植物42种、17变种和7变型(Pan,2006)。《中华人民共和国药典》2020版规定中药柴胡为柴胡(Bupleurum chinense DC.)或狭叶柴胡(Bupleurum scorzonerifolium Willd.)的干燥根(国家药典委员会,2020),但由于我国各省区均有柴胡属植物如竹叶柴胡、马尔康柴胡、马尾柴胡等种植,在其产地也作柴胡入药,使得柴胡种源混乱、临床药效难以保证(黄涵签等,2017;石丽霞等,2020)。因此,柴胡不同品种的区分和鉴别显得尤为重要。目前DNA分子标记是鉴定种质资源遗传多样性的重要技术。根据柴胡转录组数据分析柴胡SSR的分布及组成特征,以期开发出多态性高的SSR引物,对柴胡的遗传多样性分析、种质资源评价及分子标记辅助育种等具有重要意义。【前人研究进展】柴胡的研究主要集中在抗肿瘤作用(刘丹和王佳贺,2018)、化学成分(颜美玲等,2018)、基因表达量分析(杨林林等,2019)、复种模式(Liu et al.,2021)等方面,关于SSR分子标记的研究及应用较少。SSR分子标记是一类由1~6个核苷酸为重复单位组成的长度一般不超过200 bp的简单重复序列,在遗传图谱构建、基因定位克隆、品种鉴定、分子标记辅助育种等方面应用较广,是现阶段应用最广泛的分子标记技术之一(He et al.,2003;Wang et al.,2013)。SSR分子标记已在多种药用植物如黄芩(齐琳洁,2015)、苦参(段永红等,2018)、杭白芷(刘倩倩等,2018)、黄芪(刘亚令,2019)、重楼(王海明等,2019)等的遗传多样性研究及育种中得以应用。目前,有关柴胡SSR分子标记的研究及应用方面报道较少。Sui等(2010)构建了北柴胡cDNA文库,对随机挑选的3000多个克隆进行5′端单反应测序,并利用测序结果分析得到86个SSR位点。战晴晴等(2010)从30条ISSR引物和44对SSR引物中分别筛选出28条和14对多态性引物,构建了首张北柴胡遗传图谱,该图谱包含13个连锁群、80个位点,为北柴胡性状基因定位等打下基础。吴素瑞等(2015)从50对柴胡SSR引物中筛选出9对多态性高的引物,获得了供试种质的SSR特征谱带数据,将所有种质分为四大类,证实SSR分子标记可用于柴胡种质鉴别。朱楚然等(2019)对三岛柴胡进行了基因组Survey测序,并对所获得的基因组序列进行了SSR分析,检测到91377条SSR序列,对其中33条SSR序列进行扩增,筛选获得21对有效扩增引物。【本研究切入点】目前可供使用的柴胡SSR分子标记较少,且鲜见转录组SSR的分布及序列特征分析方面的研究报道。虽然杜江等(2015)对柴胡转录组进行全面分析,但对于SSR方面并未深入分析及验证。【拟解决的关键问题】基于高通量测序获得的柴胡转录组数据,深度挖掘转录组SSR位点,并对其在转录组中的分布及序列特征分析进行统计分析,为开发大量多态性良好的SSR分子标记,并用于柴胡遗传多样性分析、种质资源评价及分子标记辅助育种等研究打下基础。
1 材料与方法
1. 1 试验材料
供试材料为柴胡(Bupleurum chinense DC.)室温(15~20 ℃)保存种子及4 ℃和-20 ℃低温冻融处理12 d的种子,每种处理的种子设置3个重复,共计9个样本,放入液氮迅速冷冻保存备用。
1. 2 转录组测序
将处理过的柴胡种子送至迈维代谢生物科技有限公司,提取总RNA,并进行文库构建及测序工作。测序平台为Illumina HiSeq X Ten高通量测序平台。
1. 3 序列拼接及SSR位点搜索
使用Trinity对柴胡转录组测序Clean reads进行de novo组装,并使用微卫星标记识别工具(MicroSAtellite Identification tool,MISA)(http://pgrc.ipk-gatersleben.de/misa/)对组装得到的所有Unigenes进行SSR位点检测,搜索参数为单核苷酸最少重复10次,二核苷酸最少重复6次,三核苷酸~六核苷酸均最少重复5次。
1. 4 统计分析
以柴胡转录组中SSR位点的重复单元的类型、出现频率、SSR分布的平均距离及其基元组成来分析柴胡转录组SSR的分布及其序列特征。SSR位点的平均分布距離和出现频率相关计算公式:SSR位点的平均分布距离(kb)=总Unigenes的长度/检测到的SSR的总数;SSR的出现频率(%)=SSR数量/Unigenes的总序列数量。
1. 5 SSR引物设计及验证
根据SSR两端互补序列,使用Primer 3.0设计批量引物,引物设计参数:退火温度为57~63 ℃,上、下游引物退火温度相差小于5 ℃;扩增产物长度为100~300 bp;引物GC含量为36%~67%;引物长度为18~22 bp。随机挑选20对引物进行PCR验证,引物由金唯智生物科技有限公司合成。CBA22FE2-C5D6-49B5-95BB-86A3709C1060
2 结果与分析
2. 1 柴胡转录组中SSR出现频率和分布距离分析结果
经Trinity对柴胡转录组数据进行组装,共获得244194条Unigenes序列,其N50值为1036 bp,平均长度791 bp,总长度193138105 bp,GC含量45.39%。依照检索条件检索重复单元长度为1~6 bp的SSR,共鉴定出50303个SSR位点,只以其中的29898个单一型SSR位点为分析对象,其余的20405个复合型SSR位点不作分析。分析发现,柴胡转录组SSR的出现频率为12.24%(表1),平均分布距离6.46 kb,即平均6.46 kb就出现1个SSR位点,说明柴胡转录组中含有大量SSR位点,为后期SSR引物的开发利用提供参考。
2. 2 柴胡转录组中SSR位点的基元类型分析结果
由表1可知,柴胡转录组中SSR位点重复类型丰富,存在单核苷酸~六核苷酸重复基元类型,但重复基元类型之间数量差异较大,其中,主要重复基元类型为二核苷酸,数量最多,有17105个,占SSR总数的57.21%,其次为三核苷酸和单核苷酸,分别占SSR总数的20.75%和20.46%;四核苷酸~六核苷酸重复基元数量均较少;在柴胡转录组SSR中,六核苷酸重复基元的分布距离最高,为5518.23 kb,但出现频率最低,仅为0.01%;二核苷酸重复基元的分布距离最低,仅为11.29 kb,但出现频率最高,为7.00%。总体来说,不同重复基元类型的SSR出现频率和分布距离主要受SSR数量影响,重复基元数越多,其出现频率越高,分布距离越小。
2. 3 柴胡转录组中SSR重复基元碱基组成分析
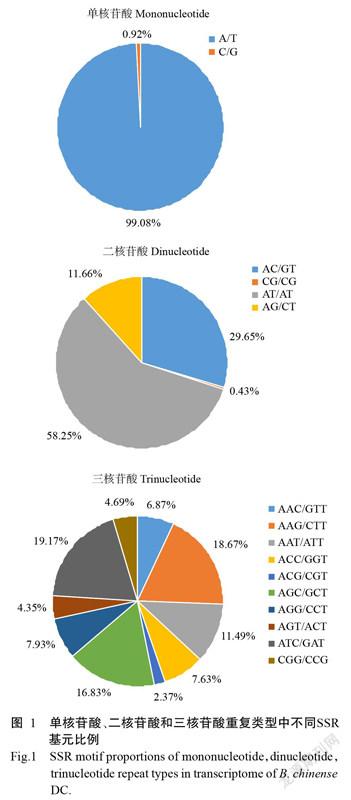
在柴胡转录组SSR位点中,共有97种基元(已考虑碱基互补、移码等因素),单核苷酸~六核苷酸出现的重复基元分别为2、4、10、29、25和27种(表2)。从出现频率来看,单核苷酸、二核苷酸和三核苷酸重复基元为优势的3种重复基元,其不同SSR基元比例如图1所示。单核苷酸重复基元以A/T为主,共6063个,占SSR总数的20.28%,占单核苷酸重复基元数的99.08%。二核苷酸重复基元以AT/AT为主,共9964个,占总SSR总数的33.33%,占二核苷酸重复基元数的58.25%,其次为AC/GT(5072个)、AG/CT(1995个)和CG/CG(74个),分别占SSR总数的16.96%、6.67%和0.25%,分别占二核苷酸重复基元数的29.65%、11.66%和0.43%。三核苷酸重复基元以ATC/GAT为主,共1189个,占SSR总数的3.98%,占三核苷酸重复基元数的19.17%,其次是AAG/CTT (1158个)和AGC/GCT(1044个),分别占SSR总数的3.87%和3.49%,分别占三核苷酸重复基元数的18.67%和16.83%,以ACG/CGT(147个)占SSR总数的比列最低,仅为0.49%,分别占三核苷酸重复基元数的2.37%。四核苷酸重复基元以TATG/CATA (143个)为主,占SSR总数的0.48%。五核苷酸和六核苷酸重复基元出现频率均不高,仅为个位数,占总SSR的比例也很低。
2. 4 柴胡转录组中不同基元重复次数分析结果
柴胡转录组SSR中,重复次数5~10次的SSR位点数量最多,共27942个,占SSR总数的93.46%,其中,重复次数为6次的SSR位点数最高,为7869个,占SSR总数的26.32%;其次是重复次数为7次的SSR位点,共4764个,占SSR总数的15.93%(图2)。单核苷酸重复类型中重复次数最多的是10次,共4201个,而二核苷酸~六核苷酸重复类型的重复次数多集中在5~9次,其中三核苷酸重复基元中重复次数最多的是5次,共3783个,且随着各基元重复次数的增加SSR数量逐渐降低(图2)。
2. 5 柴胡转录组中SSR重复序列长度分析结果
SSR重复序列长度是影响SSR多样性的重要因素,当长度<12 bp时SSR多态性极低(Temnykh,2001)。因此,本研究对序列长度≥12 bp的SSR进行分析,结果(图3)显示,柴胡转录组SSR序列长度存在较大差异,长度范围12~76 bp,平均长度15.28 bp,其中,序列长度12~20 bp的SSR数量最多,共计22656个,占SSR(≥12 bp)总数的达95.28%,且序列长度12 bp的SSR数量最多,共6262个,占SSR(≥12 bp)总数的20.94%;序列长度为21~30 bp的SSR数量为1033个,占SSR(≥12 bp)总数的4.34%;序列长度>30 bp的SSR数量为90个,占SSR(≥12 bp)总数的0.38%。不同重复类型的SSR位點中,四核苷酸的重复序列长度范围最广,为20~76 bp,平均长度为24.73 bp(表1)。
2. 6 柴胡转录组SSR引物的设计及验证
为能更好地使用挖掘的柴胡SSR位点,本研究对29898个SSR位点进行批量设计引物,为了验证设计引物的有效性,随机选取合成了包括二核苷酸重复基元~六核苷酸重复基元的20对引物(表3),分别对甘肃柴胡和冀柴1号进行扩增,扩增产物用1%琼脂糖凝胶电泳检测,结果显示,有14对引物能扩增出清晰且单一条带,引物有效扩增率为70%(图4),其中,10对SSR引物扩增产物长度与预期相符,4对SSR引物扩增产物长度超过预期,表明设计的引物大部分真实可用,且设计的引物在两种柴胡之间可通用,可用于柴胡种质鉴定及遗传多样性等方面的研究。
3 讨论
近年来,随着高通量技术快速发展,测序成本逐渐降低,转录组测序数据涵盖数据量越来越大,转录组学的研究应用越来越广。本研究从柴胡转录组测序获得的244194条Unigenes中共鉴定出单核苷酸重复基元~六核苷酸重复基元6种类型29898个SSR位点,SSR的出现频率为12.24%,平均分布距离6.46 kb。与其他药用植物相比,柴胡转录组SSR的出现频率低于半夏(13.47%)(王森等,2014)、野三七(16.86%)(李翠婷等,2014)、金钗石斛(15.78%)(李清等,2017)、穿心莲(32.36%)(李俊仁等,2018),但明显高于杜仲(2.90%)(黄海燕等,2013)和金铁锁(8.54%)(叶鹏等,2019),存在物种间差异的原因除与物种自身有关外,还可能与SSR搜索标准、转录组中Unigene数量和长度等有关。CBA22FE2-C5D6-49B5-95BB-86A3709C1060
绝大多数植物的SSR位点主要重复基元为二核苷酸和三核苷酸,但不同物种中常见重复基元类型有所不同(王东等,2014)。本研究发现,柴胡SSR中主要重复基元类型为二核苷酸,有17105个,占SSR总数的57.21%;其次为三核苷酸,占SSR总数的20.75%,四核苷酸~六核苷酸所占比例均较小,表明低级基元SSR的多态性普遍高于高级基元(Dreisigacker et al.,2004)。前人研究发现,杜仲(黄海燕等,2013)、党参(王东等,2014)和野三七(李翠婷等,2014)的主要重复基元类型均为二核苷酸,而金铁锁(叶鹏等,2019)和款冬(贺润丽等,2019)的主要重复基元则为三核苷酸。GC含量对于基因的结构和功能具有重要影响,是序列的重要特征之一。GC重复基元在多数植物中出现频率均较低(Sun et al.,2013),如野三七中仅有1个(李翠婷等,2014),党参中仅有5个(王东等,2014),霍山石斛中有73个(洪克前等,2020),本研究从柴胡SSR中鉴定出GC重复基元74个,仅占SSR总数的0.25%,且二核苷酸重复以AT/AT为主,占SSR总数的33.33%;三核苷酸重复以ATC/GAT为主,占总SSR的3.98%,表明柴胡SSR中A/T为优势重复基元。
SSR重复序列长度由基元重复次数和碱基数量决定,是影响SSR多态性的重要因素(Temnykh,2001)。柴胡转录组SSR中,重复次数5~10次的SSR位点数量最多,为27942个,占SSR总数的93.46%,表明柴胡转录组SSR多态性较高。前人研究表明,当SSR序列长度<12 bp时,多态性极低,长度为12~20 bp的SSR多态性中等,长度>20 bp的多态性较高(Temnykh,2001)。本研究柴胡转录组SSR中,序列长度12~20 bp的SSR数量最多,共计22656个,占SSR(≥12 bp)总数的达95.28%;序列长度20 bp以上的SSR数量占SSR总数的4.72%,共1123个,其中,多态性较高的低级重复基元(二核苷酸和三核苷酸)共897个,占79.9%,可用于后续SSR引物的开发。本研究设计的20对引物中,有14对引物能扩增出清晰且单一条带,引物有效扩增率为70%,其中有4对SSR引物扩增产物长度超过预期,原因可能为基因组相对转录组具有内含子,导致其长度变长,也可能是组装错误所导致(Xiang et al.,2015)。
4 结论
柴胡转录组SSR出现频率较高,类型较丰富,具有开发出高多态性SSR分子标记的潜力,将其用于柴胡的遗传多样性分析、种质资源评价及分子标记辅助育种等研究。
参考文献:
杜江. 2015. 基于RNA-Seq技术的柴胡转录组分析[D]. 太原:山西农业大学. [Du J. Analysis on the Transcriptome of Bupleuri Radix through RNA-Seq[D]. Taiyuan:Shanxi Agricultural University.]
段永红,雷海英,张旭,吴玉香,孙毅. 2018. 药用植物苦参EST-SSR标记的开发及DNA指纹图谱的构建[J]. 中国农业大学学报,23(9):21-31. [Duan Y H,Lei H Y,Zhang X,Wu Y X,Sun Y. 2018. Development of EST-SSR markers and construction of DNA fingerprint in Sophora flavescens[J]. Journal of China Agriculture University,23(9):21-31.] doi:10.11841/j.issn.1007-4333.2018. 09.03.
国家药典委员会. 2020. 中华人民共和国药典[M]. 北京:中国医药科技出版社. [Chinese Pharmacopoeia Commission. 2020. Pharmacopoeia of the Peoples Republic of China[M]. Beijing:China Medical Science Press.]
贺润丽,韩毅丽,段静静,刘计权,王莉花. 2019. 款冬转录组SSR 标记开发及其多态性研究[J]. 中草药,50(20):5026-5032. [He R L,Han Y L,Duan J J,Liu J Q,Wang L H. 2019. Development and polymorphism analysis of SSR molecular markers in Tussilago farfara transcriptome[J]. Chinese Traditional and Herbal Drugs,50(20):5026-5032.] doi:10.7501/j.issn.0253-2670.2019.20.026.
洪克前,夏維丽,李佩玲,徐函兵,谷会,陈丽. 2020. 霍山石斛叶转录组中SSR位点信息分析[J]. 中国农学通报,36(27):106-110. [Hong K Q,Xia W L,Li P L,Xu H B,Gu H,Chen L. 2020. Data mining of simple sequence repeats in Dendrobium huoshanense transcriptome[J]. Chinese Agricultural Science Bulletin,36(27):106-110.] doi:10.11924/j.issn.1000-6850.casb20200100029.
黄海燕,杜红岩,乌云塔娜,刘攀峰. 2013. 基于杜仲转录组序列的SSR分子标记的开发[J]. 林业科学,49(5):176-181. [Huang H Y,Du H Y,Wuyu T N,Liu P F. 2013. Development of SSR molecular markers based on transcriptome sequencing of Eucommia ulmoides[J]. Scientia Silvae Sinicae,49(5):176-181.] doi:10.11707/j.1001-7488.20130523.CBA22FE2-C5D6-49B5-95BB-86A3709C1060
黄涵签,王潇晗,付航,王妍,杨世海. 2017. 柴胡属药用植物资源研究进展[J]. 中草药,48(14):2989-2996. [Huang H Q,Wang X H,Fu H,Wang Y,Yang S H. 2017. Research progress on medicinal plant resources of Bupleurum L.[J]. Chinese Traditional and Herbal Drugs,48(14):2989-2996.] doi:10.7501/j.issn.0253-2670.2017.14.030.
李翠婷,張广辉,马春花,孟珍贵,陈军文,杨生超. 2014. 野三七转录组中SSR位点信息分析及其多态性研究[J]. 中草药,45(10):1468-1472. [Li C T,Zhang G H,Ma C H,Meng Z G,Chen J W,Yang S C. 2014. Analysis on SSR loci information in transcriptome of Panax vienamensis var. fuscidiscus and its polymorphism[J]. Chinese Traditional and Herbal Drugs,45(10):1468-1472.] doi:10.7501/ j.issn.0253-2670.2014.10.021.
李俊仁,陈秀珍,汤小婷,何瑞,詹若挺. 2018. 穿心莲转录组SSR位点信息分析[J]. 中国中药杂志,43(12):2503-2508. [Li J R,Chen X Z,Tang X T,He R,Zhan R T. 2018. SSR loci information analysis in transcriptome of Andrographis paniculate[J]. China Journal of Chinese Materia Medica,43(12):2503-2508.] doi:10.19540/j.cnki. cjcmm.20180329.012.
李清,李标,郭顺星. 2017. 金钗石斛转录组SSR位点信息分析[J]. 中国中药杂志,42(1):63-69. [Li Q,Li B,Guo S X. 2017. SSR information in transcriptome of Dendro-bium nobile[J]. China Journal of Chinese Materia Medica,42(1):63-69.] doi:10.19540/j.cnki.cjcmm.20161222.044.
刘丹,王佳贺. 2018. 柴胡皂苷抗肿瘤作用机制的研究进展[J]. 现代药物与临床,33(1):203-208. [Liu D,Wang J H. 2018. Research progress on antitumor mechanisms of saikosaponin[J]. Drugs & Clinic,33(1):203-208.] doi:10.7501/j.issn.1674-5515.2017.01.045.
刘倩倩,叶浩婷,李放,王荣婷,田恩伟. 2018. 杭白芷种质资源遗传多样性的SSR分析[J]. 南方农业学报,49(3):418-423. [Liu Q Q,Ye H T,Li F,Wang R T,Tian E W. 2018. SSR analysis for genetic diversity of Angelica dahu-rica var. formosana[J]. Journal of Southern Agriculture,49(3):418-423.] doi:10.3969/j.issn.2095-1191.2018.03. 02.
刘亚令,耿雅萍,解潇冬,王芳,张鹏飞. 2019. 基于SSR分子标记的药用黄芪遗传多样性与遗传结构分析[J]. 草地学报,27(5):1154-1162. [Liu Y L,Geng Y P,Xie X D,Wang F,Zhang P F. 2019. Genetic diversity and genetic structure analysis of Astragalus based on SSR molecular marker[J]. Acta Agrestia Siniga,27(5):1154-1162.] doi:10.11733/j.issn.1007-0435.2019.05.006.
齐琳洁,龙平,蒋超,袁媛,黄璐琦. 2015. 黄芩基因组SSR分子标记的开发及遗传多样性分析[J]. 药学学报,50(4):500-505. [Qi L J,Long P,Jiang C,Yuan Y,Huang L Q. 2015. Development of microsatellites and genetic diversity analysis of Scutellaria baicalensis Georgi using genomic-SSR markers[J]. Acta Pharmaceutica Sinica,50(4):500-505.] doi:10.16438/j.0513-4870.2015.04.002.
石丽霞,李科,秦雪梅,李震宇,崔连杰,李树颖,曹宇欣,王盛金. 2020. 基于糖特征图谱的不同柴胡的品种鉴别[J]. 药学学报,55(12):2968-2975. [Shi L X,Li K,Qin X M,Li Z Y,Cui L J,Li S Y,Cao Y X,Wang S J. 2020. Identification of different Bupleurum varieties based on carbohydrate-specific chromatograms[J]. Acta Pharmaceutica Sinica,55(12):2968-2975.]doi:10.16438/j.0513-4870. 2019-1054.CBA22FE2-C5D6-49B5-95BB-86A3709C1060
王东,曹玲亚,高建平. 2014. 党参转录组中SSR位点信息分析[J]. 中草药,45(16):2390-2394. [Wang D,Cao L Y,Gao J P. 2014. Data mining of simple sequence repeats in Codonopsis pilosula transcriptome[J]. Chinese Traditional and Herbal Drugs,45(16):2390-2394.] doi:10.7501/ j.issn.0253-2670.2014.16.021.
王海明,阮成江,梁玖華,韩平,闫蕊,董旭,刘祾悦,帅小春,刘贺贺. 2019. 高通量RNA-seq技术开发梵净山重楼SSR标记[J]. 分子植物育种,17(18):6059-6065. [Wang H M,Ruan C J,Liang J H,Han P,Yan R,Dong X,Liu L Y,Shuai X C,Liu H H. 2019. Development of SSR markers of Fanjingshan Paris polyphylla Smith var. chinensis based on high throughput RNA-Seq[J]. Molecular Plant Breeding,17(18):6059-6065.] doi:10.13271/j.mpb. 017.006059.
王森,张震,姜倪皓,张广辉,沙本才,杨生超,陈军文. 2014. 半夏转录组中的SSR位点信息分析[J]. 中药材,37(9):1566-1569. [Wang S,Zhang Z,Jiang N H,Zhang G H,Sha B C,Yang S C,Chen J W. 2014. SSR information in transcriptome of Pinellia ternate[J]. Journal of Chinese Medicinal Materials,37(9):1566-1569.] doi:10.13863/j. issn1001-4454.2014.09.015.
吴素瑞,高珂,赵立子,余马,杨兴旺,隋春. 2015. 利用SSR分子标记鉴别柴胡栽培种质的方法学研究[J]. 世界科学技术-中医药现代化,17(9):1806-1812. [Wu S R,Gao K,Zhao L Z,Yu M,Yang X W,Sui C. 2015. Studies on identification of Bupleurum Cultivated germplasm using SSR molecular markers[J]. World Science and Techno-logy/Modernization of Traditional Chinese Medicine and Materia Medica,17(9):1806-1812.] doi:10.11842/wst. 2015.09.010.
颜美玲,杨柳,侯阿娇,郭新月,满文静,邢绪东,韩华. 2018. 柴胡化学成分及药理作用研究进展[J]. 中医药信息,35(5):103-109. [Yan M L,Yang L,Hou A J,Guo X Y,Man W J,Xing X D,Han H. 2018. Research progress on chemical composition and pharmacological effect of Bupleurum chinense[J]. Information on Traditional Chinese Medicine,35(5):103-109.] doi:10.19656/j.cnki.1002-2406.180161.
杨林林,赵钰,韩梅,杨利民. 2019. 北柴胡和狭叶柴胡中黄酮类成分及其关键酶基因表达的组织差异分析[J]. 中草药,50(1):188-194. [Yang L L,Zhao Y,Han M,Yang L M. 2019. Analysis of tissues difference of flavonoids and key enzyme genes expression in Bupleurum chinense and B. scorzonerifolium[J]. Chinese Traditional and Herbal Drugs,50(1):188-194.] doi:10.7501/j.issn.0253-2670. 2019.01.028.
叶鹏,李显煌,陈杰,瞿绍宏,董章宏,雷瀚,刘成,辛培尧,杨生超. 2019. 金铁锁转录组 SSR 的分布及其序列特征[J]. 云南农业大学学报(自然科学版),34(5):808-814. [Ye P,Li X H,Chen J,Qu S H,Dong Z H,Lei H,Liu C,Xin P Y,Yang S C. 2019. The distribution and characteristics of SSR in transcriptome of Psammosilene tunicoides[J]. Journal of Yunnan Agricultural University(Natural Science),34(5):808-814.] doi:10.12101/j.issn.1004-390X(n).201811088.
战晴晴,隋春,魏建和,范圣此,张杰. 2010. 利用ISSR和SSR分子标记构建北柴胡遗传图谱[J]. 药学学报,45(4):517-523. [Zhan Q Q,Sui C,Wei J H,Fan S C,Zhang J. 2010. Construction of genetic linkage map of Bupleurum chinense DC. using ISSR and SSR markers[J]. Acta Pharmaceutica Sinica,45(4):517-523.] doi:10.16438/j.0513-4870.2010.04.001.CBA22FE2-C5D6-49B5-95BB-86A3709C1060
朱楚然,徐娇,都明理,王丽红,隋春,魏建和. 2019. 三岛柴胡基因组Survey分析及SSR位点挖掘[J]. 中国中药杂志,44(18):3960-3966. [Zhu C R,Xu J,Du M L,Wang L H,Sui C,Wei J H. 2019. Genome survey analysis and SSR loci mining of Bupleurum falcatum[J]. China Journal of Chinese Materia Medica,44(18):3960-3966.] doi:10.19540/j.cnki.cjcmm.20190730.103.
Dreisigacker S,Zhang P,Warburton M L,Van Ginkel M,Hoisington D,Bohn M,Melchinger A E. 2004. SSR and pedigree analyses of genetic diversity among CIMMYT wheat lines targeted to different mega environments[J]. Crop Science,44(2):381-388. doi:10.2135/cropsci2004. 0381.
He G,Meng R,Newman M,Gao G,Pittman R N,Prakash C S. 2003. Microsatellites as DNA markers in cultivated peanut(Arachis hypogaea L.)[J]. BMC Plant Biology,3(1):1-6. doi:10.1186/1471-2229-3-3.
Liu J Q,Wang Y,Liu J N,Liu X,He R L. 2021. A comparative study on different multiple cropping patterns of Bupleurum chinense[J]. Journal of Chinese Pharmaceutical Sciences,30(2):169. doi:10.5246/jcps.2021.02.014.
Pan S L. 2006. Bupleurum species:Scientific evaluation and clinical applications[M]. London:London Taylor & Francis Group.
Sui C,Wei J H,Chen S L,Chen H Q,Dong L M,Yang C M. 2010. Construction of a full-length enriched cDNA library and analysis of 3111 ESTs from roots of Bupleurum chinense DC[J]. Botanical Studies,51(1):7-16. doi:10.1111/j.1095-8339.2009.01020.x.
Sun L D,Yang W R,Zhang Q X,Cheng T R,Pan H T,Xu Z D,Zhang J,Chen C G. 2013. Genome-wide characterization and linkage mapping of simple sequence repeats in mei(Prunus mume Sieb. et Zucc.)[J]. PLoS One,8(3):e59562. doi:10.1371/journal.pone.0059562.
Temnykh S,Declerck G,Lukashova A. 2001. Computational and experimental analysis of microsatellites in rice (Oryza sativa L.):Frequency,length variation,transposon associations,and genetic marker potential[J]. Genome Research,11(8):1441-1452. doi:10.1101/gr.184001.
Wang L M,Jiao S J,Jiang Y X,Yan H D,Su D F,Sun G Q,Yan X F,Sun L F. 2013. Genetic diversity in parent lines of sweet sorghum based on agronomical traits and SSR markers[J]. Field Crops Research,149:11-19. doi:org/10. 1016/j.fcr.2013.04.013.
Xiang X,Zhang Z,Wang Z,Zhang X,Wu G. 2015. Transcriptome sequencing and development of EST-SSR markers in Pinus dabeshanensis,an endangered conifer endemic to China[J]. Molecular Breeding,35:158. doi:10.1007/s11032-015-0351-0.
(責任编辑 陈 燕)CBA22FE2-C5D6-49B5-95BB-86A3709C1060

