不同桑树品种响应干旱胁迫的比较转录组学分析
曾睿 任迎虹 祁伟亮 黄仁维 李恋龙 李鑫鑫 王飞 赵比黑
摘要:【目的】發掘响应干旱胁迫的关键抗旱基因,从转录水平上揭示桑树的抗旱分子机制,为后续开展桑树分子抗旱性育种工作提供科学依据。【方法】以干旱敏感性品种德果1号和耐旱性品种湖桑32号为研究对象,通过盆栽种植方式开展干旱胁迫及复水处理试验,采集24个桑叶样本提取总RNA后构建cDNA文库,在Illumina HiSeqTM 4000测序平台上进行高通量测序,并结合生物信息学对相关基因进行注释分析。【结果】经转录组测序,各样本的Clean reads范围为45723096~67280168条,有效碱基数(Clean bases)集中在6.86~10.09 Gb,GC含量在44.84%~46.48%(平均为45.61%),Q30在90.36%~93.07%。差异表达基因(DEGs)筛选结果显示,6个差异分组(A2 vs A1,B2 vs B1,A3 vs A1,B3 vs B1,A4 vs A1,B4 vs B1)分别筛选出3510、3399、5677、5507、5124和2734个差异表达基因;经干旱胁迫处理后,桑叶功能组基因中呈下调表达的差异表达基因明显多于呈上调表达的差异表达基因,说明桑树在生长过程中存在不同的功能基因以控制桑叶生长发育。不同抗旱性桑叶转录组测序数据中以涉及生物学过程的差异表达基因最多,且主要集中在小分子代谢过程(Small molecule metabolic process)、跨膜运输(Transmembrane transport)及碳水化合物代谢过程(Carbohydrate metabolic process)等方面;与分子功能相关的差异表达基因次之,主要涉及转移酶活性(Transferase activity)和水解酶(Hydrolase activity)等。干旱胁迫下不同抗旱性桑叶差异表达基因主要富集到59条KEGG信号通路上,可划分为代谢、遗传信息处理、环境信息处理、细胞过程和生物系统五大信号通路;其中,抗旱性桑树品种通过提高能量代谢、碳水化合物代谢、增强光合作用及脂质代谢来更好地适应干旱胁迫。综合GO功能注释分析和KEGG信号通路富集分析,得到以下可能与干旱相关的基因:LOC21410404、LOC21404884、LOC21409623、LOC21401352、LOC21398977、LOC21388561、LOC21401447、LOC21398764、LOC21385254、LOC21384661、LOC-21410404及LOC21408971。【结论】不同桑树品种的耐旱性存在显著差异,其中抗旱性桑树品种是通过提高能量代谢、碳水化合物代谢、脂质代谢及光合作用共同应对干旱胁迫。
关键词:桑树;干旱胁迫;抗旱性;转录组;差异表达基因
中图分类号: S888.2 文献标志码: A 文章编号:2095-1191(2022)03-0684-09
Comparative transcriptome analysis of drought stress responses in mulberries of differing drought resistances
ZENG Rui, REN Ying-hong QI Wei-liang, HUANG Ren-wei, LI Lian-long,
LI Xin-xin, WANG Fei, ZHAO Bi-hei
(College of Chemistry and Life Science, Chengdu Normal University, Chengdu, Sichuan 611130, China)
Abstract:【Objective】To identify drought-resistance genes operating in mulberry under drought stress and reveal the molecular mechanism of drought resistance in mulberry at the transcriptional level, so as to provide a theoretical basis to support the breeding for and the selection of drought-resistance in mulberry. 【Method】The drought-sensitive mulberry variety of Deguo 1 and drought-tolerant mulberry variety Husang 32 were used as the research materials. The drought stress and the control group of rehydration treatment were carried out through pot cultivation. Total RNA was extracted from 24 mulberry leaf samples, cDNA libraries was constructed, and then subject to high-throughput sequencing on the Illumina HiSeqTM 4000 platform. Drought-related genes were annotated and analyzed in combination with bioinformatics. 【Result】The results showed that clean reads ranged from 45723096 to 67280168, with clean bases concentrated in 6.86 Gb to 10.09 Gb, with a GC content of 44.84%-46.48% (average 45.61%) and Q30 scores of 90.36%-93.07%. The number of differentially expressed genes (DEGs) between samples (A2 vs A1, B2 vs B1, A3 vs A1, B3 vs B1, A4 vs A1, B4 vs B1) were:3510,3399,5677,5507,5124 and 2734, respectively. There were a smaller number of up-regulated genes than down-regulated genes after drought stress treatment of mulberry. DEGs between the mulberries of differing drought resistance were most enriched in biological processes, consisting mainly of small molecule metabolic process, transmembrane transport and carbohydrate metabolic process. DEGs involved in molecular function were the second most enriched, consisting mainly of transferase activity and hydrolase activity. DEGs were enriched in 59 KEGG signaling pathways, which can be divided into five categories: metabolism, genetic information processing, environmental information processing, cellular processes and biological systems. Drought-resistant mulberry varieties could better adapt to drought stress by their increased expression of genes involved in energy metabolism, carbohydrate metabolism, photosynthesis and lipid meta-bolism. GO functional annotation and KEGG signal pathway enrichment analyses were combined to obtain the drought-resistance genes:LOC21410404,LOC21404884,LOC21409623,LOC21401352,LOC21398977,LOC21388561, LOC21401447, LOC21398764, LOC21385254, LOC21384661, LOC21410404 and LOC21408971. 【Conclusion】 The higher drought tolerance of the mulberry variety Husang 32 relative to the variety Deguo 1 involved significant differential expression of genes under drought stress related with enhancements in energy metabolism, carbohydrate metabolism, photosynthesis and lipid metabolism.D469D8A7-8DA9-4B0F-AA21-212FF56397DA
Key words: mulberry (Morus alba L.); drought stress; drought-resistant; transcriptome; differentially expressed genes (DEGs)
Foundation items: Sichuan Science and Technology Project (2018JY0442); Sichuan Higher Education Talent Cultivation Quality and Education Reform Project (JG2018-888,JG2018-885); The University-level Innovation Project of Chengdu Normal University (CSCXTD2020A04)
0 引言
【研究意义】桑树(Morus alba L.)隶属于桑科(Moraceae)桑属(Morus),为典型的落叶型多年生深根性木本植物(杜伟等,2016,2017;刘丹等,2020)。桑树的深根性决定其水分主要借助于自身强大的根群系统,一旦遇到水分缺乏则会严重影响桑树的生长发育,尤其是在气温相对较高夏秋季。攀西地区是我国重要的蚕桑生产基地,蚕桑产业发展对促进当地农民增收及水土保持具有重要意义。但受制于攀西地区的特殊地理位置,常出现大规模、持续性的干旱,给桑树的生长发育带来严重威胁,进而制约着蚕桑产业的可持续发展(Liu et al.,2019c)。因此,深入全面开展桑树干旱胁迫机理研究,不仅有利于促进桑树的育种创新,对保障我国蚕桑产业的健康发展也具有重要意义。【前人研究进展】大多数植物在遇到干旱或水分不足的情況下均会借助复杂的生理代谢和细胞过程以确保及维系自身生长发育(李捷等,2019;林艳华等,2019;徐澜等,2020),其中存在大量的细胞基因转录,因此,基于转录组学从整体角度对植物细胞内基因转录及基因调控进行系统研究,对深入了解干旱胁迫下的分子机制及挖掘抗旱基因具有重要意义,进而为培育高抗逆性品种提供理论依据(Bokonon-Ganta et al.,2013)。已有研究针对马铃薯(Zhang et al.,2014)、小麦(Liu et al.,2015;Kumar et al.,2018)、油菜(Dong et al.,2017)、花生(Zhao et al.,2018)及玉米(姚启伦等,2021)等作物在干旱胁迫下的转录组学进行深入分析,鉴定出相应的干旱胁迫应答基因,并证实这些差异表达基因主要富集在氧化磷酸化、光合作用和植物代谢途径中,但有关转录组学在桑树抗旱方面的研究报道较少。Wei等(2014)基于川桑基因组数据库的全基因组分析克隆了10个MnMAPK基因,结果表明MnMAPK基因可能参与桑树的缺水胁迫途径,但未发现干旱胁迫应答的调控途径;周宏(2017)以桑树育71-1品种为材料,初步明确了桑树trihelix、bZIP、MYB和ERF转录因子家族成员在干旱胁迫中的表达变化规律;Li等(2017)通过高通量测序技术研究桑树抗旱的关联miRNA及其靶基因,证实miR156、miR172和miR39家族靶基因在干旱胁迫下发挥重要作用。此外,有研究发现MRD22基因(Wang et al.,2014)、EIL3基因(Liu et al.,2019a)、PPO1基因(Liu et al.,2019b)等在桑树干旱胁迫下的表达量有所改变。【本研究切入点】随着川桑全基因组序列的公布,越来越多学者将研究重点转移到桑树抗性基因,有关桑树耐旱分子机制的研究已有较多报道(刘丹等,2020),但鲜见基于转录组测序技术对干旱胁迫下桑树基因的表达水平进行动态跟踪分析。【拟解决的关键问题】基于高通量测序对桑树干旱胁迫及复水处理阶段的基因表达模式进行研究,发掘响应干旱胁迫的关键抗旱基因,并结合生物信息学对相关基因进行注释分析,从转录水平揭示桑树抗旱的分子机制,为后续开展桑树分子抗旱性育种工作提供科学依据。
1 材料与方法
1. 1 试验材料
供试桑树品种[干旱敏感性品种德果1号(GS),耐旱性品种湖桑32号(HS)]由成都师范学院化学与生命科学学院提供。试验试剂主要有TRIzol试剂盒(美国Invitrogen公司)及TruSeq RNA Sample Prep Kit v2(美国Illumina公司)。主要设备仪器:Agilent 2100生物分析仪(G2939AA),Illumina HiSeqTM 4000测序仪,Eppendorf Centrifuge 5418离心机。
1. 2 试验设计及取样
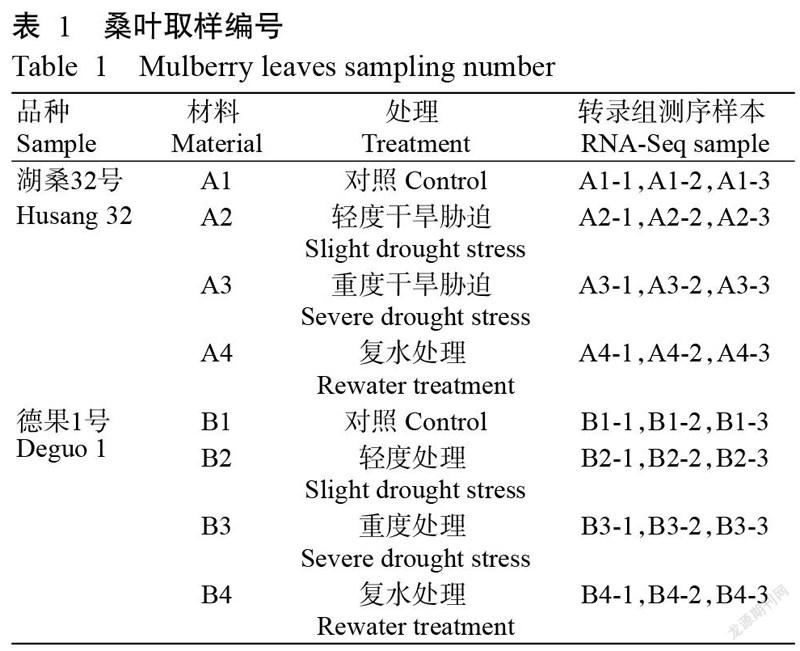
采用10 L营养土塑料盆栽种植方式,每盆种植4株桑树幼苗,于出苗后选取长势一致的德果1号和湖桑32号各24盆(株高约45 cm)。试验设4个处理(表1),每处理重复6次,水分处理分别为对照处理(CK)(正常供水85%)、轻度干旱胁迫(土壤田间含水量为65%~75%)、重度干旱胁迫(土壤田间含水量为35%~40%)和复水处理。采用烘干称重法测定含水量,每天晚上20:00取土样,当含水量达胁迫处理要求后,于次日上午8:00取植株树冠同方位、同叶位的桑叶,经液氮速冻后置于-80 ℃超低温冰箱中保存备用。取桑叶样品进行总RNA提取,重复3次。
1. 3 RNA提取与cDNA文库构建
所有桑叶样品采用TRIzol试剂盒提取总RNA,具体过程参照其说明进行操作。根据构建cDNA文库的要求进行RNA样品纯度及质量检测,并精准定量检测RNA浓度及其完整性。检测合格的RNA样品使用Oligo(dT)磁珠对mRNA进行富集,经Fragmentation缓冲液随机打断后,以此为模板借助六核苷酸随机引物进行反转录,合成双链cDNA,并在DNA聚合酶I体系下以dNTPs为原料合成cDNA第二链。使用试剂盒纯化双链cDNA,经末端修复和加poly(A)尾后连接测序接头,采用AMPure XP Beads进行片段筛选(250~300 bp),通过PCR扩增富集构建cDNA文库。cDNA文库质量检测合格后,在Illumina HiSeqTM 4000测序平台上开展高通量测序工作。D469D8A7-8DA9-4B0F-AA21-212FF56397DA
1. 4 数据分析
原始序列(Raw reads)数据提交至NCBI序列读取归档数据库,根据比对效率(Mapped ratio)及有效序列(Clean reads)在参考基因组上分布情况进行质量审查;以确认合格的Clean reads为生物信息学分析样品,进行新基因预测发掘及表达差异分析,基于GO和KEGG等数据库进行差异表达基因(Diffe-rential expressed genes,DEGs)功能注释分析,并以Origin 2018制图。
2 结果与分析
2. 1 转录组测序质量
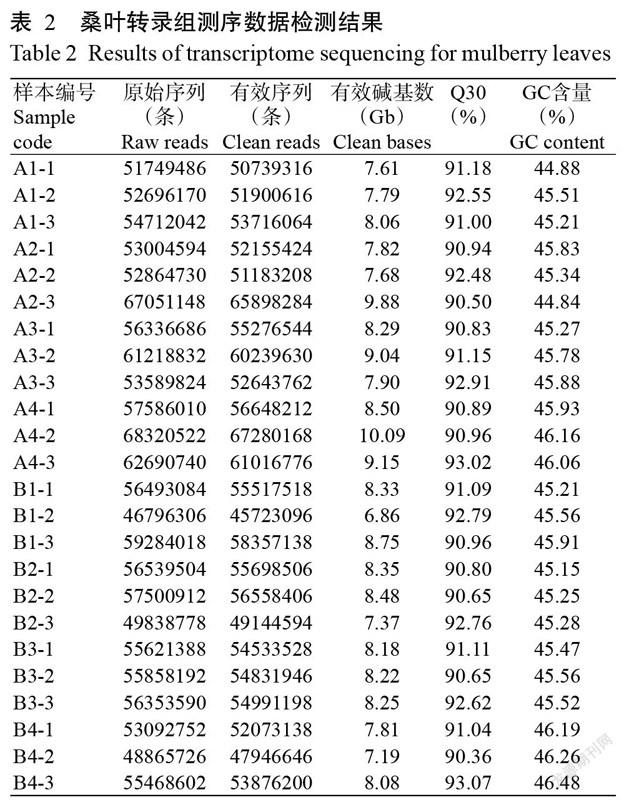
以德果1号和湖桑32号的桑叶组织为基础,选取18个干旱处理样本及6个对照样本的RNA,共同构建cDNA文库。对24个样本的转录组测序数据进行统计分析,结果显示各样本的Clean reads数量为45723096~67280168条,有效碱基数(Clean bases)集中在6.86~10.09 Gb,GC含量在44.84%~46.48%(平均45.61%),Q30在90.36%~93.07%。Clean reads与桑叶参考基因组样本的对比结果显示,二者的Mapped ratio超过75.00%,即转录组测序结果可靠,能满足后续研究的要求。
2. 2 干旱胁迫下不同抗旱性桑叶差异表达基因的表达情况
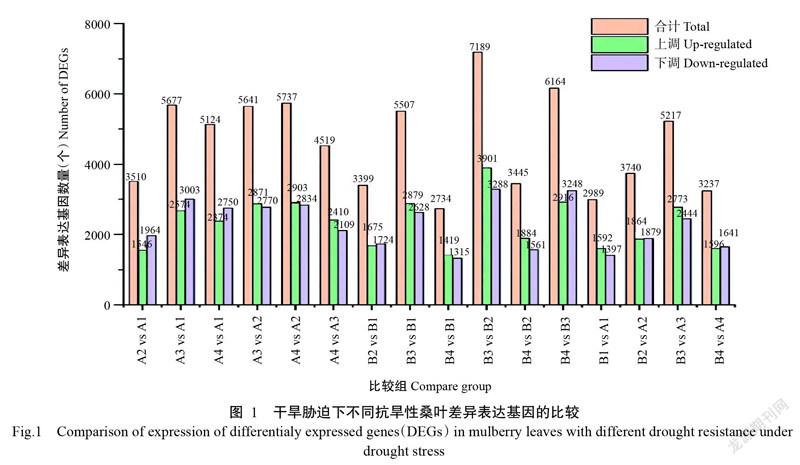
不同抗旱性桑叶响应干旱胁迫的差异表达基因表达模式见图1。与CK相比,德果1号在轻度干旱胁迫下的差异表达基因共有3399个,其中上调基因1675个、下调基因1724个;在重度干旱胁迫下的差异表达基因共有5507个,其中上调基因2879个、下调基因2628个;复水处理后共產生2734个差异表达基因,其中上调基因1419个、下调基因1315个。与CK相比,湖桑32号在轻度干旱胁迫下的差异表达基因共有3510个,其中上调基因1546个、下调基因1964个;重度干旱胁迫下的差异表达基因共有5677个,其中上调基因2674个、下调基因3003个;复水后共产生5124个差异表达基因,其中上调基因2374个、下调基因2750个。德果1号和湖桑32号在不同干旱胁迫下的基因表达水平存在明显差异,轻度干旱胁迫与重度干旱胁迫相比,2个品种桑叶的上调基因和下调基因数量均呈上升趋势,德果1号的上升幅度分别为41.82%和34.40%,湖桑32号对应的上升幅度分别是42.18%和34.60%;重度干旱胁迫与复水相比,2个品种桑叶的上调基因和下调基因数量则呈明显的下降趋势,德果1号的下降幅度分别为50.71%和49.94%,湖桑32号对应的下降幅度分别是11.22%和8.42%。经干旱胁迫处理后,桑叶功能组基因中呈下调表达的差异表达基因明显多于呈上调表达的差异表达基因,说明桑树在生长过程中存在不同的功能基因以控制桑叶生长发育。所有桑树幼苗在干旱环境下均呈现出萎蔫、发育迟缓等症状,以此应对水分的缺乏。
2. 3 桑叶差异表达基因的GO功能注释分析结果
GO功能注释分析在于表现样本体内所有差异表达基因及其产物的表达情况和属性,能真实反映6个比较组的生物学过程(Biological process)、分子功能(Molecular function)及细胞组分(Cellular component)三大功能。由图2可知,A2 vs A1注释到的差异表达基因共2141个,涉及生物学过程、分子功能、细胞组分的差异表达基因分别为1074个(占50.16%)、828个(占38.67%)和239个(占11.16%);A3 vs A1注释到的差异表达基因共3191个,涉及生物学过程、分子功能、细胞组分的差异表达基因分别为1665个(占52.18%)、1146个(占35.91%)和380个(占11.91%);A4 vs A1注释到的差异表达基因共3088个,涉及生物学过程、分子功能、细胞组分的差异表达基因分别为1554个(占50.32%)、977个(占31.64%)和557(占18.04%);B2 vs B1注释到的差异表达基因共1836个,涉及生物学过程、分子功能、细胞组分的差异表达基因分别为938个(占51.09%)、583个(占31.75%)和315个(占17.16%);B3 vs B1注释到的差异表达基因共2892个,涉及生物学过程、分子功能、细胞组分的差异表达基因分别为1419个(占49.07%)、1034个(占35.75%)和439个(占15.18%);B4 vs B1注释到的差异表达基因共1523个,涉及生物学过程、分子功能、细胞组分的差异表达基因分别为788个(占51.74%)、519个(占34.08%)和216个(占14.18%)。可见,不同抗旱性桑叶转录组测序数据中以涉及生物学过程的差异表达基因最多,且主要集中在小分子代谢过程(Small molecule metabolic process)、跨膜运输(Transmembrane transport)及碳水化合物代谢过程(Carbohydrate metabolic process)等方面;与分子功能相关的差异表达基因次之,主要涉及转移酶活性(Transferase activity)和水解酶(Hydrolase activity)等。
2. 4 桑叶差异表达基因KEGG信号通路富集分析结果
KEGG信号通路富集分析结果(表3)表明,不同抗旱性桑叶差异表达基因均富集到新陈代谢(Metabolism)、遗传信息处理(Genetic information processing)、环境信息处理(Environmental information processing)、细胞过程(Cellular processes)和生物系统(Bidogcial system)五大信号通路上,主要涉及到碳水化合物代谢、运输和分解代谢、其他氨基酸代谢、次生代谢产物生物合成、能量代谢、糖的生物合成与代谢、脂质代谢、辅助因子和维生素代谢、萜类和聚酮类化合物代谢、核苷酸代谢、环境适应、转录及翻译等。D469D8A7-8DA9-4B0F-AA21-212FF56397DA
干旱胁迫下不同抗旱性桑叶DEGs主要富集到59条KEGG信号通路上(图3),表明干旱胁迫对桑叶的淀粉和蔗糖代谢(Starch and sucrose metabolism)、环境适应(Environmental adaptation)、脂肪酸降解(Fatty acid degradation)、碳代谢(Carbon metabolism)、半乳糖代谢(Galactose metabolism)、α-亚麻酸代谢(Alpha-linolenic acid metabolism)、糖酵解/糖异生(Glycolysis/gluconeogenesis)、亚油酸代谢(Linoleic acid metabolism)、甘油脂代谢(Glycerolipid metabolism)、脂肪酸代谢(Fatty acid metabolism)、β-丙氨酸代谢(Beta-alanine metabolism)、磷酸戊糖途径(Pentose phosphate pathway)、抗坏血酸和藻酸盐代谢(Ascorbate and aldarate metabolism)、缬氨酸/亮氨酸/异亮氨酸降解(Valine/leucine/isoleucine degradation)、果糖和甘露糖代谢(Fructose and mannose metabolism)、丙酮酸代谢(Pyruvate metabolism)、不饱和脂肪酸生物合成(Biosynthesis of unsaturated fatty acids)、核糖体(Ribosome)、光合作用(Photosynthesis)、光合生物中的碳固定(Carbon fixation in photosynthetic organisms)、植物激素信号转导(Plant hormone signal transduction)、卟啉和叶绿素代谢(Porphyrin and chlorophyll metabolism)、乙醛酸和二元羧酸盐代谢(Glyoxylic acid and dicarboxylate metabolism)等有明显影响。与CK相比,轻度干旱胁迫下参与淀粉和蔗糖代谢的差异表达基因有36个,重度干旱胁迫下参与半乳糖代谢的差异表达基因有24个、参与糖酵解/糖异生的差异表达基因有39个、参与亚油酸代谢的差异表达基因有9个、参与甘油脂代谢的差异表达基因有26个、参与脂肪酸代谢的差异表达基因有27个、参与缬氨酸/亮氨酸/异亮氨酸降解的差异表达基因有20个、参与果糖和甘露糖代谢的差异表达基因有19个、参与丙酮酸代谢的差异表达基因有28个、参与不饱和脂肪酸生物合成的差异表达基因有10个。从差异表达基因在KEGG信号通路上的富集情况来看,抗旱性桑树品种通过提高能量代谢、碳水化合物代谢、增强光合作用及脂质代谢来更好地适应干旱胁迫,在逆境中保持存活。综合GO功能注释分析和KEGG信号通路富集分析,得到以下可能与干旱相关的基因:LOC21410404、LOC21404884、LOC21409623、LOC21401352、LOC 21398977、LOC21388561、LOC21401447、LOC2139 8764、LOC21385254、LOC21384661、LOC21410404及LOC21408971。
3 讨论
桑树的生物学遗传背景相当复杂,但其分子学研究基础还很薄弱。植物对干旱胁迫的响应涉及生理、生物化学和分子变化等复杂的生物学过程,桑树根系发达,能在干旱的贫瘠环境下生长,究其原因是桑树能借助次生代谢和光合作用强化、能量代谢水平提升、植物激素信号转换等有效应对干旱,但在实际种植过程中发现不同桑树品种在抗旱机制上存在明显差异。为此,本研究基于高通量测序对桑树干旱胁迫及复水处理阶段的基因表达模式进行分析,结果从6个差异分组(A2 vs A1,B2 vs B1,A3 vs A1,B3 vsB1,A4vsA1,B4 vs B1)分别筛选出3510、3399、5677、5507、5124和2734个差异表达基因,经GO功能注释分析后得到的差异表达基因分别为2141、3191、3088、1836、2892和1523个。此外,经干旱胁迫处理的绝大多数桑叶功能基因呈下調表达趋势,只有少数基因呈上调表达趋势。说明桑树在生长过程中存在不同的功能基因以控制桑叶生长发育。巩檑等(2015)研究证实,基因表达上调是叶片对水分刺激的响应,基因表达下调则是对干旱胁迫的响应。无论是动物还是植物,其新陈代谢最核心的特征就是能量代谢,以植物为例主要集中在光合作用、氧化磷酸化及碳固定、氮代谢等方面。受干旱胁迫的影响,绝大多数植物的光合速率呈下降趋势,进而抑制光合作用和叶绿素生成。如干旱胁迫下杨树叶片内涉及PS I和PS II、电子传递链基因表达水平出现不同程度的下调,而导致三羧酸循环明显降低(Jia et al.,2020)。本研究也发现桑叶在干旱胁迫环境下其光合作用及光合—触角蛋白代谢通路呈现明显的富集现象,表明光合器官受到损害,光合速率及对应的产物产出受到抑制,与李慧娟等(2019)对小麦干旱胁迫的研究结论相似。
植物的生长发育过程离不开以碳水化合物代谢为首的生化过程。本研究结果表明,测序筛选出的桑叶差异表达基因以参与碳水化合物代谢为主,其中又以涉及淀粉和蔗糖代谢的差异表达基因下调表达为主。究其原因是桑叶在干旱环境下将淀粉降解为葡萄糖以调控植物渗透调节,更好地应对干旱胁迫(Liu et al.,2016)。脂质是生物膜的重要成分,植物质膜脂肪酸主要由棕榈酸、硬脂酸、亚油酸和亚麻酸组成,而膜脂组成成分的变化能反映植物对环境胁迫的响应程度。已有研究表明,干旱胁迫会导致马铃薯叶片中细胞膜和细胞壁的稳定强化基因上调表达,包括纤维素合成酶、果胶泛酸酶及脂质结合蛋白等编码基因(Zhang et al.,2014)。本研究发现,干旱胁迫会影响桑叶中与脂肪酸降解和α-亚麻酸代谢相关的基因表达,通过保护细胞膜的流动性以缓解应对干旱胁迫,与李东等(2018)的研究结果一致,即α-亚麻酸能通过提高种子萌发过程中的α-淀粉酶活性来缓解干旱胁迫。由于作物品种及胁迫方式的不同,基因所调节的途径也各不相同。赵雅杰等(2022)研究发现,向日葵在干旱胁迫下其植物激素信号转导、蔗糖与淀粉代谢、氨基酸代谢有明显的富集现象。万丽云等(2018)对花生苗期进行干旱胁迫处理,KEGG信号通路富集分析发现花生是通过其发达根系系统、能量代谢、次生代谢和生长抑制等4个方面共同应对干旱胁迫。可见,在逆境胁迫下植物会通过基因调控作用以改变相关的代谢途径。D469D8A7-8DA9-4B0F-AA21-212FF56397DA
4 结论
不同桑树品种的抗旱性存在明显差异,其中抗旱性桑树品种是通过提高能量代谢、碳水化合物代谢、脂质代谢及光合作用共同应对干旱胁迫。
参考文献:
杜伟,陈松,刘永辉,杨建设,冉瑞法,储一宁. 20116. 13个桑树品种的农艺性状及抗逆性比较[J]. 南方农业学报,47(12):2157-2162. [Du W,Chen S,Liu Y H,Yang J S,Ran R F,Chu Y N. 2016. Comparison of agronomic characters and stress resistance in 13 mulberry varieties[J]. Journal of Southern Agriculture,47(12):2157-2162.] doi:10.3969/j:issn.2095-1191.2016.12.2157.
杜伟,杨文,吴克军,李镇刚,陈松,储一宁. 2017. 10份云南特异野生桑树种质资源的搜集与评价[J]. 南方农业学报,48(8):1504-1510. [Du W,Yang W,Wu K J,Li Z G,Chen S,Chu Y N. 2017. Collection and evaluation of ten peculiar wild mulberry resources in Yunnan[J]. Journal of Southern Agriculture,48(8):1504-1510.] doi:10. 3969/j.issn.2095-1191.2017.08.28.
巩檑,张丽,聂峰杰,陈虞超,甘晓燕,石磊,宋玉霞,郭志乾,王峰. 2015. 旱胁迫和复水处理后马铃薯转录因子的转录组分析[J]. 分子植物育种,13(8):1745-1756. [Gong L,Zhang L,Nie F J,Chen Y C,Gan X Y,Shi L,Song Y X,Guo Z Q,Wang F. 2015. Transcriptome analysis on transcription factors of potato(Solanum tuberosum)under drought stress and rehydration treatment[J]. Molecular Plant Breeding,13(8):1745-1756.] doi:10.13271/j.mpb. 013.001745.
李东,甄春燕,陈德富,陈喜文. 2018. α-亚麻酸对干旱胁迫下水稻种子萌发的影响[J]. 广西植物,38(8):1025-1031.[Li D,Zhen C Y,Chen D F,Chen X W. 2018. Effects of α-linolenic acid on germination of rice seeds under drought stress[J]. Guihaia,38(8):1025-1031.] doi:10. 11931/guihaia.gxzw201709004.
李慧娟,杨阳,常平,郑甲成,陈亮,胡银岗. 2019. 干旱胁迫下过表达TaER小麦的转录组及光合特性分析[J]. 麦类作物学报,39(8):941-949. [Li H J,Yang Y,Chang P,Zheng J C,Chen L,Hu Y G. 2019. Transcriptome and photosynthetic trait analysis of TaER over-expression wheat under drought stress[J]. Journal of Triticeae Crops,39(8):941-949.] doi:10.7606/j.issn.1009-1041.2019.08.08.
李捷,崔永涛,柏延文,王立恒,杨永义,张晓玮,朱珠. 2019. 两种枸杞对干旱胁迫的生理响应及抗旱性評价[J]. 甘肃农业大学学报,54(5):79-87. [Li J,Cui Y T,Bai Y W,Wang L H,Yang Y Y,Zhang X W,Zhu Z. 2019. Physiological response and drought resistance evaluation of two kinds wolfberries on drought stress[J]. Journal of Gansu Agricultural University,54(5):79-87.] doi:10. 13432/j.cnki.jgsau.2019.05.010.
林艳华,梁千慧,刘锦春. 2019. 喀斯特地区适生树种复羽叶栾树幼苗对干旱胁迫下异质生境的生长和光合响应[J]. 西南大学学报(自然科学版),41(8):20-26. [Lin Y H,Liang Q H,Liu J C. 2019. Response of growth and photosynthesis of Koelreuteria bipinnata,an adaptive tree species to karst regions,to the heterogeneous habitat under water stress[J]. Journal of Southwest University (Natural Science),41(8):20-26.] doi:10.13718/j.cnki.xdzk. 2019.08.004.
刘丹,向仲怀,邱长玉,林强. 2020. 桑树耐旱分子机制的研究进展与展望[J]. 蚕业科学,46(3):367-371. [Liu D,Xiang Z H,Qiu C Y,Lin Q. 2020. Research progress and prospect on molecular mechanism of mulberry resistance to drought[J]. Acta Sericologica Sinica,46(3):367-371.] doi:10.13441/j.cnki.cykx.2020.03.013.D469D8A7-8DA9-4B0F-AA21-212FF56397DA
万丽云,苏威,李蓓,雷永,晏立英,康彥平,淮东欣,陈玉宁,姜慧芳,廖伯寿. 2018. 花生苗期干旱处理后转录和代谢通路分析[J]. 中国油料作物学报,40(3):335-343. [Wan L Y,Su W,Li B,Lei Y,Yan L Y,Kang Y P,Huai D X,Chen Y N,Jiang H F,Liao B S. 2018. Molecular analysis of formation of drought tolerance traits in peanut[J]. Chinese Journal of Oil Crop Sciences,40(3):335-343.] doi:10.7505/j.issn.1007-9084.2018.03.004.
徐澜,刘艳超,安伟,高志强. 2020. 冬麦春播小麦对苗期干旱胁迫的生理响应[J]. 甘肃农业大学学报,55(6):40-47. [Xu L,Liu Y C,An W,Gao Z Q. 2020. Physiological response of winter wheat sown in spring to drought stress at seedling stage[J]. Journal of Gansu Agricultural University,55(6):40-47.] doi:10.13432/j.cnki.jgsau.2020. 06.006.
姚启伦,霍仕平,张俊军. 2021. 玉米自交系响应高温、干旱胁迫的关键基因及通路[J]. 江苏农业学报,37(1):29-37. [Yao Q L,Huo S P,Zhang J J. 2021. Key genes and pathways of maize inbred lines responding to heat and drought stress[J]. Jiangsu Journal of Agricultural Sciences,37(1):29-37.] doi:10.3969/j.issn.1000-4440.2021.01. 004.
赵雅杰,包海柱,胡树平,赵轩微,田振东,石丰源,覃一敏,高聚林. 2022. 基于转录组测序的向日葵对干旱胁迫及复水差异表达分析[J]. 分子植物育种,20(1):38-48. [Zhao Y J,Bao H Z,Hu S P,Zhao X W,Tian Z D,Shi F Y,Qin Y M,Gao J L. 2022. Analysis of differential expression of sunflower to drought stress and rewatering based on transcriptome sequencing[J]. Molecular Plant Breeding,20(1):38-48.] doi:10.13271/j.mpb.020.000038.
周宏. 2017. 桑树抗旱相关4个转录因子家族鉴定与表达分析[D]. 镇江:江苏科技大学. [Zhou H. 2017. Identification and expression analysis of drought-resistant related 4 transcription factor families in mulberry(Morus L.)[D]. Zhenjiang:Jiangsu University of Science and Technology.]
Bokonon-Ganta A H,McQuate G T,Messing R H,B Jang E. 2013. Release and establishment of the parasitoid Diachasmimorpha kraussii against the tephritid fruit fly Bactrocera latifrons in Hawaii[J]. Journal of Insect Science,13:7. doi:10.1673/031.013.0701.
Dong B,Wu B,Hong W H,Li X P,Li Z,Xue L,Huang Y F. 2017. Transcriptome analysis of the tea oil camellia (Camellia oleifera) reveals candidate drought stress genes[J]. PLoS One,12(7):e0181835. doi:10.1371/journal.pone.0181835.
Jia H X,Wang L J,Li J B,Sun P,Lu M Z,Hu J J. 2020. Comparative metabolomics analysis reveals different me-tabolic responses to drought in tolerant and susceptible poplar species[J]. Physiologia Plantarum,168(3):531-546. doi:10.1111/ppl.13036.
Kumar J,Gunapati S,Kianian S F,Singh S P. 2018. Comparative analysis of transcriptome in two wheat genotypes with contrasting levels of drought tolerance[J]. Protoplasma,255(5):1487-1504. doi:10.1007/s00709-018-1237-x.D469D8A7-8DA9-4B0F-AA21-212FF56397DA
Li R X,Chen D D,Wang T C,Wan Y Z,Li R F,Fang R J,Wang Y T,Hu F,Zhou H,Li L,Zhao W G. 2017. High throughput deep degradome sequencing reveals micro-RNAs and their targets in response to drought stress in mulberry(Morus alba)[J]. PLoS One,12(2):e0172883. doi:10.1371/journal.pone.0172883.
Liu C Y,Li J,Zhu P P,Yu J,Hou J M,Wang C H,Long D P,Yu M D,Zhao A C. 2019a. Mulberry EIL3 confers salt and drought tolerances and modulates ethylene biosynthetic gene expression[J]. PeerJ,7:e6391. doi:10.7717/peerj.6391.
Liu D,Meng S,Xiang Z H,He N J,Yang G W. 2019b. Antimicrobial mechanism of reaction products of Morus notabilis (mulberry) polyphenol oxidases and chlorogenic acid[J]. Phytochemistry,163:1-10. doi:10.1016/j.phytochem. 2019.03.026.
Liu S C,Jin J Q,Ma J Q,Yao M Z,Ma C L,Li C F,Ding Z T,Chen L. 2016. Transcriptomic analysis of tea plant responding to drought stress and recovery[J]. PLoS One,11(1):e0147306. doi:10.1371/journal.pone.0147306.
Liu Y,Ji D F,Turgeon R,Chen J,Lin T B,Huang J,Luo J,Zhu Y,Zhang C K,Lü Z Q. 2019c. Physiological and proteomic responses of mulberry trees(Morus alba L.) to combined salt and drought stress[J]. International Journal of Molecular Sciences,20(10):2486. doi:10.3390/ij ms20102486.
Liu Z S,Xin M M,Qin J X,Peng H R,Ni Z F,Yao Y Y,Sun Q X. 2015. Temporal transcriptome profiling reveals expression partitioning of homeologous genes contributing to heat and drought acclimation in wheat (Triticum aestivum L.)[J]. BMC Plant Biology,15:152. doi:10.1186/s12870-015-0511-8.
Wang H,Liu Z Y,Li F,Wang Y H,Fang R J,Zhao W G,Li L. 2014. Molecular cloning of a dehydration-responsive protein gene (MRD22) from mulberry,and determination of abiotic stress patterns of MRD22 gene expression[J]. Bioorganicheskaia Khimiia,40(2):203-210.
Wei C J,Liu X Q,Long D P,Guo Q,Fang Y,Bian C K,Zhang D Y,Zeng Q W,Xiang Z H,Zhao A C. 2014. Molecular cloning and expression analysis of mulberry MAPK gene family[J]. Plant Physiology and Biochemistry,77:108-116. doi:10.1016/j.plaphy.2014.02.002.
Zhang N,Liu B L,Ma C Y,Zhang G D,Chang J,Si H J,Wang D. 2014. Transcriptome characterization and sequencing-based identification of drought-responsive genes in potato[J]. Molecular Biology Reports,41(1):505-517. doi:10.1007/s11033-013-2886-7.
Zhao X B,Li C J,Wan S B,Zhang T T,Yan C X,Shan S H. 2018. Transcriptomic analysis and discovery of genes in the response of Arachis hypogaea to drought stress[J]. Molecular Biology Reports,45(2):119-131. doi:10.1007/s11033-018-4145-4.
(責任编辑 兰宗宝)D469D8A7-8DA9-4B0F-AA21-212FF56397DA

