两种蓝细菌中7 对毒素-抗毒素系统的分析和鉴定
林世团, 王晓雪, 陈冉
1. 中国科学院热带海洋生物资源与生态重点实验室, 中国科学院南海海洋研究所, 广东 广州 510301;
2. 中国科学院大学, 北京 100049
蓝细菌(Cyanobacteria), 也被称为蓝藻或蓝绿藻, 属于革兰氏阴性菌(Sinha et al, 2008)。蓝细菌能利用叶绿素、胡萝卜素等光合色素进行光合作用,是地球上一类能进行放氧光合作用的原核生物(Aguilera et al, 2021)。蓝细菌种类丰富, 分布广泛且形态多样, 包含6 个目2000 多个种(Stanier et al,1977)。浮游蓝细菌是海洋食物网的主要生产者, 是全球碳循环和氮循环中不可或缺的角色(Bullerjahn et al, 2014; Tang et al, 2019)。此外, 蓝细菌具有生长速度快、遗传操作简单和生理功能多样等特性, 是研究生物基因型(Do Carmo Bittencourt-Oliveira et al,2012; Gągała et al, 2014; Belykh et al, 2015; Song et al, 2015)、表型(Berrendero et al, 2011; Dojani et al,2014)和生态型(D’Agostino et al, 2014; Chandler et al,2016; Marston et al, 2016; Sohm et al, 2016)之间关系的重要模式生物。
集胞藻PCC6803(Synechocystissp. PCC6803)和聚球藻WH7803(Synechococcussp. WH7803)分别是研究淡水和海水蓝细菌的模式菌株。单细胞淡水集胞藻PCC6803 是首个完成基因组序列测定的蓝细菌(Kaneko et al, 1995, 1996)。基因组大小为3573471bp(base pair), 每个细胞具有12 个基因组拷贝(Labarre et al, 1989), 编码3317 个基因。集胞藻PCC6803 还具有7 个环形质粒, 大小在2.3~119kb 之间(Chauvat et al, 1986; Prindle, 2015)。海水蓝细菌与淡水蓝细菌亲缘关系较远, 主要成员为原绿球藻(Prochlorococcus)和聚球藻两个属。原绿球藻主要分布营养贫乏的温暖海域, 而聚球藻的生态分布更为广泛, 从赤道的温暖海域到次极地低温水域, 从富营养的河口到寡营养的海洋水域。聚球藻是一种超微型单细胞原核藻类, 大小仅为1~2μm(Partensky et al, 1999), 因而直到1979 年才得以在海洋中被发现。目前研究的海洋聚球藻的大多数为单倍体, 仅含有单个染色体。然而海洋聚球藻WH7803 的染色体具有多个拷贝,一般含有4 个染色体拷贝, 基因组大小为2366980bp。
毒素-抗毒素(toxin-antitoxin, TA)系统是原核系统中由相邻的2 个或3 个基因组成的基因元件。其中毒素基因编码毒素蛋白, 起到抑菌或杀菌功能,而抗毒素基因编码蛋白或RNA, 通过直接或间接作用拮抗毒素的毒性(Dorman et al, 2001)。毒素-抗毒素系统具有多种功能, 包括能够调节细胞代谢水平对抗外界的生长压力、参与细胞程序性死亡、参与“持留菌”形成、参与生物被膜形成以及抵御噬菌体侵染等生物过程(Wang et al, 2011)。目前依据抗毒素与毒素作用方式的不同, 将毒素-抗毒素系统分为7 种类型 (Ⅰ型至Ⅶ型)(Wang et al, 2021)。Ⅱ型毒素-抗毒素系统中毒素和抗毒素皆为蛋白质, 通过毒素和抗毒素间的蛋白-蛋白的互作抑制毒素的毒性作用。
毒素-抗毒素系统的系统发育分析表明, 蓝细菌基因组分布着大量的毒素-抗毒素系统(Akarsu et al,2019)。然而目前只有极少数蓝细菌的毒素-抗毒素系统被研究。基于蓝细菌生态位置的重要性和毒素-抗毒素功能的多样性, 我们以淡水蓝细菌集胞藻PCC6803 和海水蓝细菌聚球藻WH7803 作为研究对象, 对其基因组进行了毒素-抗毒素系统预测。并且选取集胞藻PCC6803 的5 对和聚球藻WH7803 的2 对毒素-抗毒素系统进行毒性验证并对其蛋白进行异源表达纯化。实验结果表明, 集胞藻PCC6803 的两对毒素- 抗毒素系统 BAD01932-1933 和BAA18559-New ORF7 的毒素具有明显的细胞毒性,且抗毒素能中和毒素毒性, 与预测结果相符。本文的研究结果将有助于后续对蓝细菌毒素-抗毒素系统及其生态和生物学功能的研究。
1 材料和方法
1.1 实验材料
1.1.1 菌株与培养基
实验所用的克隆模板集胞藻PCC6803 和聚球藻WH7803 基因组DNA 分别由中国科学院水生生物研究所张承才教授和香港科技大学曾庆璐教授馈赠。基因克隆和蛋白表达所用的菌株大肠杆菌(Escherichia coli) K12 BW25113, ER2738 和BL21(DE3) 均为本实验室保存, 培养条件为37℃条件下Luria-Bertani(LB)培养基培养。菌株培养使用的抗生素为羧苄青霉素和硫酸卡那霉素, 工作浓度分别为100μg·mL-1和50μg·mL-1。
1.1.2 主要试剂
DNA 限制性核酸内切酶购于NEB 公司(伊普斯威奇, 美国); 基因片段和载体同源重组使用的非连接酶依赖型单片段快速克隆试剂盒(ClonExpress®Ⅱ One Step Cloning Kit)购自诺唯赞公司(南京, 中国); DNA 凝胶回收试剂盒和质粒提取试剂盒均购买自 OMEGA 公司(诺克罗斯, 美国); DNA 提取试剂盒购于天根公司(北京, 中国)。PCR 引物由天一辉远公司(广州, 中国)合成。蛋白纯化过程中所使用的Ni-NTA 亲和柱料购自 Qiagen 公司(杜塞尔多夫,德国)。
1.2 集胞藻PCC6803 和聚球藻WH7803 基因组TA系统预测分析
集胞藻PCC6803 和聚球藻WH7803 菌株中TA的预测主要结合以下两种策略: (1)下载TADB 数据库 (Altschul, 1997)(https://bioinfo-mml.sjtu.edu.cn/TADB2/index.php)中已知的TA 蛋白序列, 通过本地 BLASTP 检索这两种菌株中和已知 TA 相似的蛋白序列(参数: -evalue 1e-5); (2)两种菌株的全部蛋白序列用InterProScan(version 5.35-74.0)对蛋白结构域(Pfam domain)进行注释, 根据已知 TA 的结构域初步筛选潜在TA。上述两种TA 预测的结果再进一步根据基因上下游“关联犯罪”原则(guilt-byassociation)(Leplae et al, 2011)进行筛选, 对于明确具有毒素或者抗毒素结构域的但是上下游又没有符合条件的基因, 利用NCBI 数据库在线工具ORF finder 对其上下游进行预测分析潜在的新的开放阅读框(open reading frames, ORFs), 再结合结构域注释确定菌株中未注释的新TA。所有备选基因通过Xtalpred 网站(https://xtalpred.godziklab.org/XtalPredcgi/xtal.pl)进行已解析结构的同类型蛋白的检索和比对分析, 根据检索结果确定备选基因类型。
1.3 分子生物学操作
分别选取预测结果中集胞藻PCC6803 的5 对和聚球藻WH7803 的2 对毒素-抗毒素系统进行验证。将这7 对毒素-抗毒素系统的毒素(BAA17887, New ORF7, BAD01872, BAD01932, BAD01997,SynWH7803_1121, SynWH7803_1701) 和抗毒素(BAA17887, BAA18559, BAD01871, BAD01933,BAD01998, SynWH7803_1122, SynWH7803_1700)以及相对应毒素-抗毒素合体的编码区采用附表1 中的引物进行扩增, 使用one-step 方法连接至 pBAD质粒。再将连接产物通过化学转化的方法, 转入感受态大肠杆菌 BW25113 中, 并使用含羧苄青霉素的 LB 平板进行重组菌株筛选。使用 pBAD-f(5′-ATGCCATAGCATTTTTATCCA-3′) 和 pBAD-r(5′-TCT GATTTAATCTGTATCAGG-3′)进行PCR 验证, 选取大小正确的克隆测序确认。用于蛋白表达的重组菌株BL21(DE3)/pET28b(+)的构建过程与上述相同。采用附表1 中的引物pUC19- pro-BAD1932-1933-f(5′-GAAAAAGAAGAATTGATGATGT-3′) 和pUC19-pro-BAD1932-1933-r(5′-CTAAAAGAGTC TTTCCACCGAT-3′)扩增毒素-抗毒素系统 BAD 1932-1933 及其启动子区, 使用one-step 方法连接至pUC19 质粒上。再将连接产物通过化学转化的方法,转入感受态大肠杆菌ER2738 中, 并使用含羧苄青霉素的LB 平板进行重组菌株筛选, 随后使用M13-f和M13-r 通用引物PCR 验证并测序。最后将测序结果正确的重组质粒通过化学转化的方法, 转入感受态大肠杆菌BW25113 中,使用M13-f 和M13-r 进行PCR 验证。

附表1 本文所用引物Tab. S1 Primers used in this study
1.4 重组菌株毒性的检测
将过夜培养的重组菌株菌液用接种环分别划线于含羧苄青霉素和0.3% L-阿拉伯糖的LB 平板, 并同时划线于不含0.3% L-阿拉伯糖, 仅含羧苄青霉素的LB 平板作为对照, 于37℃培养箱中培养过夜,拍照记录结果。
1.5 重组菌株生长曲线及菌落形成单位的测定
pBAD 质粒连接的重组菌株接种于LB 液体培养基培养过夜, 以初始浓度OD600≈0.1 转接到装有25mL 含有羧苄青霉素的 LB 液体培养基的锥形瓶,并加入 0.3% L-阿拉伯糖进行诱导表达。分别取诱导 0、2、4、6 和8h 的菌液作梯度稀释, 并取各梯度10μL 稀释液点至LB 平板, 在37℃培养箱中培养过夜, 拍照记录并计算菌落形成单位(colony-forming units, CFU)。同时分别取诱导后0、2、4、6 和8h 的菌液测定 OD600, 并使用GraphPad Prism 8 软件进行生长曲线的绘制。
1.6 质粒稳定性测定
将携带 pUC19-BAD01932-33 重组质粒和pUC19 空质粒的BW25113 菌株于含羧苄青霉素的LB 液体培养基中过夜培养, 以初始浓度OD600≈0.1转接到3mL 含羧苄青霉素的LB 液体培养基。37℃培养12h 后, 取菌液进行梯度稀释, 并分别取10μL各梯度的稀释液点在含羧苄青霉素和不含羧苄青霉素的LB 平板上, 该时间点记为0h。以0h 的菌液作为母液, 以1‰的体积比转接至3mL LB 液体培养基中, 每隔12h 取菌液进行梯度稀释, 并分别取10μL各梯度的稀释液点在含羧苄青霉素和不含羧苄青霉素的LB 平板上。在37℃培养箱中培养过夜, 拍照记录并计算CFU。并使用GraphPad Prism 8 软件进行质粒稳定性曲线的绘制。
1.7 蛋白序列的比对分析
从NCBI 网站下载所需蛋白序列, 采用Clustal X2 软件进行序列比对, 再将比对生成的文件上传至EsPript3.0 网站(http://espript.ibcp.fr/ESPript/cgi-bin/ESPript.cgi) 进行比对结果的展示。
1.8 目的蛋白的原核表达与纯化
将测序正确的重组质粒转化到表达菌株BL21(DE3)中。将转化板置于37℃恒温箱中过夜培养, 第二天挑单菌落到20mL LB 培养基中, 加入硫酸卡那霉素, 37℃恒温摇床中震荡培养过夜。次日取出过夜培养的20mL LB 培养基, 以1:100 的接种量接到500mL 培养基中, 加入硫酸卡那霉素, 37℃恒温震荡培养至 OD600达到 0.6~0.8, 加入终浓度为0.3mmol·L-1的异丙基硫代-β-D-半乳糖(IPTG), 放入37℃的恒温震荡培养箱中培养, 培养5h, 诱导目的蛋白表达。
蛋白纯化步骤由菌液离心及菌体重悬、超声破碎、高速离心、Ni-NTA 填料平衡、上清与填料结合、蛋白镍离子金属鳌合亲和层析介质(Ni-NTA)等步骤组成。具体纯化步骤如下:
1) 菌体重悬: 诱导5h 后的菌液离心去上清,沉淀用30mL Ni-NTA 缓冲液A 缓冲液重悬菌体到50mL 离心管中, 涡旋震荡充分混匀后加入终浓度为1mmol·L-1的苯甲基磺酰氟(phenylmethylsulfonyl fluoride, PMSF)和 5mmol·L-1的β-巯基乙醇(β-Mercaptoethanol, β-Me);
2) 超声破碎: 超声强度为75%, 破碎10s, 间隔5s, 冰上破碎120s。取4μL菌液加入到400mL Bradford中, 如果Bradford 变蓝, 则说明菌体已破碎好;
3) 高速离心: 将超声破碎过后的菌液 4℃,23500×g, 离心1h, 分离上清与沉淀;
4) Ni-NTA 填料平衡: 取1mL 柱料, 3434 ×g 离心60s 后去除上清。管底柱料先用2 倍柱体积的Ni-NTA 缓冲液B 平衡Ni-NTA 填料。接着再用2倍柱体积的Ni-NTA 缓冲液A 平衡填料;
5) 上清与填料结合: 蛋白高速离心结束后, 将上清倒入50mL 离心管, 用吸管吸取2mL 上清重悬平衡好的填料, 再一起转移到50mL 离心管; 4℃,低速旋转结合1h。先使用低浓度咪唑的缓冲液对结合到Ni-NTA 填料上的杂蛋白进行洗脱, 然后采用不同浓度的高浓度咪唑缓冲液进行梯度洗脱。收集的样品使用Tricine-SDS-PAGE 电泳检测蛋白的表达和洗脱情况。
2 结果
2.1 两种蓝细菌存在大量TA 系统
通过数据库检索和本地比对, 预测结果显示,集胞藻 PCC6803 含有 51 对潜在的 TA, 聚球藻WH7803 只有潜在的10 对TA(见附表2、3)。并且根据集胞藻 PCC6803 的预测结果, 在不成对的毒素基因或者抗毒素基因上下游分析寻找配对的抗毒素和毒素基因, 得到了9 个未注释的开放阅读框,命名为New ORF1-9。基于预测结果, 我们进行了不同类型 TA 的数量统计分析。分析结果表明, 集胞藻PCC6803 质粒分布有23 对, 基因组分布有 28对 TA(图 1a)。VapBC(23 对)、HicAB(9 对)和HigAB(4 对)系统是集胞藻PCC6803 基因预测的TA系统分布最多的三种类型(图 1b)。相比于集胞藻PCC6803, 聚球藻WH7803 基因组上的毒素-抗毒素系统分布较少, 仅有10 对(见附表3)。

附表2 集胞藻. PCC6803 毒素-抗毒素系统预测信息Tab. S2 Prediction information of toxin-antitoxin systems in Synechocystis sp. PCC6803

续表
依据编码基因的大小和方向, 两个基因位置是否存在重叠以及与典型TA 结构域和与同类型成员的序列相似度分析结果, 我们分别选取了集胞藻 PCC6803 预测结果中的 5 对 TA 和聚球藻WH7803 预测结果中的2 对 TA 进行进一步的分析和毒性实验验证(表1)。选取的集胞藻PCC6803 的5 对 TA 分别是: 位于质粒 pSYSM 中的BAD01871-72、BAD01932-93、BAD01997-98和位于基因组中的BAA17887、BAA18559-New ORF7。BAA17887基因编码的蛋白预测为HicAB 融合蛋白,进一步分析发现其N 端1~58 个氨基酸残基为HicA毒素蛋白, C 端 59~136 个氨基酸为HicB 抗毒素蛋白。另外,BAA18559-New ORF7中的毒素基因New ORF7在之前的报道中并没有得到注释, 为本文新注释的开放阅读框。选取的聚球藻 SynWH7803_1121-22 和SynWH7803_1700-01 两对TA 系统均位于基因组上。其余预测得到的TA 系统信息见附表2、3。
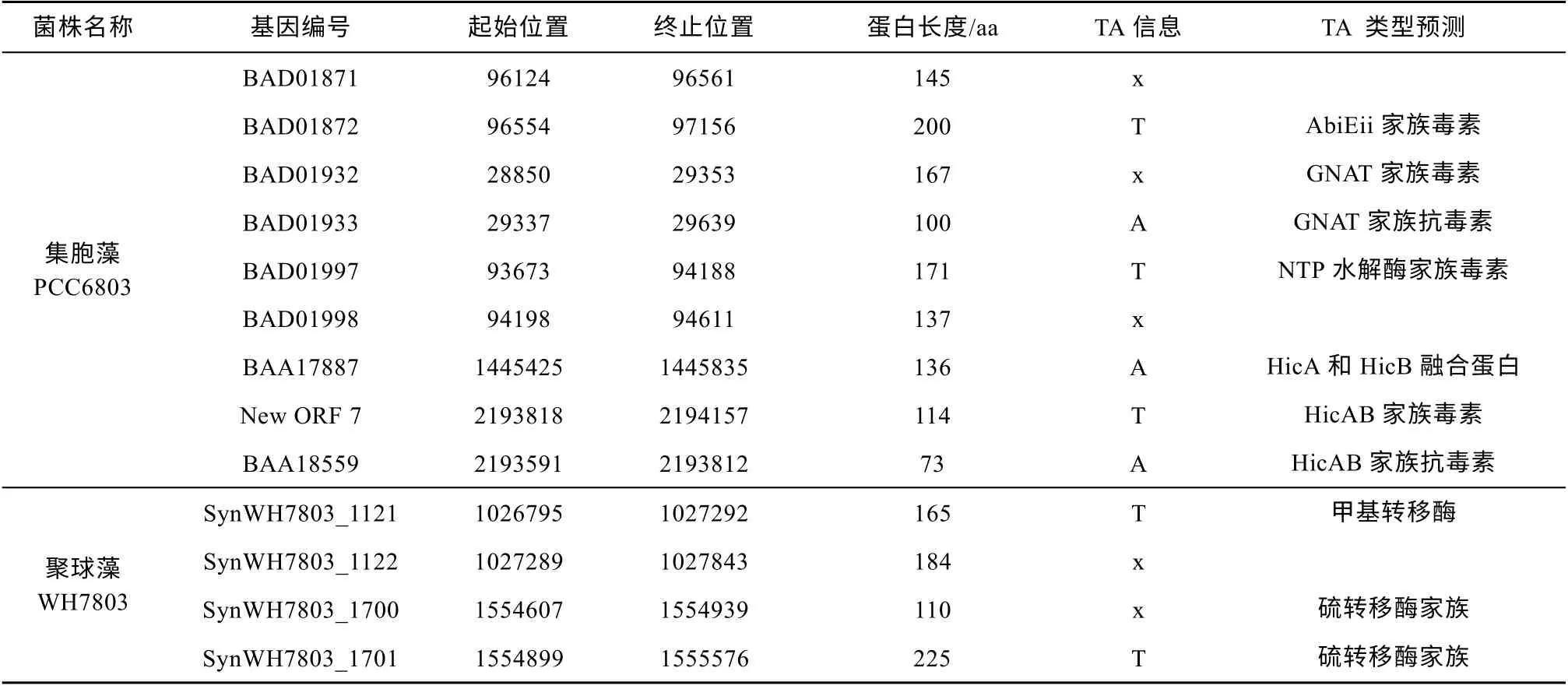
表1 本文选取的7 对毒素-抗毒素系统预测信息。Tab. 1 Prediction information of seven TA systems selected in this paper
2.2 BAD01932-33 和BAA18559-New ORF7 系统的毒素具有抑菌活性
实验结果表明, 过表达集胞藻PCC6803 毒素-抗毒素系统BAD01932-33 和BAA18559-New ORF7的毒素BAD01932 和New ORF7 时会对大肠杆菌BW25113 造成明显的生长抑制。而诱导表达合体BAD01932-1933 和BAA18559-New ORF7 可以解除毒素BAD01932 和New ORF7 的生长抑制毒性(图2a、b)。因此, BAD01932-1933 和BAA18559-New ORF7 与预测结果相符, 组成毒素-抗毒素系统。而选取的其余5 对毒素-抗毒素系统在含有50μg·mL-1卡那霉素和0.3%L-阿拉伯糖LB 平板均未表现出明显的毒性(图2c—g)。
进行毒性鉴定的 7 对毒素-抗毒素系统中,BAD01932-1933 和BAA18559-New ORF7 能够在大肠杆菌BW25113 中表现出毒性, 说明大肠杆菌中存在这两个系统与原宿主中毒素蛋白相同或相近的底物。另外5 对系统没有表现出异源宿主的毒性, 可能的原因是大肠杆菌中不存在这几个系统的底物,也有可能是这几个系统在大肠杆菌中不能表达, 所以仍需要在蓝细菌宿主中进一步确认。
2.3 BAD01932-33 具有维持质粒稳定性的生理功能
毒素-抗毒素系统具有维持质粒稳定性的生理功能在 1986 年被揭示(Gerdes et al, 1986)。BAD01932-33 分布于集胞藻 PCC6803 的质粒上,推测可能与质粒稳定性的维持有关。为了验证该设想, 我们将 BAD01932-33 构建至pUC19 质粒上,在E.coli进行质粒稳定性测定。结果表明, 传代48h后pUC19 质粒丢失95%, 而带有BAD01932-333 的pUC19 质粒在传代72h 后仍有 80% 细菌宿主携带pUC19 质粒(图3)。这说明BAD01932-33 能够明显增加pUC19 在E. coli中的稳定性。
2.4 BAD01932 和New ORF7 与同家族蛋白具有相似催化机制
鉴于选取的 7 对 TA 中 BAD01932-33 和BAA18559-New ORF7 毒素蛋白具有细胞毒性, 我们对其蛋白进行结构域和功能分析。BAD01932 的分析结果为GNAT 家族毒素蛋白。该蛋白家族目前研究得较为透彻的成员包括: 沙门氏菌(Salmonella typhimurium)的TacAT 系统(Cheverton et al, 2016),福氏志贺菌(Shigella flexneri)的 GmvAT 系统(McVicker et al, 2017), 肠出血性大肠杆菌(Enterohemorrhagic E.coli, EHEC) O157: H7 的AtaRT 系统(Jurėnas et al, 2017), 肺炎克雷伯菌(Klebsiella Pneumonia)的 KacAT 系统(Qian et al,2019)和大肠杆菌的ItaRT 系统(Wilcox et al, 2018)。我们将 BAD01932 与AtaRT、TacAT、KacAT、ItaRT和GmvAT 系统中毒素蛋白的序列比对, 结果表明BAD01932 与其他同类型的毒素成员具有一定的序列相似度, 较为保守, 其中与AtaT 的相似度最高(33%)。该家族中发挥重要的底物识别和催化功能的氨基酸残基为Tyr82、Arg92、Gly105 和Tyr141, 保守性高, 说明可能具有类似的催化机制(图4a)。
New ORF7 预测为HicAB 家族的毒素蛋白, 该蛋白家族研究得较为透彻的成员包括鼠疫杆菌(Yersinia Pestis)(Goulard et al, 2010)、类鼻疽伯克霍尔德菌(Burkholderia pseudomallei)(Butt et al, 2014)、肺炎链球菌(Streptococcus pneumoniae)(Kim et al,2018)和大肠杆菌的 HicAB 系统(Jørgensen et al,2009)。这四个物种的抗毒素蛋白HicB 单体以及HicAB 复合物的蛋白结构都已被解析, 但仅有类鼻疽伯克霍尔德菌的毒素蛋白HicA 的蛋白结构得到解析, 因此毒素蛋白HicA 的催化机制并不明确。鼠疫杆菌、类鼻疽伯克霍尔德菌、肺炎链球菌和大肠杆菌的HicAB 系统HicA 蛋白都属于同一个蛋白家族(PF07927)全长约为60 个氨基酸残基。本文新注释的New ORF7 所编码的HicA 蛋白与类鼻疽伯克霍尔德菌的HicA 蛋白相似度最高(20%), 较为特殊的是New ORF7 所编码的HicA 蛋白的N 端多出约50 个氨基酸。尽管如此Gly78 和His80 这两个在同家族成员的催化过程起重要作用的残基仍然较为保守, 这也说明它们可能具有相似的催化机制(图4b)。
2.5 BAD01932-33 和BAA18559-New ORF7 系统蛋白的表达与纯化
为了进一步验证BAD01932-33 和BAA18559-New ORF7 这两对TA 系统中毒素和抗毒素蛋白的相互作用情况, 我们采用pET28b(+)载体进行两对系统的毒素、抗毒素单独以及合体的异源表达和纯化。由于这两个系统的毒素蛋白对宿主菌株有较强的毒性作用, 我们进行了大量尝试, 均未成功构建野生型的表达载体。因此实验中只尝试了抗毒素单独以及与毒素共表达的情况。实验结果表明,BAD01932-33 的抗毒素蛋白BAD01933 单独表达以及毒素-抗毒素共表达菌株在加入IPTG 诱导之后都有目的蛋白分子量对应大小的过表达条带产生(图5a 泳道2 和图4b 泳道2), 高浓度咪唑洗脱样品中,只有单独表达抗毒素蛋白的载体有极少量可溶性蛋白能够被洗脱(图5a 泳道6 和7)。而绝大部分的抗毒素蛋白以及毒素和抗毒素共表达蛋白几乎全部在包涵体中(图5a 泳道4 和图5b 泳道4)。对诱导剂浓度、诱导时长和诱导温度的改变以及进行毒素和抗毒素的密码子优化都未能提高可溶性蛋白量。
BAA18559-New ORF7 TA 系统中抗毒素蛋白BAA18559 单独表达菌株在加入诱导剂IPTG 后有过表达条带(图5c 泳道2), 但是仍以包涵体的形式存在(图5c 泳道4), 以高浓度咪唑进行洗脱时仍没有得到抗毒素蛋白BAA18559(图5c 泳道6 和7)。而毒素蛋白New ORF7 和抗毒素蛋白BAA18559 共表达的菌株在加入诱导剂 IPTG 后都没有出现明显的过表达条带(图5d 泳道2)。这种情况的出现的原因可能是BAD01932-33 和BAA18559-New ORF7这两对TA 系统的蛋白在大肠杆菌中表达时不能正确折叠, 或者是这些蛋白的溶解度本身偏低。
3 讨论
本文采用生物信息学方法预测了淡水蓝细菌集胞藻PCC6803 和海水蓝细菌聚球藻WH7803 的毒素-抗毒素系统。预测结果表明, 集胞藻PCC6803 具有51 对潜在的TA, 而聚球藻WH7803 具有10 对TA。出现这种差别的原因可能与两种细菌所生存的环境的复杂性有关, 集胞藻PCC6803 所生活的淡水中的微生物种类和数量较多, 水体随季节变化存在较大温差, 营养成分和种类受人类活动影响大, 而聚球藻WH7803 所生活的海洋环境的温度和营养波动较小。而TA 系统的存在有利于菌体抵抗逆境和复杂生存环境, 因此更有利于集胞藻PCC6803 适应复杂多变的生态环境。由于本文仅分别预测分析了一种淡水蓝细菌和海水蓝细菌, 因此TA 系统在淡水和海水蓝细菌基因组上的分布差异性还需要后续的研究分析。
VapBC (Vap: virulence associated protein, 毒力相关蛋白)类型的TA 系统在集胞藻PCC6803 所预测的51 对TA 系统分布最高, 为23 对。在原核生物中,VapC 家族成员大多数具有PIN 结构域, 并在许多原核生物的基因组中大量存在, 如结核杆菌目前已发现超过48 对(Pandey et al, 2005; Ramage et al, 2009)。VapC 家族成员众多, 但很多成员的底物目前并不清楚。已有的研究表明, 沙门氏菌、志贺氏菌和钩端螺旋体(Leptospira interrogans)中的VapC 蛋白能切割起始tRNAfMet, 并且均从特定的位点切割反密码子环。所以VapC 能够抑制细胞蛋白翻译功能的起始(Winther et al, 2011; Lopes et al, 2014)。然而VapC 家族成员并非所有成员都通过切割tRNA 抑制蛋白的翻译, 例如结核杆菌的VapC20 能通过切割23S rRNA 中保守的Sarcin-Ricin 环(SRL)抑制蛋白的翻译(Winther et al, 2013)。
本文选取的毒素蛋白 BAD01932 和 New ORF7 在大肠杆菌BW25113 异源表达时具有细胞毒性。其中 BAD01932-33 位于集胞藻 PCC6803的质粒上, 属于一类新型Ⅱ型TA 系统。本文研究发现, BAD01932-33 能够明显增加pUC19 在E.coli中的稳定性。其毒素蛋白具有 GNAT 折叠,抗毒素蛋白具有Ribbon-Helix-Helix (RHH)折叠。GCN5-related N-acetyl-transferase (GNAT)来源于酵母菌GCN5 (general control non-depressible 5),是一种组氨酸乙酰转移酶。在乙酰化反应中, 这类酶将乙酰-CoA 作为乙酰基供体。GNAT 超家族能够对包括蛋白质、小分子代谢物和抗生素在内的一系列底物进行乙酰化修饰, 例如赋予细菌对氨基糖苷类药物产生抗性的氨基糖苷 N-乙酰基转移酶就是第一个被鉴定的GNAT 超家族成员。肠出血性大肠杆菌O157: H7 的AtaT(Jurėnas et al,2017)和沙门氏菌的TacT(Cheverton et al, 2016)这两种 GNAT 毒素的分子机理研究较为透彻,AtaT 通过将乙酰-CoA 的乙酰基转移到Met-tRNAfMet从而抑制翻译的进行。其他GNAT家族的毒素成员作用机制类似, 不同的是作用的tRNA 种类存在区别。
BAA18559-New ORF7 属于HicAB 类, 也是一种Ⅱ型 T A 系统。最早发现于流感嗜血杆菌(Haemophilus influenzae) 的菌毛基因簇(hif)当中,因此被命名为 HicAB (Haemophilus influenza contiguous)。hicAB 基因排列有一个显著的特点: 毒素基因hicA 位于抗毒素基因hicB 的前面, 这与大多数TA 系统的基因排列顺序相反。hicAB 基因在细菌中分布广泛, 常常多对存在, 具有多样性。抗毒素蛋白HicB 通常含有RNase H 样折叠(典型特征包含β1β2β3α1β4α2 模块), 而HicA 蛋白通常包含结合双链RNA 的结构域。第一对由实验证实的HicAB 成员位于大肠杆菌 K-12 基因组中(Jørgensen et al,2009)。其HicA 蛋白是核糖体不依赖型的RNA 切割酶, 能够对胞内的mRNA 和tmRNA 酶切, 进而阻断翻译过程的进行, 具有抑菌功能。而HicB 并没有表现出细胞毒性, 反而可以完全抑制HicA 的毒性。对HicAB 家族的后续研究发现, 其主要的生理功能包括维持菌株毒力(Goulard et al, 2010), 介导“持留菌”的形成(Butt et al, 2014)和协助菌体适应细胞外应激(Jørgensen et al, 2009)。
Ⅱ型 TA 的毒素蛋白和抗毒素蛋白之间存在紧密的相互作用。抗毒素蛋白要与毒素蛋白的结合底物竞争, 所以毒素-抗毒素之间的相互作用一般强于毒素-底物之间的相互作用。毒素蛋白一般具有以下特点: 比抗毒素蛋白稳定、水溶性不高和对宿主菌有毒性作用。而抗毒素分子的特点为:较不稳定、极易降解、水溶性较毒素好以及能够抑制毒素分子对宿主菌的毒性作用。因此在体外进行功能研究的时候要充分考虑毒素和抗毒素两种分子的特性。本文采用大肠杆菌表达系统异源表达的BAD01932-33 和BAA18559-New ORF7 两个系统的蛋白时发现, BAD01932-33 系统单独表达抗毒素时仅有少量可溶性蛋白, 表达合体时两种蛋白均在包涵体中, 说明蛋白的水溶性较低是该系统异源表达纯化的制约因素。而BAA18559-NewORF7 单独表达抗毒素时蛋白全部在包涵体中, 表达合体时也只有抗毒素有诱导带出现, 毒素蛋白甚至没有诱导带产生, 可能是因为HicA 蛋白的毒性太强, 导致加入诱导剂之后出现基因突变而不能正常表达。
本文分别以集胞藻PCC6803 和聚球藻WH7803为研究对象, 通过生物信息学的方法预测了二者基因组和质粒上的毒素-抗毒素系统, 并通过实验验证了其中7 对潜在的系统。本文的研究结果为后续蓝细菌毒素-抗毒素系统功能和应用研究的开展提供了理论依据。
——一道江苏高考题的奥秘解读和拓展

