基于LightGBM的蛋白质类泛素化修饰位点预测
陈焕超,魏志森,於东军,杨敬民,杨静宇
(闽南师范大学1.计算机学院;2.数据科学与智能应用福建省高校重点实验室,福建 漳州 363000;3.南京理工大学 计算机科学与工程学院,江苏 南京 210094)
翻译后修饰(Post-translational modifications,PTMs)是一种涉及蛋白质原始化学组成改变的化学修饰,它可以将化学基团添加到特殊残基或在特定位置切割序列,从而扩展从相同基因组序列生成蛋白质的最终形式范围,是增加蛋白质组多样性的关键机制[1]。PTMs在功能蛋白质组中发挥关键作用,包括调节酶的活性和定位蛋白质在细胞中的位置、介导信号转导、激酶与识别候选蛋白和蛋白质降解及泛素化调控等[2]。类泛素化(Sumoylation,SUMO)修饰位点是PTMs一种重要的类型,与许多人类疾病的发生息息相关,包括神经退化疾病、阿尔茨海默病和癌症等[2]。
传统的SUMO修饰位点检测方法利用生物湿试验,需要大量的人力物力,周期长,成本高。近十几年以来,开发计算模型预测SUMO修饰位点已经成为生物信息学领域的一个研究热点,特别是机器学习的发展促进了相关机器学习算法在生物信息学领域的应用,许多学者提出了不同的计算模型预测SUMO修饰位点,作为试验方法的一种低廉高效的补充手段。Xue等[3]基于同源蛋白质序列聚类分组比对和序列模式匹配相结合的方法,开发一个在线SUMO修饰位点预测服务器SUMOsp。Ren等[4]将SUMOsp中的同源序列聚类分组比对算法进行改进,引入了试验观察的分组先验信息。Zhao等[5]通过粒子群优化算法改进SUMOsp的同源序列聚类分组策略并取得了更好的预测性能。Xu等[6]提出基于数据集上的序列信息的概率统计预测新序列的SUMO修饰位点。Chen等[7]提出将氨基酸疏水性作为参数引入到传统的二进制编码方案中,并使用支持向量机作为分类器,其试验证明了疏水性在SUMO修饰位点预测上的鉴别能力。Teng等[8]提出位置特异性得分矩阵和生物特征相结合的特征表示,并训练支持向量机和随机森林进行预测。Xu等[9]提出结合3种不同的序列特征表示方法,基于不同特征的组合使用线性判别分析进行预测。Jia等[10]提出将序列偶合信息整合到一般的伪氨基酸组成中,并将其应用于协方差判别算法,此预测器可以自动缓解由数据不平衡所带来的误差。Sharma等[11]基于蛋白质的半球裸露结构特征训练决策树分类器预测SUMO修饰位点。最近,Qian等[12]结合氨基酸的疏水性等物化属性统计特征和氨基酸序列二元语法模式特征,训练级联森林分类器,并使用遗传算法对预测得分进行加权平均。虽然这些工作在SUMO修饰位点的预测上取得了重要的进展,但是预测精度还有较大的改进空间。
本文提出了一种新的蛋白质泛素化修饰位点预测模型,基于氨基酸的疏水性等物化属性统计与氨基酸序列二元语法模式的特征组合,训练一种轻量型梯度提升机(Light gradient boosting machine,LightGBM)分类器[13]将待预测氨基酸残基识别为修饰位点或非修饰位点,此模型记作SUMO-LGBM。在基准数据集上进行十折交叉验证试验,证明其泛化能力。
SUMO-LGBM的算法框架如图1所示。对于蛋白质序列中的某个赖氨酸(K)残基,通过统计以其为中心的大小为21的滑动窗口内氨基酸残基的物化属性统计特征(Statistics property,SP)和二元语法模式(Bi-gram and k-skip-bi-gram,BK),将其表示为一个62维的特征向量;然后,将这个特征向量输入到多个决策树集成的LightGBM分类器中,将分类器的输出值与设置的阈值进行比较,大于阈值则标记为类泛素化修饰位点,否则标记为非修饰位点。下文将对氨基酸残基的特征描述和LightGBM分类器分别进行介绍。

图1 SMUO-LGBM预测模型的算法框架
1 氨基酸残基的特征描述
将机器学习算法应用于SUMO修饰位点预测问题,蛋白质序列上的每个氨基酸残基需要表示成一个特征向量。在本文中,每个氨基酸残基由两部分特征组合表示,一部分是物化属性统计特征SP,另一部分是氨基酸序列二元语法模式特征BK。其中,SP是一个5维向量,而BK是一个57维向量,拼接得到一个62维的特征向量。在以氨基酸残基为分类对象的生物信息学问题中,经常将目标残基为中心的邻域窗口内的残基的特征作为目标残基特征向量的一部分,以增强特征向量的鉴别力[14-17]。因此,本文对蛋白质序列设置了一个滑动窗口,每个氨基酸残基由以其为中心的滑动窗口内的残基的SP和BK特征拼接而成的特征向量表示。根据Qian等[12]报告的试验结果,本文同样选择滑动窗口大小为21。
1.1 物化属性统计特征SP
根据Beauclair等[18]的统计结果,将序列中SUMO修饰位点的位置标注为0,往左n个位置标注为-n,往右n个位置标注为n,那么,在SUMO修饰位点的-1位置处,疏水性氨基酸的发生率更高,其中带有脂肪族侧链的残基为大多数,占比67.5%,而芳香族氨基酸比较少,占比5.4%;在+2位置处,酸性残基比较富集;而在+1位置处,没有特定氨基酸表达。据此,Qian等[12]以目标氨基酸残基的-1位置和+2位置处的氨基酸属性作为鉴别特征。其中,将-1位置处的氨基酸分为4类:(1)I、L、V;(2)A、F、M、P、W;(3)G、Y;(4)其余的氨基酸;分别由(0,0,0,1)、(0,0,1,0)、(0,1,0,0)和(1,0,0,0)表示;将+2位置处的氨基酸分为2类:D和E为一类,用0表示,其余为另一类,用1表示。将这2者组合起来,目标氨基酸残基可以由一个5维的特征向量表示。
1.2 氨基酸序列二元语法模式BK
在自然语言处理领域,单词序列多元语法模式(K-skip-n-gram)[19]被用于统计单词之间共现概率。由于蛋白质序列与自然语言文本序列都是一维序列,可以将每个氨基酸残基类比为文本序列的单词,从而将K-skip-n-gram应用于生物信息学领域[12]。本文采取Bi-gram和K-skip-bi-gram,其中K=1,2。Bi-gram相当于K=0时的K-skipbi-gram。由于有20种氨基酸,两两组合二肽可得20*20=400种组合,因此特定位置的二肽可以描述为

式中


在矩阵F中,每一行代表每一种二肽组合,前20列代表K=0时每个位置出现某种二肽的概率,中间19列代表K=1时每个位置出现某种二肽的概率,最后18列代表K=2时每个位置出现某种二肽的概率。这样,对于一个长度为21的氨基酸序列,根据每个位置出现的二肽,由矩阵F中取相应的元素,可以得到一个维数为20+19+18=57的特征向量。
2 LightGBM分类器
Ke等[13]提出的LightGBM算法是微软发布的一个高效、开源的梯度提升决策树(Gradient boosting decision tree,GBDT)算法[20]框架。相比于传统的GBDT,LightGBM具有更快的训练速度、更低的内存消耗、更高的准确率和支持分布式等特点,可以快速地处理海量数据,因此得到广泛应用。
LightGBM的最大特点是在传统的GBDT基础上引入了基于梯度的单边采样(Gradient-based one-side sampling,GOSS)和互斥特征捆绑(Exclusive feature bundling,EFB)这两种技术。GOSS技术根据梯度大小对训练样本进行排序,下采样时随机抛弃梯度比较小的样本,保留对信息增益有更大影响的大梯度样本。这种方法被证明在相同的采样率下比随机采样具有更高的准确率,尤其是在信息增益范围较大时。
EFB算法将样本中不同维度的互斥特征进行捆绑,用一个合成特征代替多个互斥特征,从而达到降低特征维度的目的,提高算法的效率。其中,互斥特征是指在稀疏特征空间中,不同时为零值的两个特征,比如独热编码特征中的不同维度。
3 试验与分析
在本节中,将对试验用到的数据集,评估方法以及试验结果与分析进行描述,并与现有的SUMO修饰位点预测方法进行比较。
3.1 数据集
本文使用Qian等[12]构建的基准数据集对提出的方法进行评估。此数据集从UniProt数据库[21]获取510个蛋白质序列中以赖氨酸为中心的长度为21的肽段。这些肽段的中心赖氨酸残基如果被试验标注为SUMO修饰位点则作为正样本,否则作为负样本。为了去除冗余数据,对数据集中的数据进行序列同一性的样本筛选,确保数据集中任意两个样本的相似度小于40%,最终得到755个正样本和9 944个负样本。
3.2 评估方法
在本文的试验中,引入6种常用的指标用于评估本文提出的方法以及与其他方法进行比较,包括准确率(Accuracy,Acc)、特异性(Specificity,Sp)、灵敏度(Sensitivity,Sn,也称为召回率Recall)、马修斯相关系数(Matthews correlation coefficient,MCC)、精确率(Precision)和F1指数。这些指标定义如下
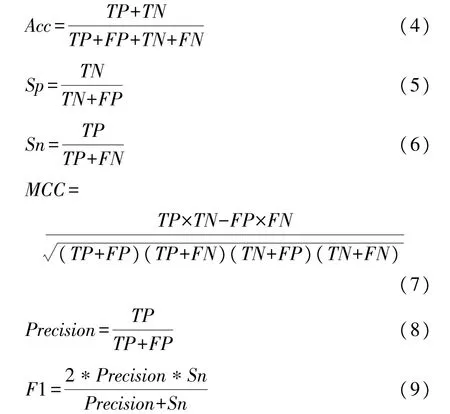
式中:TP为被正确分类的正样本的数量,FP为被错误分类的负样本的数量,TN为被正确分类的负样本的数量,FN为被错误分类的正样本的数量。由于数据集的两类样本分布不平衡,单一使用Acc、Sp、Sn评价无法很好地评估整体性能,因此,本文主要关注AUC、AUPR、MCC这3个能反映分类器整体性能的指标,其中AUC是接收者操作特征曲线(Receiver operating characteristic curve,ROC曲线)下包围的面积,AUPR表示精度召回率曲线(Precision recall curve,PR曲线)下包围的面积,通常认为这两个面积越大,分类效果越好[22]。
本文将使用10折交叉验证和留一法交叉验证来评估提出的方法在数据集上的性能,通过使用不同的评价指标来观察参数对性能的影响,并选择MCC、AUC和AUPR作为最终的评价指标。
3.3 试验结果与分析
3.3.1 分类算法对比为了建立一个有效的预测模型,K近邻算法(K-nearest neighbor,KNN)、决策树(Decision tree,DT)、朴素贝叶斯(Naive bayesian,NB)、随机森林(Random forest,RF)、逻辑回归(Logistics regression,LR)、极端随机树(Extremely randomized trees,ET)和LightGBM被用于构建分类模型。各种算法的参数通过十折交叉验证进行优化,其中,KNN的K值设置为10,极端随机树、逻辑回归、决策树和随机森林对正负类样本设置不同的权重,使得两类样本的权重之和相等。在基准数据集上进行十折交叉验证,各种分类算法构建的分类器的性能指标展示在表1中。为了更直观地比较不同分类器的性能,图2和图3分别展示了数据集上十折交叉验证的平均ROC曲线和PR曲线。

表1 不同分类算法的性能比较 %
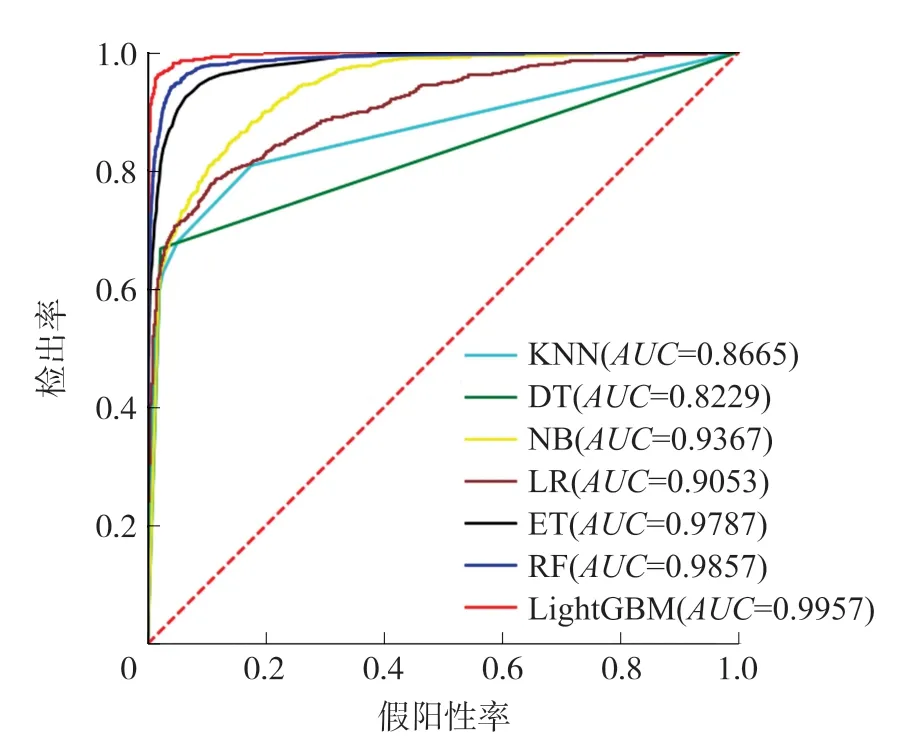
图2 不同分类器的ROC曲线比较
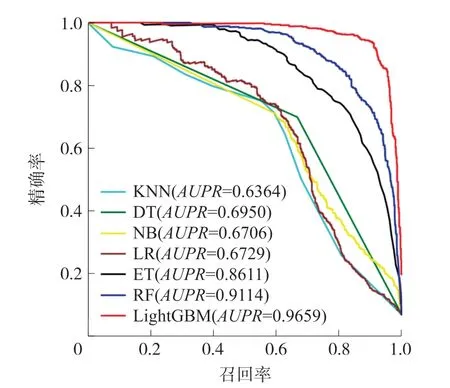
图3 不同分类器的PR曲线比较
从表1可以看出,LightGBM在所有评估指标上均取得了最好的结果,其中,在F1指数、MCC、AUC和AUPR这几个反映全局性能的指标上分别达到了92.21%、91.64%、99.57%和96.59%,比排在第二的RF高出了11.21%、11.94%、1%和5.45%。这说明,与其他算法相比,LightGBM在分类准确率和整体性能上均表现出更好的结果。另外,从表1也可以发现,相比于其他4种算法,LightGBM、RF和ET这3种算法在MCC等全局性能指标上均获得了较为明显的更好的结果,这说明了树分类器的集成算法在这一数据集上具有更好的分类性能。同样,从图2和图3的ROC曲线和PR曲线中也能得出以上结论。在图2的ROC曲线中,对于任意一个假阳性率(False positive rate,FPR)值,LightGBM的ROC曲线均位于其他算法的曲线之上,而在图3的PR曲线上,对于任意一个召回率值,LightGBM的PR曲线均位于其他算法的曲线之上,这说明LightGBM在性能上显著地战胜了其他所有算法。同时,LightGBM、RF和ET这3种算法的曲线比较靠近,且明显超越其他4种算法,进一步验证了这类树分类器的集成算法具有更好的分类性能。
3.3.2 特征分析
为了验证SP和BK这两种特征表示方法的有效性,本文对单独使用SP和BK特征,以及两种特征的组合分别进行了测试,同时与蛋白质序列常用的另外两种特征表示方法进行了比较,即氨基酸组成(Amino acid composition,AAC)[23]和氨基酸二进制编码(Binary encoding,BE)[24]。基于以上的特征及特征组合分别训练LightGBM分类器,在基准数据集上进行十折交叉验证,对MCC、AUC和AUPR这几个重要指标进行比较,结果展示在表2中。
从表2可以看出,在所有单特征表示中,BK在MCC、AUC和AUPR上分别取得了88.75%、99.44%和95.69%,显著地高于其他所有的特征。而SP和BE的表现比较接近,SP取得了更高的M C C值,但是在AUC和AU P R上均弱于BE。另外,SP+BK的特征组合进一步提高了性能,比BK在M C C和F1上分别提升了2.89%和2.79%,在AUC和AUP R上有略微提高。这说明BK和SP特征具有一定的互补性,两种特征的组合具有更好的鉴别性,能够有效地提高预测模型的性能。
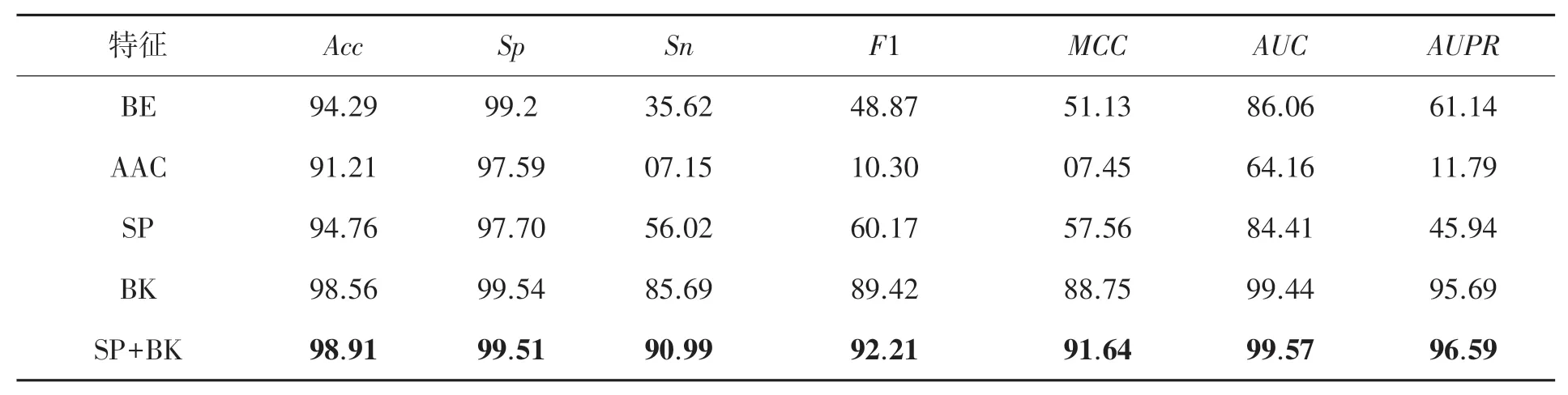
表2 不同特征的性能比较 %
3.3.3 与现有方法的比较
为了证明方法的有效性,将本文提出的SUMO-LGBM 与 pSumo-CD[10]、HseSUMO[11]、SUMO-Forest-FM[12]、SUMO-Forest-CM[12]等现有方法进行了比较,结果展示在表3中,表中现有方法的数据均来自于文献[12]。其中,除HseSUMO外,其他预测器与本文使用相同的基准数据集。

表3 SUMO-LGBM预测器与其他方法的比较
如表3所示,在所有方法中,本文提出的SUMO-LGBM取得了最好的Acc、Sp、MCC和AUC。其中,MCC值为91.64%,超过第二高的SUMO-Forest-FM预测器2.1%;AUC值为99.57%,与SUMO-Forest-CM相近,超过SUMO-Forest-FM预测器1.4%。
为了更直观地说明SUMO-LGBM模型的性能提升,SUMO-LGBM、SUMO-Forest-CM和SUMOForest-FM的ROC曲线和PR曲线分别展示在图4和图5中。由图4可知,3种模型的ROC曲线是非常接近的,这也验证了SUMO-LGBM在AUC上只取得了轻微的提升。而从图5可以发现,SUMO-LGBM的PR曲线显著地高于另外两个模型的PR曲线。这说明,在相同的召回率下,SUMO-LGBM模型具有更高的精确率,从而具有更好的预测性能。
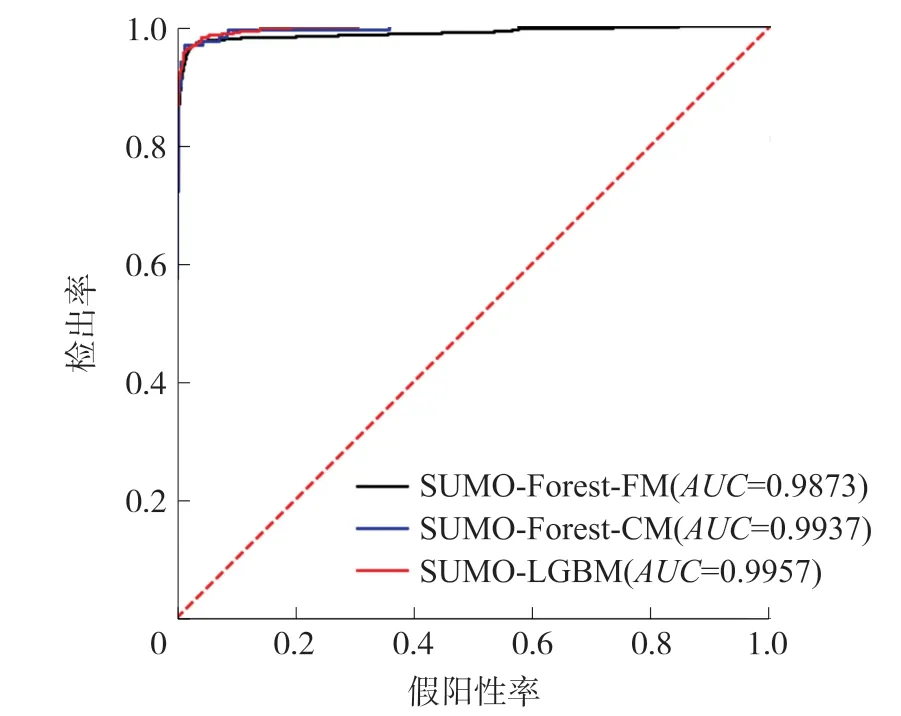
图4 与最新预测器的ROC曲线比较

图5 与最新预测器的PR曲线比较
4 结束语
本文基于SUMO修饰位点邻域内氨基酸残基的物化属性和二肽统计规律,利用LightGBM算法出色的分类能力,提出了一种新的蛋白质SUMO修饰位点预测模型SUMO-LGBM。在基准数据集上的试验结果证明了本文所提模型的有效性和鲁棒性,取得了当前领先的预测性能,可作为试验方法识别SUMO修饰位点的一种辅助手段。
由于当前的基准数据集在规模上还无法满足训练大通量预测模型的要求,在未来的研究工作中,一方面要探索更具鉴别性的特征和更复杂的机器学习模型的应用,另一方面需要继续收集数据,扩充基准数据集的规模以支持更复杂模型的训练和评估。

