基于转录组测序分析弥漫型胃癌特征及其核心基因LUM的表达
李戴牟 吴留成 梁榜辉 潘佳宇 王婷安 韦尉元 金钦文 凌通 黄名威 覃宇周
在我国乃至全球范围内,胃癌的发病率和致死率长期以来居高不下[1-2]。尽管胃癌在临床治疗上已经取得较大进展,但其早期症状隐匿,筛查手段也较局限,同时缺乏敏感性高、特异性强的生物标志物,因此整体预后仍然不理想。深入研究胃癌发生发展的分子机制及其相关基因有助于发掘更有效的靶点。胃癌是异质性极高的肿瘤,不同组织学类型的胃癌生物学行为及预后差别巨大。根据常用的Lauren分型,胃癌一般分为肠型、弥漫型和介于两者之间的混合型。胃癌中约30%为弥漫型,通常弥漫型胃癌具有更强的恶性生物学行为和更少的靶向治疗选择[3-4],加之疾病进展迅速,容易侵犯浆膜造成腹膜转移,因此成为临床治疗的一个挑战[5-7]。本研究利用转录组测序分析弥漫型胃癌中的优势基因集,并结合TCGA数据库对优势基因集的表达及其与Lauren分型进行相关性分析,同时对核心基因LUM(Lumican)的表达进行扩大样本量验证,为弥漫型胃癌寻找新的预测标志物和治疗靶点提供依据。
1 资料与方法
1.1 一般资料
选取2021年1月—2021年12月在广西医科大学附属肿瘤医院首次诊断并经术后病理确诊为胃癌的29例患者为研究对象,其中5例弥漫型胃癌样本用于转录组测序,24例胃癌样本用于验证转录组测序结果。所有癌组织及其配对癌旁正常组织样本手术切除后立即速冻于液氮,并置于-80℃冰箱保存。样本与病例资料收集经广西医科大学附属肿瘤医院伦理委员会批准(批件号:LW2022011)。
1.2 转录组测序
1.2.1 总RNA质检、文库构建及Illumina测序 根据RNeasy Mini Kit[天根生化科技(北京)有限公司]提供的标准操作流程提取总RNA,经NanoDrop ND-2000分光光度计及Agilent Bioanalyzer 4200质检合格后用于后续的文库构建。经去除rRNA、打断、双链cDNA合成、降解第二链、末端修复、3'末端加A、连接接头、扩增实验等过程后,采用链特异性建库和PE150测序策略,最终获得原始数据。每个样品的下机数据量不低于10 Gb,由上海华盈生物医药科技有限公司完成。
1.2.2 数据质量评估、预处理及比对分析 原始数据应用测序质量Q值进行评估,经过碱基识别及误差过滤后,使用Seqtk软件进行预处理,经去除接头序列、所属物种的ribosome RNA reads、长度小于25的低质量reads和3'端质量低于Q20(碱基错误率小于0.01)的碱基等步骤,最终获得后续分析使用的clean reads。clean reads经HISAT2软件进行基因组mapping分析,参数设置为默认参数,参考基因组版本为GRCh38。
1.2.3 基因差异表达分析 使用edgeR软件中的TMM算法对fragment counts进行归一化,根据假设检验模型计算P-value,经多重假设检验校正后得到FDR值(Q.value)。使用FPKM(Fragments Per Kilobase of exon model per Million mapped reads)表示基因表达水平,通过 FPKM计算差异倍数(Fold-change,FC),过滤掉在50%样本中表达量低于1的基因,差异基因筛选标准:FC>2且Q.value<0.05。火山图绘制涉及R语言分析报告包:ggplot2包、ggrepel包。
1.2.4 差异基因的富集分析 通过GO数据库,从生物过程、细胞组分、分子功能三个层面上分析差异基因的功能,再利用KEGG分析差异基因的通路。涉及R语言分析包:clusterProfiler包、org.Hs.eg.db包、ggplot2包。
1.2.5 差异基因的蛋白互作网络 使用cytoscape软件中的stringApp,以默认参数输出差异基因的蛋白互作网络[8-9];使用MCODE,以默认参数进行聚类功能模块分选[10];使用cytoHubba的Degree算法评估网络并计算每个差异基因的得分[11]。
1.2.6 差异基因功能簇分析 通过MSigDB Collections数据库中获取h.all.v7.2.symbols.gmt(Hallmarks)数据,提取上皮-间充质转化(epithelial-mesenchymal transition,EMT)通路中的基因信息。
1.3 RT-qPCR检测LUM的表达水平
采用TransZol Up Plus RNA Kit试剂盒(北京全式金生物技术股份有限公司)提取胃癌组织及其癌旁正常组织样本的总RNA,用Trans Script Uni All-in-one First-strand cDNA Synthesis Supermix for qPCR试剂盒(北京全式金生物技术股份有限公司)逆转录成cDNA,最后使用Perfectstart Green qPCR Supermix试剂盒(北京全式金生物技术股份有限公司)在ABI Prism 7500仪器中进行实时荧光定量PCR反应。按照试剂盒说明书配制反应体系,采用推荐的两步法反应条件进行反应:94℃预变性30 s,随后94℃变性5 s、60℃退火34 s依次循环,于退火时采集荧光信号,共计循环40次。引物序列由北京擎科生物科技有限公司合成:LUM上游5'-TGGCTGATAGTGGAATACCTGGAA-3',下游5'-ATGCTTGATCTTGGAGTAGGATAATGG-3';β-actin上游 5'-GTCATTCCAAATATGAGATGCGT-3',下游 5'-GCATTACATAATTTACACGAAAGCA-3'。检测结果采用2-△Ct和2-△△Ct法计算。
1.4 基于TCGA数据库的转录水平及临床资料分析
通过TCGA数据库(https://portal.gdc.cancer.gov/)获取STAD(胃癌)项目下完整的RNAseq数据和临床数据,FPKM格式的RNAseq数据经log2(FPKM+1)转化后用于后续分析。通过cBioPortal数据库(http://www.cbioportal.org/)获取胃癌(TCGA,Nature 2014)临床资料[12],作为Lauren分型资料的补充,筛选出Lauren分型中的弥漫型、肠型和混合型样本ID号,通过样本ID号匹配RNAseq数据和临床资料数据。
1.5 统计学方法
采用SPSS 20.0软件、GraphPad Prism 8.0软件和R 4.1.1软件进行统计学分析及绘制结果图。在TCGA数据库的资料分析中,涉及主要R语言分析包:ggpubr包、ggplot2包、survival包、survminer包,当基因的表达量符合正态性和方差齐性要求时,两组间比较采用独立样本t检验,多组间采用单因素方差分析进行差异比较;当基因的表达量不符合正态性和方差齐性的要求,两组间比较采用Wilcoxon秩和检验,多组间采用Kruskal-Wallis检验进行差异比较。采用Kaplan-Meier法计算生存率,组间比较采用log-rank检验。在胃癌组织及其配对正常组织的RT-qPCR结果中,资料不满足正态性和方差齐性的要求,配对样本采用Wilcoxon符号秩检验进行差异比较,事后两两比较采用Bonferroni法。以双侧P<0.05为差异有统计学意义。
2 结果
2.1 转录组测序的差异基因分析
以FC>2且Q.value<0.05为筛选条件,弥漫型胃癌样本中癌组织与配对正常组织共有175个差异基因,其中上调基因91个,下调基因84个,见图1。
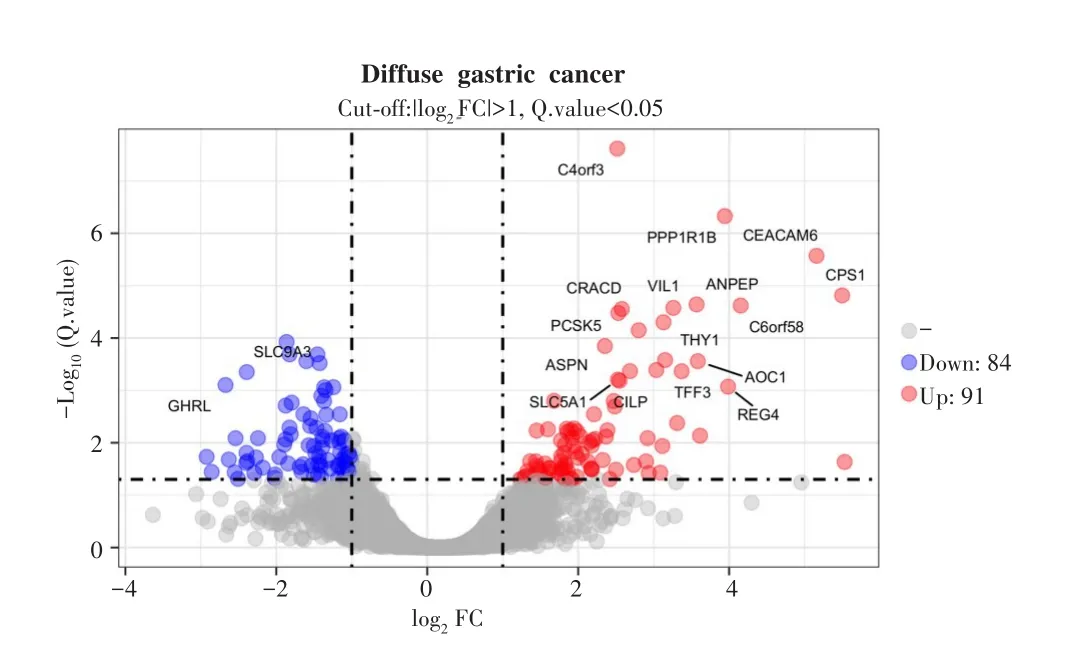
图1 弥漫型胃癌中癌组织与配对正常组织的差异基因火山图Fig.1 Volcano map of differentially expressed genes in diffuse gastric cancer tissues and matched normal tissues
2.2 GO和KEGG富集结果
差异基因经富集分析后,分别选取GO分析中生物过程、细胞组件分子功能和KEGG通路中排名前5的条目进行展示,见图2。其中,富集的生物过程包括细胞外结构组织、细胞外基质组织、骨骼发育、平滑肌收缩、胶原原纤维组织;富集的细胞组件包括含胶原蛋白的细胞外基质、细胞顶端部分、顶端质膜、细胞外基质成分、胶原三聚体的复合物;富集的分子功能包括细胞外基质结构成分、赋予抗压能力的细胞外基质结构成分、生长因子结合、碳水化合物跨膜转运蛋白活性、血小板衍生生长因子结合。GO富集分析提示弥漫型胃癌存在多种方式参与细胞外基质相关的细胞过程改变。富集的KEGG通路包括蛋白质消化和吸收、血小板活化、矿物质吸收、补体和凝血级联、胃酸分泌,提示弥漫型胃癌中存在多种信号通路共同调控恶性进展。
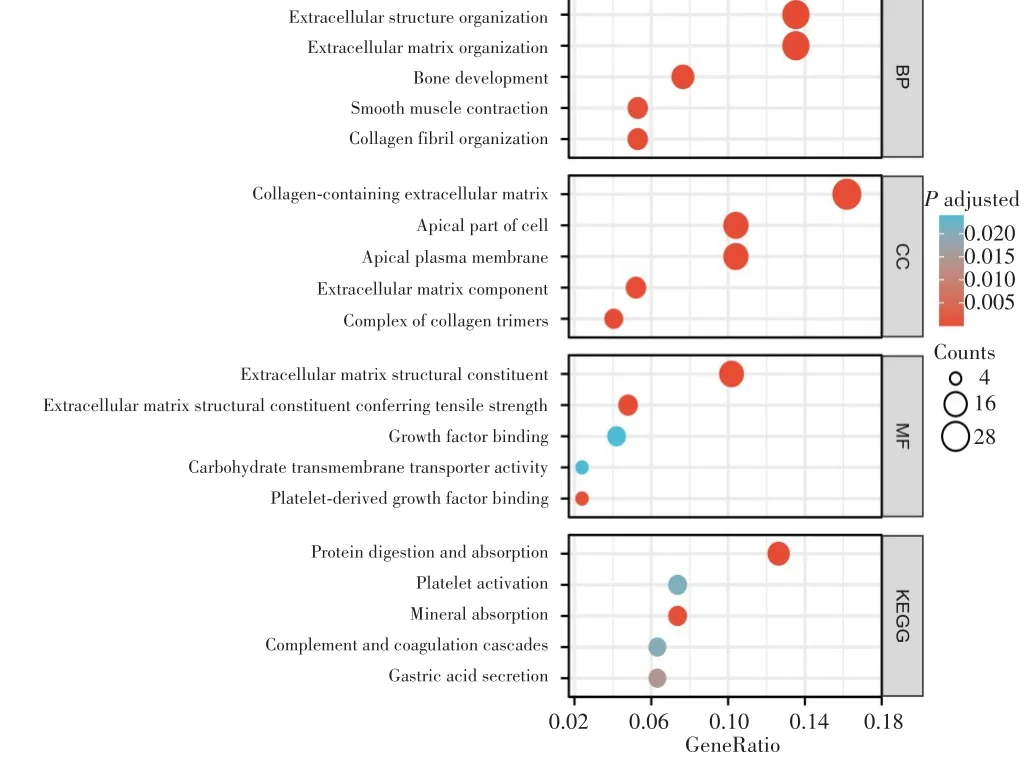
图2 差异基因的GO和KEGG分析结果Fig.2 GO and KEGG analysis of differentially expressed genes
2.3 差异基因的蛋白互作网络构建
将175个差异基因用于绘制蛋白互作网络(图3),差异基因共可富集出4个功能基因集,其中最大功能基因集包含的差异基因数远大于另外3个,且大多数由cytoHubba评分高的基因组成,推测该基因集在弥漫型胃癌的功能调控中占主导地位。最大基因集包括LUM、BGN、COL1A2、ADAMTS2、COL3A1、COL4A1、COL12A1、COL5A2、SPARC、NID2、COL1A1、CDH11和COL6A3,其中LUM是最大基因集中的核心基因,也同时是cytoHubba中的高评分基因。结合Hallmarks进一步分析,结果显示,12/13的基因参与EMT过程,因此称该基因集为EMT相关基因集。
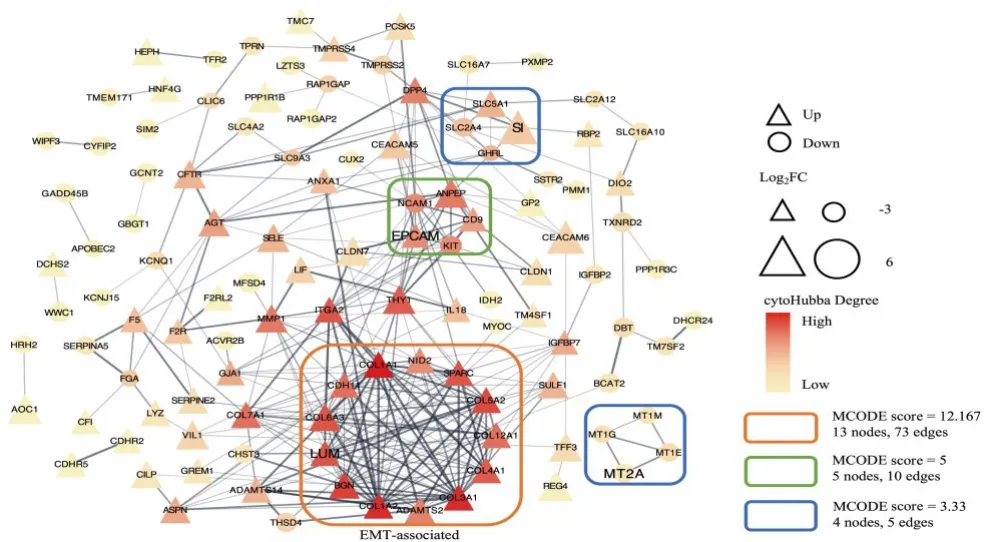
图3 差异基因的蛋白互作网络Fig.3 Protein-protein interaction network of differentially expressed genes
2.4 EMT相关基因的表达
分析TCGA数据库中EMT相关基因集的表达,结果显示,在弥漫型胃癌中,COL6A3较癌旁正常组织、肠型胃癌组织和混合型胃癌组织高表达(P<0.05);LUM、COL1A2、SPARC、COL1A1、CDH11、COL3A1、NID2、BGN和COL4A1较癌旁正常组织和肠型胃癌组织高表达(P<0.05),但与混合型胃癌比较差异无统计学意义(P>0.05);ADAMTS2、COL12A1和COL5A2较癌旁正常组织高表达(P<0.05),但与肠型和混合型胃癌组织比较差异无统计学意义(P>0.05),见图4。这一结果进一步确定了COL6A3、LUM、COL1A2、SPARC、COL1A1、CDH11、COL3A1、NID2、BGN和COL4A1是弥漫型胃癌中EMT相关的关键基因。
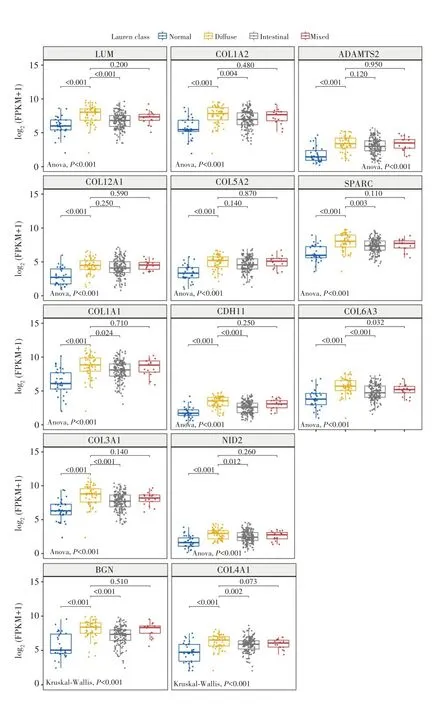
图4 EMT相关基因在胃癌组织中的表达Fig.4 Expression of EMT-associated genes in gastric cancer tissues
2.5 预后相关基因的筛选及表达量验证
基于TCGA数据库中的55例弥漫型胃癌患者的临床随访资料,分析EMT相关基因表达水平与弥漫型胃癌总生存期的关系。结果显示,LUM基因高表达的弥漫型胃癌患者总生存期缩短(P=0.043),其他EMT相关基因表达水平与弥漫型胃癌总生存期无关,见图5A。说明在EMT相关基因集中,LUM是弥漫型胃癌潜在的预后相关核心基因。
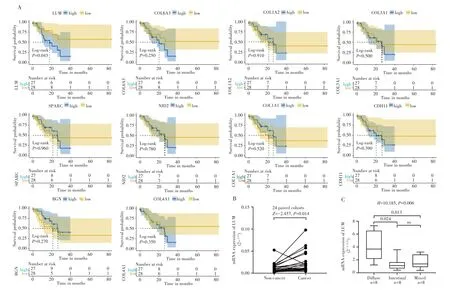
图5 EMT相关基因的预后分析及核心基因LUM表达量的验证Fig.5 Prognostic analysis of EMT-associated gene and verification of hub gene LUM expression
收集24例初诊胃癌患者样本,对LUM的mRNA相对水平表达量进行验证,结果与测序结果一致,均表现为LUM在胃癌组织中高表达(P=0.014),见图5B;且LUM在弥漫型胃癌组织中的表达高于肠型和混合型(均P<0.05),见图5C。
3 讨论
2015年,亚洲癌症研究组(ACRG)在Nature Medicine上将胃癌分为4种分子亚型:微卫星不稳定型(MSI)、上皮间质转化型(MSS/EMT)、p53活跃型(MSS/p53+)和p53不活跃型(MSS/p53-),其中弥漫型胃癌主要存在于MSS型中,且预后最差的MSS/EMT型中超过80%属于弥漫型[13]。一项关于弥漫型胃癌的蛋白质组学研究基于差异蛋白的聚类分析将弥漫性胃癌分为3种亚型:PX1(细胞周期型),PX2(EMT型)和PX3(免疫过程增强亚型),再次证实了EMT过程在弥漫型胃癌中的重要地位[14]。这些分子表征加深了对弥漫型胃癌内分子异质性的理解,表明弥漫型胃癌在基因背景稳定下可能通过EMT相关机制推动癌细胞的恶性生物学行为。
EMT是恶性肿瘤的特征之一,常以渐进、可逆的形式发生,并推动细胞获得干性特征、转移潜能和治疗抗性[15]。细胞外基质由于能提供肿瘤细胞黏附和迁移的支持结构,在肿瘤发展过程中往往受到一系列信号通路调控,因此其结构成分和机械性能发生重塑,从而影响包括EMT在内的各种关键细胞事件[16-17]。本研究的GO分析结果提示弥漫型胃癌中存在多种方式参与细胞外基质相关的生物学过程,其核 心 基 因 集 中COL6A3、LUM、COL1A2、SPARC、COL1A1、CDH11、COL3A1、NID2、BGN和COL4A1的表达均高于正常组织,且在弥漫型胃癌中的表达高于肠型。在 EMT 相关基因集中,COL6A3[18]、COL1A2[19]、COL3A1[20]、COL1A1[21]和 COL4A1[22]是胶原蛋白家族基因,主要参与细胞外基质组成,诱导肿瘤细胞发生EMT转变。SPARC可作为肿瘤抑制或启动因子,在肿瘤进展的不同阶段重塑细胞外基质,并通过EMT过程促进肿瘤侵袭和转移[23]。CDH11[24]和 BGN[25]也可以通过不同通路参与EMT过程。因此认为,本研究中的EMT相关基因集可能作为弥漫型胃癌转移和预后的生物标志物。
进一步基于TCGA数据库和临床样本进行验证,发现LUM是EMT相关基因集中的核心基因,且在弥漫型胃癌中高表达,且LUM是EMT相关基因集中唯一影响弥漫型胃癌总生存期的基因,因此推测LUM是弥漫型胃癌的核心基因。LUM是参与细胞外基质组织构成和细胞外信号传导的关键介质,也是Ⅱ类富含亮氨酸重复序列的小分子蛋白聚糖的家族成员(small leucine-rich proteoglycans,SLRPs),在肝脏、心脏、肾脏和肠等多种组织中广泛表达,可促进癌细胞增殖、侵袭和转移[26-30]。近年来在多种癌症中进行了关于LUM机制的研究,例如,在膀胱癌细胞中敲除LUM可通过失活MAPK信号通路传导抑制细胞增殖和迁移[31]。在乳腺癌中,LUM能抑制CD44表达,并下调包括FAK、ERK1/2 MAPK 42/44和AKT等整合素下游通路[32]。在头颈鳞状细胞癌的耐药研究中,化疗耐受组中LUM的表达显著上调,而沉默LUM表达可显著增强顺铂介导的细胞凋亡[33]。在胃癌中,LUM能通过激活整合素β1-FAK信号通路促进细胞增殖、侵袭和迁移[34]。综合以上研究结果,说明LUM可能作为癌基因促进弥漫型胃癌的发生进展。
综上所述,本研究从转录组测序、RT-qPCR以及TCGA数据库角度均证实LUM在弥漫型胃癌中表达增强,且弥漫型胃癌的转录组特征表现为EMT相关基因集处于优势地位,其核心基因LUM高表达患者总生存期缩短,因此LUM可能是弥漫型胃癌潜在的预后标志物和治疗靶点。但本研究受限于单中心小样本,优势基因集在弥漫型胃癌中的作用及机制仍需进一步研究证实。

