种子耐脱水性的生理及分子机制研究进展
宋松泉,刘军,唐翠芳,程红焱,王伟青,张琪,张文虎,高家东
种子耐脱水性的生理及分子机制研究进展
宋松泉1,2*,刘军1*,唐翠芳3,程红焱2,王伟青2,张琪1,张文虎1,高家东1
1广东省农业科学院农业生物基因研究中心/广东省农作物种质资源保存与利用重点实验室,广州 510640;2中国科学院植物研究所,北京 100093;3深圳前海国垦大地基金管理有限公司,广州 510630
耐脱水性是指生物体或组织在丧失所有或几乎所有细胞水分的状态下而不产生不可逆损伤的存活能力。种子的耐脱水性是植物在长期进化过程中保证物种生存和繁衍的适应性机制,在植物种子(质)资源保存中起关键作用。种子的耐脱水性是一个复杂的性状,其分子机理至今尚不清楚。为此,本文综述了种子耐脱水性的生理及分子机制的研究进展。研究发现,正常性种子的耐脱水性是在发育过程中逐渐形成的,在生理成熟期达到峰值;顽拗性种子在整个发育过程中对脱水敏感,不具有成熟脱水的发育阶段。成熟的正常性种子在吸胀初期保持对重新脱水的耐性,随着萌发进程,种子的耐脱水性逐渐下降,最后完全丧失;在萌发初期,种子的耐脱水性可以重建,不同组织具有不同的耐脱水性。种子和胚的耐脱水性程度与其线粒体的呼吸活性下降呈负相关性,顽拗性种子的呼吸活性高于正常性种子。脱水过程中,耐脱水性胚(轴)的H2O2含量、超氧阴离子自由基(·O2-)的产生速率和硫代巴比妥酸活性产物的含量显著低于脱水敏感性胚(轴),而活性氧清除(包括酶促和非酶促)系统的活性明显高于脱水敏感性胚(轴)。种子成熟过程中,胚胎发育晚期丰富(LEA)蛋白、小分子量热休克蛋白和非还原性棉子糖家族寡聚糖的积累与耐脱水性的形成密切相关。B3转录因子的AFL亚家族(包括ABI3(ABA INSENSITIVE 3)、FUS3(FUSCA3)和LEC2(LEAFY COTYLEDON 2))通过正向调控贮藏物和保护性蛋白的积累增加种子(胚)的耐脱水性。在整个种子发育过程中,DNA甲基化水平显著增加,随后在种子萌发过程中逐渐降低;与发育早期阶段的胚和幼苗相比,成熟胚具有较高水平的基因组甲基化。在种子中,平行的ABA和DOG1(DELAY OF GERMINATION 1)信号转导途径激活棉子糖家族寡聚糖的合成、LEA基因和HSP基因的表达,从而调控耐脱水性的起始和向休眠转变。最后,本文提出了该领域需要进一步研究的科学问题,包括利用种子及其组织的不同耐脱水性重建其模式研究系统;种子的萌发能力、耐脱水性和休眠特性都是在发育过程中起始和完成的,它们之间的相互关系仍不清楚;种子中同时存在核心ABA信号途径和DOG1信号途径,这两条途径在ABI3或者ABI3下游汇合,在种子脱水过程中哪条途径优先响应?又是如何协调?本文将为全面理解种子耐脱水性的生理及其分子机制、提高农作物的胁迫抗性与产量、改善资源库的贮藏条件和长期保存植物种子(质)资源提供参考。
抗氧化系统;耐脱水性;遗传调控;种质资源的长期保存;代谢活性;保护性物质
0 引言
被子植物的种子发育起始于双受精,终止于成熟脱水(maturation drying)。在胚胎发生过程中,受精的卵细胞发育成为胚,而2个极核与另一个精细胞融合发育成胚乳。早期的成熟过程包括细胞扩大和贮藏物积累;在成熟后期,种子感受未知的生理和/或环境信号,开始丧失水分,进入代谢不活跃或者静止状态[1]。种子的贮藏活力和寿命主要取决于种子的含水量和贮藏温度,成熟干燥的种子能在低温和低含水量条件下长期存活。因此,种子的耐脱水性是植物种子(质)资源长期保存的关键,而种质资源的保存又是农作物优良种质创制和新品种选育的前提。
成熟脱水是种子发育的最后阶段,它能提高种子的萌发能力,终止贮藏物的积累,以及使种子胚进入“玻璃态(glassy state)”,从而导致代谢静止、增加贮藏寿命和对逆境条件的抵抗能力[2]。耐脱水性(desiccation tolerance)是指生物体或组织在丧失所有或几乎所有细胞水分(包括自由水和束缚水)的状态下而不产生不可逆损伤的存活能力[3]。种子的耐脱水性是植物(特别是一年生和二年生植物)在环境胁迫中保证物种生存和繁衍的适应性机制。在种子成熟后期,大多数物种的种子开始丧失水分,收获干燥后,达到5%—10%含水量(除了特别说明外,均以鲜重为基础)或低于0.1 g·g-1(H2O/DW,干重为基础),该程度的水分丧失不会引起正常性种子(orthodox seed)死亡,因此,被认为耐脱水性[2]。耐脱水性也包括种子成功保持重新水合和萌发的能力[3-4]。
根据种子在发育后期是否具有成熟脱水过程以及成熟后对脱水和低温的反应,将种子分为正常性种子和顽拗性种子(recalcitrant seed)。正常性种子在母体植株上经历成熟脱水,种子脱落时含水量较低,一般能被进一步干燥,含水量达到5%以下而不发生伤害,并根据贮藏温度和种子含水量能预测其贮藏寿命。顽拗性种子不经历成熟脱水,种子脱落时含水量相对较高,在整个发育过程中不耐脱水,通常对低温敏感,在适合正常性种子贮藏的条件下,顽拗性种子的贮藏寿命通常只有几天到几周(取决于物种)[5-7]。此外,还有一类种子(如番木瓜()、大薸())表现出中间性贮藏行为(intermediate storage behavior),在相对低的含水量条件下能够存活(0.2 g·g-1),但不能像正常性种子(<0.1 g·g-1)一样耐受更低的水分丧失,在脱水状态下,对低温敏感或不敏感[4, 6, 8]。
研究表明,许多过程或机制可以授予或提高种子的耐脱水性,不同的过程可能在不同的水合水平对水分丧失起保护作用,包括细胞内脱分化与代谢关闭、活性氧(reactive oxygen species,ROS)的产生与清除以及保护性分子等作用[7, 9-11]。近年来,随着多组学(-omics)和现代分子生物学技术的进步,种子耐脱水性的分子机制研究取得了重要进展[4, 11-14]。本文主要从种子发育过程中耐脱水性的变化、耐脱水性与代谢活性下降、抗氧化系统和保护性物质在种子耐脱水性中的作用以及耐脱水性的遗传调控等方面进行综述,以期为深入研究种子耐脱水性的分子机制、改善作物的耐旱性以及种质资源的长期保存提供参考。
1 种子发育过程中耐脱水性的变化
1.1 耐脱水性的产生
种子耐脱水性是在发育过程中逐渐形成的,在生理成熟期达到峰值。玉米()种子的鲜重和干重随着发育进程逐渐增加,分别在授粉后45和50 d达到最大值;而种子的含水量则随着发育逐渐下降[15-16]。在种子鲜重达到峰值之前,含水量的下降主要是由于贮藏物的积累,而在种子干重达到峰值之后,含水量的下降是由成熟脱水所引起。在授粉后45 d,玉米种子开始形成萌发能力,随着发育种子的萌发能力逐渐增强,在授粉后65 d达到峰值。玉米种子在授粉后24 d不耐脱水,此后开始形成耐脱水性,且随着发育逐渐提高;直至授粉后40 d,种子的耐脱水性(以萌发率表示)达到75%[16]。玉米胚在授粉后35—40 d开始形成耐脱水性,授粉后52 d达到100%耐脱水性[17]。玉米胚形成耐脱水性的时间要晚于整粒种子[16-17]。此外,种子和胚形成耐脱水性的时间进程除了与物种的遗传特性有关外,还与种子发育时的环境条件有关。
研究表明,耐脱水性的形成与脱水速率有关。从授粉后26 d的法国菜豆(French bean)或普通菜豆(common bean)豆荚中离体的种子,若脱水在几小时内完成,种子则不会存活;然而,若在一周内缓慢脱水,种子则能够萌发[2]。Huang等[18]发现快速脱水对玉米胚的伤害大于缓慢脱水。如玉米胚从2.53 g·g-1缓慢脱水到0.16 g·g-1时,其存活率为94%;而从2.53 g·g-1快速脱水到0.17 g·g-1时,其存活率仅为5.3%。50%玉米胚因快速脱水而死亡的含水量(water content50,W50)为0.39 g·g-1,而在缓慢脱水的玉米胚中未发现W50;其原因可能是在缓慢脱水过程中,种子或胚中会形成一些保护性化合物,从而增加它们的存活率[2]。与正常性种子(胚)对脱水速率的反应相反,缓慢脱水对顽拗性黄皮()胚轴的损伤反而更大。如当含水量从0.82 g·g-1快速脱水到0.13 g·g-1时,黄皮胚轴的存活率为100%;而含水量从0.82 g·g-1缓慢脱水到0.24 g·g-1时,其胚轴的存活率仅为52.4%。缓慢脱水和快速脱水引起的黄皮胚轴W50分别为0.24和0.09 g·g-1[18]。Black等[2]提出,在缓慢脱水时,顽拗性胚轴处于中间含水量的时间较长,但此时发生的代谢是不受调控的,可能发生自由基(free radical)介导的破坏性反应。
1.2 耐脱水性的丧失
成熟种子吸胀初期保持对重新脱水的耐性,随着萌发进程,种子的耐脱水性逐渐下降,最后完全丧失;但耐脱水性丧失的时间进程与物种的遗传特性和种子吸胀/萌发时的环境条件有关。玉米种子吸胀过程中,胚在吸胀初期的含水量迅速增加,随后出现一个缓慢的吸水时期(平台期),然后进一步迅速增加[17]。玉米胚在吸胀初期耐脱水;吸胀28 h后,胚根伸出,胚的耐脱水性下降;吸胀72 h时,胚的耐脱水性完全丧失[17]。在豌豆()种子萌发过程中,种子和胚轴的耐脱水性逐渐丧失,10%和50%的种子和胚轴被脱水致死的含水量明显增加,种子的耐脱水性明显高于胚轴[19]。吸胀15 h的豌豆种子脱水到0.1 g·g-1时,能保持其最大的存活能力,随着吸胀时间的延长,种子的耐脱水性降低,当吸胀时间为48 h时,种子的存活率为0[20]。
Wang等[13]根据胚根从种皮伸出的长度(小于1 mm(S1)、2—3 mm(S2)和4—5 mm(S3)),评估了豌豆种子在S1—S3萌发时期胚轴不同组织的耐脱水性。S1时期,上胚轴、下胚轴和胚根在脱水后的存活率分别为93.3%、94.6%和37.9%;S2时期,分别下降到65.9%、37.5%和0;S3时期,上胚轴的存活率仅为20%,下胚轴和胚根则完全丧失耐脱水性。2,3,5-三苯基氯化四氮唑(2,3,5-triphenyltetrazolium chloride)染色表明,在同一萌发时期,萌发豌豆种子的不同胚轴组织表现出不同的耐脱水性:上胚轴(epicotyl,E)>下胚轴-E(hypocotyl-epicotyl,将下胚轴分为两部分,与上胚轴连接的一部分)>下胚轴-R(hypocotyl-radicle,与胚根连接的一部分)>胚根(radicle,R)[13]。经聚乙二醇(polyethylene glycol,PEG)处理,能重新诱导S2时期豌豆种子不同胚轴组织的耐脱水性,即显著增加上胚轴和下胚轴、稍微增加胚根脱水后的存活率[13]。此外,在花椰菜()种子萌发过程中,也观察到子叶的耐脱水性>下胚轴>胚根[2]。
在发育过程中,尽管一些顽拗性种子由于贮藏物的积累,其种子或胚(轴)的含水量会降低,但在成熟和脱落时,其含水量仍然较高,在整个发育过程中和脱落后不耐脱水。如顽拗性荔枝()[21]、非洲鹧鸪花()[5]、箭毒木()[22]和黄皮[23-24]。箭毒木[22]和黄皮[23]胚轴的耐脱水性显著高于完整的种子。
2 耐脱水性与代谢活性下降
Pammenter等[7]提出,正常性种子具有一些保护性过程或机制赋予种子的耐脱水性,其中,重要的过程是代谢关闭和细胞内脱分化;然而,顽拗性种子不经历细胞内脱分化或任何明显的代谢关闭,在其脱落时,胚(轴)保持代谢活性,从发育模式逐渐转变为与萌发有关的过程。在水分缺乏条件下,细胞存活的一个重要因素是代谢活性和呼吸速率的降低。代谢活性与种子或胚(轴)的脱水敏感性有关,因此,代谢活性的降低是耐脱水性的特征[25]。
线粒体是种子中的主要细胞器,主要功能是为细胞提供能量,以及为其他大分子生物合成提供碳骨架。代谢活性下降主要表现为线粒体的功能降低。Leprince等[3]提出,形成耐脱水性的一个重要因素是减少对ATP的需求。随着玉米种子耐脱水性的形成,胚线粒体的耗氧速率、细胞色素c氧化酶(cytochrome c oxidase,CCO;线粒体的标记酶)和NAD+-苹果酸脱氢酶(NAD+-malate dehydrogenase,NAD+-MDH;线粒体的主要功能酶)活性下降,表明线粒体的活性降低[15]。
随着脱水程度的提高,箭毒木胚轴的存活率迅速下降,而未成熟玉米胚的存活率则缓慢降低[26]。为了解它们对脱水的反应,对其线粒体的呼吸速率、CCO活性和CCO活性滞后期(CCO activity latency,线粒体外膜完整性的指标)在脱水过程中的变化进行测定。新采收的箭毒木胚轴线粒体的基础呼吸速率(basic respiratory rate,状态I)和呼吸速率(respiratory rate,状态Ⅲ)显著高于新采收的玉米胚线粒体[26]。箭毒木胚轴线粒体的基础呼吸速率在脱水早期迅速降低,而玉米胚线粒体则是随着脱水缓慢下降。当箭毒木胚轴和玉米胚的含水量降低50%时,其线粒体的基础呼吸速率分别下降70%和17%。箭毒木胚轴和玉米胚线粒体呼吸速率的变化与基础呼吸速率类似,即箭毒木胚轴线粒体的呼吸速率随着脱水迅速下降,而玉米胚线粒体的呼吸速率则相对缓慢降低;在整个脱水过程中,箭毒木胚轴线粒体的呼吸速率持续高于玉米胚线粒体[26]。
呼吸速率由细胞色素(cytochrome,Cyt)c途径、交替氧化酶(alternative oxidase,AOX)途径和其他耗氧途径组成。利用Cyt c途径抑制剂氰化钾(KCN)和AOX途径抑制剂水杨基羟肟酸(salicylhydroxamic acid,SHAM)分析脱水过程中不同呼吸途径在呼吸速率中的变化。箭毒木胚轴线粒体中,KCN抑制的呼吸速率占整个呼吸速率的32.5%,并随着脱水而迅速下降,当胚轴含水量为0.400 g·g-1时,其呼吸速率降低到0。玉米胚线粒体中,KCN抑制的呼吸速率占整个呼吸速率的66.7%,在脱水初期下降,随后上升。玉米胚线粒体由KCN抑制的呼吸速率远高于箭毒木胚轴的线粒体[26]。另外,在箭毒木胚轴线粒体中,SHAM抑制的呼吸速率占整个呼吸速率的58%,在脱水早期增加,随后维持在一个相对恒定的水平。与箭毒木胚轴线粒体相反,玉米胚线粒体中,由SHAM抑制的呼吸速率占整个呼吸速率的25%,随着脱水,直到胚轴含水量为0.113 g·g-1,呼吸速率略有下降;当胚含水量为0.101 g·g-1时,呼吸速率迅速下降到0。玉米胚线粒体中由SHAM抑制的呼吸速率远低于箭毒木胚轴的线粒体[26]。除了KCN和SHAM抑制的耗氧(呼吸)途径外,在箭毒木胚轴和玉米胚线粒体中还存在其他的耗氧途径,它们在整个呼吸速率中所占的比例随着脱水程度的提高而增加,但箭毒木胚轴线粒体的其他耗氧速率在脱水过程中低于玉米胚线粒体[26]。
在对3种咖啡物种种子进行转录组和蛋白质组研究中,Stavrinides等[27]表明,种子中与能量产生相关的基因表达减少。下调的基因与线粒体电子传递链(electron transport chain,ETC)复合物(ATP合酶(ATP synthase)、NADH脱氢酶复合物I(NADH dehydrogenase complex I,CⅠ)、辅酶Q-细胞色素bc1复合物(ubiquinol-cytochrome bc1 complex,CⅢ)、CCO(CⅣ))以及三羧酸循环(tricarboxylic acid cycle)(异柠檬酸脱氢酶(isocitrate dehydrogenase)、丙酮酸脱氢酶(pyruvate dehydrogenase)、琥珀酰辅酶A连接酶(succinyl-CoA ligase)、二氢硫辛酸脱氢酶(dihydrolipoyl dehydrogenase))有关,并且,在线粒体能量代谢调控方面,耐脱水性和脱水敏感性种子之间存在显著差异。内膜移位酶亚基(translocase inner membrane subunit,TIM44-2)和线粒体剪接因子(mitochondrial splicing factor,OTP439)在脱水敏感的中粒咖啡()种子中表现出更高的表达,分别参与基础细胞过程(如蛋白输入)和细胞器转录后过程。在耐脱水的小粒咖啡()和欧基尼奥伊德斯种咖啡()种子中,一些调控能量过程的重要基因(如、和)表达量下调。在脱水敏感的中粒咖啡种子成熟后期,一些与线粒体呼吸ETC复合物有关的基因表达量增加。因此,维持高能量代谢可能是顽拗性种子脱水敏感性的原因之一。脱水时,耐脱水种子表现出呼吸速率稳定下降,脱水敏感性种子则急剧下降;同时,中粒咖啡的呼吸速率高于小粒咖啡和欧基尼奥伊德斯种咖啡[27]。在顽拗性欧洲板栗()种子中,也观察到类似结果,脱水敏感的子叶比脱水耐性的胚轴表现出更高的呼吸速率[28]。
3 抗氧化系统对种子脱水的保护作用
3.1 活性氧的产生
植物线粒体ETC由复合物Ⅰ(complex Ⅰ,CI)、复合物Ⅱ(complex Ⅱ,CⅡ;琥珀酸脱氢酶(succinate dehydrogenase))、复合物Ⅲ(complex Ⅲ,CⅢ)和复合物Ⅳ(complex Ⅳ,CⅣ)组成。电子在传递给末端氧化酶之前,常常会漏出呼吸链,并与O2反应生成超氧阴离子自由基(superoxide anion radical,·O2-),这是线粒体ROS的主要来源。线粒体ETC的4种复合物中,CⅠ和CⅢ是产生ROS的部位[29]。此外,植物的ETC还包括5种在哺乳动物线粒体中不存在的酶——1种AOX和4种NAD(P)H脱氢酶,这些脱氢酶与黄素蛋白(flavoprotein)的特性非常相似,也是产生ROS的潜在位点[29]。
ROS是植物氧化还原代谢的组成性产物,在植物组织中不断地生成,参与细胞中多个正常的生理过程[30-32]。在多数情况下,·O2-是ROS的主要类型,易于转化为过氧化氢(H2O2)和其他过氧化物,也易于参与Fenton反应,产生羟基自由基(hydroxyl radical,·OH)[33]。当产生的ROS超过抗氧化保护性系统的清除活性时,它们在组织中的含量增加,并产生氧化胁迫[34]。在分子水平上,氧化胁迫表现为脂质过氧化作用增强、膜完整性被破坏、酶失活、蛋白和核酸被氧化降解以及抗氧化库的耗尽[11-12]。玉米种子发育过程中,在形成耐脱水性的同时,脱水胚的H2O2含量略有下降;但在吸胀过程中,随着耐脱水性的丧失,H2O2的含量显著增加(2.2倍)[17]。在箭毒木种子脱水过程中,种子和胚轴中·O2-的产生速率逐渐增加。如当胚轴含水量从1.42 g·g-1降低到0.26 g·g-1时,·O2-的产生速率从2.85 nmol·g-1DW增加到6.88 nmol·g-1DW。胚轴的H2O2含量也随着脱水而显著增加,但种子的H2O2含量在脱水初期和中期明显增加,后期略有下降[22]。脂质过氧化对膜的结构和功能具有很大的伤害,被认为是种子生活力丧失的主要原因之一[1]。硫代巴比妥酸活性产物(thiobarbituric acid reactive substance,TBARS)是脂质过氧化的产物。玉米种子发育过程中,随着耐脱水性的形成,胚中TBARS的含量显著下降(3.7倍);成熟玉米种子吸胀过程中,随着耐脱水性的丧失,胚中TBARS的含量显著增加(2.7倍)[17]。此外,在脱水过程中,顽拗性荔枝种子[21]、黄皮[23]、非洲鹧鸪花[5]以及箭毒木[22]种子和胚轴中的TBARS含量显著增加。
3.2 抗氧化酶系统
酶促抗氧化系统包括超氧化物歧化酶(superoxide dismutase,SOD)、愈创木酚过氧化物酶(guaiacol peroxidase)、抗坏血酸过氧化物酶(ascorbate peroxidase,AsA-POD)、谷胱甘肽过氧化物酶(glutathione peroxidase)、过氧化氢酶(catalase,CAT)、单脱氢抗坏血酸还原酶(monodehydroascorbate reductase,MDHAR)、脱氢抗坏血酸还原酶(dehydroascorbate reductase,DHAR)和谷胱甘肽还原酶(glutathione reductases,GR)[35-36]。这些酶在·O2-、H2O2和单线态氧(single oxygen,1O2)的解毒中起重要作用[5, 33]。
黄皮胚轴和子叶中的SOD活性随着种子含水量的下降先增加再降低;当种子生活力完全丧失时,胚轴和子叶中的SOD活性分别比未脱水的对照下降了12%和73%;胚轴的SOD活性比子叶高,在种子脱水初期,胚轴SOD活性的增加高于子叶[23]。脱水过程中,非洲鹧鸪花胚轴的SOD、AsA-POD、CAT、GR和DHAR活性下降[5]。箭毒木种子的SOD、CAT和DHAR活性在脱水初期增加,然后随着进一步的脱水而降低,而AsA-POD和GR的活性随着脱水逐渐降低;然而,在胚轴中,这些酶的活性在脱水初期增加,然后随着进一步的脱水而下降[22]。以上结果表明,抗氧化酶的活性与耐脱水性的变化密切相关。
从发育的玉米胚中分离线粒体,脱水使授粉后21 d和27 d胚线粒体中的SOD、AsA-POD和MDHAR活性以及授粉后21 d胚线粒体中的DHAR活性显著降低,但对授粉后50 d胚线粒体中的SOD、AsA-POD、MDHAR和DHAR活性没有影响;相反,脱水增加授粉后21、27和50 d胚线粒体中的GR活性[15]。王伟青等[24]通过研究黄皮种子脱水过程中线粒体ROS清除酶活性的变化,发现胚轴线粒体中的SOD、AsA-POD和GR活性随着脱水迅速下降,但DHAR活性在脱水初期增加,随后下降;子叶线粒体中的SOD、DHAR和GR活性在脱水初期增加,随后下降,AsA-POD活性随着脱水逐渐降低。胚轴线粒体的SOD、DHAR和AsA-POD活性显著高于子叶线粒体,而子叶线粒体的GR活性明显高于胚轴线粒体[24]。以上结果表明,线粒体中的抗氧化酶活性与胚(轴)的耐脱水性密切相关。
蛋白质组研究表明,在比较耐脱水的豌豆下胚轴-E中,一些与依赖还原型谷胱甘肽(reduced glutathione,GSH)解毒作用有关的蛋白和过氧化物酶高丰度积累;与此一致,脱水后下胚轴-E中积累的H2O2水平非常低[13]。PEG处理后,胚根和下胚轴-R中2种谷胱甘肽过氧化物酶(GPX1和GPX8L)和3种过氧化物酶(PER15A、PER15B和PER73)的丰度均增加,而GPX8L和PER73也在PEG处理前于下胚轴-E中高丰度积累。结果表明,依赖GSH的解毒酶和过氧化物酶活性的增加可能有助于耐脱水性在胚根和下胚轴中的重建[13]。WANG等[16]也发现玉米种子的成熟脱水和成熟前强迫脱水均导致一些与ROS解毒相关的蛋白丰度增加,如谷胱甘肽S-转移酶(glutathione S-transferase,GST;胚乳中)、1-Cys-过氧化物酶-PER1(胚乳中)、线粒体醛脱氢酶2(mitochondrial aldehyde dehydrogenase 2,胚中)和乳酰谷胱甘肽裂解酶(lactoylglutathione lysase,也称为乙二醛酶I(glyoxalase I))。
此外,氧化还原过程参与成熟种子的代谢调控和耐脱水性的建立[37]。氧化还原状态能够被抗氧化酶和含有二硫键的化合物谷胱甘肽、过氧化氧化还原蛋白(peroxiredoxin,Prx)、硫氧还蛋白(thioredoxin)和谷氧还蛋白(glutaredoxin)调控[38]。Ratajczak等[39]发现,正常性挪威槭()和顽拗性欧亚槭()种子中存在氧化还原电位的差异。在种子发育过程中,1-Cys-Prx在挪威槭种子中被还原,在欧亚槭种子中被氧化。
3.3 非酶抗氧化系统
非酶抗氧化系统包括低分子量抗氧化剂α-生育酚(α-)、类胡萝卜素(carotenoid)、AsA和谷胱甘肽[40-41]。还原型AsA能够直接与ROS相互作用,并参与其他低分子量抗氧化剂生育酚和谷胱甘肽的还原。生育酚是细胞膜的普遍保护剂,参与ROS的猝灭,从而防止脂质的非酶促氧化[40, 42]。谷胱甘肽可以直接作为一种ROS的猝灭分子或作为与ROS解毒有关酶的电子供体(例如GST)[43],与ROS相互作用时,伴随着巯基的氧化和谷胱甘肽二硫化物(GS-SG)的转化。GR存在于干种子中,当种子水合时被迅速激活,将氧化的谷胱甘肽还原为巯基形式[41]。谷胱甘肽的还原态与氧化态的比例被认为是种子生活力的一种标记[43-44]。
2.5—10 mmol·L-1AsA处理可显著提高非洲鹧鸪花胚轴的耐脱水性和胚轴中SOD、AsA-POD、CAT和GR的活性,降低胚轴电解质渗漏率和TBARS含量[5]。AsA和甘露醇(mannitol)处理也能降低黄皮种子的脂质过氧化作用,提高种子的生活力[23]。
4 保护性物质在耐脱水性中的作用
正常性种子耐脱水性的分子机制之一是保护性物质的积累,包括胚胎发育晚期丰富(late embryogenesis abundant,LEA)蛋白、小分子量热休克蛋白(small heat shock proteins,sHSP)和非还原性棉子糖(raffinose)家族寡聚糖等[4, 45-46]。
4.1 胚胎发育晚期丰富蛋白
LEA蛋白最初在棉籽发育后期被发现[47],普遍分布于植物界,从藻类(algae)、苔藓(moss)、蕨类植物(fern)到被子植物(angiosperm)和复苏植物(resurrection plant)[4, 48-49]。LEA蛋白的特征是富含甘氨酸残基,但半胱氨酸和色氨酸残基的含量非常低(甚至缺乏),丙氨酸、谷氨酸、赖氨酸/精氨酸和苏氨酸残基也比较丰富。由于这些主要的结构特征,LEA蛋白在较大的温度范围内是稳定的,并被高度水合。在细胞脱水过程中,LEA蛋白作为分子伴侣(molecular chaperon)起作用,即通过形成密集的氢键稳定其他蛋白和细胞膜的结构,它们也能稳定变性蛋白并促进其重新折叠[50]。LEA蛋白能隔离细胞脱水过程中积累的离子化合物,保护膜蛋白和酶免受离子浓度增加的毒害作用[50]。尽管LEA蛋白在所有的细胞组分中均能被检测到,但它们主要定位于细胞质中[51]。在水溶液中,大多数LEA蛋白以无序和随机卷曲的结构存在[50],然而,脱水导致它们重新折叠,形成具有两亲性α-螺旋(amphiphilic α-helix)结构。LEA蛋白的另一个特征是具有柔性结构元件(flexible structural element)(如聚脯氨酸II螺旋(polyproline Ⅱ helix)),能增加与DNA、RNA或者其他蛋白的结合[50, 52]。
在拟南芥()基因组中已经鉴定了51个编码LEA蛋白的基因[52]。依据Pfam蛋白结构域数据库,LEA蛋白可以分为8个家族:脱水素(dehydrin,DHN)、LEA-1–LEA-6和种子成熟蛋白(seed maturation protein,SMP)[52-53]。LEA蛋白和一些热休克蛋白(heat shock protein)属于兼职功能蛋白(moonlighting protein),它们具有几种相关的生理功能[53-54]。LEA基因在植物种子中的表达水平比营养器官中高。51个拟南芥LEA基因中,有21个仅在种子中表达,直接参与种子的形成[52]。在茶()中共检测到48个LEA基因,其中39个基因在种子成熟过程中起重要作用[55]。
沉默拟南芥第4组LEA蛋白中的3个蛋白足以引起水分缺乏敏感性[56]。玉米胚中LEA蛋白EMB564在胚成熟过程中积累,在萌发过程中下降,在耐脱水胚的脱水过程中增加[17]。玉米种子的成熟脱水和成熟前强迫脱水均引起LEA蛋白的高丰度积累[16]。蛋白保守结构域分析发现,1个脱水素、4个第1组LEA蛋白和5个第5组LEA蛋白在比较耐脱水性的豌豆胚轴中积累[13]。经PEG处理后,6个LEA蛋白在胚根和下胚轴-R中的丰度增加。在这些蛋白中,第1组(SLE2A)和第5组(SLE3)LEA蛋白以及DHN在PEG处理前也在上胚轴和下胚轴-E中高丰度积累。以上结果表明,第1组和第5组LEA蛋白可能在豌豆胚轴耐脱水性中起保护作用[13]。
顽拗性海榄雌()种子与正常性种子的比较研究表明,脱水敏感性种子缺乏LEA蛋白[57]。与顽拗性中粒咖啡比较,在中间性耐脱水的小粒咖啡和欧基尼奥伊德斯种咖啡种子成熟后期,一种专一的表达水平和蛋白含量明显增加[27]。Delahaie等[58]在脱水敏感的栗豆树()和耐脱水性的蒺藜苜蓿()研究中得出类似结果。比较蛋白组分析表明,6个LEA多肽SBP65、MP2、PM25、LEAm、EM1和EM6在脱水敏感的栗豆树中丰度较低[58]。在顽拗性茶种子转录组研究中,发现所有的LEA蛋白均被下调,表明这些基因的转录水平不足以导致脱水敏感性[59]。
通过对脱水敏感的栗豆树种子研究表明,2种脱水素BudCar5和DHN的同源物大量积累,但在耐脱水的蒺藜苜蓿中几乎不存在。在栗豆树种子中,脱水素占LEA蛋白的83%;而在蒺藜苜蓿种子中,脱水素仅占LEA蛋白的20%[58]。此外,在拟南芥种子中,脱水素的沉默不影响耐脱水性,1个或2个LEA基因的缺失也不影响耐脱水性[60]。以上结果表明,并不是所有的LEA蛋白都与耐脱水性的形成有关。
4.2 热休克蛋白
热休克蛋白系统的激活是生物体对胁迫因子最普遍的反应之一。根据其分子量,真核生物的HSP可分为六类:Hsp100、Hsp90、Hsp70、Hsp60、Hsp40和sHSP。在整个HSP超家族中,sHSP是分子量在12—43 kD范围内变化最大和最不均一的一组蛋白,它们具有高度保守的C端α-晶状体ACD),该结构域含有80—100个氨基酸残基[61]。与LEA蛋白类似,sHSP在种子成熟后期积累并存在于干种子中[36, 45, 61]。在种子中,sHSP具有促进新合成蛋白的折叠、三级结构受损多肽的重新折叠和抗氧化保护的功能[36, 45, 61-62]。sHSP最重要的特征之一是能够形成大的寡聚复合物(100—1 000 kD),在胁迫条件下,其大小可以达到5 000 kD[61]。显然,只有相对较大的寡聚复合物才具有高的伴侣活性,即能够与受损或错误折叠的蛋白相互作用并稳定其结构[61]。
拟南芥热胁迫转录因子(heat stress transcription factor,HSF)家族由A、B和C 3种类型的21个成员组成[63]。脱水触发向日葵()种子中和表达,相应的mRNA水平与水分丧失的程度相关[61]。研究表明,在蓖麻()种子成熟过程中,41个编码细胞质、线粒体和微粒体HSP基因进行表达,这些HSP含有α-晶状体[64]。Kaur等[62]报道,水稻()种子成熟后期的丰度显著增加。17.4 kD类型I HSP3在玉米胚成熟过程中积累,在萌发过程中下降,在耐脱水胚的脱水过程中增加[17]。玉米种子的成熟脱水和成熟前强迫脱水均引起sHSP的高丰度积累[16]。此外,与LEA基因类似,HSP基因的表达受关键休眠调控因子DOG1(DELAY OF GERMINATION 1)的介导[65]。
4.3 非还原性碳水化合物
非还原性碳水化合物主要有蔗糖(sucrose)、棉子糖和水苏糖(stachyose),它们的积累通过取代水分子来稳定细胞膜和细胞质蛋白的结构,是脱水过程中种子存活的重要方式之一[12]。相反,还原性单糖(reductive monosaccharide)的存在与种子生活力呈负相关[45]。可溶性糖主要是己糖的葡萄糖、果糖及其磷酸化衍生物,其丰度在种子成熟后期逐渐减少,而非还原性寡聚糖的积累增加[45-66]。在脱水过程中,寡糖和LEA蛋白能取代水分,成为磷脂极性头部磷酸基团的氢键伙伴(hydrogen-bonding partner)[45],导致膜从液晶相(liquid-crystalline phase)向凝胶相(gel phase)转变;同时,由于在水合外壳(hydrate shell)丧失后,脂质头部之间的范德华(van der Waals)相互作用加强,脂质的流动性降低[67]。脱水细胞的细胞质变为“玻璃态”,无细胞质向固态转变。因此,细胞质的黏性增加,水分和氧的扩散受到抑制,以及所有可能的化学反应速率显著降低[67-68]。在脱水过程中,用棉子糖和水苏糖代替水也能维持膜和蛋白稳定所需的氢键[36, 69]。由于多种细胞结构的物理化学性质发生变化,正常性种子能够在几十年内保持生活力[36]。然而,细胞质转变成为玻璃态的分子机制还不清楚,可能涉及一系列编码不同功能蛋白的基因[11]。
拟南芥转录组研究表明,与野生型相比,编码棉子糖途径的关键酶(包括蔗糖合酶(sucrose synthase)、UDP-D-半乳糖-4-差向异构酶(UDP-D-galactose-4- epimerase)、肌醇半乳糖苷合酶(galactinol synthase)和水苏糖合酶(stachyose synthase))基因的转录水平在脱水敏感的突变体种子中被显著下调[69];另一方面,转化酶(invertase)的基因被上调,表明D-葡萄糖和D-果糖没有被代谢成为棉子糖和水苏糖,这一结果被脱水敏感的突变体种子具有高水平的D-葡萄糖和D-果糖所证实[69]。Jing等[70]的研究得出类似结果,与野生型和耐脱水性增加的突变体种子相比,拟南芥突变体中肌醇半乳糖苷合酶和棉子糖合酶(raffinose synthase)的过表达与高浓度的肌醇半乳糖苷、棉子糖和水苏糖有关。在正常性象牙花()种子中也发现棉子糖的积累增加[71]。在耐脱水欧洲山毛榉()种子中,棉子糖含量增加以及蔗糖与棉子糖的比值降低[72]。糖的积累也是高度耐脱水性复苏植物的特征[73-74]。以上结果表明,在种子成熟后期,细胞质中,与棉子糖途径相关基因表达的增加和可溶性非还原糖的积累可能是种子形成耐脱水性的关键作用之一。
5 耐脱水性的遗传调控
5.1 转录水平的基因表达调控
耐脱水性和脱水敏感性种子在其成熟过程中的基因表达模式存在一些差异[27, 69],这些差异主要受ABA水平和信号转导途径的调控,涉及种子成熟和休眠过程的许多转录因子,但也有不依赖于ABA信号的机制[11-12, 75-76]。在发育的种子中,ABA含量通常在发育中期达到峰值,成熟脱水时下降。ABA的应用赋予未成熟合子胚(如大麦)和敏感体细胞胚(如紫花苜蓿和油菜)的耐脱水性。对ABA不敏感和不能合成ABA的拟南芥突变体产生脱水敏感性种子。在外源ABA存在时,调控种子成熟的转录激活因子ABI3(ABA INSENSITIVE 3)的异位表达导致胡萝卜()非胚性细胞的耐脱水性[2]。
在种子发育和成熟过程中,B3转录因子的AFL亚家族在ABA信号转导中起关键作用。AFL来源于、()和()一组基因的首字母,包括与CCAAT转录因子的NF-YB亚基结合的一个直系同源物(())[77-78],这些转录因子可以控制数千个与种子发育阶段有关的基因表达。AFL转录因子的主要特征是具有一个由七股链的β-折叠组成的B3 DNA结合结构域,它排列成一个开放的桶状,2个短的α-螺旋酶(α-helicase)位于桶的两端[79]。B3结构域与RY基序结合,能够激活属于成熟特异性的基因。此外,ABI3的活性还需要另一个结构域(B2结构域)的参与,该结构域与ABA反应元件结合[80]。AFL基因的突变引起种子贮存物缺乏,降低耐脱水性程度,以及导致难以获得休眠特性[81]。
最初鉴定的植物B3转录因子是玉米VP1(VIVIPAROUS-1),它是拟南芥ABI3转录因子的直系同源物。玉米突变体种子对ABA不敏感,表现为种子在穗上萌发,因此不能达到静止状态[81]。同样,拟南芥突变体种子也不能完成成熟程序。因此,它们具有低耐脱水性以及不能获得休眠[82]。一些突变体种子由于缺乏叶绿素分解代谢而保持绿色,导致生活力和贮藏寿命降低[36]。因此,ABI3很可能与花青素(anthocyanin)和叶绿素的积累减少有关。苜蓿和拟南芥共表达网络研究表明,ABI3与耐脱水性相关基因的关联程度最高[83]。因此,该转录因子是种子发育过程中的一个重要的转录调控因子。Delahaie等[58]将苜蓿突变体的耐脱水性缺失与LEA蛋白的积累减少相联系。拟南芥ABI3不仅是一个与耐脱水性形成相关的LEA蛋白的正调控因子(图1),而且也是营养组织专一的LEA蛋白的抑制因子[84]。To等[82]在拟南芥、和3突变体的表达分析中提出,ABI3调控另一个AFL转录因子FUS3,特别是在胚轴和子叶中。此外,ABI3也被其他AFL调控,包括它本身[82](图1)。、和突变体种子含有较低的贮藏蛋白和脂质含量[45, 85-86]。
Braybrook等[80]证明LEC2在种子发育最初阶段表达,直到成熟中期仍然保持活性。LEC2通过诱导其他AFL转录因子(如ABI3和FUS3)的表达,直接和间接地激活与种子成熟以及与脂质和种子贮藏蛋白(seed storage protein,SSP)积累相关的基因表达[80]。在种子发育早期阶段,需要LEC2激活FUS3表达;在成熟后期阶段,需要LEC2来维持ABI3的稳定表达水平[82]。通过正调控FUS3和ABI3的表达,LEC2也阻止花青素和叶绿素的积累[82]。LEC2直接调控WRI1(WRINKLED 1)的表达,在种子成熟过程中,WRI1在脂肪酸生物合成中起关键作用(图1),但WRI1似乎仅仅在胚的下胚轴中被LEC2转录诱导[87]。LEC2还调控与贮藏物积累相关的另一个基因的表达,该基因编码油素蛋白(oleosin)[80, 88]以及编码2S和12S贮藏蛋白[80](图1)。因此,的突变可能引起种子贮藏物的减少。此外,LEC2通过诱导EEL(ENHANCED EM LEVEL)碱性亮氨酸拉链(basic leucine zipper,bZIP)转录因子的表达,进而间接影响和的表达[80]。在拟南芥中,bZIP转录因子是这些EM蛋白的负调控因子,EEL转录因子与EM正调控因子ABI5竞争其启动子位点。在成熟后期和脱水过程中,当LEC2含量较低时,EEL的数量下降,耐脱水性相关蛋白EM1和EM6的表达被ABI5诱导。因此,LEC2在种子耐脱水性形成中的主要作用似乎是调控与贮藏物积累相关的基因[11]。LEC1激活LEC2和FUS3,而LEC2也激活LEC1和FUS3[89-90]。

FUS3抑制TTG1(TRANSPARENT TESTA GLABRA 1)转录因子,一个与脂肪酸和贮藏蛋白生物合成相关基因的负调控因子;以及正调控脂肪酸生物合成诱导因子WRI1(WRINKLED 1);因此,FUS3间接地正向影响贮藏物的积累。FUS3也在子叶的两侧调控ABI3表达。LEC2调控其他B3转录因子FUS3和ABI3,阻止花青素和叶绿素的积累,以及通过正调控WRI1和OLE1参与增加脂肪酸的生物合成和贮藏;LEC2也正调控2S和12S贮藏蛋白的表达。ABI3调控胚轴和子叶中FUS3的表达,以及通过正调控HSFA9转录因子间接参与热休克保护性蛋白的积累;ABI3是胚胎发生晚期丰富(LEA)保护性蛋白的主要调控因子
另一个关键的与发育种子中贮藏物积累相关的B3转录因子是FUS3。Wang等[91]研究表明拟南芥FUS3的直接和间接靶基因与营养库的活性、脂质定位、贮藏、代谢过程和种子油体的生物发生有关。TTG1(TRANSPARENT TESTA GLABRA 1)是拟南芥中抑制种子油和贮藏蛋白积累的转录因子[92],FUS3对贮藏物相关基因的间接转录调控与对TTG1的负调控有关(图1),通过抑制TTG1,导致编码2S贮藏蛋白前体基因的表达[92]。TTG1与花青素积累相关,在成熟种子中,花青素与脂肪酸生物合成过程竞争相同的碳源,抑制与脂肪酸碳链延长相关的还原酶基因的表达[91-92]。WRI1是发育种子中脂肪酸生物合成化合物的正调控因子[93],FUS3与LEC2共同诱导WRI1表达,通过抑制TTG1和增强WRI1的表达,间接促进贮藏脂质的生物合成。
5.2 表观遗传调控
表观遗传学(epigenetics)研究基因组中可遗传的改变,这些改变不伴随DNA序列的任何变化[94-95]。表观遗传事件主要依赖于胞嘧啶残基的DNA甲基化(DNA methylation)、组蛋白的翻译后共价修饰(covalent post-translational modification),如乙酰化(acetylation)、甲基化、泛素化(ubiquitylation)和小分子RNA的合成[94-95]。
研究发现,DNA甲基化在种子成熟后期的耐脱水性形成中起重要作用[41, 96]。DNA甲基化是DNA序列中一种普遍的胞嘧啶共价修饰,可以分为维持型(maintenance type)和从头型(de novo type)2种类型[97-98]。在所有真核生物中,修饰位点通常定位于CG区域(CG consensus),但在植物中甲基化也可能发生在CHG和CHH区域的胞嘧啶,其中H是除G以外的任何核苷酸[99]。甲基化反应由位点专一的DNA胞嘧啶甲基转移酶(DNA cytosine methyltransferase)催化,在DNA链中产生5-甲基胞嘧啶残基[99]。在从头甲基化中,底物是非甲基化的DNA分子。维持甲基化是对新合成的DNA链进行修饰,与母体DNA互补,以保持现有的胞嘧啶甲基化模式[99]。
植物中至少有3个DNA甲基转移酶家族,包括甲基转移酶1(methyltransferases 1,MET1)、染色质甲基化酶3(chromomethylase 3,CMT3)和域重排甲基转移酶2(domain rearranged methyltransferases 2,DRM2)家族。MET1在CG位点参与维持甲基化,而CMT3在CHG和CHH位点催化维持甲基化[99]。DRM2催化从头甲基化,在植物所有器官和组织中均有表达。靶甲基化位点的识别依赖于与靶DNA位点互补的小分子干扰RNA(small interfering RNA)。在识别和相互作用后,RNA指导的DNA甲基化(RNA-directed DNA methylation,RdDM)在CG位点发生,较小程度上在CHG和CHH位点发生[98]。在种子形成过程中和成熟后期,通过RdDM机制在CHH位点发生甲基化[100]。在种子形成过程中,CG和CHG位点的DNA甲基化通常保持稳定,而mCHH甲基化水平在整个种子发育过程中显著增加,随后在种子萌发过程中逐渐降低[98, 100]。在整个植物生命周期中,低甲基化的基因组区域富含编码转录因子、贮藏蛋白和脂肪酸代谢酶的基因[101]。与发育早期的胚和幼苗相比,成熟胚具有较高的基因组甲基化水平,主要是由于CHH位点的高水平甲基化造成的[12]。
An等[102]分别在CG、CHG和CHH位点鉴定了40、66和2 136个含有差异甲基化区域的基因,在CG、CHG和CHH位点中检测到66%、45%和9%的甲基化。因此,在种子成熟过程中,CHH的甲基化水平从6%增加到11%,而含有CHH区域的基因表达被显著减少。这些基因主要与DNA复制和细胞分裂有关[102]。在成熟后期,种子脱水引起核DNA的甲基化[96, 103],通常与基因转录的长期抑制有关[99]。Michalak等[103]发现正常性野生梨()种子在成熟完成后,总DNA的甲基化水平立即增加。
尽管胞嘧啶甲基化是一种相对稳定的表观遗传修饰,但它能被酶促去甲基化(enzymatic demethylation)控制[104]。去甲基化是通过切除甲基化核苷酸,随后插入非甲基化胞嘧啶完成[105]。参与植物DNA去甲基化的酶包括糖基化酶(demeter,DME)、沉默抑制子1(repressor of silencing 1,ROS1)/类糖基化酶1(demeter-like 1,DML1)、DML2和DML3[106]。
除了DNA甲基化外,基因表达的表观遗传调控也可能依赖于组蛋白的翻译后共价修饰和染色质重塑(chromatin remodeling)[97]。这种组蛋白的翻译后修饰(如乙酰化、甲基化和泛素化)在种子形成和休眠调控中起重要作用[99, 107-108]。此外,小分子干扰RNA和/或长非编码RNA(long non-coding RNA)也能触发休眠或者萌发种子的表观遗传变化[12]。
5.3 ABA和DOG1对种子成熟的调控
种子成熟的调控除了依赖AFL转录因子网络外[107],还依赖ABA的种子成熟调控因子DOG1和DOG4,它们在胚胎发生、萌发抑制、贮藏物积累和耐脱水性中也起重要作用[65, 108-109]。研究表明,ABA激活LEA蛋白合成,从而影响发育种子的耐脱水性和对不利环境因子的耐性[45, 85]。脱水敏感突变体的种子直接在母体植株上萌发[45, 86, 110]。转录因子DOGL4(DOG1-like 4)诱导70多个种子成熟专一基因的表达,包括编码贮藏蛋白如白蛋白(albumin)、cruciferin和油素蛋白的基因,被认为是种子贮藏化合物积累的主要调控因子[109]。
ABA通过刺激FUS3和LEC1转录因子促进种子成熟和休眠,从而正调控贮藏物的生物合成[89]。外源ABA增强FUS3表达,FUS3也诱导ABA增加。因此,FUS3和ABA相互作为正调控因子起作用[11, 89]。拟南芥和玉米种子不积累贮藏蛋白,不形成耐脱水性,不进入休眠,可能直接在母体植株上萌发[111]。
Nakabayashi等[112]提出DOG1和ABA以平行的途径在种子休眠调控中起作用(图2),这两条途径在ABI3或ABI3下游汇合。核心ABA信号转导组分主要由PYR/PYL/RCAR(pyrabactin resistance 1/pyrabactin resistance 1-like/ regulatory components of ABA receptor)蛋白、A组2C类蛋白磷酸酶(group A type 2C protein phosphatase,PP2C)、亚类Ⅲ蔗糖非发酵-1-相关蛋白激酶2(subclass Ⅲ sucrose nonfermenting-1-related protein kinase 2,SnRK2)和ABF(ABA-responsive element(ABRE)-binding factor/ABRE-binding protein(AREB))转录因子组成[85, 113-114](图2)。ABA信号转导与种子成熟相关的调控基因表达有关[115]。
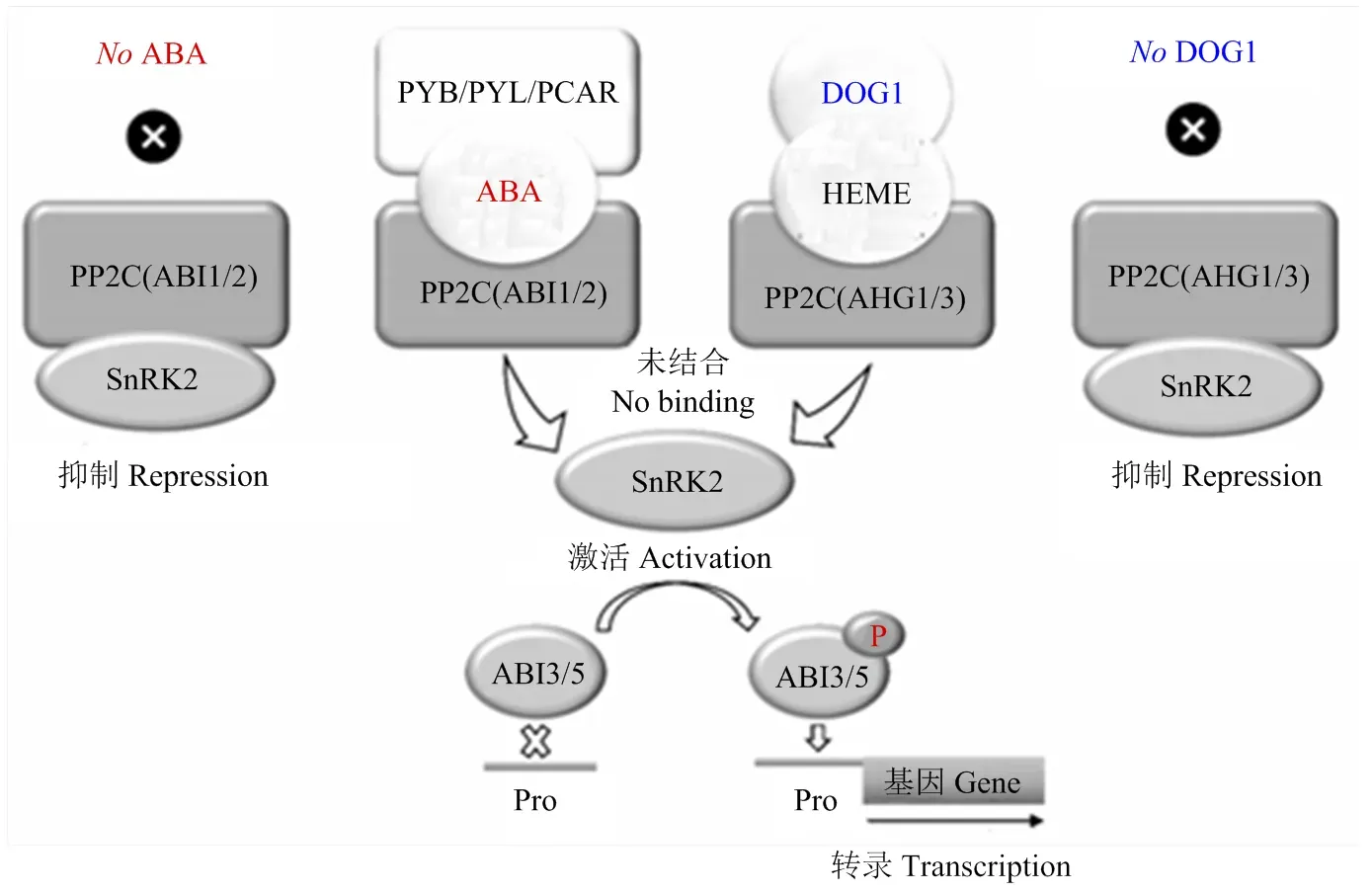
DOG1信号转导途径的关键组分是血红素分子和由AHG1和AHG3编码的PP2C。PCAR-ABA-PP2C和/或DOG1-血红素-PP2C的三重复合物阻断PP2C与SnRK2的结合。活化的SnRK2使ABI3和ABI5磷酸化,ABI3和ABI5与ABA控制的基因的启动子(Pro)结合。在种子中,平行的ABA和DOG1信号转导途径激活棉子糖家族寡聚糖(RFO)的合成、LEA和HSP的表达,从而调控耐脱水性的起始和向休眠转变。PYB/PYL/PCAR,pyrabactin resistance (PYR)/PYR-like/regulatory component of abscisic acid receptor;AHG,ABA过敏感萌发;PP2C,A组2C类蛋白磷酸酶;SnRK2,亚类Ⅲ蔗糖非发酵-1-相关蛋白激酶2
PP2C被称为ABA信号转导的关键负调控因子,由ABI1和AHG1(ABA hypersensitive germination 1)亚家族组成[116]。在低水平ABA条件下,PP2C通过抑制SnRK2阻断ABA信号转导,从而阻止由ABI3和ABI5介导的调控事件。相反,ABA水平的增加诱导PP2C与受体结合,形成一个三重的RCAR-ABA- PP2C复合物,抑制PP2C磷酸酶活性,从而诱导转录因子ABI3和ABI5的磷酸化,诱导ABA依赖的基因表达[117](图2)。ABI3、ABI4和ABI5是萌发拟南芥种子重建耐脱水性所必需的[118]。
DOG1诱导ABI3/ABI5介导的和表达,增加含氮化合物的积累以及促进种子成熟和随后的耐脱水性[65]。DOG1信号转导途径的关键组分蛋白磷酸酶AHG1和AHG3是PP2C家族成员,抑制蛋白激酶SnRK2[116](图2)。此外,DOG1能与血红素结合。DOG1与AHG1和/或AHG3结合导致SnRK2的释放和ABI5的磷酸化。DOG1控制多个种子成熟基因的表达,包括和以及棉子糖家族寡聚糖生物合成的基因[12]。因此,由ABA和DOG1调控的信号转导途径的激活抑制PP2C家族蛋白磷酸酶的活性,从而抑制ABA依赖的基因表达的转录因子[110]。
6 展望
种子是农业的“芯片”,是农业科学与技术的载体。正常性种子具有耐脱水性,在丧失高达95%水分后仍然能够保持其生活力。耐脱水性确保了种子在不利环境条件下长期存活,是种子安全贮藏和植物种质资源长期保存的基础。正常性种子的耐脱水性在发育过程中形成并逐渐增加,在萌发过程中又逐渐丧失,PEG可以诱导萌发种子耐脱水性的重建;顽拗性种子在整个发育过程中不耐脱水,对水分丧失高度敏感;中间性种子具有一定的耐脱水性;以及种子的不同组织其耐脱水性不同。利用种子的这些特性建立和完善耐脱水性的模式研究系统,可以深入研究种子耐脱水性的分子机理。
种子的耐脱水性是一种复杂的性状,其形成机制包括代谢活性下降,ROS的产生与清除活性,LEA蛋白、HSP和非还原性棉子糖家族的保护作用,以及AFL亚家族转录因子网络及其调控。值得注意的是,种子的萌发能力、耐脱水性和休眠特性都是在发育过程中起始和完成的,它们之间的相互关系还不清楚。研究表明,大多数具有生理休眠的种子是耐脱水的,但其休眠特性与耐脱水性之间的关系尚不清楚。此外,AFL转录因子调控不同类型种子的成熟程序,但耐脱水性和脱水敏感性种子之间的表达模式存在一些差异,产生这些差异的原因也不清楚[11]。
种子中同时存在核心ABA信号途径和DOG1信号途径,这两条途径在ABI3或ABI3下游汇合(图2)。在种子脱水过程中,哪一条途径优先响应以及这两条途径怎样协调也不清楚。
组学技术已经应用于种子耐脱水性的研究,在构建新的耐脱水性研究体系的基础上,结合细胞学、生理学和各种组学技术(包括转录组、翻译组、蛋白质组和代谢组)研究,可能产生一些新的知识。这些新知识将有助于更全面地理解种子耐脱水性的机制,为提高农作物的胁迫抗性与产量,改善资源库的贮藏条件和长期保存植物种质资源提供参考。
[1] Bewley J D, Bradford K J, Hilhorst H W M, NONOGAKI H.Seeds: Physiology of Development, Germination and Dormancy.3rd ed.New York: Springer, 2013.
[2] Black M,Bewley J D, Halmer P.The Encyclopedia of Seed.Science, Technology and Uses.Oxfordshire: CAB International, 2006.
[3] Leprince O, Buitink J.Desiccation tolerance: From genomics to the field.Plant Science, 2010, 179: 554-564.
[4] Oliver M J, Farran t J M, Hilhorst H W M, MUNDREE S, WILLIAMS B, BEWLEY J D.Desiccation tolerance: Avoiding cellular damage during drying and rehydration.Annual Review of Plant Biology, 2020, 71: 435-460.
[5] Song S Q, Berjak P, Pammenter N.Desiccation sensitivity ofSond.axes and antioxidant role of ascorbic acid.Acta Botanica Sinica, 2004, 46: 803-810.
[6] 傅家瑞, 宋松泉.顽拗性种子生物学.北京: 中国科学文化出版社, 2004.
Fu J R, Song S Q.Recalcitrant Seed Biology.Beijing: China Science and Culture Press, 2004.(in Chinese)
[7] Pammenter N W, Berjak P.Physiology of desiccation-sensitive (recalcitrant) seeds and the implications for cryopreservation.International Journal of Plant Science, 2014, 175: 21-28.
[8] Kan J, Song S Q.Effects of dehydration, chilling, light, phytohormones and nitric oxide on germination ofseeds.Seed Science and Technology, 2008, 36: 38-45.
[9] Berjak P, Pammenter N W.Implications of the lack of desiccation tolerance in recalcitrant seeds.Frontiers in Plant Science, 2013, 4: 478.
[10] Berjak P, Pammenter N W.Recalcitrant seeds//Benech- Arnold R L, Snchez R A, eds.Handbook of Seed Physiology: Applications to Agriculture.New York: Haworth Press, 2014: 305-345.
[11] Kijak H, Ratajczak E.What do we know about the genetic basis of seed desiccation tolerance and longevity? International Journal of Molecular Science, 2020, 21: 3612.
[12] Smolikova G, Leonova T, Vashurina N, FROLOV A, MEDVEDEV S.Desiccation tolerance as the basis of long-term seed viability.International Journal of Molecular Sciences, 2021, 22: 101.
[13] Wang W Q, Wang Y, Song X J, ZHANG Q, CHENG H Y, LIU J, SONG S Q.Proteomic analysis of desiccation tolerance and its re-establishment in different embryo axis tissues of germinated pea seeds.Journal of Proteome Research, 2021, 20: 2352-2363.
[14] Xu X, Legay S, Sergeant K, ZORZAN S, LECLERCQ C C, CHARTON S, GIAROLA V, LIU X, CHALLABATHULA D, RENAUT J, HAUSMAN J F, BARTELS D, GUERRIERO E.Molecular insights into plant desiccation tolerance: Transcriptomics, proteomics and targeted metabolite profiling in.The Plant Journal, 2021, 107: 377-398.
[15] Wu J H, Wang W Q, Song S Q, CHENG H Y.Reactive oxygen species scavenging enzymes and down-adjustment of metabolism level in mitochondria associated with desiccation-tolerance acquisition of maize embryo.Journal of Integrative Plant Biology, 2009, 51: 638-645.
[16] Wang W Q, Ye J Q, Rogowska-Wrzesinska A, WOJDYLA K, JENSEN O N, MØLLER I M, SONG S Q.Proteomic comparison between maturation drying and prematurely imposed drying ofseeds reveals a potential role of maturation drying in preparing proteins for seed germination, seedling vigor, and pathogen resistance.Journal of Proteome Research, 2013, 13: 606-626.
[17] Huang H, MØller I M, Song S Q.Proteomics of desiccation tolerance during development and germination of maize embryos.Journal of Proteomics, 2012, 75: 1247-1262.
[18] Huang H, Song S Q, Wu X J.Response of Chinese wampee axes and maize embryos on dehydration at different rates.Journal of Integrative Plant Biology, 2009, 51: 67-74.
[19] 宋松泉, Berjak P, Pammenter N W.Temporal pattern of changes in desiccation tolerance during imbibition ofseeds.云南植物研究, 2009, 31: 239-246.
Song S Q, Berjak P, Pammenter N W.Temporal pattern of changes in desiccation tolerance during imbibition ofseeds.Acta Botanica Yunnanica, 2009, 31: 239-246.(in Chinese)
[20] Wang W Q, Cheng H Y, MØller I M, SONG S Q.The role of recovery of mitochondrial structure and function in desiccation tolerance of pea seeds.Physiologia Plantarum, 2012, 144: 20-34.
[21] Song S Q, Fu J R.Studies on desiccation sensitivity and peroxidation of membrane lipids in lychee (Sonn.) seeds.Chinese Science Bulletin, 1992, 37: 1470-1473.
[22] Cheng H Y, Song S Q.Possible involvement of reactive oxygen species scavenging enzymes in desiccation sensitivity ofseeds and axes.Journal of Integrative Plant Biology, 2008, 50: 1549-1556.
[23] 宋松泉, 傅家瑞.黄皮种子脱水敏感性与脂质过氧化作用.植物生理学报, 1997, 25: 163-168.
Song S Q, Fu J R.Desiccation-sensitivity and lipid peroxidation in Chinese wampee [(Lour.) Skeels] seeds.Acta Phytophysiologica Sinica, 1997, 25: 163-168.(in Chinese)
[24] 王伟青, 程红焱, 刘树君, 宋松泉.黄皮种子线粒体呼吸速率和活性氧清除酶对脱水的响应及其生态学意义.植物生态学报, 2012, 36: 870-879.
Wang W Q, Cheng H Y, Liu S J, SONG S Q.Response of respiratory rate and reactive oxygen species scavenging enzyme activity in seed mitochondria ofdehydration and its ecological significance.Chinese Journal Plant Ecology, 2012, 36: 870-879.(in Chinese)
[25] Obroucheva N V, Sinkevich I A, Lityagina S V.Physiological aspects of seed recalcitrance: A case study on the tree.Tree Physiology, 2016, 36: 1127-1150.
[26] Song S Q, Tian M H, Kan J, CHENG H Y.The response difference of mitochondria in recalcitrantaxes and orthodoxembryos to dehydration injury.Journal of Integrative Plant Biology, 2009, 51: 646-653.
[27] Stavrinides A K, Dussert S, Combes M C, FOCK-BASTIDE I, SEVERAC D, MINIER J, BASTOS-SIQUEIRA A, DEMOLOMBE V, HEM S, LASHERMESW P, JOËT T.Seed comparative genomics in three coffee species identify desiccation tolerance mechanisms in intermediate seeds.Journal of Experimental Botany, 2020, 71: 1418-1433.
[28] Leprince O, Buitink J, Hoekstra F A.Axes and cotyledons of recalcitrant seeds ofMill.exhibit contrasting responses of respiration to drying in relation to desiccation sensitivity.Journal of Experimental Botany, 1999, 50: 1515-1524.
[29] MØller I M.Plant mitochondria and oxidative stress: Electron transport, NADPH turnover, and metabolism of reactive oxygen species.Annual Review of Plant Physiology and Plant Molecular Biology, 2001, 52: 561-591.
[30] BaillyC.The signalling role of ROS in the regulation of seed germination and dormancy.Biochemical Journal, 2019, 476: 3019-3032.
[31] Del Río L A.ROS and RNS in plant physiology: An overview.Journal of Experimental Botany, 2015, 66: 2827-2837.
[32] Mittler R.ROS are good.Trends in Plant Science, 2017, 22: 11-19.
[33] Demidchik V.Reactive oxygen species and their role in plant oxidative stress//Shabala S, ed.Plant Stress Physiology.CABI: Wallingford, 2017.
[34] Mullineaux P M, Baker N R.Oxidative stress: Antagonistic signaling for acclimation or cell death? Plant Physiology, 2010, 154: 521-525.
[35] Jeevan KUMAR S P, Rajendra P S, Banerjee R, THAMMINENI C.Seed birth to death: Dual functions of reactive oxygen species in seed physiology.Annals of Botany-London, 2015, 116: 663-668.
[36] Sano N, Rajjou L, North H M, DEBEAUJON I, MARION-POLL A, SEO M.Staying alive: Molecular aspects of seed longevity.Plant Cell and Physiology, 2016, 57: 660-674.
[37] Colville L, Kranner I.Desiccation tolerant plants as model systems to study redox regulation of protein thiols.Plant Growth Regulation, 2016, 2: 241-255.
[38] Foyer C H, Noctor G.Redox homeostasis and antioxidant signaling: A metabolic interface between stress perception and physiological responses.The Plant Cell, 2005, 17: 1866-1875.
[39] Ratajczak E, Małecka A, Ciereszko I, STASZAK A M.Mitochondria are important determinants of the aging of seeds.International Journal of Molecular Science, 2019, 20: 1568.
[40] Chen D, Li Y, Fang T, SHI X, CHEN X.Specific roles of tocopherols and tocotrienols in seed longevity and germination tolerance to abiotic stress in transgenic rice.Plant Science, 2016, 244: 31-39.
[41] Kurek K, Plitta-Michalak B, Ratajczak E.Reactive oxygen species as potential drivers of the seed aging process.Plants, 2019, 8: 174.
[42] Kranner I, Minibayeva F V, Beckett R P, SEAL C E.What is stress? Concepts, definitions and applications in seed science.New Phytologist, 2010, 188: 655-673.
[43] Roach T, Nagel M, Börner A, EBERLE C, KRANNER I.Changes in tocochromanol and glutathione reveal differences in the mechanisms of seed ageing under seed bank conditions and controlled deterioration in barley.Environmental and Experimental Botany, 2018, 156: 8-15.
[44] Shvachko N A, Khlestkina E K.Molecular genetic bases of seed resistance to oxidative stress during storage.Vavilov Journal Genetics and Breeding, 2020, 24: 451-458.
[45] Leprince O, Pellizzaro A, Berriri S, BUITINK J.Late seed maturation: Drying without dying.Journal of Experimental Botany, 2017, 68: 827-841.
[46] Marques A, Buijs G, Ligterink W, HILHORST H.Evolutionary ecophysiology of seed desiccation sensitivity.Functional Plant Biology, 2018, 45: 1083.
[47] Dure L I I I, Galau G A.Developmental biochemistry of cottonseed embryogenesis and germination: XIII.Regulation of the biosynthesis of the principal storage proteins.Plant Physiology, 1981, 68: 187-194.
[48] Battaglia M, Covarrubias A A.Late embryogenesis abundant (LEA) proteins in legumes.Frontiers in Plant Science, 2013, 4: 190.
[49] Costa M C D, Cooper K, Hilhorst H W M, FARRANT J M.Orthodox seeds and resurrection plants: Two of a kind? Plant Physiology, 2017, 175: 589-599.
[50] Amara I, Zaidi I, Masmoudi K, LUDEVID M D, PAGÈS M, GODAY A, BRINI F.Insights into late embryogenesis abundant (LEA) proteins in plants: From structure to the functions.American Journal of Plant Sciences, 2014, 5: 3440-3455.
[51] Candat A, Paszkiewicz G, Neveu M, GAUTIER R, LOGAN D C, AVELANGE-MACHEREL M H, MACHEREL D.The ubiquitous distribution of late embryogenesis abundant proteins across cell compartments inoffers tailored protection against abiotic stress.The Plant Cell, 2014, 26: 3148-3166.
[52] Hundertmark M, Hincha D K.LEA (late embryogenesis abundant) proteins and their encoding genes in.BMC Genomics, 2008, 9: 118.
[53] Artur M A S, Zhao T, Ligterink W, SCHRANZ E, HILHORST H W M.Dissecting the genomic diversification of late embryogenesis abundant (LEA) protein gene families in plants.Genome Biology and Evolution, 2019, 11: 459-471.
[54] Chen C, Zabad S, Liu H, WANG W, JEFFERY C.MoonProt 2.0: An expansion and update of the moonlighting proteins database.Nucleic Acids Research, 2018, 46: D640-D644.
[55] Jin X, Cao D, Wang Z, MA L, TIAN K, LIU Y, GONG Z, ZHU X, JIANG C, LI Y.Genome-wide identification and expression analyses of the LEA protein gene family in tea plant reveal their involvement in seed development and abiotic stress responses.Scientific Reports, 2019, 9: 14123.
[56] Olvera-Carrillo Y, Campos F, Reyes J L, GARCIARRUBIO A, COVARRUBIAS A A.Functional analysis of the group 4 late embryogenesis abundant proteins reveals their relevance in the adaptive response during water deficit in.Plant Physiology, 2010, 154: 373-390.
[57] Farrant J M, Pammenter N W, Berjak P.Seed development in relation to desiccation tolerance: A comparison between desiccation-sensitive (recalcitrant) seeds ofand desiccation-tolerant types.Seed Science Research, 1993, 3: 1-13.
[58] Delahaie J, Hundertmark M, Bove J, LEPRINCE O, ROGNIAUX H, BUITINK J.LEA polypeptide profiling of recalcitrant and orthodox legume seeds reveals ABI3-regulated LEA protein abundance linked to desiccation tolerance.Journal of Experimental Botany, 2013, 64: 4559-4573.
[59] Jin X, Liu D, Ma L, GONG Z, CAO D, LIU Y, LI Y, JIANG C.Transcriptome and expression profiling analysis of recalcitrant tea (L.) seeds sensitive to dehydration.International Journal of Genomics, 2018, 2018: 5963797.
[60] Dussert S, Serret J, Bastos-Siqueira A, MORCILLO F, DÈCHAMP E, ROFIDAL V, LASHERMES P, ETIENNE H, JOËT T.Integrative analysis of the late maturation programme and desiccation tolerance mechanisms in intermediate coffee seeds.Journal of Experimental Botany, 2018, 69: 1583-1597.
[61] Kalemba E M, Pukacka S.Possible role of LEA proteins and sHSPs in seed protection: A short review.Biology Letters, 2007, 44: 3-16.
[62] Kaur H, Petla B P, Kamble N U, SINGH A, RAO V, SALVI P, GHOSH S, MAJEE M.Differentially expressed seed aging responsive heat shock protein OsHSP18.2 implicates in seed vigor, longevity and improves germination and seedling establishment under abiotic stress.Frontiers in Plant Science, 2015, 6: 713.
[63] Nover L, Bharti K, Döring P, MISHRA S K, GANGULI A, SCHARF K D.and the heat stress transcription factor world: How many heat stress transcription factors do we need? Cell Stress and Chaperones, 2001, 6: 177.
[64] Neto V G, Barbosa R R, Carosio M G A, FERREIRA A G, FERNANDEZ L G, DE CASTRO R D, LIGTERINK W, HILHORST H, RIBERIRO P R.Sequence analysis ofsmall heat shock protein (sHSP) subfamily and its role in abiotic stress responses.Industrial Crops and Products, 2020, 152: 112541.
[65] Dekkers B J W, He H, Hanson J, WILLEMS L A J, JAMER D C L, CUEFF G, RAJJOU L, HILHORST H W M, BENTSINK L.Thegene affects() expression and genetically interacts withduringseed development.The Plant Journal, 2016, 85: 451-465.
[66] Baud S, Dubreucq B, Miquel M, ROCHAT C, LEPINIEC L.Storage reserve accumulation in: Metabolic and developmental control of seed filling.The Arabidopsis Book, 2008, 6: e0113.
[67] Buitink J, Leprince O.Intracellular glasses and seed survival in the dry state.Comptes Rendus Biologies, 2008, 331: 788-795.
[68] Walters C.Orthodoxy, recalcitrance and in-between: Describing variation in seed storage characteristics using threshold responses to water loss.Planta, 2015, 242: 397-406.
[69] González-Morales S I, Chávez-Montes R A, Hayano- Kanashiro C, ALEJO-JACUINDE G, RICO-CAMBRON T Y, DE FOLTER S, HERRERA-ESTRELLA L.Regulatory network analysis reveals novel regulators of seed desiccation tolerance in.Proceedings of the National Academy of Sciences of the United Stated America, 2016, 113: E5232-E5241.
[70] Jing Y, Lang S, Wang D, XUE H, WANG X F.Functional characterization of galactinol synthase and raffinose synthase in desiccation tolerance acquisition in developingseeds.Journal of Plant Physiology, 2018, 230: 109-121.
[71] Hell A F, Kretzschmar F S, Simões K, HEYER A G, BARBEDO C J, BRAGA M R, CENTENO D C.Metabolic changes on the acquisition of desiccation tolerance in seeds of the brazilian native tree.Frontiers in Plant Science, 2019, 10: 1356.
[72] Pukacka S, Ratajczak E, Kalemba E.Non-reducing sugar levels in beech () seeds as related to withstanding desiccation and storage.Journal of Plant Physiology, 2009, 166: 1381-1390.
[73] Ingram J, Chandler J W, Gallagher L, SALAMINI F, BARTELS D.Analysis of cDNA clones encoding sucrose-phosphate synthase in relation to sugar interconversions associated with dehydration in the resurrection plantHochst.Plant Physiology, 1997, 115: 113-121.
[74] Peters S, Mundree S G, Thomson J A, FARRANT J M, KELLER F.Protection mechanisms in the resurrection plant(Baker): Both sucrose and raffinose family oligosaccharides (RFOs) accumulate in leaves in response to water deficit.Journal of Experimental Botany, 2007, 58: 1947-1956.
[75] Yoshida T, Mogami J, Yamaguchi-Shinozaki K.ABA- dependent and ABA-independent signaling in response to osmotic stress in plants.Current Opinion in Plant Biology, 2014, 21: 133-139.
[76] Liu S, Lü Z, Liu Y, LI L, ZHANG L.Network analysis of ABA-dependent and ABA- independent drought responsive genes in.Genetics and Molecular Biology, 2018, 41: 624-637.
[77] Fatihi A, Boulard C, Bouyer D, BAUD S, DUBREUCQ B, LEPINIEC L.Deciphering and modifying LAFL transcriptional regulatory network in seed for improving yield and quality of storage compounds.Plant Science, 2016, 250: 198-204.
[78] Carbonero P, Iglesias-Fernández R, Vicente- Carbajosa J.The AFL subfamily of B3 transcription factors: Evolution and function in angiosperm seeds.Journal of Experimental Botan, 2017, 68: 871-880.
[79] Yamasaki K, Kigawa T, Seki M, SHINOZAKI K, YOKOYAMA S.DNA-binding domains of plant-specific transcription factors: Structure, function, and evolution.Trends in Plant Science, 2013, 18: 267-276.
[80] Braybrook S A, Stone S L, Park S, BUI A Q, LE B H, FISCHER R L, GOLDBERG R B, HARADA J J.Genes directly regulated by LEAFY COTYLEDON 2 provide insight into the control of embryo maturation and somatic embryogenesis.Proceedings of the National Academy of Sciences of the United Stated America, 2006, 103: 3468-3473.
[81] Grimault A, Gendrot G, Chaignon S, GILARD F, TCHERKEZ G, THÈVENIN J, DUBREUCQ B, DEPÈGE-FARGEIK N, ROGOWSKY P M.Role of B3 domain transcription factors of the AFL family in maize kernel filling.Plant Science, 2015, 236: 116-125.
[82] To A, Valon C, Savino G, GUILLEMINOT J, DRVIC M, GIRAUDAT J, PARCY F.A network of local and redundant gene regulation governsseed maturation.The Plant Cell, 2006, 18: 1642-1651.
[83] Righetti K, Vu J L, Pelletier S, VU B L, GLAAB E, LALANNE D, PASHA A, PATEL R V, PROVART N J, VERDIER J, LEPRINCE O, BUITINKA J.Inference of longevity-related genes from a robust coexpression network of seed maturation identifies regulators linking seed storability to biotic defense-related pathways.The Plant Cell, 2015, 27: 2692-2708.
[84] Bies-Ethève N, Gaubier-Comella P, Debures A, LASSERRE E, JOBET E, RAYNAL M, COOKE R, DELSENY M.Inventory, evolution and expression profiling diversity of the LEA (late embryogenesis abundant) protein gene family in.Plant Molecular Biology, 2008, 67: 107-124.
[85] Chen K, Li G, Bressan R A, SONG C, ZHU J, ZHAO Y.Abscisic acid dynamics, signaling, and functions in plants.Journal of Integrative Plant Biology, 2020, 62: 25-54.
[86] Jo L, Pelletier J M, Harada J J.Central role of the LEAFY COTYLEDON 1 transcription factor in seed development.Journal of Integrative Plant Biology, 2019, 61: 564-580.
[87] Baud S, Mendoza M S, To A, HARSCOËT E, LEPINIEC L, DUBREUCQ B.WRINKLED1 specifies the regulatory action of LEAFY COTYLEDON 2 towards fatty acid metabolism during seed maturation in.The Plant Journal, 2007, 50: 825-838.
[88] Che N, Yang Y, Li Y, WANG L, HUANG P, GAO Y, An C.Efficient LEC2 activation of OLEOSIN expression requires two neighboring RY elements on its promoter.Science in China Series C-Life Sciences, 2009, 52: 854-863.
[89] Braybrook S A, Harada J J.LECs go crazy in embryo development.Trends in Plant Science, 2008, 13: 624-630.
[90] Graeber K, Nakabayashi K, Miatton E, LEUBNER- METZGER G, SOPPE W J J.Molecular mechanisms of seed dormancy.Plant Cell and Environment, 2012, 35: 1769-1786.
[91] Wang F, Perry S E.Identification of direct targets of fusca3, a key regulator ofseed development.Plant Physiology, 2013, 161: 1251-1264.
[92] Chen M, Zhang B, Li C, KULAVEERASINGAM H, CHEW F T, YU H.TRANSPARENT TESTA GLABRA 1 regulates the accumulation of seed storage reserves in.Plant Physiology, 2015, 169: 391-402.
[93] Yamamoto A, Kagaya Y, Usui H, HOBO T, TAKEDA S, HATTORI T.Diverse roles and mechanisms of gene regulation by theseed maturation master regulator FUS3 revealed by microarray analysis.Plant Cell and Physiology, 2010, 51: 2031-2046.
[94] Berr A, Shen W H.Molecular mechanisms in epigenetic regulation of plant growth and development//Pua E C, Davey M R, eds.Plant Developmental Biology Biotechnological Perspectives.Springer: Berlin/Heidelberg Press, 2010: 325-344.
[95] Cabej N R.Epigenetic Principles of Evolution.Elsevier Inc.: Amsterdam, The Netherlands, 2019: 733-781.
[96] Plitta-Michalak B P, Naskret-Barciszewska M Z, Kotlarski S, TOMASZEWSKI D, TYLKOWSKI T, BARCISZEWSKI J, CHMIELARZ P, MICHALAK M.Changes in genomic 5-methylcytosine level mirror the response of orthodox (L.) and recalcitrant (L.) seeds to severe desiccation.Tree Physiology, 2018, 38: 617-629.
[97] Lebedeva M A, Tvorogova V E, Tikhodeyev O N.Epigenetic mechanisms and their role in plant development.Russian Journal of Genetics, 2017, 53: 1057-1071.
[98] Bouyer D, Kramdi A, Kassam M, HEESE M, SCHNITTGER A, ROUDIER F, COLOT V.DNA methylation dynamics during early plant life.Genome Biology, 2017, 18: 1-12.
[99] Bartels A, Han Q, Nair P, STACEY L, GAYNIER H, MOSLEY M, HUANG Q, PEARSON J, HSIEH T F, An Y Q, XIAO W.Dynamic DNA methylation in plant growth and development.International Journal of Molecular Science, 2018, 19: 2144.
[100] Kawakatsu T, Nery J R, Castanon R, ECKER J R.Dynamic DNA methylation reconfiguration during seed development and germination.Genome Biology, 2017, 18: 1-12.
[101] Chen M, Lin J Y, Hur J, PELLETIER J M, BADEN R, PELLEGRINI M, HARADA J J, GOLDBERG R B.Seed genome hypomethylated regions are enriched in transcription factor genes.Proceedings of the National Academy of Sciences of the United States of America, 2018, 115: E8315-E8322.
[102] An Y Q C, Goettel W, Han Q, BARTELS A, LIU Z, XIAO W.Dynamic changes of genome-wide DNA methylation during soybean seed development.Scientific Reports, 2017, 7: 1-14.
[103] Michalak M, Barciszewska M Z, Barciszewski J, PLITTA B P, CHMIELARZ P.Global changes in DNA methylation in seeds and seedlings ofafter seed desiccation and storage.PLoS ONE, 2013, 8: e70693.
[104] Li Y, Kumar S, Qian W.Active DNA demethylation: Mechanism and role in plant development.Plant Cell Report, 2018, 37: 77-85.
[105] Zhu J K.Active DNA demethylation mediated by DNA glycosylases.Annual Review of Genetics, 2009, 43: 143-166.
[106] Liu R, Lang Z.The mechanism and function of active DNA demethylation in plants.Journal of Integrative Plant Biology, 2020, 62: 148-159.
[107] Lepiniec L, Devic M, Roscoe T J, BOUYER D, ZHOU D X, BOULARD C, BAUD S, DUBREUCQ B.Molecular and epigenetic regulations and functions of the LAFL transcriptional regulators that control seed development.Plant Reproduction, 2018, 31: 291-307.
[108] Nonogaki H.Seed germination and dormancy: The classic story, new puzzles, and evolution.Journal of Integrative Plant Biology, 2019, 61: 541-563.
[109] Sall K, Dekkers B J W, Nonogaki M, KATSURAGAWA Y, KOYARI R, HENDRIX D, WILLEMS L A J, BENTSINK L, NONOGAKI H.DELAY OF GERMINATION 1-LIKE 4 acts as an inducer of seed reserve accumulation.The Plant Journal, 2019, 100: 7-19.
[110] Soppe W J J, Bentsink L.Seed dormancy back on track; its definition and regulation by DOG1.New Phytologist, 2020, 228: 816-819.
[111] Gutierrez L, Wuytswinkel O V, Castelain M, BELLINI C.Combined networks regulating seed maturation.Trends in Plant Science, 2007, 12: 294-300.
[112] Nakabayashi K, Bartsch M, Xiang Y, MIATTON E, PELLENGAHR S, YANO R, SEO M, SOPPE W J J.The time required for dormancy release inis determined by DELAY OF GERMINATION1 protein levels in freshly harvested seeds.The Plant Cell, 2012, 24: 2826-2838.
[113] Cutler S R, Rodriguez P L, Finkelstein R R, ABRAMS S R.Abscisic acid: Emergence of a core signaling network.Annual Review of Plant Biology, 2010, 61: 651-679.
[114] Dejonghe W, Okamoto M, Cutler S R.Small molecule probes of ABA biosynthesis and signaling.Plant Cell and Physiology, 2018, 59: 1490-1499.
[115] Xu P, Cai W.Function ofBnABI3 ings1, an allele of AtABI3, in seed development and stress response.Frontiers in Plant Science, 2019, 10: 67.
[116] Nishimura N, Tsuchiya W, Moresco J J, HAYASHI Y, SATOH K, KAIWA N, IRISA T, KIOOSHITA T, SCHROEDER J I, YATES J R, HIRAYAMA T, YAMAZAKI T.Control of seed dormancy and germination by DOG1-AHG1 PP2C phosphatase complex via binding to heme.Nature Communications, 2018, 9: 2132.
[117] Dekkers B J W, Bentsink L.Regulation of seed dormancy by abscisic acid and delay of germination 1.Seed Science Research, 2015, 25: 82-98.
[118] Maia J, Dekkers B J W, Dolle M J,LIGTERINK W, HILHORST H W M.Abscisic acid (ABA) sensitivity regulates desiccation tolerance in germinatedseeds.New Phytologist, 2014, 203: 81-93.
Research progress on the physiology and its molecular mechanism of seed desiccation tolerance
SONG SongQuan1,2*, LIU Jun1*, TANG CuiFang3, CHENG HongYan2, WANG WeiQing2, ZHANG Qi1, ZHANG WenHu1, GAO JiaDong1
1Agro-Biological Gene Research Center, Guangdong Academy of Agricultural Sciences/Guangdong Provincial Key Lab for Crop Germplasm Resources Preservation and Utilization, Guangzhou 510640;2Institute of Botany, Chinese Academy of Sciences, Beijing 100093;3Shenzhen Qianhai Guoken Earth Fund Management Co., Ltd, Guangzhou 510630
Dehydration tolerance (DT) is defined as the ability of an organism or tissue to survive the removal of all, or almost all the cellular water without irreversible damage.DT of seeds is an adaptive mechanism to ensure the survival and reproduction of plant species in the long-term evolution process, and plays a key role in the conservation of plant seeds and germplasm resources.However, the DT of seeds is a complex trait, and its molecular mechanism is not now largely understood.Therefore, in the present paper, the research progresses on the physiological and molecular mechanisms of seed DT were reviewed.It was found that the DT of orthodox seeds was gradually formed during development, and reached the peak at physiological maturity.Recalcitrant seeds do not undergo the development stage of maturity dehydration, and are very sensitive to dehydration throughout development.Mature orthodox seeds maintained their resistance to re-dehydration at the initial stage of imbibition.With the time course of germination, the DT decreased gradually, and finally lost completely.The DT of seeds and embryos can be re-established during the early stage of germination, and of different tissues is different.The DT of seeds and embryos was inversely correlated with the decrease in mitochondrial respiratory activity.Respiratory activity of recalcitrant axis mitochondria was higher than that of orthodox embryo ones.During dehydration, the H2O2content, the production rate of superoxide anion radical (·O2-) and the content of thiobarbituric acid reactive substance in desiccation-tolerant embryos (axes) were significantly lower than those of desiccation-sensitive embryos (axes), while the reactive oxygen species scavenging system in desiccation-tolerant embryos (axes), including enzymatic and non-enzymatic activities, was significantly higher than that in desiccation-sensitive embryos (axes).During the maturation of seeds, the accumulation of late embryogenesis abundant (LEA) proteins, small heat shock proteins and non-reducing oligosaccharides is closely related to the formation of DT.The AFL subfamily of B3 transcription factors (including ABI3 (ABA INSENSITIVE 3), FUS3 (FUSCA3) and LEC2 (LEAFY COTYLEDON 2)) increase the DT of seeds and embryos by positively regulating the accumulation of storage materials and protective proteins.The level of DNA methylation increased significantly throughout seed development and then decreased gradually during seed germination.Compared with embryos during the early stage of development and seedlings, mature embryos had a higher level of genomic methylation.In seeds, the parallel ABA and DOG1 (DELAY OF GERMINATION 1) signaling pathways activate synthesis of raffinose family oligosaccharides, and expression of LEA and HSP (heat shock protein) genes, thus regulating the onset of DT and transit to dormancy.Finally, the scientific issues that require to be further studied in this field are proposed, including the re-establishment of their model research system by using seeds and their tissues with different DT.Germinability, DT and dormancy characteristics of seeds are initiated and completed during development, and the relationship among them is still now unclear.There are both core ABA signaling pathway and DOG1 signaling pathway in seeds, and they converge at the ABI3 or downstream of ABI3.Which pathway will response preferentially and how these two pathways coordinate during dehydration of seeds? This paper will provide a reference for comprehensively understanding of the physiology and molecular mechanism of seed DT, increasing the stress resistance and yield of plant crops, improving the storage conditions of the resource bank and long-term preserving plant seed (germplasm) resources.
antioxidant system; desiccation tolerance; genetic regulation; long-term conservation of germplasm resource; metabolic activity; protective substance
2021-08-12;
2021-10-08
国家科技支撑计划(2012BAC01B05)、广东省农作物种质资源保存与利用重点实验室项目(2020B121201008)、国家自然科学基金(31871716)
通信作者宋松泉,E-mail:sqsong@ibcas.ac.cn。通信作者刘军,E-mail:liujun@gdaas.cn
(责任编辑 李莉)

