基于免疫信息学方法设计针对猪急性腹泻综合征冠状病毒S、M及E蛋白的多表位疫苗
李世念,刘婉宁,陈亚萍,王金涛
(黑龙江八一农垦大学动物科技学院,大庆 163319)
猪急性腹泻综合征(swine acute diarrhea syndrome,SADS)由猪急性腹泻综合征冠状病毒(Swine acute diarrhea syndrome coronavirus,SADS-CoV)引起,可经粪-口传播,引起动物的亚临床、轻度或致死性呼吸道和胃肠道疾病[1]。临床突出表现为被感染的母猪轻度腹泻,大多数在2 d内恢复,被感染的5日龄内新生仔猪急性腹泻、急性呕吐,体重迅速下降,导致急性死亡,死亡率高达90%以上[2]。2017年,中国广东省清远地区在世界范围内首次发现该病,导致该地区4个猪场24 693头仔猪死亡,造成了严重的经济损失[3]。鉴于SADS-CoV是国内在猪身上新出现的冠状病毒,目前对SADS-CoV的认识及研究尚浅,又尚无商业化疫苗可用[4],一旦全国性暴发将极有可能给养猪业带来致命的威胁,因此,为了科学有效地防控SADS,其疫苗开发就显得尤为重要。
现有研究表明,SADS-CoV基因组全长约27.17 kb,包括5′-UTR、3′-UTR,5个非结构蛋白ORF1a、ORF1b、NS3a、NS7a和NS7b,以及4个结构蛋白刺突蛋白(S)、膜糖蛋白(M)、核衣壳蛋白(N)和包膜蛋白(E)[5]。其中S蛋白作为结合和进入宿主细胞的关键蛋白,其功能发挥分别依赖于受体结合域S1亚基和融合域S2亚基,其在细胞附着、受体结合、种间传播、介导病毒入侵和感染,诱导机体产生中和抗体中发挥重要作用,而且是疫苗研发中的重要蛋白[6]。M蛋白作为一种跨膜蛋白,其在机体内数量多,在膜融合、病毒复制方面发挥重要作用,还能介导机体产生干扰素-α(IFN-α)及刺激机体产生免疫保护,同时该蛋白高度保守,又能诱导产生病毒中和抗体,在开发基因工程疫苗领域具有广阔前景[7]。E蛋白在冠状病毒表面含量最少,亲水性较强,其不仅在病毒包膜与粒子及冠状病毒的病毒样颗粒形成中发挥重要作用,在病毒感染和组装期间也具有重要功能。除此之外,还具有离子通道活性,可与宿主蛋白相互作用[8]。正是基于S、M和E蛋白的重要性,且位于病毒粒子表面,S、M及E蛋白被认为是疫苗开发最有吸引力的目标蛋白[9]。基于此,本研究利用免疫信息学方法预测并设计S、M和E蛋白潜在的优势表位并构建多表位疫苗,旨在为SADS-CoV多表位疫苗的设计提供一种新的方法,为SADS-CoV多表位疫苗的研发提供理论依据及数据支持。
1 材料与方法
1.1 SADS-CoV S、M及E蛋白序列检索及T、B淋巴细胞表位预测
以SADS-CoV的主要结构蛋白S蛋白(GenBank登录号: QBP43300.1)、 M蛋白(GenBank登录号:AVM80442.1)、E蛋白(GenBank登录号:QEH62671.1)为靶点用于表位筛选和设计针对SADS-CoV的多表位疫苗。运用Immunomedicine Group(http:∥imed.med.ucm.es/Tools/antigenic.pl)、IEDB(http:∥tools.iedb.org/bcell/)对其B淋巴细胞表位进行预测;运用IEDB(http:∥tools.iedb.org/mhci/)预测蛋白T淋巴细胞MHCⅠ类分子结合表位;运用NetMHCIIpan 4.0 Serve(https:∥services.healthtech.dtu.dk/service.php?NetMHCIIpan-4.0)预测蛋白T淋巴细胞MHCⅡ类分子结合表位[10]。
1.2 表位的保守性及致敏性预测
运用IEDB(http:∥tools.iedb.org/conservancy/)对MHCⅠ、MHCⅡ类分子及B淋巴细胞表位进行保守性分析,具有高保守性的表位被列入候选名单以供进一步分析。 此外,多表位疫苗设计还应关注其致敏性,运用AllerTOP v 2.0(http:∥www.ddg-pharmfac.net/AllerTOP)对其致敏性进行分析。
1.3 多表位疫苗的构建
使用选定的MHCⅠ、MHCⅡ类分子和B淋巴细胞优势表位构建疫苗氨基酸序列。不同的优势表位使用柔性linker串联在一起,MHCⅠ类分子使用AAY,MHCⅡ类分子使用GPGPG,B淋巴细胞使用KK,为了提高疫苗的免疫原性,将β-防御素Ⅰ作为免疫增强剂通过构建体N-端的EAAAK加入[11]。
1.4 多表位疫苗抗原性及理化性质预测
运用ANTIGENpro(http:∥scratch.proteomics.ics.uci.edu/)和VaxiJen v 2.0(http:∥www.ddg-pharmfac.net/vaxijen/VaxiJen/VaxiJen.html)对最终构建的多表位疫苗进行抗原性分析;运用Expasy ProtParam(https:∥web.expasy.org/protparam/)预测多表位疫苗的理化性质。
1.5 多表位疫苗糖基化及二级结构预测
运用NetNGlyc(https:∥services.healthtech.dtu.dk/service.php?NetNGlyc-1.0)预测多表位疫苗的N-糖基化位点;运用SOPMA(http:∥npsa-prabi.ibcp.fr/cgi-bin/npsa_automat.pl?page=npsa_sopma.html)分析多表位疫苗的二级结构。
1.6 多表位疫苗三级结构建模、优化
运用ANTIGENpro(http:∥scratch.proteomics.ics.uci.edu/)对多表位疫苗进行同源建模,运用3D StructureViewer(https:∥srv.mbi.ucla.edu/Viewer/?)可视化模型结构,用3Drefine(http:∥sysbio.rnet.missouri.edu/3Drefine/)改进模型质量,此过程同时将模型中的粗结构进行优化,为此运用SWISS-MODE(https:∥swissmodel.expasy.org/assess)生成Ramachandran图用于评估模型优化前后的质量。
1.7 多表位疫苗三级结构突出表位预测
为验证多表位疫苗是否具有强体液免疫和细胞免疫的能力,运用ElliPro(http:∥tools.iedb.org/ellipro/)分析多表位疫苗三级结构中连续和不连续表位。
1.8 多表位疫苗与免疫受体的分子对接
运用ClusPro 2.0(https:∥cluspro.org)进行蛋白质-蛋白质对接,检测设计的多表位疫苗与研究较彻底的Toll样受体3[12](TLR3)(PDB ID:2A0Z)的亲和力。
1.9 多表位疫苗密码子优化、反向翻译及基因克隆
运用JCat(http:∥www.jcat.de/)进行密码子优化和反向翻译,生成多表位疫苗的cDNA序列,结果由GC含量和密码子适应指数(CAI)分数组成,可用于评估蛋白质表达水平。此外,通过SnapGene计算机软件将优化后的多表位疫苗cDNA序列插入到pET-32a(+)载体中,以确保其在大肠杆菌K-12中稳定表达。
2 结 果
2.1 SADS-CoV S、M及E蛋白序列检索及T、B淋巴细胞表位分析
从GenBank获得的S蛋白核苷酸序列全长3 393 bp,共编码1 130个氨基酸,M蛋白核苷酸序列全长687 bp,共编码228个氨基酸,E蛋白核苷酸序列全长228 bp,共编码75个氨基酸。IEDB预测MHCⅠ类分子结合表位时,等位基因距离趋近于0,被认为是优良的等位基因,选择等位基因距离越趋近于0的SLA-1*0401和SLA-20401。NetMHCIIpan 4.0 Serve预测MHCⅡ类分子结合表位时,DRB等位基因被认为是MHCⅡ类分子经典的基因蛋白表达产物,等位基因选择DRB1*0801和DRB1*0301。结果显示,当选择不同等位基因时预测结果不尽相同,但仍有重叠部分,选择评分较高且两两重叠的表位区域作为优势表位。筛选出的S、M、E蛋白T淋巴细胞MHCⅠ、MHCⅡ类分子优势表位见表1。
B淋巴细胞表位结果显示,Immunomedicine Group预测出的S、M、E蛋白B淋巴细胞表位分别为43、7和2个;IEDB预测出的S、M、E蛋白B淋巴细胞表位分别为34、7和2个。因此,需选择重叠部分,同时因较短的肽不易形成抗原表位,故重叠区域还应排除掉5个氨基酸以下的短肽,使预测结果具有较高的可信度,筛选出的S、M、E蛋白B淋巴细胞优势表位见表2。
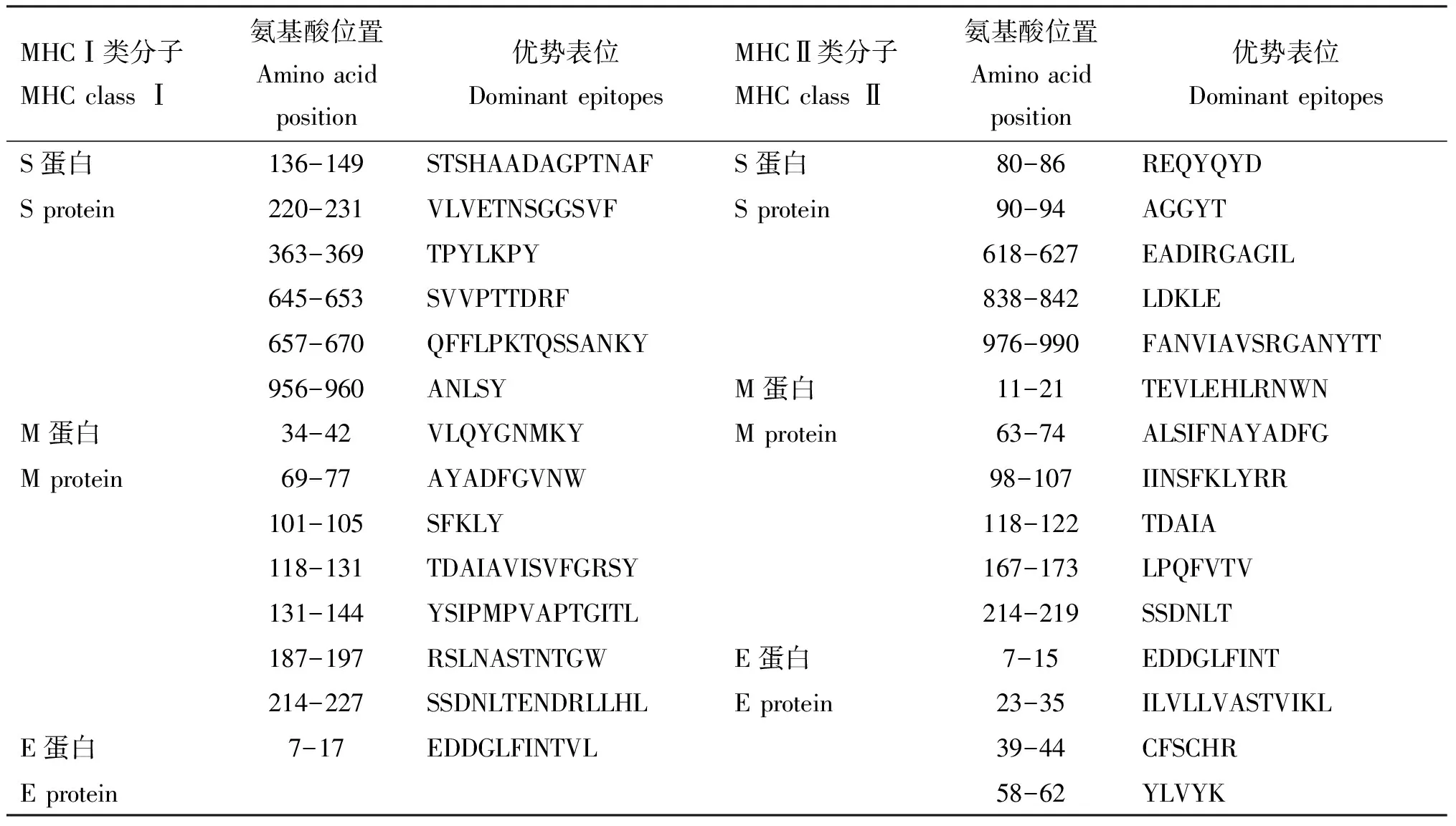
表1 SADS-CoV S、M及E蛋白MHCⅠ、MHCⅡ类分子优势表位筛选结果
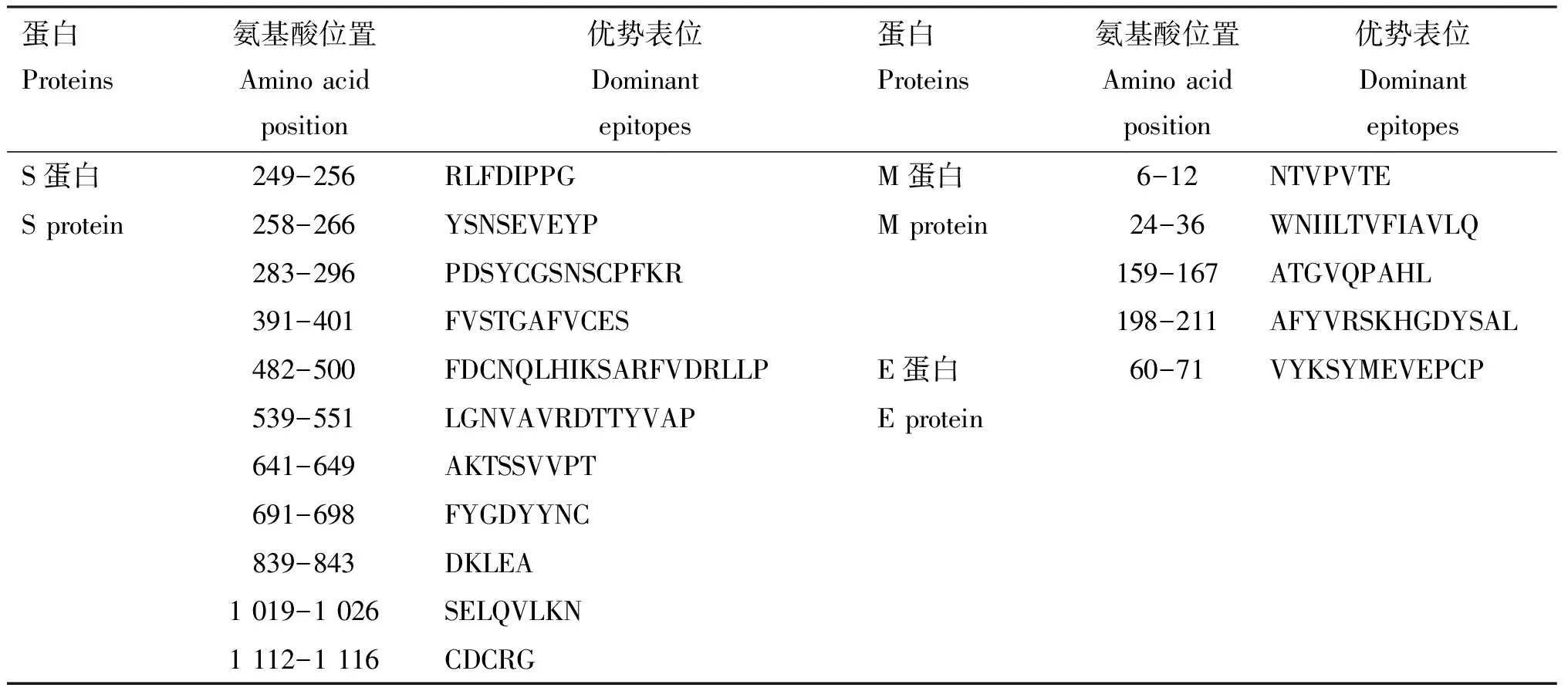
表2 SADS-CoV S、M及E蛋白B淋巴细胞优势表位筛选结果
2.2 表位保守性及致敏性分析
运用IEDB筛选保守性100%的优势表位以供下一步分析。通过筛选非致敏性优势表位,得到SADS-CoV S蛋白MHCⅠ类分子优势表位位置为136-149、363-369和956-960位氨基酸,MHCⅡ类分子优势表位位置为90-94和618-627位氨基酸,B淋巴细胞优势表位位置为283-296和553-551位氨基酸;SADS-CoV M蛋白MHCⅠ类分子优势表位位置为63-77、131-144和187-197位氨基酸,MHCⅡ类分子优势表位位置为118-122和14-227位氨基酸,B淋巴细胞优势表位位置为6-12、24-36、159-167和214-219位氨基酸;SADS-CoV E蛋白MHCⅠ类分子优势表位位置为7-17位氨基酸,MHCⅡ类分子优势表位位置为23-35、39-44和58-62位氨基酸,B淋巴细胞优势表位位置为60-71位氨基酸。
2.3 多表位疫苗的构建
选择上述优势表位进行多表位疫苗构建,构建的多表位疫苗氨基酸序列为:GIINTLQKYYCRV-RGGRCAVLSCLPKEEQIGKCSTRGRKCCRRKKE-AAAKSTSHAADAGPTNAFAAYTPYLKPYAAY-ANLSYAAYALSIFNAYADFGVNWAAYYSIPMP-
VAPTGITLAAYRSLNASTNTGWAAYEDDGLFIN-
TVLGPGPGAGGYTGPGPGEADIRGAGILGPGPG-
ALSIFNAYADFGGPGPGTDAIAGPGPGSSDNL-TENDRLLHLGPGPGILVLLVASTVIKLGPGPG-CFSCHRGPGPGYLVYKKKPDSYCGSNSCPFKRK-KLGNVAVRDTTYVAPKKNTVPVTEKKWNII-
LTVFIAVLQKKATGVQPAHLKKVYKSYMEV-
EPCP,多表位疫苗盒如图1所示,其中GIINTL-QKYYCRVRGGRCAVLSCLPKEEQIGKCSTRG-RKCCRRKK为β-防御素Ⅰ,EAAAK为N-端接头,AAY为MHCⅠ类分子linker,GPGPG为MHCⅡ类分子linker,KK为B淋巴细胞linker。
2.4 多表位疫苗抗原性及理化性质分析
ANTIGENpro和VaxiJen 2.0预测构建的多表位疫苗抗原性分别为0.897和0.654,表明构建的多表位疫苗具有良好的抗原性。ExPASy ProtParam预测结果显示,多表位疫苗由336个氨基酸组成,蛋白质分子质量为35.30 ku,理论等电点为9.36,分子式为C1590H2475N429O455S13。哺乳动物网织红细胞体外半衰期30 h,酵母体内半衰期>20 h,大肠杆菌体内半衰期>10 h;不稳定系数为47.70,脂肪指数为28.20,亲水性平均值为-0.147,预测结果将其归为稳定亲水蛋白。
2.5 多表位疫苗糖基化及二级结构预测
NetNGlyc结果显示,多表位疫苗具有1个潜在的N-糖基化位点(图2)。SOMPA预测多表位疫苗二级结构显示, α-螺旋、 β-折叠、 无规则卷曲和β转角分别占22.11%、20.35%、50.88%和6.67%(图3)。
2.6 多表位疫苗三级结构建模、优化
ANTIGENpro基于同源建模构建多表位疫苗的三级结构,所得模型运用3D StructureViewer服务器查看结果如图4A所示。Swissmode服务器生成Ramachandran图,模型得分3.81,冲突得分113.75,Ramachandran图优势区域90.00%,异常区域5.00%(图4B)。3Drefine服务器优化蛋白质模型(图4C)。优化后的模型得分3.62,冲突得分74.54,Ramachandran图优势区域91.92%,异常区域3.89%(图4D)。 与优化前相比,优化后Ramachandran图得到了更好的结果。

图1 SADS-CoV多表位疫苗盒Fig.1 SADS-CoV multi-epitope vaccine kit

h,α-螺旋;e,β-折叠;t,β-转角;c,无规则卷曲h,Alpha helix;e,Beta sheet;t,Beta turn;c,Random coil图3 多表位疫苗二级结构预测Fig.3 Prediction of secondary structure of multiepitope vaccine
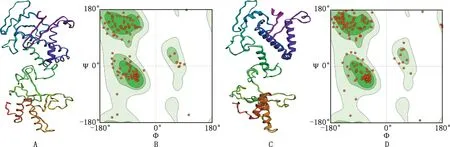
A,多表位疫苗的三级结构;B,Ramachandran作图验证三级结构;C,多表位疫苗优化后的三级结构;D,Ramachandran作图验证优化后的三级结构A,Tertiary structure of multiepitope vaccine;B,Ramachandran plots to validate the tertiary structure;C,Tertiary structure optimized by the multi-epitope vaccine;D,Ramachandran plots validates the optimized tertiary structure图4 多表位疫苗三级结构的建模、优化Fig.4 Modeling and optimization of tertiary structure of multiepitope vaccine
2.7 多表位疫苗三级结构突出表位预测
从优化后的多表位疫苗的三级结构模型中预测得到10个线性表位和9个不连续表位,线性表位结合位置的最高得分为0.81(图5A,箭头所示),不连续表位结合位置的最高得分0.78(图5B,箭头所示),得分远高于0.5的设定阈值,证实设计的多表位疫苗具有较好的免疫原性。
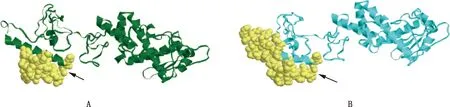
A,评分最高的线性表位结合位置;B,评分最高的不连续表位结合位置A,The linear epitope binding position with the highest score;B,The binding position of discontinuous epitope with the highest score图5 B细胞优势表位图Fig.5 Map of dominant epitopes of B cells
2.8 多表位疫苗构建体与免疫受体的分子对接
使用ClusPro 2.0进行多表位疫苗和TLR3分子对接,共产生10个模型,其中0号模型所得能量分数为-1 064.2,在预测的所有模型中能量分数最少,为最佳对接复合体,具有最高的亲和力(图6)。

A,TLR3;B,多表位疫苗A,TLR3;B,Multiepitope vaccine图6 疫苗蛋白和TLR3的对接Fig.6 Docking of vaccine protein and TLR3
2.9 多表位疫苗密码子优化、反向翻译及基因克隆
密码子优化序列的CAI改进值为0.96,多表位疫苗GC含量为54.07%。总体而言,多表位疫苗属于适合在大肠杆菌中表达的类别。将多表位疫苗嵌入到原核表达载体pET-32a(+)限制性酶切位点BamH Ⅰ和XhoⅠ中,最终生成一个完整的克隆构建体,其序列长度为1 008 bp。
3 讨 论
SADS-CoV作为新近发现的第4种猪肠道冠状病毒[13],自2017-2019年在中国广东、福建两省暴发以来,相继使多个猪场遭受了严重的打击,并给当地养猪业带来了重大的经济损失[14-15]。陈仲德等[16]将前期收集的发病猪病变组织进行灭活处理后作为灭活苗对妊娠母猪进行口服,新生仔猪获得母源抗体,使疫情得到了一定的控制。但2019年,SADS-CoV在广东省某猪场再次暴发,宏基因组测序结果表明其已发生突变,致使毒力变强[17],这提示对该疫病早期控制和预防已变得十分必要。然而现有的研究结果虽然在病毒的起源、致病性、诊断方法和检测技术等方面有了一定进展,但依旧存在许多尚未解决的问题,疫苗接种一直是预防和控制传染病的有效措施[18],因此,为防止SADS-CoV的持续传播,迫切需要有效的疫苗。
传统疫苗的抗原表位筛选耗时耗力,而随着免疫信息学的发展,计算机可依据不同运算法则对基因序列进行深层次信息的筛选,从基因序列源头对抗原表位进行预测,即为“反向疫苗学”,因具有更高的准确性故被广泛使用[19]。多表位疫苗正是伴随着反向疫苗学应运而生,相对于传统弱毒疫苗,其安全性更高,其将病毒蛋白上的多个B淋巴细胞或T淋巴细胞表位整合到特定载体上,构建的多表位疫苗可能有效刺激机体引发体液和细胞免疫,增强宿主保护性免疫应答,从而有利于控制病毒的感染[20]。多表位疫苗在抑制病毒复制和对抗免疫逃逸株等方面都具有独特优势,是目前疫苗研究的一个热点。 设计开发的多表位嵌合抗原用于候选疫苗和诊断剂,不仅成本较低,且刺激强烈的免疫应答[21]。
本试验正是基于反向疫苗筛选策略,通过免疫信息学方法筛选出SADS-CoV S、M及E蛋白优势表位,优势表位经柔性linker连接以延长蛋白在体内的作用时间,有效避免新结合表位的产生而被引入[22]。EAAAK接头连接到构建体的N-端以促进持久的免疫反应,通过有效分离减少与其他蛋白质区域的连接并提高稳定性,AAY和GPGPG接头连接可提高溶解度并使相邻的结构域易于接近和自由发挥作用,并促进抗原的免疫加工,KK接头以增强蛋白质折叠、稳定和表达[23]。本研究预测分析结果显示,构建的SADS-CoV多表位疫苗具有较高的保守性和非致敏性,且为稳定亲水蛋白,可为蛋白表达纯化提供理论依据。同时抗原性决定了抗原与B细胞和T细胞受体结合能力,促进免疫应答和记忆细胞形成[24],运用ANTIGENpro、VaxiJen v 2.0预测多表位疫苗的抗原性确保其与免疫受体相互作用,结果显示构建的多表位疫苗高出设定阈值0.3,证明多表位疫苗具有较好的抗原性。而对疫苗N-糖基化位点预测尤为重要,其在蛋白质稳定方面发挥着重要作用[25],本试验结果显示,多表位疫苗存在N-糖基化位点。二级结构预测主要有α-螺旋、β-折叠、β-转角及无规则卷曲结构,其中α-螺旋、β-折叠结构规则不易形变,较难嵌合抗体,一般不作为抗原表位[26]。本试验预测中N-端α-螺旋出现频率较少,预测结果显示,α-螺旋、β-折叠、无规则卷曲和β-转角分别占22.11%、20.35%、50.88%和6.67%,但预测结果发现其结构较为分散。而β-转角、无规则卷曲结构较易发生扭转,结构向外凸出,多出现在蛋白质抗原表面,利于与抗体嵌合,因此其形成优势表位的可能性较大[27]。预测结果显示无规则卷曲和β-转角约占比60%,显示出多表位疫苗较好的抗原性。除此之外,多表位疫苗的生物学功能发挥还依赖其三级结构,通过预测其三级结构上的连续与不连续表位,判断多表位疫苗的免疫原性[28],多表位疫苗的三级结构由ANTIGENpro服务器建模,之后用3Drefine模型进行优化。最终优化后的多表位疫苗三级结构在B淋巴细胞引起的体液免疫中共产生10个线性表位,9个不连续表位,预测得分均在0.8左右,证实了设计的多表位疫苗具有良好免疫原性。同时多表位疫苗和免疫受体分子之间的相互作用对于触发免疫反应十分重要,将优化后的多表位疫苗模型与TLR3分子对接,以检查免疫反应时的结合能力[29]。在分子对接分析中观察到多表位疫苗和TLR3之间能稳定相互作用,并且结合所得的能量分数较少,为-1 064.2,具有较高的亲和力。最后将多表位疫苗进行密码子优化,使用SnapGene克隆到大肠杆菌载体,确保多表位疫苗在原核表达载体pET-32a(+)内能够准确翻译并且稳定表达[30]。本研究应用一系列免疫信息学工具,构建了可对抗SADS-CoV的有效疫苗,但仍需试验验证来评估设计构建的多表位疫苗免疫原性和安全性。后续试验将对构建的多表位疫苗进行高通量克隆、表达纯化重组蛋白、免疫动物,并进行免疫学评价,以确定所构建多表位疫苗的实际应用价值。
4 结 论
本研究运用免疫信息学方法成功预测并筛选出了SADS-CoV S、M及E蛋白的优势表位,通过对优势表位的保守性、致敏性进一步评估,构建了SADS-CoV的多表位疫苗。通过对多表位疫苗理化性质、抗原性、N-糖基化位点、二级结构、三级结构建模及优化、分子对接、密码子优化、反向翻译和计算机克隆等进行综合分析,成功构建了SADS-CoV多表位疫苗,为SADS-CoV多表位疫苗的研发提供了理论依据及数据支持。

