哺乳动物合成基因组学研究进展
何博,付宗恒,吴毅,赵广荣
(天津大学化工学院,系统生物工程教育部重点实验室,教育部合成生物学前沿科学中心,天津 300072)
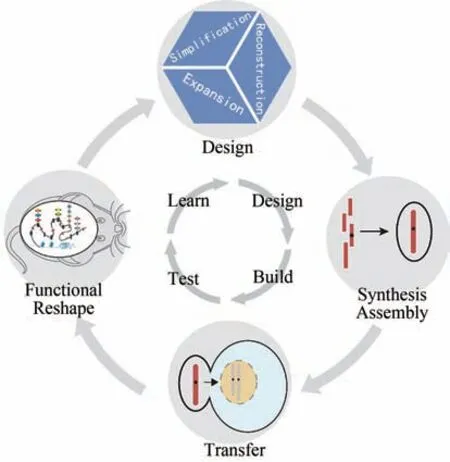
DNA 是原核生物和真核生物的主要遗传物质,指导细胞的各种生命活动。不同于原核生物遗传物质裸露于细胞质中,真核生物以染色体的形式将其庞大、复杂的遗传信息组织起来。按照合成生物学的经典理念--了解事物工作原理的最佳方法就是设计重构[1],“编写”基因组可以进一步理解基本生命过程。
随着多组学的不断进步以及DNA 合成组装技术的快速发展,“编写”生物基因组也逐渐成为现实[2-3],通过对基因组进行理性设计、从头化学合成、组装、转移以及功能重塑,实现生命遗传物质的人工合成及重塑。近十多年来合成基因组学在较低等生物的基因组设计与合成中取得了一系列重要进展和里程碑式的突破,合成基因组学的研究对象实现了由病毒到原核生物再到真核生物的发展,设计深度也由简单的复制序列发展为多元化甚至颠覆性的基因组重塑。目前已经实现支原体[4-5]、大肠杆菌全基因组[6-7]的设计再造,以及酿酒酵母Ⅱ号[8]、Ⅲ号[9]、Ⅴ号[10]、Ⅵ号[11]、Ⅹ号[12]和Ⅻ号[13]染色体的全化学合成。
高等动物基因组的设计与合成被视为合成基因组学领域的下一个任务和挑战。通过高等动物基因组的设计与合成加深对高等生物基因组的理解,同时开发在药物研发、疾病治疗等方面的潜在应用场景,其潜在应用可包括复杂疾病模型、人源化动物、抗病毒超级细胞系、器官移植等。然而相对于大肠杆菌、酿酒酵母等低等模式生物,哺乳动物细胞的基因组更为庞大,具有复杂的表观遗传修饰以及染色体三维结构,除此之外,基因组注释不完善以及复杂的时空调控机制也增加了哺乳动物基因组的设计难度。通过设计-合成-检验-学习的循环发展,哺乳动物合成基因组学也将推动技术的进步同时加深对基因组的进一步理解,进而指导哺乳动物基因组的设计。
2016年6 月,由来自美中英等多个国家的跨学科团队[2]提出基因组编写计划(Genome Projectwrite,GP-write),将促进基因组规模编写DNA 新技术的开发,有望如20 年前人类基因组计划(Genome Project-read,GP-read)一样对研究生物学以及改善人类健康方面产生变革式的影响。基因组编写计划正在分阶段进行,以评估相关研究的可行性和应用价值。重新编码人类细胞基因组需要对合成构建和测试人工基因组的技术进行突破式的重大改进。在染色体或基因组尺度的DNA合成、组装以及活细胞内测试的技术路线中,关键步骤仍存在技术难题,目前大多数合成基因组编写技术仍处于实验室开发阶段[14],如化学合成寡核苷酸需进一步降低成本并提高化学合成更长的寡核苷酸链精确度;开发新的组装宿主和分子生物学工具用于高GC 含量和直接重复序列等复杂序列的组装;开发染色体规模的高效率、高通量的转移方法;需要基础研究进一步阐明基因型-表型的关系以及序列-表观遗传调控指导基因组结构的机制等。通过推动创新和增加需求,基因组编写计划可进一步推动新技术的开发和改进。
基因组编写计划等相关项目的逐步开展,可为哺乳动物基因组设计提供理论指导,为哺乳动物中大片段DNA 的操纵提供新方法,将极大推动哺乳动物合成基因组学的发展。
1 哺乳动物基因组的设计
设计构建不同于天然基因组的合成基因组或染色体,一方面能检验和提高对基因组生物学知识的理解和认识,另一方面可以赋予生物体新的功能,揭示表型与基因型的关系。合成基因组的设计原则依据对基因组重塑的方式可以分为:简化(simplification)、扩展(expansion)、重构(reconstruction)等(图1)。基因组简化设计原则指导探索不同条件下支持生命活动所需的最少基因集合及其对应的生理功能,包括基因水平的简化[5]和基因间区的简化等。合成基因组的扩展设计原则旨在通过向天然基因组中增添DNA 序列从而使生命体获得新的表型和功能,包括在脱氧核苷酸水平将天然碱基对扩展至更多种非天然碱基对,以及在基因水平引入外源基因或路径等[15]。合成基因组的重构设计原则根据对天然基因组的设计再造维度从小到大(从密码子、基因表达盒内部元件、多基因排列到染色体或整个基因组结构),可包括密码子重新编码(recoding)[16]、元件模块化(modularization)、基因成簇化(gene clustering)、染色体结构重排(rearranging)等[17-23]。此外,研究表明DNA 的转录、翻译、调控等也受到表观遗传修饰等的影响[24],这也将是合成基因组设计应考虑的因素之一。

图1 基因组的设计原则Fig.1 Design principles for genomes
1.1 微生物基因组设计为哺乳动物基因组设计提供参考
合成基因组学在支原体、大肠杆菌、酿酒酵母等低等生物中的创新性设计取得了一系列具有重大科学意义的成果。不仅证明全化学合成基因组可以替代天然基因组作为生物体的遗传物质调控生命正常功能,而且通过“自上而下”的设计再造方式检验和完善了对现有生命系统的认知和理解。
JCVI-syn1.0[4]实现了丝状支原体(Mycoplasma mycoides)基因组的全化学合成,JCVI-syn3.0[5]项目根据基因组简化设计原则,探索生命所需最基本的基因,使用了全基因组设计和完整的化学合成方法,将JCVI-syn1.0 长达1.079 Mb 的合成型基因组简化至531 kb,只保留了473 个基因。根据基因组扩增设计原则,可在基因组中引入非天然碱基对[15,25]或异源基因,如Luo等[26]通过引入异源的代谢路径实现了在酿酒酵母中全合成大麻素及其非天然类似物。根据基因组重构设计原则,英国剑桥大学Jason W.Chin 团队利用开发的在大肠杆菌中可迭代地循环替换约100 kb 基因组的技术[27],完成了对大肠杆菌18 214 个密码子的重新编码[7],创建仅具有61 个密码子的4 Mb 基因组的大肠杆菌变体(a variant ofEscherichia coli),空出来的密码子可以用来编码新氨基酸,从而创造具有新功能的蛋白,赋予生命体新功能。2021 年,该团队敲除合成型菌株密码子TCG、TGA、TAG的tRNAs 和释放因子,从而使菌株获得对噬菌体的抵抗能力,同时实现了对密码子的再利用,赋予合成型菌株新的功能[28]。
在真核基因组设计领域,Sc2.0 项目[29]是全世界第一个真核生物基因组设计合成的国际合作项目,该项目旨在对酿酒酵母基因组进行系统性设计和化学合成再造以获得具有生物活性的合成型酿酒酵母基因组,Sc2.0 采用“自下而上”的基因组合成策略以及模块化组装、多轮迭代替换的方式构建酿酒酵母染色体,已完成酿酒酵母Ⅱ、Ⅲ、Ⅴ、Ⅵ、Ⅹ和Ⅻ号染色体的全化学合成。Sc2.0 项目通过计算机辅助真核基因组设计[30],根据基因组简化设计原则删除tRNA、转座子等不稳定元件,提升基因组稳定性,同时根据基因组重构设计原则引入多个LoxPsym 位点增加基因组可操作性,构建了一个人工基因组重排系统(SCRaMbLE),可实现染色体结构重排并在实验室条件下加速酿酒酵母的基因型进化和表型进化。现阶段哺乳动物基因组的设计仍处于萌芽初期,未来基因组设计旨在将更多元更具颠覆性的设计引入到染色体级别的DNA 序列设计中。
1.2 基因组学数据库为哺乳动物基因组设计提供支撑
设计更复杂的哺乳动物基因组需要全面分析更多的数据信息,包括更加完善的基因组功能元件注释、转录组、蛋白组、代谢组、DNA-蛋白质结合位点数据、表观遗传修饰数据以及基因组三维结构数据等。
近年来随着ENCODE、gnomAD、GWAS等一系列项目的推进,可为哺乳动物基因组的设计提供支持。DNA 元件百科全书(The ENCyclopedia Of DNA Elements,ENCODE)项目旨在鉴定人类基因组序列中的所有功能性元件[31-32]。截至2020年ENCODE[33]确 定 了20 225 个 蛋 白 质 编 码 基 因、37 595个非编码蛋白质基因、2 157 387个开放染色质区域,750 392 个组蛋白修饰区域、1 224 154 个转录因子和染色质相关蛋白结合区域,845 000 个RNA结合蛋白占据的RNA子区域(RNAsubregions),还鉴定出超过130 000 个远距离相互作用的染色质基因座(chromatin loci),并注释了约90 万个人类基因组中的和约30 万个小鼠基因组中的调控元件信息。2019年,UCSU发布了人类调控元件及相关靶基因的数据库--GeneHancer track[34],它集成了7 个全基因组数据库(ENCODE、Ensembl、FANTOM5、 VISTA、 dbSUPER、 EPDNew 和UCNEbase)包括超过100 万个调控元件。与此同时,高通量测序技术的发展促进对个体基因组的测序工作及基因组突变的相关研究。基因组聚合数据库(GenomeAggregation Database,gnomAD)汇总了15 708个来自不同个体的完整基因组和125 748个外显子组,整理并分析不同类型的变异对功能或表型的潜在影响[35]。全基因组关联研究(genomewide association studies,GWAS)鉴定发现人类基因组中不同个体之间的变异位点90%以上都位于非编码区[36]。发现和解析不同人种和不同个体之间基因组多样性丰富了对基因组的认识,可帮助识别引起疾病的基因突变并确定潜在的药物靶标,是哺乳动物基因组设计合成的重要数据资源。
尽管小鼠、人等哺乳动物基因组测序工作已基本完成[37-39],新的测序技术对基因组测序数据进行不断的补充[40],大量的基因组注释工作也取得持续性进展,但对基因组的注释工作仍不完善。人类基因组中蛋白质编码序列所占比例不到2%[41],因此在解析基因编码区的同时不断完善非编码区的注释才能为哺乳动物基因组的深度设计提供支撑。在简化、扩展、重构等设计原则指导下,基因组的高效设计一般除了需要从已有的生物信息数据库[14]获取基本的元件信息,还需通过计算机来实现基因组规模的序列设计。如Sc2.0 借助计算机辅助设计合成型酿酒酵母染色体[29],JCVI 3.0 借助计算机辅助设计结合DBT(Design-Build-Test)循环成功构建出简化的支原体基因组[5]。人工智能已在蛋白质结构预测领域取得突破性进展[42-43],AlphaFold2预测的蛋白质结构能达到原子水平的准确度,确定了覆盖几乎整个人类蛋白质组(98.5%的所有人类蛋白)的蛋白质结构,该工作的研究者期望这一精准的预测算法可以让蛋白质结构解析技术促进基因组学的发展。随着各种生物信息数据库以及基因组注释的不断完善,人工智能有望在高等生物基因组设计、改善表型预测等方面发挥重要作用。
1.3 哺乳动物基因组设计进展
设计构建独立于基因组的合成型染色体是哺乳动物基因组设计的一个重要策略。设计构建的染色体可从其传代稳定性、承载能力、复制能力、穿梭能力、表达能力等几个方面来进行评估。染色体基本功能元件使其在细胞中稳定存在并发挥功能,其基本功能元件包括着丝粒、端粒、自主复制起始位点、启动子、增强子、沉默子等在分配、复制、转录、翻译等不同阶段发挥重要功能的元件。其中,端粒是维持线性染色体稳定的重要元件,而构建环形染色体可在不使用端粒的情况下实现染色体的稳定。
高等哺乳动物着丝粒区域长、序列复杂,且高级重复(higher-order repeat,HOR)单元阵列长度个体之间差异极大[44]。目前人类基因组中只有8 号、X 和Y 染色体的着丝粒序列测序组装完成[45],天然着丝粒序列由复杂的高度重复序列构成,对于测序和合成都是巨大的挑战,这些因素限制了高等哺乳动物着丝粒作为通用元件的使用。不同于酿酒酵母染色体上序列明确的点着丝粒区域,研究表明哺乳动物染色体依靠组蛋白H3 变体CENP-A 从表观遗传的角度确定着丝粒的位置[46-52],合成型哺乳动物染色体着丝粒区域的设计构建是哺乳动物合成基因组学研究的重要内容,其设计构建可分为自下而上(bottom-up)和自上而下(top-down)两种。着丝粒区域自下而上构建又称从头构建(de novo),指化学合成包含天然着丝粒序列或其他能发挥着丝粒功能的非天然序列[图2(a)、(b)]。1997 年,Harrington 等[53]使用人17号染色体的2.7 kb重复序列阵列,产生约173 kb大小载体,其在细胞内自发形成6~10 Mb 的稳定HAC,并在6 个月内能正常复制、分配至子代细胞。Oshimura 课题组[54]开发了可诱导型着丝粒合成型哺乳动物染色体Alphoidtet-O-HAC,将6000 拷贝的42 bp 四环素操纵子(tet-O)序列插入兆碱基大小的合成DNA 阵列中,利用tet-O 被tet 阻遏物(tet-R)结合的高亲和力和特异性,通过tet-R 融合蛋白来靶向HAC 中的tet-O 序列使着丝粒功能失活并 诱 导HAC 丢 失。Kouprina 等[55-56]进 一 步 将Alphoidtet-O-HAC 开发成一个用于将大片段DNA 向哺乳动物细胞转移的工具,通过在CHO 细胞中的Alphoidtet-O-HAC 上进行DNA 片段多轮迭代组装实现大片段功能区域的组装,可用于纠正人类细胞的遗传缺陷[57]。Pesenti 课题组[58]利用人21 号染色体上着丝粒序列从头构建了α21-ITetO和α21-ⅡLacO/Gal4HAC,分析和研究在HAC 形成初期的复杂的DNA 重排。2019 年Ben E.Black 团队[46]开发了一种从头构建哺乳动物染色体载体的新方法,该方法绕过传统自下而上载体对天然alpha satellite、CENP-B boxes 序列的需要,利用被称为新型着丝粒(neocentromere)的非重复序列(4q21)代替重复的着丝粒序列,并在载体上添加LacO重复序列,由于LacI-LacO相互作用,经诱导表达的LacI-HJURP被定位至LacO 序列上,随后HJURP 募集CENP-A,被附近的DNA 包裹,形成着丝粒。这项工作避开了对原着丝粒大片段重复DNA 的合成组装,对动物人工染色体载体设计构建具有重要意义。自上而下的载体是通过同源重组在天然染色体臂上引入人工端粒或LoxP位点对天然染色体进行截短得到,被称为微染色体(minichromosome)。鸡淋巴瘤细胞DT40同源重组能力强,是微染色体的构建的主要工具[59][图2(c)]。基于人类14 号、21 号、X、Y 染色体都被改造成微染色体[60]。

图2 合成型哺乳动物染色体着丝粒区域(a)基于卫星(satellite)重复序列自下而上构建的着丝粒区域;(b)基于非天然重复序列自下而上构建的着丝粒区域;(c)自上而下截短的人工染色体Fig.2 Centromeric regions of synthetic mammalian chromosomes(a)Bottom-up centromere region based on satellite repeat sequence;(b)Bottom-up centromere region based on non-natural repetitive sequences;(c)Top-down artificial chromosomes from truncated natural chromosomes
从头构建(de novo)的着丝粒稳定性依赖HT1080 细胞,其低Suv39h1 甲基转移酶表达量有利于合成型着丝粒功能的实现[51],这也就导致最初着丝粒功能的实现局限在极少数的细胞系。包含从头构建着丝粒序列的载体在细胞中会发生3种不同的变化:①整合到染色体臂上;②发生多聚化形成一个能自我复制以及随传代稳定分裂的独立HAC(细胞群体可以包含两者的混合物);③整体或部分丢失。造成包括功能DNA 序列在内的不可预测的改变,限制了其应用。自上而下构建的微染色体的“微”是相对于天然染色体而言,其自身的大小也在Mb 级别,无法在大肠杆菌、酿酒酵母等模式微生物中进行组装和编辑,且其转移的方式也受到极大的限制。如何设计构建合成型哺乳动物染色体载体,使其大小上能通过常规转染方式进行转移,在体内以稳定的游离形式存在且序列与初始保持一致,是合成型哺乳动物染色体载体设计接下来面临的任务和挑战。
除了设计独立于哺乳动物基因组的合成型染色体,设计构建大片段DNA 整合到哺乳动物细胞基因组上也是在哺乳动物细胞中操纵大片段DNA的一个重要策略。该策略可利用大片段操纵技术实现大片段的敲入等,从而将基因组设计的重点放在基因组研究内容的设计上。2018 年,基因组编写计划(GP-write)宣布第1 个大型试点项目:能够抵抗天然病毒以及潜在的辐射、冷冻、衰老和癌症的“超安全”(“ultra-safe”)版本的人类细胞系(明确限于在细胞中研究)。超安全细胞系的构建预计将需要对人类基因组进行至少40 万处更改,基于简化和密码子重新编码的设计原则,从所有20 000 多个人类基因中删除特定的冗余密码子,通过“重新编码”所有基因的冗余密码子并删除对其进行解码的tRNA 来实现抗病毒功能[16,61],通过改造p53 等抑癌基因的突变热点序列降低突变率或增加抑癌基因的拷贝数从而降低细胞癌变的风险,团队期望在10 年内完成该项工作。由于全基因组关联研究(GWAS)绘制人类疾病和性状相关变异的图谱发现大多数变体(90%以上)位于非编码区,因此关于非编码区如启动子、增强子、CTCF 结合位点等调控区域以及内含子等的设计工作具有重要意义。此外,由于逆转座子导致的基因组不稳定性使干细胞治疗存在潜在风险[62],设计合成删除逆转座子的简化基因组可提高基因组的稳定性。大多数的非编码序列具有功能,盲目的删减可能有潜在的研究风险。在开展研究前需要对于涉及的基因组进行广泛而深入的调研,对于注释不明的序列应采取相对保守的设计。Bradley团队[63]构建的人源化抗体转基因小鼠,对小鼠自身的免疫球蛋白基因座采取倒置失活而不是直接敲除,是因为小鼠免疫球蛋白重链基因座中含有其他有重要功能的基因以及非编码RNA,盲目删除存在巨大的未知风险。尽管现阶段哺乳动物基因组的设计充满挑战,但是哺乳动物基因组设计-合成-检验-学习的循环发展也会提升对基因组序列与功能关系的理解。
目前对于哺乳动物基因的设计仍局限在局部小范围的设计,仍不能像低等生物基因组的设计一样实现整个基因组的全局性设计。哺乳动物基因组的复杂性如精密的基因时空调控系统、基因间相互作用网络、表观遗传调控、细胞间相互作用等也给基因组设计工作引入了更多的挑战。哺乳动物基因组信息量庞大,现有的计算机软件辅助设计工具无法满足合成型哺乳动物基因组的设计,有效的辅助设计工具还有待开发。虽然哺乳动物基因组的设计面临诸多挑战,但低等生物基因组的设计思路可为高等生物基因组的设计提供参考,并随着高等生物基因组注释不断完善以及设计合成哺乳动物基因组的研究陆续开展,哺乳动物基因组的设计将立足于生物医学研究和生物技术开发的重要需求朝更具开创性、更多元化的方向发展。
2 大片段DNA细胞内组装
大片段DNA(百kb 到Mb 级别DNA)组装技术是合成基因组学的核心技术之一。较小的DNA片段通常可以在体外通过酶切连接、Gibson 组装等方法进行组装,而高分子量的DNA 分子通常通过同源重组(homologous recombination)在体内进行组装。大肠杆菌、枯草芽孢杆菌和酿酒酵母是体内DNA 组装的主要微生物宿主,其中酿酒酵母由于其较高的同源重组能力而成为同时组装多个DNA片段的首选底盘[64]。
2.1 大片段DNA在原核细胞内的组装
革兰氏阳性细菌枯草芽孢杆菌(Bacillus subtilis)和革兰氏阴性细菌大肠杆菌(E.coli)是许多生物技术和合成生物学应用中经常使用的宿主,可用于组装DNA。枯草芽孢杆菌是天然的感受态细胞,具有吸收外源DNA 的能力[65],可将胞外DNA 转化到细胞质中。然后通过RecA 介导的同源重组将导入的DNA 整合到枯草芽孢杆菌基因组中。枯草芽孢杆菌基因组(B.subtilisgenome,BGM)可直接 作 为DNA 克 隆 的 载 体。2005 年Itaya 等[66]将3.5 Mb 的蓝藻PCC6803 基因组整合到4.2 Mb 的枯草芽孢杆菌基因组上形成一个7.7 Mb 的杂合基因组。Itaya 等[67]开 发 了 多 米 诺 骨 牌 法(domino method)组装技术进一步提高在枯草芽孢杆菌中组装的可操作性,其通过两种抗生素标记基因的交替使用,在BGM载体中进行多轮多米诺片段延伸来组装超大DNA片段,成功将163 kb的小鼠线粒体基因组和134.5 kb的水稻叶绿体基因组整合到BGM载体上。
大肠杆菌中天然的RecA 重组系统由于效率较低,未被作为组装工具广泛使用。1998 年Zhang等[68]引入了λ 噬菌体中λRed 基因,开发了λRed/ET 重组系统,可用于在大肠杆菌中实现片段、载体以及基因组之间的同源重组。2012年Fu等[69]将Rac噬菌体的RecET 重组系统用于在大肠杆菌中组装大型DNA 片段,将发光肠杆菌的所有巨型合成酶基因(每个长度为10~52 kb)直接克隆到大肠杆菌表达载体上。λRed 系统更适用于线性和环状DNA 分子之间的重组,而Rac RecET 系统更适用于两个线性DNA分子之间的重组。
在原核细胞中组装的大分子DNA 经过扩增、提取、纯化后,再通过合适的技术转移至哺乳动物细胞中。
2.2 大片段DNA在酿酒酵母细胞内的组装
酿酒酵母具有强大的同源重组能力,因此在合成基因组学为组装大片段DNA 和分子构建的工具被广泛使用。Gibson 等[70]在酿酒酵母细胞中将25个超过20 kb的DNA片段组装成完整的生殖支原体基因组。人工合成酵母基因组计划(Sc2.0)[29]旨在化学合成世界上首个真核生物基因组,利用酿酒酵母高效同源重组实现合成型染色体通过多轮顺序迭代替换野生型酿酒酵母染色体。酿酒酵母合成型染色体的构建是一个多层次逐级组装的过程,首先将化学合成的单链DNA体外组装为750 bp的双链DNA砖块(building blocks),接着继续在体外进一步组装成2~3 kb 的小模块(minichunks)或8~10 kb 的大模块(megachunks),然后一次性将6~12个模块导入酵母细胞中,利用同源重组完成大片段DNA 的组装同时替换对应的野生型染色体序列。Zhou 等[71]将CRISPR/Cas9 和酵母同源重组结合开发了CasHRA 技术并成功构建了1.03 Mb MGE-syn1.0(大肠杆菌最小基因组),CasHRA 通过酿酒酵母原生质体转化向酿酒酵母中同时引入2~3 个数十万碱基对的大型环状DNA,之后环状DNA 被gRNA-Cas9 蛋白复合物切割线性化暴露同源臂,最终在酵母体内完成组装。Mitchell 等[72]利用酿酒酵母成功组装101 kb 人类基因HPRT1,并通过电转将总共114 kb 的YCP 转移至小鼠胚胎干细胞进行基因组位点特异性整合,该研究对在酿酒酵母中DNA 组装的一些细节做了分析,如选择的目标序列的复杂程度对组装的影响、合适的同源臂长度、一轮组装中使用的DNA 片段数与组装效率之间的关系等,可为在酿酒酵母中组装DNA片段的相关研究提供参考。Postma等[73]探索在酿酒酵母中组装的DNA 片段的数量和大小、效率和保真度等的极限,利用酿酒酵母强大的同源重组能力,可通过一次转化实现多达44 个DNA 片段从头组装成50 kb 和100 kb 的模块化的酿酒酵母人工染色体(yeast artificial chromosome,YAC)。2018 年,Shao 等[74]将酿酒酵母的16 条染色体合成为一条染色体,产生有正常功能的单染色体酵母,证明理论上在酿酒酵母中单条染色体的承载能力可达12 Mb。在酿酒酵母中,通常具有不少于40 bp 重叠序列的DNA 片段就能被有效连接起来,但酿酒酵母本身对序列的耐受能力、序列的GC 含量和序列的重复程度都是影响DNA 片段能否正确组装以及组装效率的重要因素。在酿酒酵母中组装细菌基因组的工作表明[75-76],酿酒酵母可以组装并维持稳定GC 含量在32%~38%之间的细菌基因组序列,但高GC 含量可能会限制在酵母中组装的DNA 的长度[77]。尽管高等哺乳动物基因组上均匀分布有能作为酵母复制起始位点元件的序列,但对于高GC 含量的难以组装的序列也可以考虑在合成的序列中额外添加酿酒酵母复制起始位点序列[78],以提高组装的成功率。在酿酒酵母中组装的DNA 大分子可通过酿酒酵母原生质体与哺乳动物细胞融合的方式直接转移到目的宿主中。
目前酿酒酵母被广泛用作大片段从头组装的工具细胞,而除了酿酒酵母以及前文提到的可用于组装的原核细胞,基因组染色体或合成型染色体上大片段DNA 的敲除(knock-out)或敲入(knock-in)通常在DT40(鸡淋巴瘤细胞)[54,79-82]中完成,如Kuroiwa 团队截取分别位于3 条人类染色体(chr2、chr14 和chr22)中的抗体基因,在DT40 细胞中通过Cre/LoxP 系统整合到人工染色体载体上[82],通过微细胞介导的转移将包含人免疫球蛋白基因座的微染色体转移到小鼠ES细胞[80]和牛成纤维细胞中[81],最终构建产人源化抗体的小鼠和牛。由于目前DNA 大片段的转移效率普遍较低,因此在选择和开发用于DNA 大片段重组的哺乳动物的底盘时需要考虑在哺乳动物细胞中组装完成的大片段DNA 是否能进行有效的转移操作。在DT40 细胞中操纵后的染色体可通过微细胞介导的转移技术(MMCT)转移至目的细胞。除了DT40 细胞,中国仓鼠卵巢细胞系(CHO)也是MMCT技术常用的供体细胞,可考虑将其开发成大片段DNA 重组整合的工具细胞[79]。相比于其他动物细胞,DT40 细胞的同源重组能力较强,非同源末端 连 接(non-homologous end joining,NHEJ)能力较低[83],有望被开发成组装大片段DNA 的动物细胞。
不同于细胞中天然的DNA,化学合成的DNA是裸露的DNA,未经过表观遗传修饰,而细胞内的DNA 会发生不同的表观遗传修饰。在原核细胞中,DNA 腺嘌呤甲基化在DNA 复制、修复等许多过程中起着至关重要的作用,由甲基化酶和限制性核酸内切酶组成的限制性修饰系统是细菌进化出的对抗病毒侵袭的重要方法。真核细胞中表观遗传修饰比原核细胞更加复杂,除了DNA 的修饰还包括组蛋白的修饰,可通过修饰组蛋白调节基因表达,在哺乳动物细胞胚胎发育以及整个生命过程中起着重要作用[84]。体内-体外、供体-受体细胞中DNA 表观遗传修饰的差异可能会影响合成型基因组的序列稳定性与功能实现。2009 年Lartigue等[85]将从酿酒酵母中组装的丝状支原体(Mycoplasma mycoides)完整基因组提取后直接移植到山羊支原体(Mycoplasma capricolum)细胞后,由于酿酒酵母与支原体表观遗传修饰系统不兼容,外源性丝状支原体基因组会被限制酶降解,而通过体外供体基因组甲基化或受体细胞限制酶失活,可避免降解。在酵母、DT40等异源细胞中组装完的DNA大分子的表观遗传修饰与目的哺乳动物细胞中不同,可能影响合成型哺乳动物染色体的稳定性与功能。
原核生物如大肠杆菌生长传代的速度比酿酒酵母稍快,且更利于组装的DNA 分子的富集、提取纯化,但其同源重组的能力远不及酿酒酵母,很难一次性组装数十个DNA 片段,且其自身承载外源DNA 大分子的能力也受到很大的限制,不能满足大片段DNA 组装的需求。动物细胞的同源重组能力较弱,难以满足从头组装大片段DNA 的需求。目前大片段DNA 的组装技术主要依赖酿酒酵母体内同源重组,但同源重组利用相同重叠序列来实现片段之间组装的原理也导致了该技术的局限性,即难以有效组装复杂的重复序列。通过前期设计避免同源臂区域出现重复序列,可一定程度减轻重复序列对同源重组组装的干扰,但是对于着丝粒等大段高度重复区域,仍缺少有效的组装方法。大片段DNA 的组装朝着容量更大、效率更高、成本更低的方向发展,对于难以用现有组装方法精确组装的长片段重复序列和复杂的高GC含量序列等还需开发新方法。
3 大片段DNA的转移
大片段DNA 的高效转移方法是其在目的细胞发挥功能的前提和基础。细胞膜由磷脂双分子层和镶嵌蛋白组成,带有净负电荷,DNA 等大分子无法直接通过。可以借助物理、化学或生物方法实现DNA 的转移(表1)。①物理方法:通过电穿孔、基因枪、显微注射等方法将核酸直接导入细胞质或细胞核内。②化学方法:使用脂质体包裹DNA、PEG 介导等使其能通过细胞膜。③生物方法:采用经过遗传学改造的病毒转移非病毒基因至细胞内(又称为转导)、细胞间融合等(图3)。
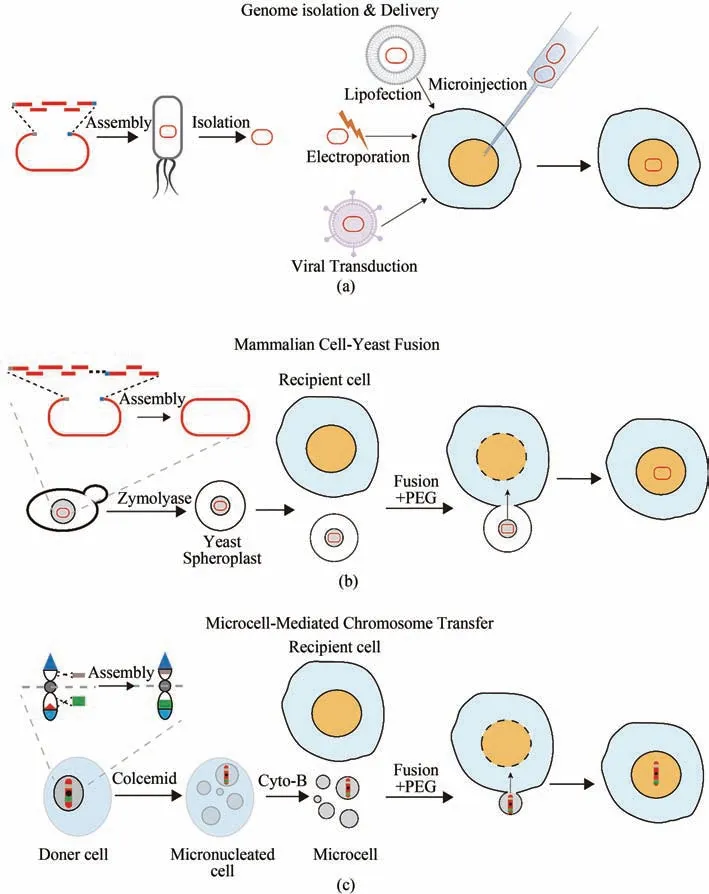
图3 哺乳动物细胞大片段DNA的转移方法(a)由酿酒酵母组装的DNA分子在大肠杆菌中富集后提取纯化到体外并通过显微注射、脂质体转染、电穿孔转移或病毒介导的转染等方法转移到宿主细胞中;(b)由酿酒酵母组装的DNA分子通过PEG介导的酵母原生质体与哺乳动物细胞融合转移到宿主细胞中;(c)在供体细胞中通过自上而下构建的人工染色体以微细胞介导的染色体转移方式转移到宿主细胞Fig.3 Method for delivering large fragments of DNA to mammalian cells(a)DNA molecules assembled by S.cerevisiae are enriched in E.coli,extracted and purified in vitro and transferred to host cells by microinjection,lipofection,electroporation,or virus-mediated transduction;(b)DNA molecules assembled by S.cerevisiae are transferred to host cells through the PEG-mediated fusion of yeast protoplasts and mammalian cells;(c)Top-down artificial chromosomes constructed in donor cell are transferred to host cell through the microcell-mediated chromosome transfer

表1 哺乳动物细胞大片段DNA的转移方法Tab.1 Method for delivering large fragments of DNA to mammalian cells
传统的DNA 转移方法都需要在体外操纵DNA分子,但是大片段DNA 分子受体外操纵的剪切力后极易发生断裂,完整性被破坏[96]。目前,针对Mb 级别的DNA 整合,一般通过多次导入来实现,2014 年Murphy 团队[97-98]通过Velocigen 技术分9 步将约1 Mb 的人免疫球蛋白重链区敲入小鼠基因座中。但多级组装费时费力效率低,开发大片段DNA 一次转移的方法可有效解决该问题。基于琼脂糖包埋和膜融合的转移技术避免了DNA 的体外直接操纵,理论上能实现更大尺度的DNA转移。
3.1 琼脂糖包埋介导的大片段DNA转移
将大片段DNA 包埋至琼脂糖中,避免剪切力等外界因素对DNA 完整性的影响,后续使用脉冲场凝胶电泳分离提纯大片段DNA,该方法分离得到的大片段DNA 的纯度以及完整性能用于显微注射等后续实验。Lartigue 等[99]使用琼脂糖包埋方法将1.1 Mb 的Mycoplasma mycoides基因组分离,并通过PEG 介导转移至Mycoplasma capricolum细胞中。Montoliu 等[100-101]利用琼脂糖包埋法从酿酒酵母中分离出人工染色体,经脉冲场凝胶电泳纯化后,将大于300 kb 的DNA 显微注射至细胞中。Lee等[102]使用琼脂糖包埋方法将2.3 Mb的YAC 分离纯化,随后通过聚L-赖氨酸(PLL)或聚乙烯亚胺(PEI)中和DNA 的负电荷,形成更紧凑的分子,进一步避免大片段DNA 受剪切力导致的断裂,提高其完整性,最终通过脂质体转染将DNA引入小鼠胚胎干细胞。
琼脂糖包埋法只在分离提纯过程中保护DNA不受剪切力的影响,但是转移还要依赖脂质体转染、PEG、显微注射等技术工具,受转移方法本身的承载上限以及效率的影响。
3.2 MMCT介导的大片段DNA细胞间转移
微细胞介导染色体转移(microcell-mediated chromosome transfer,MMCT)技术[103],是目前较常用的一种染色体转移技术。MMCT 过程分为三步:①核化,通过有丝分裂抑制剂将携带人工染色体的DT40、CHO、A9 细胞周期同步至M 期;②核去除,通过细胞松弛素破坏细胞骨架,密度梯度离心,制备单染色体的微核细胞;③融合,通过PEG、电融合、仙台病毒介导,使微核细胞与目的细胞融合,完成染色体的转移。虽然MMCT 被广泛使用,但操作步骤烦琐,通过PEG介导的MMCT 融合效率极低,一般在10−6~10−5[93,104]。提高微核的生成效率以及微核与目的细胞的融合效率可以改进MMCT 技术。2016 年Liskovykh 等[105]使 用 TN-16、 Griseofulvin 和Latrunculin B 分别替代Colcemid 和Cytochalasin B破坏细胞骨架,并在供体细胞中过表达Collagen/Laminin促进黏附,使MMCT的转移效率提高了近6 倍;2016 年Teruhiko 等[106]在供体细胞CHO 表达
鼠类白血病病毒(murine leukemia retroviruses,MLVs)的包膜蛋白(EnvΔR)与目的细胞膜表面受体蛋白Pit-2(sodium-dependent phosphate transporter)结合进而促进膜融合的发生,使MMCT 的效率提高了26.5倍。
3.3 酿酒酵母-哺乳动物细胞融合介导的大片段DNA细胞间转移
酿酒酵母具有强大的同源重能力,可用于从头组装Mb级别的合成型染色体。通过PEG 介导的酿酒酵母原生质体-哺乳动物细胞融合能实现大片段DNA 转移。1993 年Klapholz 等[107]通过PEG 介导的细胞融合将YAC 转移至哺乳动物细胞,在该过程中酿酒酵母通过酶解去除细胞壁,形成原生质体,在PEG 作用下与目的细胞发生膜融合,使YAC 转 移 至 目 的 细 胞。2013 年Li 等[95,108]通 过PEG 介导的酵母原生质体-哺乳动物细胞融合,将在酿酒酵母中组装的1.4 Mb 大小的人T 细胞受体基因转移至小鼠胚胎干细胞,产生人源化T细胞受体小鼠。但是,PEG 介导的酵母向哺乳动物细胞转移大尺度DNA 效率极低,在10−6~10−5。其低效的原因之一是通过融合转移的大片段DNA 大多数位于动物细胞质中,不能稳定到达细胞核内发挥功能。为了提高DNA 转移至细胞核的效率,2017 年Brown 等[94]用秋水仙素、Nocodazole 等有丝分裂微管形成抑制剂处理受体细胞,使细胞处于M 期,此时细胞核膜消失,融合后细胞重新进入周期,处于胞质中的YAC 被新生核膜包裹,进入细胞核内,转移效率提高了近300倍。目前,通过酿酒酵母原生质体-哺乳动物细胞融合技术转移大片段DNA 的同时,酿酒酵母基因组也会转移至动物细胞中,并可能发生随机整合,存在对动物细胞基因组造成扰动的风险。
综合比较以上3种方法,MMCT和酿酒酵母原生质体-哺乳动物细胞融合过程都需借助PEG,高PEG 浓度和长接触时间能显著提高转移效率,但同时PEG 对细胞的毒性增加[94]。由于PEG 介导的融合机制尚不清楚,无法针对其进行有理论指导的改进。生理过程中的膜融合例如病毒感染、突触小泡融合、骨骼肌细胞的融合、精子-卵子的融合等过程,有望为MMCT 以及酿酒酵母原生质体-哺乳动物细胞融合提供理论指导。膜融合介导的转移不仅能绕过DNA 的体外操纵步骤,实现大片段DNA 的完整转移,更重要的是将从头合成的合成型动物染色体直接转移至目的细胞,极大简化了操作步骤。对膜融合介导的转移效率的优化研究,有望打通合成型动物染色体的合成组装与功能研究之间的鸿沟,为动物合成基因组学提供关键研究工具。
4 哺乳动物大片段DNA 设计与操纵的应用前景
哺乳动物合成基因组学采用自下而上的策略,引入多元化设计,以不同于常规传统方法的新方式揭示基因功能和调控以及疾病发生发展的机制。为了更好地了解人类疾病,数十年来研究者们创建出来多种动物模型,基因组改造的范围从单个核苷酸、特定氨基酸、单个外显子、单基因、多基因、大片段基因座到染色体级。哺乳动物大片段DNA 设计与操纵可在人类医药健康领域发挥重要的作用。
4.1 涉及大片段DNA的疾病动物模型
合成基因组学的大片段操纵技术可为研究人类重要基因及基因异常导致的严重疾病提供技术支持。编码人α-珠蛋白和β-珠蛋白两个亚基的血红蛋白基因的异常或缺失会导致地中海贫血,Suzuki等[109]将包含五个功能基因(ε、Gγ、Aγ、δ和β)大小约130 kb 的人β-珠蛋白基因簇引入HAC,随后转移至小鼠胚胎干细胞中并研究其表达模式。合成基因组学的技术可设计α-珠蛋白和β-珠蛋白基因编码区域及调控区域的变体来替代小鼠和小鼠细胞系中天然存在的α-球蛋白基因和β-珠蛋白基因,以研究它们对血红蛋白产生的影响并揭示地中海贫血的致病机理。相比于传统的研究方法,设计合成的变体可以从新的角度解析致病机理,同时为疾病治疗提供新思路,并进一步使我们对基因表达的总体调控方式有所了解。
大片段DNA 操纵技术还可用于构建非整倍体性综合征模型和染色体大片段变异导致的癌症模型。21 三体综合征(Trisomy 21)也称为唐氏综合征(Down syndrome),是最常见的染色体异常疾病。O′Doherty 等[110]将几乎完整的人21 号染色体转移到小鼠中构建出人唐氏综合征的小鼠模型,以研究非整倍体染色体疾病。大部分癌症细胞的基因组呈非整倍性,包括染色体结构与数目的异常。染色体的不稳定性可能在直肠癌等癌症的发生发展中起一定作用[110],通过合成型哺乳动物染色体构建癌症模型,可研究癌细胞中引发染色体不稳定的机制以及基因组的非整倍体异常如何促进癌症的发展。
大片段操纵技术不仅能构建动物模型,而且有望在已经明确致病机制的多基因遗传病和涉及大片段基因区域的疾病的治疗中发挥作用。2010 年Oshimura团队[111]将大小约2.4 Mb的肌营养不良蛋白基因完整序列引入至人工染色体载体上,通过MMCT转移至杜氏肌肉营养不良(Duchenne muscular dystrophy,DMD)模型小鼠和人类DMD患者的诱导性多能干细胞(iPS)中,纠正了由肌营养不良蛋白的缺失或突变引发的疾病表型。
4.2 人源化免疫系统
将Mb 级别的人免疫球蛋白基因序列引入小鼠中,为研究人免疫反应、开发单克隆抗体提供模型。20 世纪90 年代,多个团队将人免疫球蛋白基因座整合至小鼠基因组上[112-118],培育出能够产生人免疫球蛋白的小鼠,这些人源化抗体小鼠模型是药物研发领域的重要进步,但是第1代人源化抗体小鼠主要有3个问题:①随机插入引起基因组不稳定,人免疫球蛋白基因区域中的调控序列与宿主自身调控分子相互作用弱,导致其B细胞的发育和功能受损;②人源化抗体恒定区与小鼠B细胞受体相互作用受损,影响B细胞谱系发育;③全人源化抗体与Fc 受体结合能力受损,导致亲和选择下降和抗体血清浓度降低。这三个问题都可以通过免疫球蛋白基因的原位替换来解决。2014 年Murphy 团队[97-98]使用同源重组技术,Bradley 团队[63]使用重组酶介导的盒式交换技术,均在mES细胞中实现了Mb 级别的原位迭代替换,培育出第2 代人源化抗体小鼠。与第1 代人源化抗体小鼠不同,这些小鼠的B细胞发育以及免疫系统均没有损伤,这些新的小鼠模型为治疗性抗体的发现提供了强大的通用平台。与引入天然的免疫球蛋白基因序列不同,2021 年Xu 等[119]从羊驼、单峰驼、骆驼中选出并合成30 个VHHs 基因片段,通过CRISPR/Cas9 技术替换约2.5 Mb 的小鼠免疫球蛋白重链V 基因区,生成能产生驼科纳米抗体的纳米小鼠(nanomouse),并能产生针对SARS-CoV-2的纳米抗体,展现出重新设计免疫球蛋白基因座的独特优势重要应用价值。
此外,将人免疫球蛋白基因引入牛等大型哺乳动物中,可快速、高效、大量生产针对未知病毒(新抗原)的人源化抗体,在突发性疾病预防上具有独特的应用前景。2002 年Kuroiwa 团队[81]构建了包含人免疫球蛋白重链和λ链的人工染色体载体,之后通过MMCT、体细胞核移植,培育出表达人源化抗体的牛,但是牛源抗体仍然占主要部分。将人源免疫球蛋白基因调控区域替换成牛源序列能够大幅提高人源化抗体的产量。2013 年Kuroiwa 等[82]将人抗体重链基因恒定区替换为牛源恒定区,人源抗体量最高可达9 g/L。随后,2015 年Kuroiwa 等[120]将人源免疫球蛋白基因类型转换元件换成牛源序列,全人源抗体平均产量为5 g/L,最高可达15 g/L。2016年,针对中东呼吸综合征(MERS),SAB公司利用人源抗体牛生产全人源化IgG,最高可达15 g/L[121-122]。2018 年,该研究已经进行健康志愿者人体实验,有望在未来进入临床试验[123]。
异种器官移植有望解决器官短缺的重大医学问题。2017 年,Yang 等[124]通过CRISPR/Cas9 基因编辑技术失活了猪原代细胞中所有的25 个猪内源性逆转录病毒(porcine endogenous retroviruses,PERVs)基因,并通过体细胞核移植,最终培育出内源性逆转录病毒失活的猪。2019年Yang团队[125]使CRISPR/Cas9 和转座子系统在42 个等位基因上设计改造猪基因组,并生产了无内源性逆转录病毒、异种抗原基因敲除且携带9个抗免疫排斥功能人源基因的转基因猪。利用大片段DNA 操纵技术构建抗免疫排斥人源化染色体,结合高效的内源基因组编辑技术,有望加速异种器官移植的研究。
4.3 涉及大片段DNA的代谢动物模型
由于物种之间的差异,小鼠、大鼠等动物模型无法真实反映人体相关代谢情况,可以通过引入完整的人源代谢通路,实现在人源化的动物模型中模拟人体环境进行相关研究。完整的人源代谢通路往往包含较大的基因组区域,或涉及多个位点的大片段基因,很难通过常规技术将完整的人源代谢通路引入小鼠中。哺乳动物细胞大片段DNA 操纵技术的发展可为人源代谢通路的引入提供技术支持。2013 年Oshimura 团队[126]以自上而下构建的微染色体为载体将大小约700 kb 的天然人源CYP3A(cytochrome P450 family 3 subfamily A)基因区域引入小鼠中,构建人源化动物模型。CYP3A 基因编码参与药物代谢的酶,并与大约50%的市售药物的代谢有关[127],人源化的CYP3A模型动物可用于预测CYP3A 相关的药代动力学和新药在人体内的毒性。2019 年Kazuki 团队[128]将UGT2(UDP glucuronosyltransferase family 2)的编码基因区域(约1.5 Mb)引入大鼠中,构建了人源化药物糖基化代谢模型动物。
设计和操纵哺乳动物大片段DNA 在多基因以及染色体疾病的研究上有独特的优势。将多个功能相关基因成簇化排列,设计合成有特定功能的大型DNA 区域,一步直接改造目的细胞,将避免分步改造操作烦琐、效率低等问题;承载大片段DNA 的合成型哺乳动物染色体不仅能用来构建疾病模型,结合干细胞治疗技术,有望用于临床上治疗复杂遗传性疾病。
5 展望
在哺乳动物细胞中设计与操纵大片段DNA 具有重要的科学意义,对高等哺乳动物基因组的设计再造将推动对高等生物的理解,在人类医学健康领域具有广阔的运用前景。包括设计-组装-转移在内的大片段DNA 操纵技术是哺乳动物合成基因组学研究的基础。与现有微生物合成基因组学领域相比,多细胞真核水平的动物合成基因组学在设计和操纵上仍具有挑战。
当前,哺乳动物基因组设计的挑战主要体现在由于对基因组解析不够完善,基因组的复杂性如精密的基因时空调控系统、基因间相互作用网络、表观遗传调控、细胞间相互作用、多细胞发育等,设计再造哺乳动物基因组向更大尺度的进展受到阻碍。利用计算机辅助基因组设计,并随着ENCODE、GWAS 等基因组注释项目的不断推进,未来哺乳动物合成基因组学可进一步实现更大尺度的、更加多元化的设计,在更深入理解哺乳动物基因组的同时提供新的见解,进一步揭示高等真核生物基因组的奥秘。着丝粒是合成型哺乳动物染色体在细胞内稳定存在及正确分配的保障,目前新型合成型着丝粒可以绕过传统合成型着丝粒对天然微卫星序列以及CENP-B结合序列的需要,为哺乳动物染色体着丝粒序列研究提供了一个新思路。
依托现有的技术,能初步实现大片段DNA 的组装、转移,但存在操作难、效率低的问题。大片段DNA 的组装主要依靠酿酒酵母细胞内的同源重组,对于难以组装的复杂序列目前仍没有有效的方法精准组装,Mb 级别DNA 难以快速高效组装。现有的DNA 大片段组装主要基于技术相对成熟的酿酒酵母同源重组,但复杂的DNA 序列,尤其是高等哺乳动物细胞中常见的高度重复序列在酿酒酵母细胞中存在不稳定现象,限制了酵母组装在哺乳动物基因组学中的应用。开发新的组装底盘以及调控酿酒酵母同源重组,使其在完成高效组装的同时又能稳定高度重复序列,是DNA 大片段组装技术发展方向之一。大片段DNA 的跨物种转移技术是哺乳动物合成基因组学研究中的重大挑战,需在体外纯化DNA 的转移技术因大片段DNA 的不稳定而受到限制,大片段DNA 细胞间转移技术如MMCT、酿酒酵母原生质体融合等都有较大的使用局限性,如MMCT 技术只能将DT40、A9 等能形成微核的细胞中的人工染色体转移到哺乳动物细胞中,而酿酒酵母原生质体融合方法转移的大片段DNA 通常只能是在酿酒酵母细胞中能被成功组装、在酿酒酵母细胞中能稳定存在的序列,因此该方法不适用于转移自上而下截短的染色体。化学、材料等学科的发展为经纯化过的DNA 向细胞内的转移提供新载体工具,对生理过程中膜融合现象的研究能为细胞间转移提供新思路。此外,由于哺乳动物细胞较低等生物细胞更为复杂,在其中操纵大片段DNA 可能导致许多与预期不同的结果,如编辑过程中的脱靶效应、大片段的意外删除或重排等,因此在哺乳动物细胞中快速找到并修正这些与预期不同的错误也是操纵工作的一大难点。需技术的进一步优化和开发推动领域的发展。相关技术以及新成果的不断涌现,可为大片段DNA 在哺乳动物细胞中的设计和操纵提供参考。
对于大片段DNA 在哺乳动物细胞中的存在形式,除了设计独立于哺乳动物基因组的合成型染色体,设计构建大片段DNA 整合到哺乳动物细胞基因组上也是在哺乳动物细胞中操纵大片段DNA的一个重要策略。整合策略可根据研究内容的具体需求进行非特异性整合或位点特异性原位整合,且整合到宿主基因组后引入的大片段DNA 能像内源DNA 一样稳定存在。而独立于宿主基因组的合成型染色体相比于将大片段整合到基因组上能很大程度地减少对宿主基因组的扰动,此外利用合成型染色体策略的瓶颈主要为转移步骤,而大片段整合策略的效率除受到转移步骤的制约外还受到大片段DNA 整合到基因组上效率的制约,综合来看两种存在形式的策略各有利弊。对基因组大尺度的改造还应考虑到对后续研究如表观遗传调控、发育生物学等的影响。
哺乳动物合成基因组学处于发展的初期,虽然哺乳动物细胞中大片段DNA“设计-组装-转移”的技术路线仍充满挑战,但技术难题正逐个被攻破。哺乳动物合成基因组学自下而上的研究在染色体疾病模型构建、异种器官移植、人源化免疫系统构建等研究领域具有重要应用。可以预见,随着哺乳动物合成基因组学的发展,对高等生命系统的理解和认识将进一步加深,并为人类医药健康做出贡献。

