红花花青素还原酶基因ANR的克隆及表达分析
鲁丹丹 谭政委 李 磊 余永亮 许兰杰 杨红旗 董 薇 梁慧珍
(河南省农业科学院芝麻研究中心,河南 郑州 450002)
红花(CarthamustinctoriusL.)属于菊科红花属一年生双子叶草本植物,是一种非常古老的多用途经济作物。红花种子的亚油酸含量很高,可用于榨油,幼苗可作为蔬菜食用,花可以用于制作染料和化妆品等,干燥花还有活血通经、散瘀止痛的功效[1]。在我国,红花作为药物已有2 100多年的历史,大量研究表明,红花花中主要的活性化合物是黄酮类化合物,如羟基红花黄色素A、红花素和黄酮醇等[2]。红花的品质也直接受这些物质含量的影响。
黄酮类化合物是植物中普遍存在的次级代谢产物,是以黄酮为母核而衍生的一系列化合物,它在植物体内既可以与糖结合成苷类或碳糖基,也能以游离形式存在。黄酮类化合物复杂的结构多样性决定了其功能的多样性,如抵御紫外线照射[3],防御虫害、病害[4-6]等,而食物中的黄酮类化合物通过人体吸收后可以维护身体健康[7]。植物体内黄酮的生物合成途径一直是科学界广泛关注的课题,在模式植物拟南芥中,黄酮的生物合成途径已基本明确,但植物体内黄酮代谢极其复杂多变,因而其他物种中黄酮的合成和调控机制还亟待解决。近年来,越来越多红花黄酮生物合成途径的基因被鉴定克隆,如脂肪酸去饱和酶基因(fatty and desaturase,FAD2)[8]、查尔酮合成酶基因(chalcone synthase,CHS)[9-10]、黄烷酮3-羟化酶基因(flavanone 3-hydroxylase,F3H)[11]、UDP-糖基转移酶基因(UDP-glycosyl transferase, UGT)[12]、花青素合成酶基因(anthocyanidin synthase,ANS)[13]、查尔酮异构酶基因(chalcone isomerase,CHI)[14-15]等。
花青素又称花色素,是重要的黄酮类化合物,也是植物体内普遍存在的天然色素。花青素使植物不同组织部位在不同的pH值条件下呈现不同的颜色。原花青素是花青素在花青素还原酶的催化下形成的产物,是类黄酮代谢途径下游的次级代谢产物。原花青素具有强抗氧化、消除自由基的作用,是强力的金属螯合剂,还可以保护和稳定维生素C[16]。花青素还原酶是合成原花青素即缩合单宁的关键酶,它可以将有色的花青素(矢车菊素、天竺葵素、翠雀素)还原合成表儿茶素,通过进一步向液泡转运聚合成为原花青素,对花青素含量具有一定的负调控作用[17-18],因而其表达也对植物花瓣、种皮等呈色有重要影响。1997年,Albert等[19]在拟南芥中克隆到了第一个花青素还原酶基因,随后在桑树[20]、越橘[21]、油茶[22]、蛇根草[23]、芒果[24]、花生[25]、红掌[26]、甘薯[27]、笃斯越橘[28]、川桑[29]、苗药艾纳香[30]、葡萄[31]等植物中也相继克隆出了ANR基因,而在红花中花青素还原酶基因相关研究还鲜见报道。本研究以前期红花花期转录组测序的Unigene序列为参考序列设计引物,以红花花冠总RNA反转录出cDNA为模板,通过PCR扩增及测序分析,获得红花花青素还原酶基因ANR的全长序列;通过生物信息学的分析方法对该序列的理化性质、保守结构域、疏水/亲水性、二级结构及三级结构进行分析,并对该序列进行系统进化分析;另外检测该基因在不同花色红花品种的不同组织及不同花期的表达量,以期为解析红花类黄酮生物合成的分子机制及应用分子技术的方法提高红花黄酮类化合物的含量,培育优质红花品种提供理论基础。
1 材料与方法
1.1 试验材料
供试材料为河南省农业科学院芝麻研究中心提供的红色大果球红花品种及白色红花品种,于2018年11月下旬种植于河南省农业科学院现代农业研究开发基地。待幼苗长至3~5片真叶时,采取不同花色红花幼嫩根、茎、叶,现蕾后,取苞片以及不同开花时期的花瓣置于液氮中,每个样品取3个重复,-80℃冰箱保存备用。
1.2 总RNA提取及cDNA第一链合成
液氮研磨样品,按照华越洋生物公司的Quick RNA Isolation Kit试剂盒说明书提取红花样品的总RNA。利用1.2%琼脂糖凝胶电泳检测提取的总RNA的质量和完整性,浓度的测定利用 NanoDrop 2000分光光度计进行。利用反转录试剂盒(大连TaKaRa, PrimeScriptTMRT reagent Kit with gDNA Eraser)将总RNA反转录为cDNA第一链,反转录操作步骤详见其说明书(Code No. RR047A)。
1.3 CtANR基因的克隆
以花瓣基本伸出苞片时的红花花器官cDNA为模板,根据转录组测序的Unigene基因序列设计引物(表1),利用KOD酶进行PCR扩增。扩增体系总体积 20 μL,包含 2×PCR buffer for KOD FX 10 μL、2 mmol·L-1dNTPs 4 μL、cDNA 模板 2 μL、正反向引物各 0.6 μL、KOD FX 0.4 μL和ddH2O 2.4 μL。反应程序如下:94℃预变性 2 min;98℃变性 10 s,52℃退火 30 s,68℃延伸 2 min,共35个循环;68℃延伸 5 min。经琼脂糖凝胶电泳检测获得预期大小的片段,利用生工生物工程上海(股份)有限公司Sangon Biotech SanPrep柱式DNA胶回收试剂盒(Lot.: AA24KA4369)回收目的片段,连接到TaKaRa pMD19-T载体上,热击法转化TaKaRa DH5α感受态细胞,涂布于含氨苄青霉素抗性的LB培养基[胰蛋白胨(Tryptone) 10 g·L-1;酵母提取物(Yeast extract) 5 g·L-1;氯化钠(NaCl) 10 g·L-1;用NaOH调节该培养基的pH至7.4]上,37℃培养16 h。挑取单菌落,PCR检测后,阳性菌落送往河南尚亚生物技术有限公司测序。
1.4 CtANR基因序列的生物信息学分析
将CtANR基因FASTA格式的核苷酸序列导入DNAMAN6.0软件,利用软件自带的翻译功能对CtANR基因编码的氨基酸序列进行预测,使用NCBI中的CD-Search(https://www.ncbi.nlm.nih.gov/Structure/cdd/wrpsb.cgi)对氨基酸序列的保守结构域进行鉴定。使用ProtParam(https://web.expasy.org/protparam/)对CtANR蛋白质的相对分子质量、等电点、稳定性等进行分析。使用ProtScale(https://web.expasy.org/protscale/)在线工具对红花ANR蛋白的疏水性和亲水性进行预测分析。CtANR蛋白的二级结构及三级结构的预测分别使用在线工具SOPMA(https://npsa-prabi.ibcp.fr/cgi-bin/npsa_automat.pl?page=/NPSA/npsa_sopma.html)及SWISSMODLE(https://swissmodel.expasy.org/interactive)。通过NCBI数据库中的Blastp进行CtANR同源序列的搜索,并使用DNAMAN软件进行红花和其他植物的ANR氨基酸同源序列比对分析。使用MEGA 5.05软件中的Neighbor-joining构建关于CtANR蛋白的系统化进化树,并通过Bootstrap方法对进化树进行检测,Bootstrap值设置为1 000。系统发育树中序列的模体结构采用MEME(https://meme-suite.org/meme/tools/meme)在线网站进行分析。
1.5 实时荧光定量PCR(qRT-PCR)
通过实时荧光定量PCR(quantitative real-time PCR, qRT-PCR)技术检测CtANR基因在红花不同组织及花发育不同时期的表达情况,利用Primer Premier5设计荧光定量PCR引物,内参基因Ct60S引物序列参考文献[32](表1)。qRT-PCR在德国艾本德公司的Eppendorf Mastercycler ep Realplex 2.2 Detection System上进行,试剂使用大连TaKaRa公司的 TB Green®Premix Ex TaqTMⅡ(Tli RNaseH Plus)。反应体系为10 μL:5 μL TB GreenPremix Ex TaqⅡ(Tli RNaseH Plus)(2×),1.5 μL cDNA,0.4 μL正向引物(10 μmol·L-1),0.4 μL反向引物(10 μmol·L-1),2.7 μL无酶ddH2O。首先将相同组分混匀配成混合液,之后分别加入不同体系中,以保证反应条件的一致性,再加入差异组分。反应程序:95℃预变性3 min; 95℃变性10 s,55℃退火30 s;72℃延伸28 s,45 个循环。通过熔解曲线分析监测PCR扩增的特异性,反应程序为以0.1℃·s-1的速度从55℃逐渐升温到94℃。每个样品的检测平行重复3次。通过2-ΔΔCT法对基因进行表达水平分析,利用Microsoft Excel 2007中的统计分析方法T-检验对不同开花时期、不同组织CtANR的表达量进行显著性分析。

表1 试验中所用CtANR基因引物序列、名称及用途Table 1 Oligonucleotide primer names, sequences and function in the amplification of CtANR genes
2 结果与分析
2.1 红花ANR基因的克隆
以大果球红花品种花瓣总RNA反转录合成的cDNA为模板,根据红花转录组测序的Unigene基因序列设计引物CtANR-F/R(表1)进行PCR扩增,扩增得到的条带在1 000 bp左右的位置上(图1)。将该条带切胶回收后与载体pMD19-T连接,转化至DH5α感受态细胞后,将菌液均匀涂布在含有氨苄青霉素抗性的培养基上,37℃培养16 h后,随机挑选单菌落进行PCR鉴定,将阳性菌落送往上海生工生物工程有限公司测序。测序结果用MEGA5.05软件进行分析,结果显示,目的片段大小1 020 bp,碱基序列与预期一致,将其命名为CtANR。
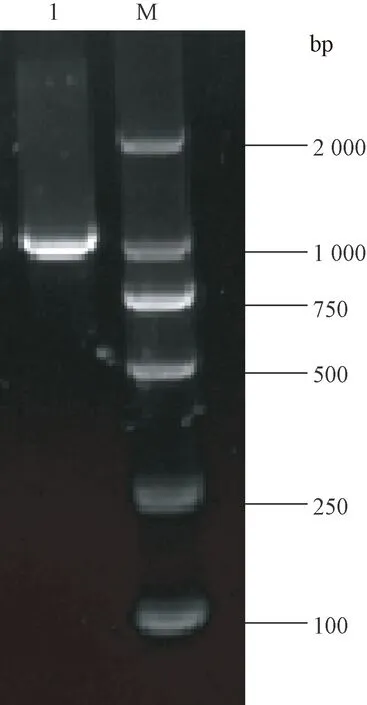
注: M:DL2000 DNA marker; 1:CtANR基因PCR扩增产物。Note: M:DL2000 DNA marker. 1:PCR product of CtANR gene.图1 CtANR基因的扩增Fig.1 Amplification of CtANR from Carthamus tinctorius L.

注: A: CtANR基因的核苷酸序列及其推导的氨基酸序列;B: CtANR蛋白的保守结构域。Note: A:Nucleotide and deduced amino acid sequences of CtANR. B: Conserved domains of CtANR protein.图2 CtANR基因的核苷酸序列和推导的氨基酸序列及序列特征Fig.2 Nucleotide and deduced amino acid sequences of CtANR and its sequence character
2.2 红花ANR基因的序列分析
经NCBI的BLAST比对后,发现该片段含有一个长为1 020 bp的完整开放阅读框(open reading frame, ORF),起始密码子为ATG,终止密码子为TAA。利用DNAMAN6.0软件对该序列进行分析发现,该基因编码339个氨基酸(图2-A),通过Prot Param软件在线预测CtANR蛋白的氨基酸组成及比例,Glu数量最多,占9.1%,Trp数量最少,占0.6%(表2)。CtANR基因编码的蛋白质分子质量为37 958.28,理论等电点(pI)为5.87,包括5 318个原子,分子式为C1697H2649N445O512S15。带负电荷残基(Asp+Glu)43个,正电荷残基(Arg+Lys)35个,在哺乳动物体内的半衰期为30 h,脂肪系数为87.70,亲水性指数为-0.242,不稳定指数为41.63,为不稳定的亲水性蛋白。
2.3 红花ANR基因的生物信息学分析
利用ExPASy-Prot Scale在线工具的Hphob./Kyte & Doolittle分析CtANR蛋白的亲水/疏水性(正值表示疏水性,负值表示亲水性),分析结果表明,负值数量多于正值数量,且198位苏氨酸(Pro,P)具有最高分值(2.2),疏水性最强,285位脯氨酸(Glu,E)具有最低分值(-2.6),亲水性最强(图3)。
利用SOPMA在线工具对CtANR蛋白的二级结构预测发现,该蛋白编码的339个氨基酸残基中42.18%的氨基酸组成α-螺旋,无规则卷曲占36.28%,延伸链占13.57%,仅有7.96%的氨基酸组成β-折叠(图4-A)。
为了进一步了解红花ANR蛋白的结构,利用SWISS-MODEL对红花ANR蛋白进行三维结构的建模分析(图4-B)。结果表明,其三维结构模型与蛋白质二级结构预测的结果一致。
用NCBI的CD-Search分析红花ANR蛋白的功能结构域,结果显示该蛋白含有3个功能位点,即活性位点4个,烟酰胺腺嘌呤二核苷酸磷酸(nicotinamide adenine dinucleotide phosphate, NADP)结合位点10个,底物结合位点10个,属于NADPH结合位点超家族(NADB-Rossmann superfamily)(图2-B)。
2.4 红花ANR蛋白的系统进化分析
通过NCBI Blastp查找红花ANR的同源氨基酸序列,并通过DNAMAN6.0软件将红花ANR和其他植物中的ANR进行氨基酸多序列比对。分析表明红花ANR的氨基酸序列和其他植物的ANR在氨基酸水平上同源性很高,其中红花ANR与刺菜蓟ANR的相似

表2 CtANR氨基酸成分Table 2 Amino acid composition of CtANR
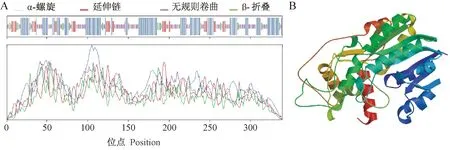
图4 CtANR蛋白的二级结构和三级结构Fig.4 Secondary and tertiary structure of CtANR protein
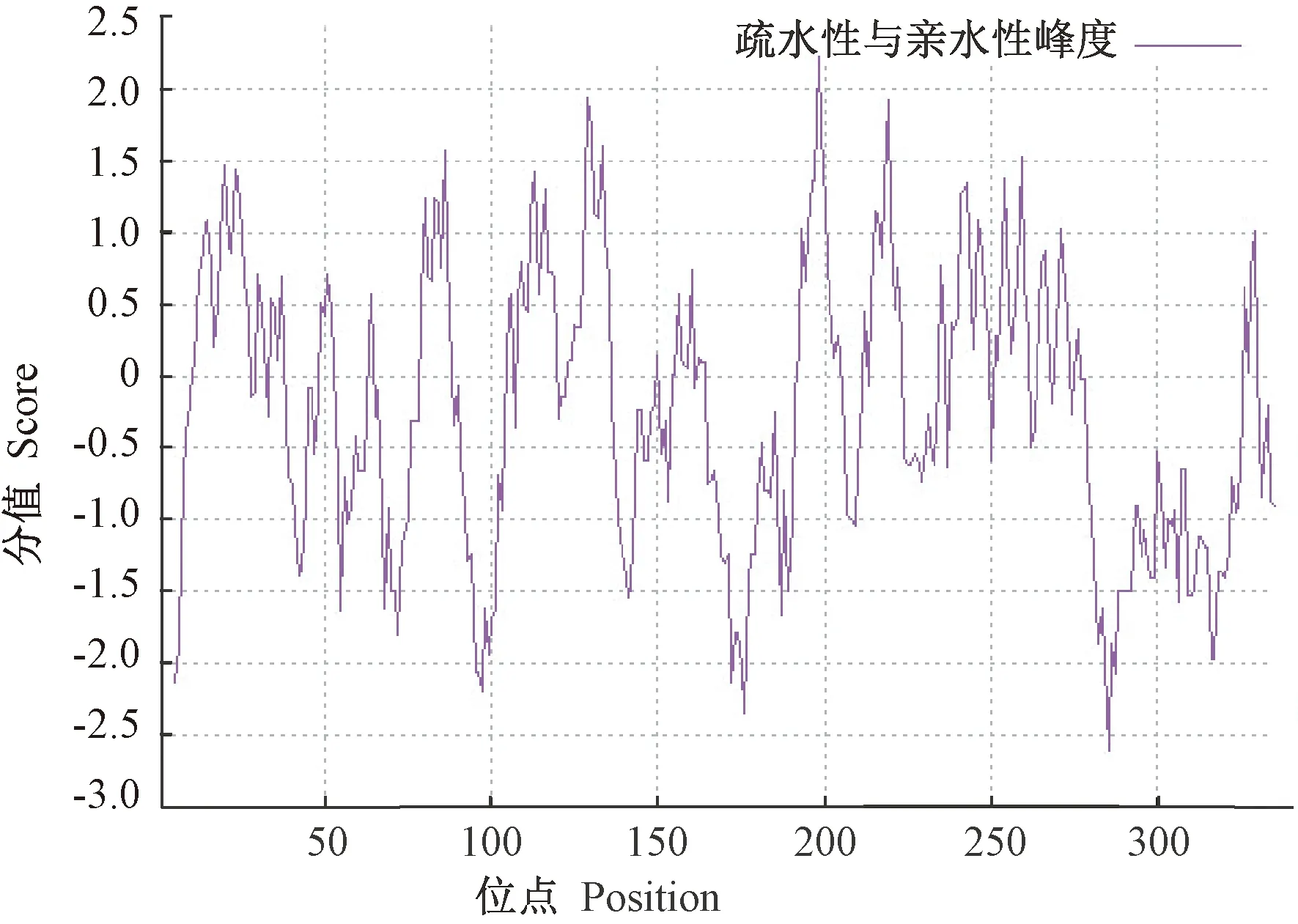
图3 CtANR蛋白疏水性/亲水性预测Fig.3 Hydrophobicity or hydrophilia prediction of CtANR protein
性为83.98%,与莴苣ANR的相似性为79.46%,与向日葵ANR的相似性为76.56%,与黄花蒿ANR的相似性为70.24%,与薇甘菊ANR的相似性为71.08%(图5)。此外,与其他植物ANR类似,红花ANR也含有NADP辅酶结合位点(GGAGYI/LR/VATPL/TA/CGLV/S)、活性位点(105T/130S/170Y/174K)和底物结合位点(90L/92H/131V132V/135S/199C200G201L/217S/243P)。序列比对表明,ANR的辅酶结合位点及活性位点都是比较保守的,而底物结合位点存在较大变异,如红花ANR在214位和231位有底物结合位点S和Ⅰ(图5),其他植物ANR则不含有这两个底物结合位点。
从NCBI数据库中选取其他植物ANR蛋白序列与红花ANR蛋白序列进行系统进化树分析,从进化树(图6)可以看出,CtANR蛋白与同为菊科的刺菜蓟进化关系最近,首先聚为一支,与菊科莴苣、黄花蒿,豆科花生、大豆、赤豆聚为一个大分支,亲缘关系较近,而与蔷薇科苹果、梅花、欧洲甜樱桃,葡萄科葡萄等的亲缘关系较远。除葡萄、陆地棉、榴莲外,其他各科不同物种各自聚成一组,表明不同物种的ANR蛋白具有明显的种属特性。模体结构分析显示,基序3、6、8比较保守,在系统进化树的17个物种中都含有,而基序1比较特异,只在梅花和欧洲甜樱桃中有。此外,聚合在系统进化树同一分支内的不同植物ANR的模体基序是相似的,关系越近,模体基序越相似。红花ANR蛋白的模体基序和其他植物ANR蛋白的非常相似,且与刺菜蓟、莴苣的模体基序最相似(图6)。

注: Ct: 红花; Aa: 黄花蒿; Mm: 薇甘菊; Ls: 莴苣; Ha: 向日葵; Cc: 刺菜蓟。方框标志为 NADP辅酶结合位点, *为活性位点,#为底物结合位点。Note: Ct: Carthamustinctorius. Aa: Artemisia annua(PWA68657.1). Mm: Mikania micrantha(KAD3069085.1). Ls: Lactuca sativa(XP_023746445.1). Ha: Helianthus annuus(XP_022035717.1). Cc: Cynara cardunculusvar. scolymus(XP_024992787.1). Coenzyme NADP binding sites are framed. Active sites are marked with*. Substrate binding sites are marked with #.图5 红花和其他植物的ANR蛋白多序列比对Fig.5 Multiple sequence alignment of ANR protein from Carthamus tinctorius and other plants
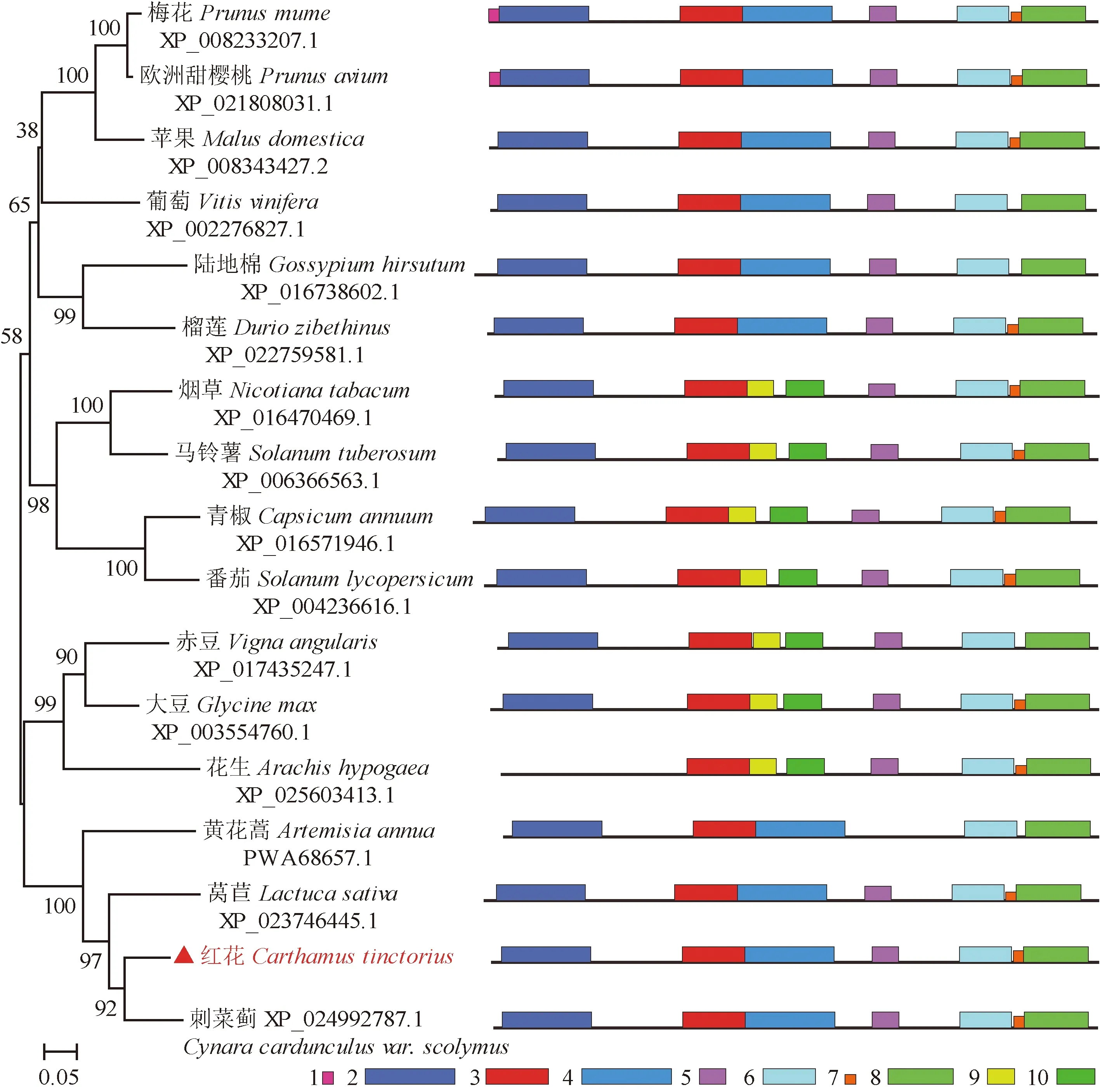
图6 不同植物基于ANR氨基酸序列的邻接法系统进化树及模体分析Fig.6 A neighbor-joining phylogenetic tree and motif prediction of different plants based on amino acid sequence of ANR
2.5 红花ANR基因的表达分析
利用qRT-PCR技术检测不同花色红花品种开花各时期即花组织形成初期(S1)、花组织形成中后期(S2)、花瓣基本伸出期(S3)、盛花期(S4)、花凋谢期(S5)及不同组织[初期果球(F)、苞片、根、茎、叶]中CtANR基因的表达量。熔解曲线分析表明,内参基因Ct60S和目标基因CtANR都只出现了一个特异峰(图7),表明引物具有很好的特异性,排除了引物二聚体和非特异性扩增产物对结果带来影响的可能。定量数据分析表明,红色和白色红花品种中CtANR基因均在果球形成初期表达量最低,其次是根和茎,在花中的表达量相对较高。另外,在花发育的不同时期,红色红花品种中CtANR基因的表达量呈现先降低后升高再降低的趋势,而白色红花品种中CtANR基因的表达量呈现先降低后升高的表达趋势。其次,红色和白色红花品种中CtANR基因的表达量在花发育的S1、S3和S5时期差异极显著(P<0.01),S4、苞片、根、叶中差异显著(P<0.05),其他时期、其他组织中差异不显著(图8)。
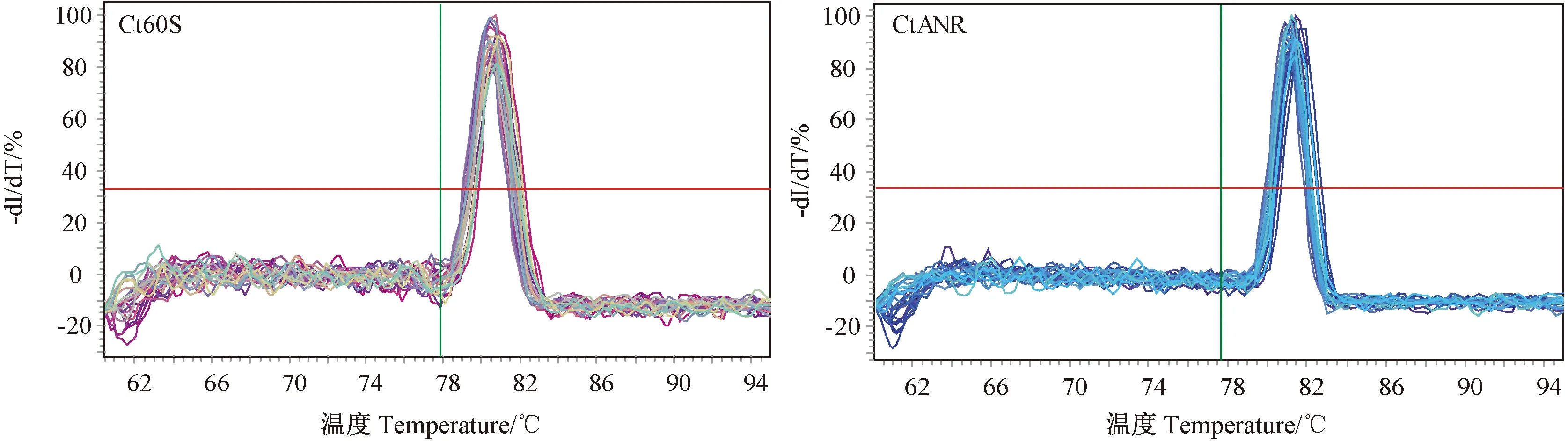
图7 Ct60S和CtANR基因的熔解曲线Fig.7 The melting curves for Ct60S and CtANR

注:*表示差异显著(P<0.05);**表示差异极显著(P<0.01);R:根;S: 茎;L:叶;B: 苞片;F:初期果球;S1~S5:开花各时期。Note: * denoted significant difference at 0.05 level. ** denoted extremely significant difference at 0.01 level. R: Root. S: Stem. L: Leaf. B: Bract. F: Initial fruit ball. S1-S5: Flowering periods.图8 CtANR基因各时期相对表达分析Fig.8 Relative expression analysis of CtANR gene in each period
3 讨论
花青素还原酶是类黄酮物质合成途径中与原花青素相关的一个关键酶,它催化花青素合成表儿茶素、儿茶素、没食子酸酯衍生物等类黄酮化合物。本研究根据课题组前期转录组测序的Unigene基因序列,设计特异性引物,从红花花冠中克隆到红花花青素还原酶基因CtANR,并对其理化性质及氨基酸序列进行了分析。通过生物信息学分析,发现红花ANR蛋白和大多数植物ANR蛋白一样都属于亲水性蛋白[23-24,28],且其二级结构和笃斯越橘ANR蛋白的二级结构类似,主要由α-螺旋组成,仅含有少量β-折叠[28],而与芒果ANR蛋白二级结构主要以无规则卷曲和β-折叠为主,α-螺旋较少[24]相反,表明红花ANR蛋白的功能更类似于笃斯越橘ANR蛋白。
氨基酸多序列同源比对发现CtANR和其他植物ANR的同源性较高,相似性达70.24%~83.98%,桑树及紫花苜宿中ANR蛋白的序列比对也得到了相似的结果,表明植物中ANR蛋白的氨基酸序列较为保守[20,33]。这一点在模体结构分析中也得到了验证,模体结构分析发现,不同植物ANR蛋白基序大多较为保守,如在系统进化树的17个物种中都含有基序3、6、8,16个物种中都含有基序2和5。此外,系统进化树及模体结构分析还发现,亲缘关系越近的种属,其ANR蛋白的模体结构越相似,CtANR蛋白的模体基序和刺菜蓟、莴苣的最相似,由此推测CtANR蛋白也具有相似的功能。
前人研究表明,ANR基因存在组织表达特异性[25],本研究组织表达分析也表明,CtANR基因在红花不同组织部位均有表达,为组成型基因,且在果球形成初期表达量最低,根和茎中表达量也较低,而在花中的表达量相对较高。此外,研究还发现MaANR在桑椹中的表达量最高,在茎中的表达量最低,且在桑椹不同发育时期,MaANR的表达量呈现先升后降的趋势[20],川桑ANR基因在叶片中的表达量最高,其次是果、皮、花,根中表达量较低[29],药用植物黑果枸杞中ANR在果实中的表达量较高,其次是幼叶,成熟叶及根和茎中表达量较低,且随着果实的成熟,ANR表达量下降,呈现先升后降的趋势[34],表明植物ANR基因的表达受发育阶段和环境因子的影响,在不同植物、不同组织及不同时期的表达量不同。
在不同花色的红花品种中,CtANR基因的表达量在花发育的S1、S3和S5时期差异极显著(P<0.01),S4、苞片、根、叶中差异显著(P<0.05),而在其他时期、其他组织中差异不显著。另外,在花发育的前期,也是花色变化较大的阶段,ANR基因的表达量出现骤降,而后随着花的进一步发育,红色红花品种ANR基因的表达量在花瓣基本伸出期(S3)表达量最高,随后逐渐降低,白色红花品种ANR基因的表达量逐渐升高,在花衰落期(S5)表达量最高,表明CtANR基因可能参与了红花花色的形成,但其表达量不是决定红花花色的主要因素,后期仍需检测花青素含量和其他颜色红花品种不同花期CtANR基因的表达量及克隆其他花色基因做进一步验证。目前国内外对于红花花色形成相关基因的研究较少,因此对于花色基因在红花花色形成中的作用仍有待进一步研究。
4 结论
本研究从红花花冠中克隆到花青素还原酶基因CtANR,全长1 020 bp,编码339个氨基酸,为不稳定的亲水性蛋白,且与刺菜蓟ANR亲缘关系最近,与梅花、欧洲甜樱桃ANR亲缘关系较远。CtANR基因在果球形成初期表达量最低,根和茎中表达量也很低,而在花中的表达量相对较高。在花发育的不同时期,红色红花品种和白色红花品种中CtANR基因的表达分别呈现降低-升高-降低的趋势和降低-升高的趋势。不同花色的红花品种中,CtANR基因的表达在花发育的S1、S3和S5时期差异极显著(P<0.01)。CtANR基因可能参与了红花花色的形成,但其表达量不是决定红花花色的主要因素,后期仍需检测花青素含量和其他颜色红花品种不同花期CtANR基因的表达量及克隆其他花色基因做进一步验证。

