茶树单体和聚合态儿茶素生物合成的研究进展
刘亚军,王培强,蒋晓岚,庄菊花,高丽萍,夏涛
茶树单体和聚合态儿茶素生物合成的研究进展
刘亚军1,2,王培强3,蒋晓岚1,庄菊花4,高丽萍1,2*,夏涛1*
1. 安徽农业大学茶树生物学与资源利用国家重点实验室,安徽 合肥 230036;2. 安徽农业大学生命科学学院,安徽 合肥 230036;3. 青岛农业大学园艺学院,山东 青岛 266109;4. 贵州大学茶学院,贵州 贵阳 550025
儿茶素类化合物,主要包括单体儿茶素和聚合态儿茶素,是茶树()多酚主要组成部分,是绿茶“茶味”决定性成分。茶树儿茶素类化合物合成与积累具有显著的组织器官特异性,鲜叶中主要积累单体儿茶素,根中以积累聚合态儿茶素为主。类黄酮代谢途径下游的无色花青素还原酶(LAR)和花青素还原酶(ANR)是决定茶树儿茶素类化合物类型的关键酶类。本文主要综述了茶树儿茶素类化合物合成、积累、转录调控研究进展,重点关注LAR、ANR以及无色花青素双加氧酶(LODX)功能和基因转录调控的最新研究进展,并对儿茶素合成途径待解决问题提出了自己的观点。
儿茶素;原花青素;无色花青素还原酶;花青素还原酶;无色花青素双加氧酶
儿茶素类化合物(Catechins)属于2-苯基苯并吡喃类化合物,具有典型黄烷-3-醇(Flavan-3-ol)的结构特征(图1)。不同的黄烷-3-醇类化合物的差异在于B环羟基化数目多少、C环-3-位是否被没食子基团取代、C环2,3位置的连接基团空间结构、是否形成聚合物等。按照C环2,3位置连接基团空间结构的差异进行分类,茶树儿茶素分为顺式(-)儿茶素[或称表型(-)儿茶素]和反式(-)儿茶素(或称非表型儿茶素)。茶树中儿茶素单体主要是顺式儿茶素,主要包括表没食子儿茶素没食子酸酯(EGCG)、表儿茶素没食子酸酯(ECG)、表没食子儿茶素(EGC)和表儿茶素(EC)4种,还有少量反式儿茶素,如儿茶素(C)和没食子儿茶素(GC)。如果考虑儿茶素旋光构型,茶树中儿茶素单体主要是左旋的顺式儿茶素(-)-EGCG、(-)-ECG、(-)-EGC、(-)-EC和右旋的反式儿茶素(+)-C和(+)-GC[1]。
茶树中也存在聚合态儿茶素。聚合态儿茶素通常被称为缩合单宁(Condensed tannins,CT)或原花青素(Proanthocyanidins,PAs或PCs)[2]。根据延伸单元之间连接方式的不同,原花青素可分A型(通过C4-C8以及C2-O7连接)和B型(通过C4-C8和/或C4-C6连接)[3]。目前,本课题组利用LC-MS检测到茶树中24种原花青素都是B型结构,其中大多数是以二聚体形式存在[4]。
茶叶界普遍认为,单体儿茶素类物质与茶饮料的收敛性关系密切,尤其是鲜叶中含量最高的EGCG。而原花青素由于含量远低于单体儿茶素且难以定量检测,所以其与品质的关系长期被忽略。最近的研究表明,原花青素很可能是夏秋茶苦涩的关键化合物[4]。研究原花青素的合成及其调控机理,对于解决茶树夏秋茶苦涩问题具有指导作用。
近几年,类黄酮下游代谢途径中几个关键酶功能有了新的发现,本文结合苜蓿()、拟南芥()等植物类黄酮代谢途径最新研究进展,综述了茶树单体儿茶素和聚合态儿茶素的合成及其调控的研究进展。
1 茶树单体和聚合态儿茶素积累的组织和种质特异性
栽培种茶树主要分为南方种(var.)和北方种(var.)。茶组植物与其他山茶属植物中单体儿茶素积累存在显著差异,特别是酯型儿茶素,在非茶组茶属植物中几乎检测不到[5]。茶组植物中,栽培种茶树中儿茶素积累区别于其他茶组植物,如秃房茶()、四球茶()、大厂茶()等四室或五室茶类。四室或五室茶类相比于栽培种,B环三羟基儿茶素(EGC和EGCG)含量极低,主要和类黄酮-3',5'-羟基化酶基因()低表达有关[6-7]。对比四球茶和舒茶早栽培种鲜叶,舒茶早幼嫩叶中EGCG含量约为90 mg·g-1(干重),而四球茶对应发育时期鲜叶EGCG仅为9 mg·g-1(干重),因为三羟基儿茶素的缺乏,四球茶鲜叶儿茶素总量只有舒茶早的1/3[7]。茶组植物毛叶茶()主要积累非表型儿茶素,如GCG、GC、C[8-9]。栽培种鲜叶GCG含量极低,较难被检测到;相比于北方种茶树,南方种茶树鲜叶EC和ECG相对偏高[10]。
茶树不同发育时期鲜叶和不同组织器官中儿茶素类型和积累差异明显(图2)。鲜叶中,主要积累单体儿茶素,6种单体儿茶素(C,GC,EC,EGC,ECG,EGCG)占鲜叶干重的12%~24%,其中酯型儿茶素约占总儿茶素的80%[10-11]。不同发育时期的鲜叶中,儿茶素总量在第一叶中最高,尤其是酯型儿茶素积累明显。随着鲜叶的发育,酯型儿茶素ECG和EGCG含量显著降低,而对应非酯型儿茶素EC和EGC显著上升,一般3~4叶中非酯型儿茶素含量最高,这主要是与酯型儿茶素合成酶ECGT酶活性降低和单宁水解酶CsTA上升有关[12-13]。在成年茶树的老叶中,酯型与非酯型儿茶素都显著下降。鲜叶中也有少量原花青素积累,一般占鲜叶干重的0.3%~2.0%,老叶中积累相对较多[4]。
在茶树不同组织器官中,单体儿茶素在鲜叶中含量最高,其次是嫩茎,根中最低。而原花青素在根中含量最高,叶中含量低。从单体儿茶素组成种类看,鲜叶中儿茶素种类最多,茎中儿茶素种类与鲜叶中相似,根中只检测到了EC。鲜叶中积累的主要是低聚的原花青素,如EC-EGC、EC-EGCG,目前LC-MS在鲜叶中检测到22种原花青素[4]。茶树鲜叶和茎中,当原花青素聚合度较小时,B环二羟基和三羟基的结构单元同时存在,C2-形式儿茶素是主要的结构单元;当原花青素聚合度大于5,主要的聚合延伸单元为B环二羟基EC[14]。
在茎中,原花青素含量随着茎的发育而升高。在木质化程度较低的嫩茎中,单体以及原花青素的种类与叶中类似,其中单体儿茶素的含量占干重的3%~10%,原花青素占干重的1%~3%。木质化程度较高的茎中积累的主要是EC及EC类型花青素,平均聚合度达到3[14]。
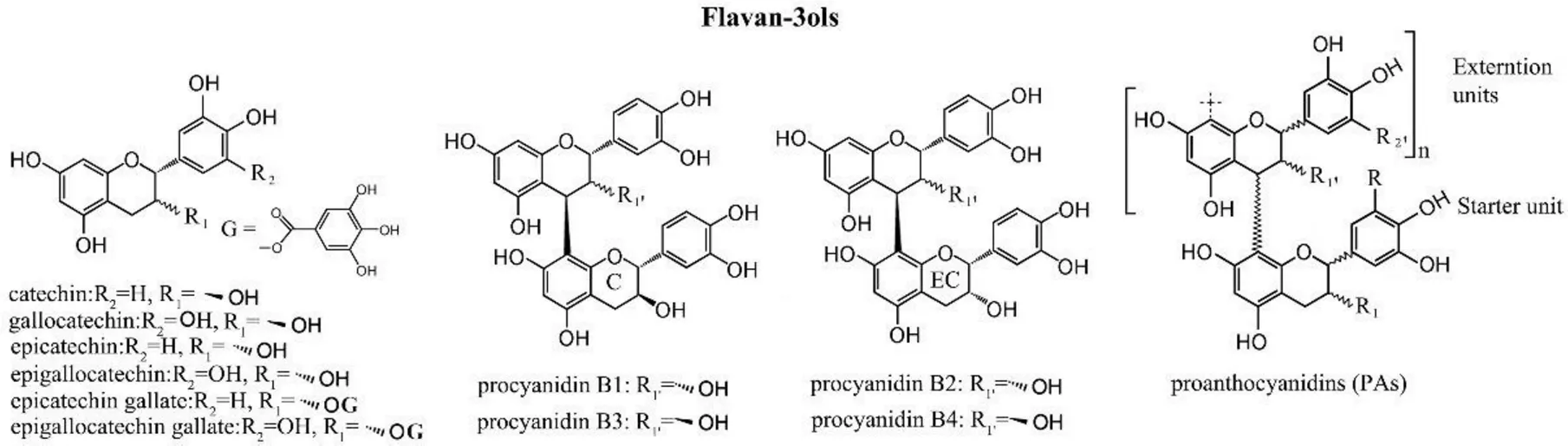
图1 茶树主要黄烷醇类化合物的结构
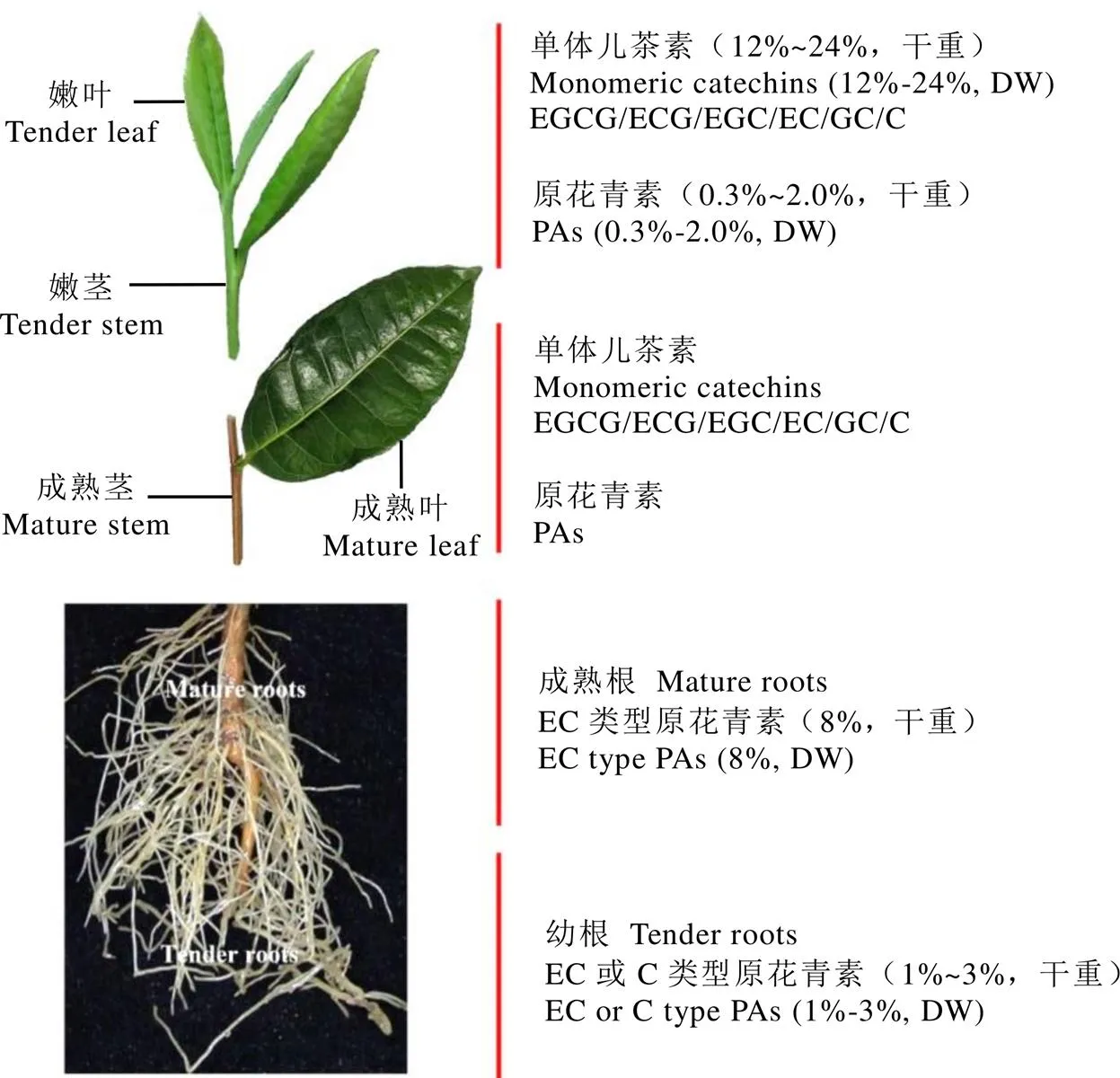
图2茶树黄烷醇类化合物器官积累特异性
相比之下,根中积累的黄烷醇类化合物种类相对单一,主要是EC以及EC类型原花青素。在木质化程度较低的根中仅积累少量的EC和C,原花青素占干重的1%~3%。木质化成度高的老根中积累的主要是EC及EC类型原花青素,含量能达到干重的8%左右。老根中原花青素的聚合度达到12,平均聚合度为5左右,但是结构单元单一,EC是主要的结构单元,且以B型C4-C8连接为主[14]。
2 儿茶素类物质组织和亚细胞定位
关于儿茶素类物质的定位研究相对较少。无损质谱成像表明,不同类型儿茶素在叶片组织内的分布不同。酯型儿茶素EGCG在全叶片积累,而非酯型儿茶素EC和EGC主要分布于叶片两边靠近叶脉的部位[15]。原位杂交实验表明,儿茶素合成相关基因、、和在叶原基和幼叶组织中表达明显,而在芽顶端分生组织中未检测到明显表达[16],说明儿茶素在鲜叶中合成,呈现明显的叶发育相关性。香草醛-盐酸染色鲜叶切片显示,儿茶素在叶片初展的鲜叶栅栏组织、维管束中均具有较明显积累[17]。亚细胞层面,幼嫩叶片叶绿体中有儿茶素类化合物积累,而在较为成熟的叶片叶绿体中积累不明显,主要积累在液泡中。儿茶素类化合物在幼嫩茎的表皮薄壁细胞中有少量积累,主要在韧皮部和木质部导管壁。在幼嫩的根中,儿茶素类化合物主要积累于中柱鞘部位。在含叶绿体的愈伤组织中,叶绿体和液泡均有儿茶素类化合物积累[17]。
3 茶树中儿茶素合成关键酶的研究进展
茶树鲜叶积累的主要是表型儿茶素EC、EGC、ECG、EGCG,而非表型儿茶素C或GC积累量极少,该科学问题在茶多酚代谢研究领域一直备受关注。儿茶素合成关键酶位于类黄酮代谢途径末端,一般认为二氢黄酮醇-4-还原酶(Dihydroflavonol-4-reductase,DFR)催化二氢黄酮醇生成无色花青素,无色花青素一方面可以被无色花青素还原酶(Leucoanthocyanidin reductase,LAR)催化形成儿茶素,也可以被花青素合成酶(Anthocyanin synthase,ANS)催化形成花青素,生成的花青素可以经花青素还原酶(Anthocyanin reductase,ANR)催化形成表儿茶素。然而,随着研究的不断深入,关于这些酶的功能有了新的发现,特别是LAR、ANR和ANS的功能。
3.1 LAR的研究进展
LAR酶功能研究报道始于二十世纪八十年代,Stafford等[18]利用花旗松()粗酶,以NADPH作为供氢体,催化无色花青素生成反式儿茶素(C)。2003年,Tanner等[19]利用原核表达获得了重组的豆科植物山蚂蝗属钩骨()DuLAR蛋白,并利用体外酶反应证明其可以催化无色矢车菊色素(3,4--leucocyanidin)生成对应的儿茶素C;2004年,Punyasiri等[20]第一次从茶树的粗酶中检测到了DFR和LAR酶的活性,当以二氢黄酮醇类(DHK、DHQ、DHM)为底物时,粗酶反应又可生成对应的产物阿福豆素(Afzelechin)、C和GC,间接证明了茶树中DFR/LAR双酶的功能。
2007年,Dixon课题组鉴定了苜蓿中、和等3个类黄酮代谢途径中的关键基因[21]。在苜蓿和烟草中过表达发现,尽管花青素含量明显降低,但转基因植物中并没有检测到C。2013年该研究团队进一步研究了1条茶树基因功能,原核表达得到的CsLAR重组酶,在体外可以催化无色花青素生成(+)-C,但是将在烟草中过表达之后却生成了大量的EC,只检测到了少量的C[22]。
2017年,本课题组对茶树中LARs功能进行了研究,通过原核表达获得了3条CsLARs重组酶,并通过“DFR+LAR双酶反应”和“饲喂”的方式验证了3条CsLARs在体外的催化功能,均可以催化二氢黄酮醇生成儿茶素C;将3条基因过表达到烟草中,转基因烟草花瓣颜色均变浅,同时生成了大量的EC[23]。CsLARs在植物体内和体外呈现出不同的催化功能。2016年,Dixon课题组在苜蓿中发现了1个MtLAR的新底物4-(-cysteinyl)-EC,MtLAR催化4-(-cysteinyl)-EC生成EC[24],表明植物体内可能存在LAR的另外1个底物。之后在葡萄原花青素合成过程的研究中,发现葡萄也积累了4-(-cysteinyl)-EC和4-(-cysteinyl)-C,VvLAR1和VvLAR2除了可以催化无色花青素以外,也可以催化4-(-cysteinyl)-EC和4-(-cysteinyl)-C生成EC和C[25]。
百脉根()是一种积累C类型儿茶素的植物,单体儿茶素主要以(+)-C为主。本课题组对百脉根的LAR功能研究发现,转基因烟草生成了大量的EC,再次说明不同植物LAR的酶功能是一定的,其催化产物取决于底物类型[26]。结合文献和本课题组的研究发现,LAR既可以参与表型儿茶素的合成,也可以参与非表型儿茶素的合成。如果LAR的底物是C类型无色花青素(C-typeleucocyanidin),生成的产物是C类型儿茶素;如果LAR的底物是EC类型无色花青素(EC-typeleucocyanidin),则生成的产物就是EC类型儿茶素(图3)。
3.2 ANR的研究进展
植物原花青素在植物物种中普遍存在,其在植物中合成机制一直是植物类黄酮代谢途径待解决的难题。寻找原花青素合成末端合成酶,一直未停止。通过关联性分析发现,多个植物中ANR基因表达与原花青素的积累高度正相关[27-29]。
ANR的研究报道最早追溯到1997年,Albert等[30]在拟南芥中筛到了1个突变体,发现突变体拟南芥的种子在成熟前期,种皮表面积累了大量的红色物质,由于这个颜色与Banyuls葡萄酒的颜色类似,因此将这个突变体命名为Banyuls,简称为BAN。
2003年,Dixon课题组的Xie等[31]在苜蓿中找到了基因的同源序列,将苜蓿和拟南芥中的过表达到烟草中,发现转基因后的烟草花色变浅,甚至变为白色,同时利用对二甲氨基肉桂醛(Dimethylaminocinnamaldehyde, DMACA)的化学显色方法检测到转基因烟草花中积累了可以使DMACA变蓝色的物质;利用原核表达获得的MtBAN重组蛋白,可以催化花青素生成表儿茶素。根据以上催化功能,Xie等[31]将基因编码蛋白命名为花青素还原酶(ANR),这是花青素还原酶被首次提出,也澄清了BAN基因被认为是同源基因的误解,他提出ANR酶功能是催化花青素形成表型儿茶素。
在ANR基因从拟南芥中被分离鉴定后,葡萄、柿子、草莓等植物中的基因功能相继被鉴定[27-28,32]。在体外ANR都可以催化花青素单体生成表儿茶素,而在ANR转基因后的植物体内都会积累可使DMACA变蓝色的原花青素类似物,但是均未检测到真正的原花青素物质含量上升,因此推测ANR在植物体内和体外的功能不同。是否ANR酶和LAR酶类似,存在多底物?产物的形成取决于底物类型?2016年,Dixon课题组研究了苜蓿的、以及双突变体,发现突变体种子的种皮与野生型种子的种皮颜色一样,而和/突变体的种子为红褐色;检测突变体的代谢物,发现中可溶性原花青素含量下降,而不可溶性原花青素含量升高,突变体中可溶性原花青素含量与不可溶性原花青素均降低[24],这暗示ANR可能在原花青素的合成过程中扮演着重要的角色。
针对这个问题,本课题组进一步研究了茶树CsANR功能。茶树CsANR原核重组酶在体外添加底物花青素Cyanidin和NAPDH,生成了(-)-C和两种对应旋光异构体儿茶素(+)-EC、(-)-EC[33]。将茶树在烟草中过量表达,发现过表达烟草中积累了大量的原花青素类似物。利用LC-MS分析转基因烟草酚类化合物,意外发现转基因烟草中一类儿茶素的中间体(中间态)含量显著上升[34]。我们根据该化合物的特性,称其为儿茶素碳正离子。儿茶素碳正离子在植物体内极不稳定,可以与EC类型无色花青素(EC-typeleucocyanidin)动态相互转变[35]。这类碳正离子具有亲电特性,可以被甲醇、乙醇、半胱氨酸(含有巯基)、儿茶素单体等亲核试剂捕捉形成结合物[34]。有研究表明,在苜蓿中分离鉴定的4-(-cysteinyl)-EC可能来源于半胱氨酸与EC的结合反应[24-25]。在本课题组研究中,以半胱氨酸为亲核试剂去捕捉这类中间体,可以形成4-(-cysteinyl)-EC[34]。另外,当以儿茶素单体作为亲核试剂去捕捉这类中间体时,形成了原花青素二聚体和三聚体。据此,我们推测这类儿茶素中间体是原花青素聚合延伸单元,也初步确定原花青素的合成关键步骤是以儿茶素单体为起始单元,以亲核试剂儿茶素中间体为延伸单位的非酶促反应。
至此,基本明确茶树CsANR功能,其催化花青素生成EC类型无色花青素(或EC类型儿茶素中间体),LAR催化该类型的中间体生成EC(图3)。
3.3 ANS的研究进展
从茶树CsLAR功能研究可以得出,CsLAR可以催化花青素生成EC类型无色花青素和C类型无色花青素,合成EC和C。从合成途径上看,EC类型无色花青素合成源头也来自于C类型无色花青素。EC和C合成途径共享底物,但茶树鲜叶积累了大量表型儿茶素EC、EGC、ECG、EGCG,非表型儿茶素C或GC积累极少。推测是茶树体内存在非表型儿茶素向表型儿茶素转变的途径,非表型儿茶素只是茶树儿茶素代谢途径中的中间产物。
花青素合成酶(Anthocyanidin synthase,ANS)是类黄酮代谢途径中负责花青素合成的重要酶,能够催化无色花青素生成花青素。在催化机理上,花青素合成酶属于双加氧酶类,其蛋白具有依赖Fe2+的2-酮戊二酸双加氧酶家族基因的结构域,因此也被称为无色花青素双加氧酶(Leucoanthocyanidin dioxygenase,LDOX)[36]。很长时间的认知误区都认为ANS和LDOX是同一个酶。在拟南芥中,可以互补(Tannin deficient seed 4)突变体缺失花青素和原花青素的性状,证明既可以参与花青素的合成,也可以参与原花青素的合成[37]。有研究表明,ANS的功能并不只局限于催化无色花青素合成花青素,也可以执行黄酮醇合成酶(Flavonol synthase,FLS)的活性,催化二氢黄酮醇生成黄酮醇,也可以催化(+)-C生成矢车菊色素(Cyanidin)和一种氧化型的二聚体原花青素[38-39]。这暗示了ANS和LDOX功能可能有所不同。
2018年,Dixon课题组对苜蓿中MtANS和MtLDOX的功能进行了区分[40]。氨基酸比对分析表明,苜蓿中报道的MtLDOX与MtANS一致性较低,只有40%。通过原核表达获得MtLDOX与MtANS重组酶,体外酶反应表明,两者都可以催化无色花青素生成花青素,同时也能够以(+)-C为底物,生成矢车菊色素。酶动力学实验表明,MtLDOX比MtANS对底物(+)-C更具有亲和力。在苜蓿体内,两者的功能差异表现明显。突变基因以后,苜蓿小苗失去了合成花青素的功能;而突变后,对苜蓿苗中花青素的合成几乎没有影响,这说明植物体内ANS的功能主要是合成花青素。
此外,通过检测苜蓿与突变体中原花青素的种类,研究人员发现突变体中的原花青素大都是以C为起始单元(Starter unit),说明突变,植物体内积累了较多的C。而在的突变体中,原花青素大都是以EC为延伸单元(Extension unit)进行聚合;双突变体中的原花青素,延伸单元和起始单元都是C类型[40]。通过以上试验结果可以推测,在苜蓿体内,ANS主要控制花青素的合成;LDOX可以催化(+)-C形成原花青素类似物,因此是否突变并不影响花青素的合成。此外,在百脉根、棉花()和银叶山蚂蝗()等植物中儿茶素单体都是以(+)-C为主,原花青素也都以(+)-C为起始单位,推测可能是这些植物体中缺少基因。栽培种茶树鲜叶中积累大量的顺式儿茶素,而非反式儿茶素,因此我们推测茶叶中可能也存在类似功能的LDOX,但是目前尚未见相关报道。
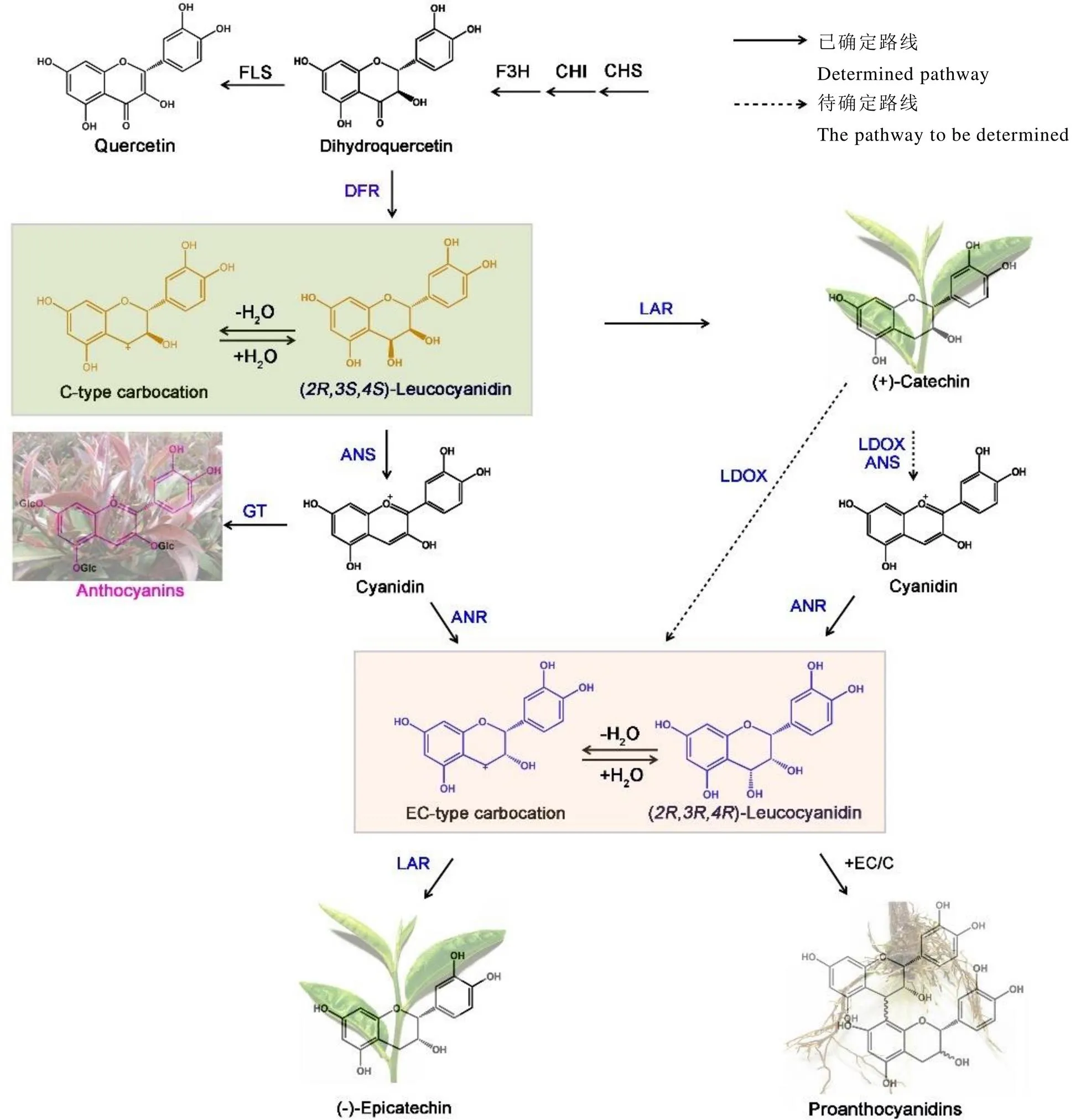
注:CHS:查耳酮合成酶;CHI:查耳酮异构酶;F3H:二氢黄酮3-羟化酶;DFR:二氢黄烷醇4-还原酶;LAR:无色花色素还原酶;ANS:花青素合成酶;LDOX:无色花青素双加氧酶;ANR:花青素还原酶;GT:糖基转移酶
结合目前其他植物中黄烷-3-醇合成途径的最新研究进展[35],我们提出了新的茶树儿茶素生物合成途径(图3)。
4 茶树儿茶素及原花青素合成调控
植物类黄酮结构基因表达受到多种转录因子调控,如NAC、ZIP、MYB、bHLH等[41],目前研究主要集中在R2R3-MYB、bHLH、WD40构成的三联复合体对类黄酮代谢的调控方面[42]。茶树酚类物质生物合成的代谢调控研究,特别是转录因子R2R3-MYB的功能鉴定,已取得了一定的进展[43-47]。
根据R2R3-MYB的C端保守基序不同,126条拟南芥R2R3-MYB可分为25个亚组[48],位于进化树中同一亚组的MYB往往具有相似的功能。为了预测茶树R2R3-MYB潜在的调控功能,将茶树和拟南芥R2R3-MYB共同构建进化树。通过进化树和MYB基序特点,对茶树R2R3-MYB进行分组归类(图4和表1)。根据预测,参与茶树酚类物质生物合成调控的R2R3-MYB可能位于进化树的第4、5、6、7、15等亚组[43,46]。
第4亚组的R2R3-MYB预测为负调控植物酚酸代谢和单宁的生物合成。例如拟南芥AtMYB4抑制基因的表达[53];金鱼草基因转烟草植株,苯丙烷代谢途径中的、和基因表达显著下调[54]。茶树的第4亚组MYB同样被证明是负调控因子,双荧光素酶试验结果表明,CsMYB4a显著抑制、、和等基因启动子的活性[55]。转烟草转录组以及代谢组分析表明,茶树CsMYB4a同时抑制转基因烟草莽草酸途径、苯丙烷合成途径、类黄酮合成途径以及木质素途径[55]。
有关促进儿茶素以及原花青素合成的MYB多位于第5亚组。研究最为深入的是拟南芥TT2,其可与bHLH转录因子(TT8、GL3、EGL3)以及WD40蛋白TTG1一起形成三联复合体MBW,激活类黄酮合成下游靶基因(如、、、、、和等)表达,调控单体及原花青素的生物合成[56]。第5亚组不同MYB对原花青素合成调控也有所不同,如MtMYB5和MtMYB14均调控原花青素的合成,但作用靶基因有明显的差异[57]。
茶树第5亚组MYB至少有5个,包括TT2分支的CsMYB5a、CsMYB5b和CsMYB5c,以及AtMYB5分支的CsMYB5d和CsMYB5e。主要在嫩梢中高表达,且随着发育程度增加而降低,与茶树嫩梢中儿茶素类物质的积累规律一致。而主要在根中高表达,可能与根中原花青素高积累相关[46]。在和过表达烟草中,这两个转录因子都调控原花青素的合成,不同的是CsMYB5a促进原花青素积累的同时降低花青素的含量;而CsMYB5e促进EC类型儿茶素中间体的合成,但不影响花青素的积累[46]。全基因组关联分析也表明(基因编号W12g025045)与茶树第三叶片的儿茶素EC积累相关性较高[58]。基因表达分析表明,CsMYB5a主要促进、和的表达,而CsMYB5e主要促进的表达,其次是[46]。与CsMYB5a类似,CsMYB5b同样通过调控和的共表达调控原花青素的积累。不同的是,CsMYB5b受糖诱导显著表达[45]。
第6亚组的R2R3-MYB转录因子预测为调控花青素的合成。在模式植物拟南芥中,R2R3-MYB家族的AtPAP1、AtPAP2、AtMYB113、AtMYB114与bHLH家族的TT8、GL3、EGL3,以及WD40蛋白(TTG1)形成三联复合体MBW,调控花青素合成关键基因和的表达[59],其中拟南芥以及其他植物直系同源基因调控植物花青素合成最为明显。茶树同源基因()与紫色芽叶品种如紫娟中花青素高积累高度相关,正调控茶鲜叶中花青素的合成[60-61]。茶树和拟南芥转基因烟草后发现,和均可以显著上调烟草中和的表达,从而促进代谢流流向类黄酮途径,特别是花青素糖苷的合成[61]。也就是说茶树第6亚组的()与功能相似,促进花青素的合成。
茶树黄烷醇类化合物合成呈现明显的组织表达特异性。叶中积累单体儿茶素,根中积累原花青素,这是茶树酚类积累最大的特点。控制组织表达特异性的转录因子除了CsMYB5家族转录因子外,有研究表明TCP转录因子控制叶发育的同时也调控儿茶素的合成[62]。TCP转录因子是植物中调控多个组织细胞增殖、细胞分化的一类转录因子[62]。茶树2条CIN-type转录因子CsTCP3和CsTCP4,1条PCF-type转录因子可以与CsTT8结合,调节三联复合体MBW,激活靶基因和的表达,从而调控茶鲜叶儿茶素合成[63]。LBD(Lateral organ boundaries domain)转录因子是植物特有的转录因子,调控多个植物组织生长和发育[64]。茶树LBD家族CsLOB_3和CsLBD36_2可以与儿茶素合成有关类黄酮途径基因、和启动子结合,暗示有些LBD家族转录因子同时调控叶片发育和叶片中儿茶素的合成[65]。
除了上述转录因子,通过基因表达和化合物关联分析,MADS、C2H2-Zinc finger、WRKY、C2H2、C3H、NAC和ERF家族成员基因表达与茶树儿茶素类化合物的积累高度关联[66-68]。此外,有文献表明茶树儿茶素代谢途径还受到MicroRNA的调控[69],如介导了氮素营养对儿茶素代谢的调控[70]。
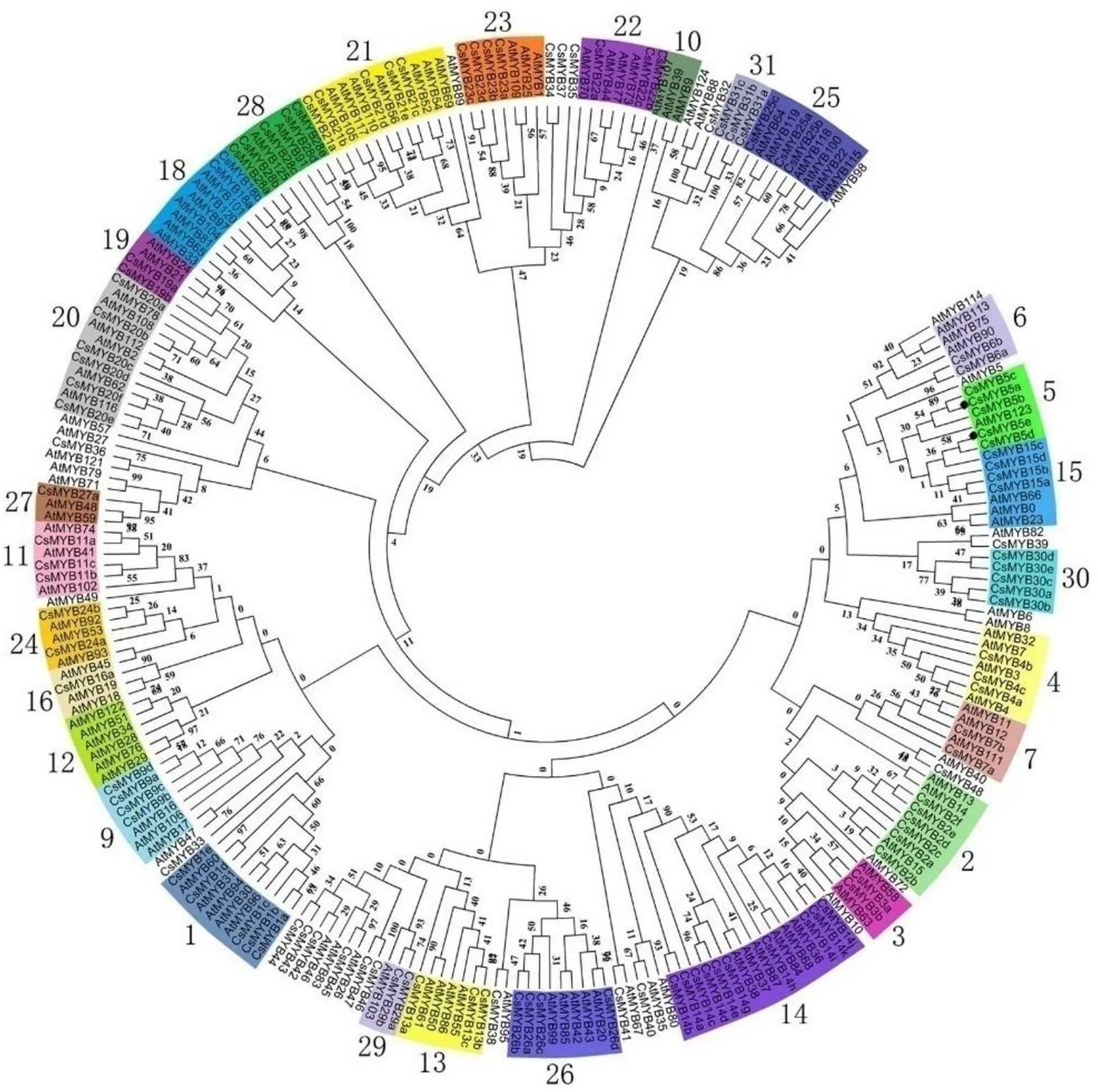
注:图中涉及拟南芥和茶树基因编号见表1
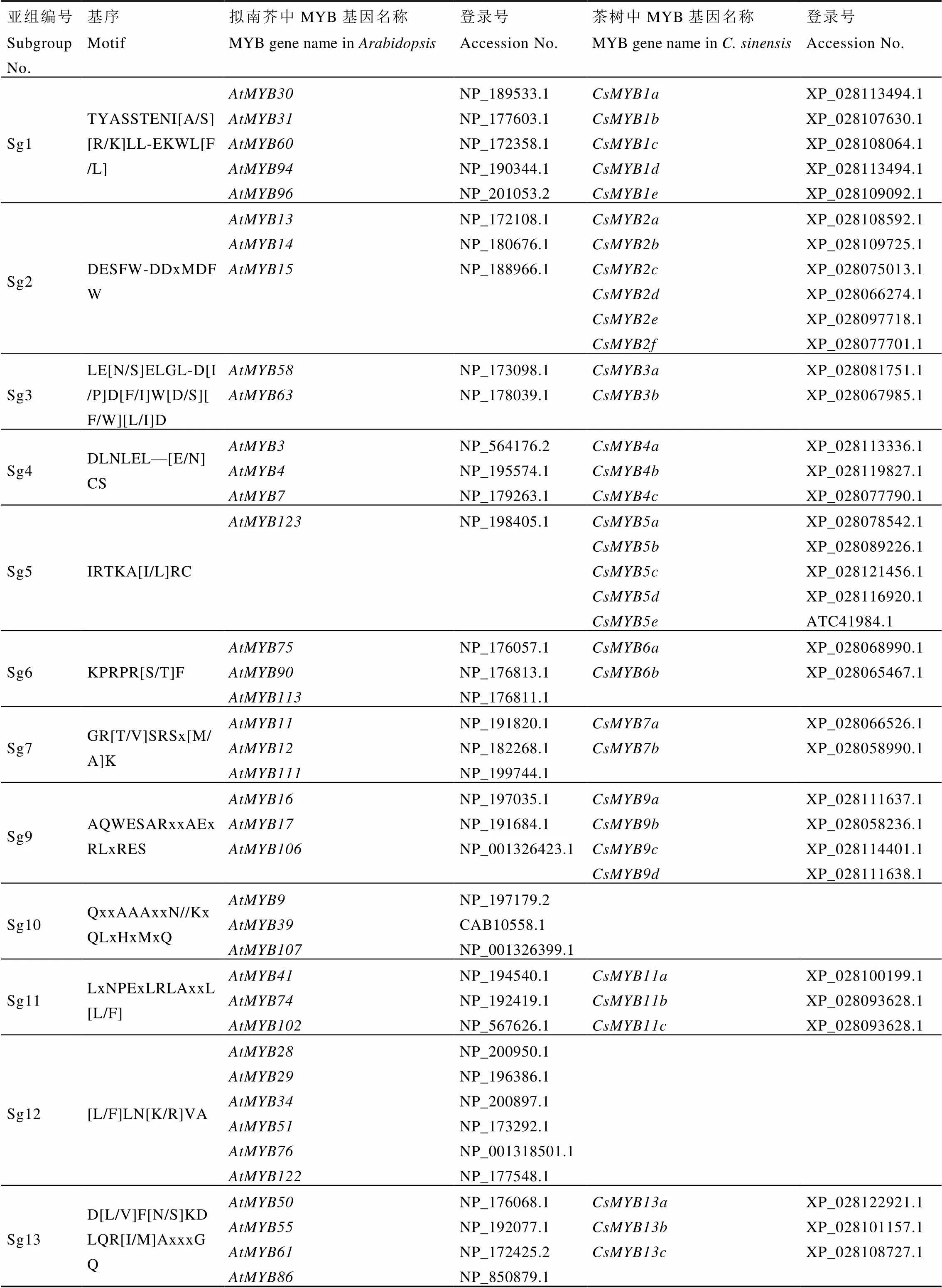
表1 基于拟南芥的同源蛋白分析茶树R2R3-MYB转录因子分组和基序
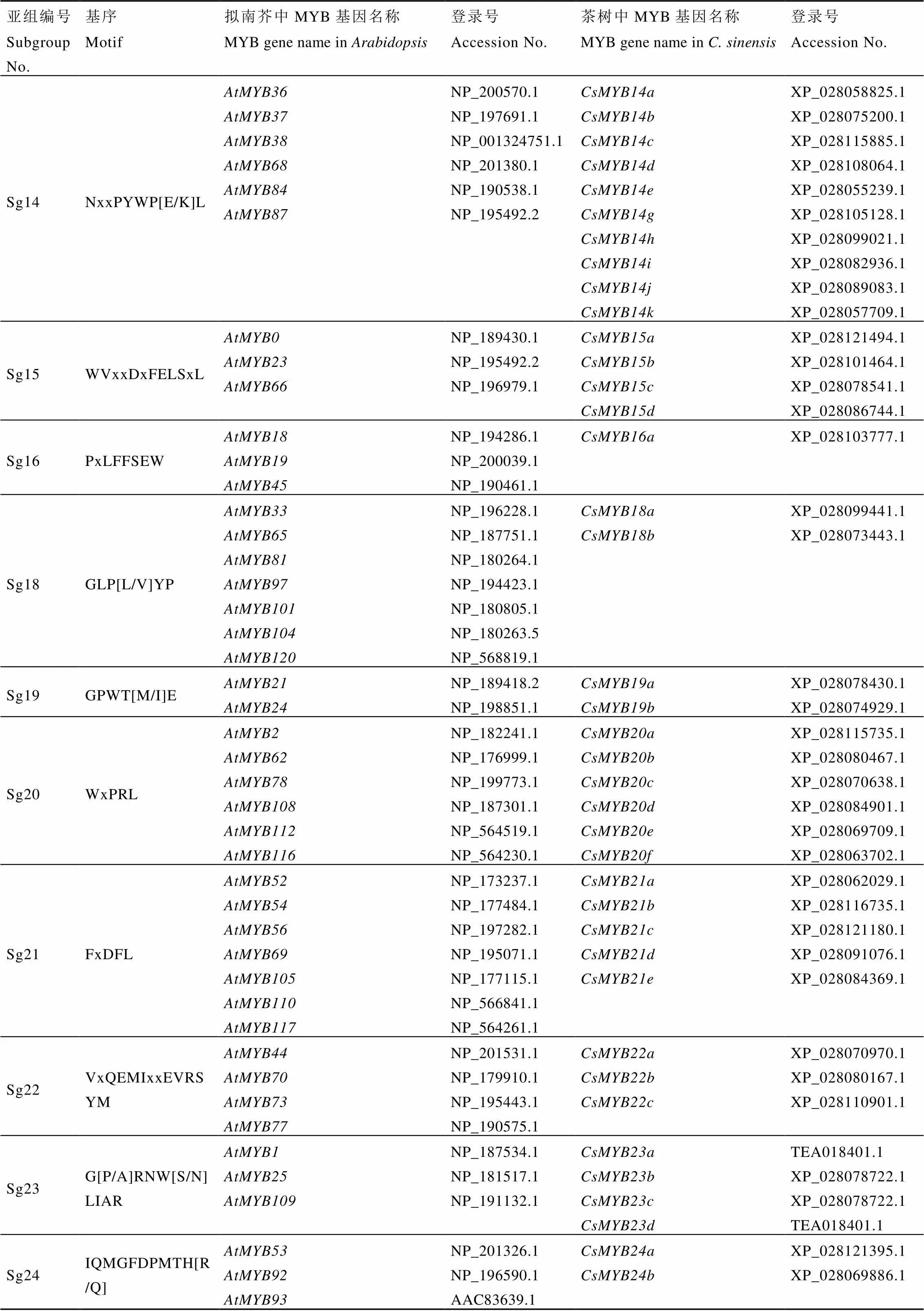
续表1
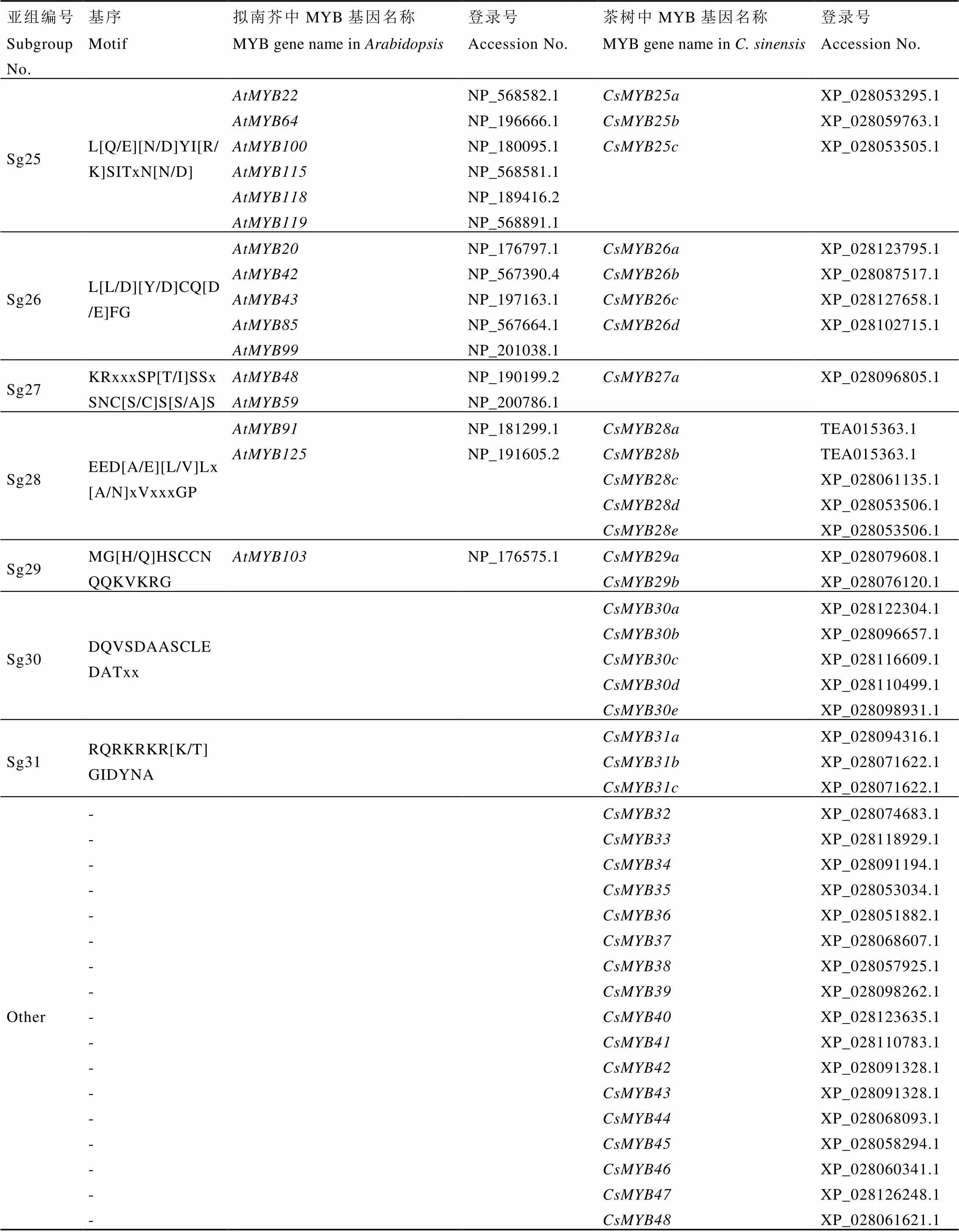
续表1
注:R2R3-MYB分组1—31根据参考文献信息命名[49-50];基序类型通过MEME工具确认[51];茶树MYB基因编码来自NCBI,其余来自茶树TPIA茶树基因组数据库平台[52]
Note: R2R3-MYB subgroup numbers from 1 to 31were named according to the references[49-50]. Motifs were confirmed using MEME tools[51]. The CsMYB accession number were provided from NCBI, the others from TPIA tea plant genome platform (http://tpdb.shengxin.ren)[52]
茶树儿茶素合成表现出显著的组织表达特异性。此外,茶鲜叶中儿茶素的合成还受到蔗糖[45]、红蓝光[71]、温度[72]等因子影响,但上述因子调控儿茶素的调控网络有待进一步研究。
5 总结与展望
茶树非酯型儿茶素和原花青素的合成途径基本清晰。茶树LAR控制茶树单体儿茶素的EC和EGC形成,进一步在酯型儿茶素合成酶的作用下合成大量的ECG和EGCG。ANR酶催化EC类型儿茶素中间体的形成,促进原花青素合成或后续通过LAR酶合成单体儿茶素。
在茶树儿茶素类化合物合成、调控、转运方面,仍然有多个科学问题待解决:(1)鲜叶儿茶素积累模式。茶鲜叶为何主要积累的是儿茶素单体而不是高聚合态原花青素?虽然鲜叶中也有少量低聚原花青素积累。(2)黄烷醇类化合物的远距离运输。茶树根中积累了大量的高聚合态原花青素,是根中合成还是转运而来?虽然幼嫩的根中原花青素合成关键基因都有表达,但他们的表达量与高积累原花青素的量不相匹配[73]。例如,目前茶树中检出两条原花青素合成延伸单位合成关键酶ANR的基因和,其根中表达量分别约是鲜叶中的1/15和1/10[73]。化合物含量和组织化学定位表明,幼根中维管束鞘部位积累了大量黄烷醇类化合物,幼嫩茎中维管束木质部和韧皮部都积累了大量黄烷醇类化合物[17]。这就暗示茶树体内可能存在叶茎根的远距离运输。(3)茶树细胞内转运机制。研究表明CsGSTF1参与了茶树黄酮醇和花青素的胞内运输积累[56,74]。体外亲和力试验表明,3条CsGSTs重组酶对黄酮醇糖苷和花青素糖苷有较高的亲和力,但是对儿茶素类化合物EC亲和力极低;3条CsGSTs转基因烟草叶片黄酮醇含量也较对照有显著升高,但是花瓣中EC含量却没有上升,暗示茶树中可能存在其他转运蛋白参与单体儿茶素的胞内运输或其他转运方式[74]。组织化学定位观察到茶树鲜叶细胞中存在多个小液泡积累儿茶素类化合物[17,75],暗示茶树儿茶素亚细胞转运可能通过小液泡转运机制。Dixon等[76]提出苜蓿中原花青素可能是在Pre-vacuolar vesicle中合成,再由Pre-vacuolar vesicle转移到细胞中心大液泡。(4)儿茶素合成的调控。虽然调控茶树黄烷醇类化合物的MYB转录因子通过模式植物异源表达基本得以确定,但是茶树缺乏有效转基因体系和突变体库。因此,调控这些MYB的上游转录因子和其他转录因子,特别是控制儿茶素组织表达特异性的关键转录因子在茶树体内功能验证也无法深入。所以茶树儿茶素合成调控研究,虽然早已在路上,但仍任重道远。
[1] 宛晓春, 夏涛. 茶树次生代谢[M]. 北京: 科学出版社, 2015.
Wan X C, Xia T. Secondary metabolism of tea plant [M]. Beijing: Science Press, 2015.
[2] Gu L, Kelm M, Hammerstone J F, et al. Fractionation of polymeric procyanidins from lowbush blueberry and quantification of procyanidins in selected foods with an optimized normal-phase HPLC-MS fluorescent detection method [J]. Journal of Agricultural and Food Chemistry, 2002, 50(17): 4852-4860.
[3] Kennedy J A, Taylor A W. Analysis of proanthocyanidins by high-performance gel permeation chromatography [J]. Journal of Chromatography A, 2003, 995(1/2): 99-107.
[4] Zhuang J H, Dai X L, Zhu M Q, et al. Evaluation of astringent taste of green tea through mass spectrometry-based targeted metabolic profiling of polyphenols [J]. Food Chemistry, 2020, 305: 125507. doi: 10.1016/j.foodchem.2019.125507.
[5] Xia E H, Zhang H B, Sheng J, et al. The tea tree genome provides insights into tea flavor and independent evolution of caffeine biosynthesis [J]. Molecular Plant, 2017, 10(6): 866-877.
[6] Liu Y J, Zhao G F, Li X, et al. Comparative analysis of phenolic compound metabolism among tea plants in the sectionof the genus[J]. Food Research International, 2020, 135: 109276. doi: 10.1016/j.foodres.2020.109276.
[7] Jin J Q, Liu Y F, Ma C L, et al. A novelallele with 14 bp deletion is associated with high catechin index trait of wild tea plants and has potential use in enhancing tea quality [J]. Journal of Agricultural and Food Chemistry, 2018, 66(40): 10470-10478.
[8] Yang X R, Ye C X, Xu J K, et al. Simultaneous analysis of purine alkaloids and catechins in,andvar.by HPLC [J]. Food Chemistry, 2007, 100(3): 1132-1136.
[9] Jin J Q, Chai Y F, Liu Y F, et al. Hongyacha, a naturally caffeine-free tea plant from Fujian, China [J]. Journal of Agricultural and Food Chemistry, 2018, 66(43): 11311-11319.
[10] Jin J Q, Ma J Q, Ma C L, et al. Determination of catechin content in representative Chinese tea germplasms [J]. Journal of Agricultural and Food Chemistry, 2014, 62(39): 9436-9441.
[11] Jiang X L, Liu Y J, Li W W, et al. Tissue-specific, development-dependent phenolic compounds accumulation profile and gene expression pattern in tea plant [] [J]. Plos One, 2013, 8(4): e62315. doi: 10.1371/journal.pone.0062315.
[12] Liu Y J, Gao L P, Liu L, et al. Purification and characterization of a novel galloyltransferase involved in catechin galloylation in the tea plant () [J]. Journal of Biological Chemistry, 2012, 287(53): 44406-44417.
[13] Dai X L, Liu Y J, Zhuang J H, et al. Discovery and characterization of tannase genes in plants: roles in hydrolysis of tannins [J]. New Phytologist, 2020, 226(4): 1104-1116.
[14] Jiang X L, Liu Y J, Wu Y H, et al. Analysis of accumulation patterns and preliminary study on the condensation mechanism of proanthocyanidins in the tea plant[J]. Scientific Reports, 2015, 5: 8742. doi: 10.1038/srep08742.
[15] Liao Y Y, Fu X M, Zhou H Y, et al. Visualized analysis of within-tissue spatial distribution of specialized metabolites in tea () using desorption electrospray ionization imaging mass spectrometry [J]. Food Chemistry, 2019, 292: 204-210.
[16] Wang X Z, Guo L N, Gao L P, et al. Molecular evidence for catechin synthesis and accumulation in tea buds () [J]. Journal of Agricultural and Food Chemistry, 2018, 66(1): 63-69.
[17] Liu Y J, Gao L P, Xia T, et al. Investigation of the site-specific accumulation of catechins in the tea plant ((L.) O. Kuntze) via vanillin-HCl staining [J]. Journal of Agricultural and Food Chemistry, 2009, 57(21): 10371-10376.
[18] Stafford H A, Lester H H. Flavan-3-ol biosynthesis: the conversion of (+)-dihydroquercetin and flavan-3,4-cis-diol (leucocyanidin) to (+)-catechin by reductases extracted from cell suspension cultures of douglas fir [J]. Plant Physiology, 1984, 76(1): 184-186.
[19] Tanner G J, Francki K T, Abrahams S, et al. Proanthocyanidin biosynthesis in plants. Purification of legume leucoanthocyanidin reductase and molecular cloning of its cDNA [J]. Journal of Biological Chemistry, 2003, 278(34): 31647-31656.
[20] Punyasiri P A N, Abeysinghe I S B, Kumar V, et al. Flavonoid biosynthesis in the tea plant: properties of enzymes of the prominent epicatechin and catechin pathways [J]. Archives of Biochemistry and Biophysics, 2004, 431(1): 22-30.
[21] Pang Y, Peel G J, Wright E, et al. Early steps in proanthocyanidin biosynthesis in the model legume[J]. Plant Physiology, 2007, 145(3): 601-615.
[22] Pang Y Z, Abeysinghe I S B, He J, et al. Functional characterization of proanthocyanidin pathway enzymes from tea and their application for metabolic engineering [J]. Plant Physiology, 2013, 161(3): 1103-1116.
[23] Wang P Q, Zhang L J, Jiang X L, et al. Evolutionary and functional characterization of leucoanthocyanidin reductases from[J]. Planta, 2018, 247: 139-154.
[24] Liu C G, Wang X Q, Shulaev V, et al. A role for leucoanthocyanidin reductase in the extension of proanthocyanidins [J]. Nature Plants, 2016, 2: 16182. doi: 10.1038/nplants.2016.182.
[25] Yu K J, Jun J H, Duan C Q, et al. VvLAR1 and VvLAR2 are bifunctional enzymes for proanthocyanidin biosynthesis in grapevine [J]. Plant Physiology, 2019, 180(3): 1362-1374.
[26] Zhang L J, Wang P Q, Ma X, et al. Exploration of the substrate diversity of leucoanthocyanidin reductases [J]. Journal of Agricultural and Food Chemistry, 2020, 68(13): 3903-3911.
[27] Fujita A, Soma N, Goto N, et al. Anthocyanidin reductase gene expression and accumulation of flavan-3-ols in grape berry [J]. American Journal of Enology & Viticulture, 2005, 56(4): 336-342.
[28] Fischer T C, Mirbeth B, Rentsch J, et al. Premature and ectopic anthocyanin formation by silencing of anthocyanidin reductase in strawberry (×) [J]. New Phytologist, 2014, 201(2): 440-451.
[29] Ghag S B, Shekhawat U K S, Ganapathi T R. Silencing ofgene reduces proanthocyanidin content in transgenic banana plants [J]. Plant Cell, Tissue and Organ Culture, 2015, 121(3): 693-702.
[30] Albert S, Delseny M, Devic M., a novel negative regulator of flavonoid biosynthesis in theseed coat [J]. The Plant Journal, 1997, 11(2): 289-299.
[31] Xie D Y, Sharma S B, Paiva N L, et al. Role of anthocyanidin reductase, encoded byin plant flavonoid biosynthesis [J]. Science, 2003, 299(5605): 396-399.
[32] Akagi T, Ikegami A, Suzuki Y, et al. Expression balances of structural genes in shikimate and flavonoid biosynthesis cause a difference in proanthocyanidin accumulation in persimmon (.) fruit [J]. Planta, 2009, 230(5): 899-915.
[33] Qian Y M, Zhao X Q, Zhao L, et al. Analysis of stereochemistry and biosynthesis of epicatechin in tea plants by chiral phase high performance liquid chromatography [J]. Journal of Chromatography B, 2015, 1006: 1-7.
[34] Wang P Q, Liu Y J, Zhang L J, et al. Functional demonstration of plant flavonoid carbocations proposed to be involved in the biosynthesis of proanthocyanidins [J]. The Plant Journal, 2020, 101(1): 18-36.
[35] Jun J H, Lu N, Docampo-Palacios M, et al. Dual activity of anthocyanidin reductase supports the dominant plant proanthocyanidin extension unit pathway [J]. Science Advances, 2021, 7(20): eabg4682. doi: 10.1126/sciadv.abg4682.
[36] Saito K, Kobayashi M, Gong Z, et al. Direct evidence for anthocyanidin synthase as a 2-oxoglutarate-dependent oxygenase: molecular cloning and functional expression of cDNA from a red forma of[J]. The Plant Journal, 1999, 17(2): 181-189.
[37] Abrahams S, Lee E, Walker A R, et al. Thegene encodes leucoanthocyanidin dioxygenase (LDOX) and is essential for proanthocyanidin synthesis and vacuole development [J]. The Plant Journal, 2003, 35(5): 624-636.
[38] Wilmouth R C, Turnbull J J, Welford R W, et al. Structure and mechanism of anthocyanidin synthase from[J]. Structure, 2002, 10(1): 93-103.
[39] Wellmann F, Griesser M, Schwab W. Anthocyanidin synthase fromcatalyzes the conversion of (+)-catechin to cyanidin and a novel procyanidin [J]. FEBS Letters, 2006, 580(6): 1642-1648.
[40] Jun J H, Xiao X R, Rao X L, et al. Proanthocyanidin subunit composition determined by functionally diverged dioxygenases [J]. Nature Plants, 2018, 4: 1034-1043.
[41] Meraj T A, Fu J Y, Raza M A, et al. Transcriptional factors regulate plant stress responses through mediating secondary metabolism [J]. Genes, 2020, 11(4): 346. doi: 10.3390/genes11040346.
[42] Deng Y X, Lu S F. Biosynthesis and regulation of phenylpropanoids in plants [J]. Critical Reviews in Plant Sciences, 2017, 36(4): 257-290.
[43] Zhao L, Gao L P, Wang H X, et al. The R2R3-MYB, bHLH, WD40, and related transcription factors in flavonoid biosynthesis [J]. Functional & Integrative Genomics, 2013, 13: 75-98.
[44] Sun B M, Zhu Z S, Cao P R, et al. Purple foliage coloration in tea (L.) arises from activation of the R2R3-MYB transcription factor CsAN1 [J]. Scientific Reports, 2016, 6: 32534. doi: 10.1038/srep32534.
[45] Wang P Q, Ma G L, Zhang L J, et al. A sucrose-induced MYB (SIMYB) transcription factor promoting proanthocyanidin accumulation in the tea plant () [J]. Journal of Agricultural and Food Chemistry, 2019, 67(5): 1418-1428.
[46] Jiang X L, Huang K Y, Zheng G S, et al. CsMYB5a and CsMYB5e fromdifferentially regulate anthocyanin and proanthocyanidin biosynthesis [J]. Plant Science, 2018, 270: 209-220.
[47] Wang W L, Wang Y X, Li H, et al. Two MYB transcription factors (CsMYB2 and CsMYB26) are involved in flavonoid biosynthesis in tea plant [(L.) O. Kuntze] [J]. BMC Plant Biology, 2019, 19: 36. doi: 10.1186/s12870-018-1582-0.
[48] Cho W, Nam J W, Kang H J, et al. Zedoarondiol isolated from the rhizoma ofis involved in the inhibition of iNOS, COX-2 and pro-inflammatory cytokines via the downregulation of NF-κB pathway in LPS-stimulated murine macrophages [J]. International Immunopharmacology, 2009, 9(9): 1049-1057.
[49] Stracke R, Werber M, Weisshaar B. The R2R3-MYB gene family in[J]. Current Opinion in Plant Biology, 2001, 4(5): 447-456.
[50] Dubos C, Stracke R, Grotewold E, et al. MYB transcription factors in[J]. Trends in Plant Science, 2010, 15(10): 573-581.
[51] Bailey T L, Boden M, Buske F A, et al. MEME SUITE: tools for motif discovery and searching [J]. Nucleic Acids Research, 2009, 37(s2): W202-W208.
[52] Xia E H, Li F D, Tong W, et al. Tea Plant Information Archive: a comprehensive genomics and bioinformatics platform for tea plant [J]. Plant Biotechnology Journal, 2019, 17(10): 1938-1953.
[53] Xie G, Schepetkin I A, Quinn M T. Immunomodulatory activity of acidic polysaccharides isolated fromL. [J]. International Immunopharmacology, 2007, 7(13): 1639-1650.
[54] Tamagnone L, Merida A, Parr A, et al. The AmMYB308 and AmMYB330 transcription factors from antirrhinum regulate phenylpropanoid and lignin biosynthesis in transgenic tobacco [J]. Plant Cell, 1998, 10(2): 135-154.
[55] Li M Z, Li Y Z, Guo L L, et al. Functional characterization of tea () MYB4a transcription factor using an integrative approach [J]. Frontiers in Plant Science, 2017, 8: 943. doi: 10.3389/fpls.2017.00943.
[56] Baudry A, Heim M A, Dubreucq B, et al. TT2, TT8, and TTG1 synergistically specify the expression ofand proanthocyanidin biosynthesis in[J]. The Plant Journal, 2004, 39(3): 366-380.
[57] Liu C G, Jun J H, Dixon R A. MYB5 and MYB14 play pivotal roles in seed coat polymer biosynthesis in[J]. Plant Physiology, 2014, 165(4): 1424-1439.
[58] Zhang W Y, Zhang Y J, Qiu H J, et al. Genome assembly of wild tea tree DASZ reveals pedigree and selection history of tea varieties [J]. Nature Communications, 2020, 11(1): 3719. doi: 10.1038/s41467-020-17498-6.
[59] Hofmann T, Glabasnia A, Schwarz B, et al. Protein binding and astringent taste of a polymeric procyanidin, 1,2,3,4,6-penta--galloyl---glucopyranose, castalagin, and grandinin [J]. Journal of Agricultural and Food Chemistry, 2006, 54(25): 9503-9509.
[60] Wei K, Wang L Y, Zhang Y Z, et al. A coupled role forandin anthocyanin hyperaccumulation in purple tea [J]. The Plant Journal, 2019, 97(5): 825-840.
[61] He X J, Zhao X C, Gao L P, et al. Isolation and characterization of key genes that promote flavonoid accumulation in purple-leaf tea (L.) [J]. Scientific Reports, 2018, 8: 130. doi: 10.1038/s41598-017-18133-z.
[62] Manassero N G, Viola I L, Welchen E, et al. TCP transcription factors: architectures of plant form [J]. Biomolecular Concepts, 2013, 4(2): 111-127.
[63] Yu S, Li P, Zhao X, et al. CsTCPs regulate shoot tip development and catechin biosynthesis in tea plant () [J]. Horticulture Research, 2021, 8: 104. doi: 10.1038/s41438-021-00538-7.
[64] Shuai B, Reynaga-Pena C G, Springer P S. The lateral organ boundaries gene defines a novel, plant-specific gene family [J]. Plant Physiology, 2002, 129(2): 747-761.
[65] Zhang X Y, He Y Q, He W D, et al. Structural and functional insights into the LBD family involved in abiotic stress and flavonoid synthases in[J]. Scientific Reports, 2019, 9: 15651. doi: 10.1038/s41598-019-52027-6.
[66] Wei C L, Yang H, Wang S B, et al. Draft genome sequence ofvarprovides insights into the evolution of the tea genome and tea quality [J]. PNAS, 2018, 115(18): E4151-E4158.
[67] Guo F, Guo Y F, Wang P, et al. Transcriptional profiling of catechins biosynthesis genes during tea plant leaf development [J]. Planta, 2017, 246(6): 1139-1152.
[68] Zhang S Y, Liu J J, Zhong G X, et al. Genome-wide identification and expression patterns of the C2H2-Zinc finger gene family related to stress responses and catechins accumulation in[L.] O. Kuntze [J]. International Journal of Molecular Sciences, 2021, 22(8): 4197. doi: 10.3390/ijms22084197.
[69] Sun P, Cheng C Z, Lin Y L, et al. Combined small RNA and degradome sequencing reveals complex microRNA regulation of catechin biosynthesis in tea () [J]. Plos One, 2017, 12(2): e0171173. doi: 10.1371/journal.pone.0171173.
[70] Fan K, Fan D M, Ding Z T, et al. Cs-miR156 is involved in the nitrogen form regulation of catechins accumulation in tea plant (L) [J]. Plant Physiology and Biochemistry, 2015, 97: 350-360.
[71] Zheng C, Ma J Q, Ma C L, et al. Regulation of growth and flavonoid formation of tea plants () by blue and green light [J]. Journal of Agricultural and Food Chemistry, 2019, 67(8): 2408-2419.
[72] Xiang P, Wilson I W, Huang J X, et al. Co-regulation of catechins biosynthesis responses to temperature changes by shoot growth and catechin related gene expression in tea plants (L) [J]. The Journal of Horticultural Science & Biotechnology, 2021, 96(2): 228-238.
[73] Wang W Z, Zhou Y H, Wu Y L, et al. Insight into catechins metabolic pathways ofbased on genome and transcriptome analysis [J]. Journal of Agricultural and Food Chemistry, 2018, 66(16): 4281-4293.
[74] Liu Y J, Jiang H, Zhao Y, et al. Threeglutathione S-transferases are involved in the storage of anthocyanins, flavonols, and proanthocyanidins [J]. Planta, 2019, 250(4): 1163-1175.
[75] Suzuki T, Yamazaki N, Sada Y, et al. Tissue distribution and intracellular localization of catechins in tea leaves [J]. Bioscience, Biotechnology, and Biochemistry, 2003, 67(12): 2683-2686.
[76] Dixon R A, Sarnala S. Proanthocyanidin biosynthesis: a matter of protection [J]. Plant Physiology, 2020, 184(2): 579-591.
Research Progress on the Biosynthesis of Monomeric and Polymeric Catechins in
LIU Yajun1,2, WANG Peiqiang3, JIANG Xiaolan1, ZHUANG Juhua4, GAO Liping1,2*, XIA Tao1*
1. State Key Laboratory of Tea Plant Biology and Utilization, Anhui Agricultural University, Hefei 230036, China; 2. School of Life Science, Anhui Agricultural University, Hefei 230036, China; 3. College of Horticulture, Qingdao Agricultural University, Qingdao 266109, China; 4. College of Tea Science, Guizhou University, Guiyang 550025, China
Catechin compounds, mainly including monomeric catechins and polymericcatechins, are the main components of tea polyphenols inand the decisive components of the "taste" of green tea. The synthesis and accumulation of tea catechin compounds have remarkable tissue- and organ- specificity. The leaves mainly biosynthesize monomeric catechins, and roots mainly accumulate polymeric catechins. The downstream enzymes of flavonoid metabolism pathway, leucoanthocyanidin reductase (LAR) and anthocyanin reductase (ANR) are the key enzymes determining the types of catechins. This paper mainly reviewed the research progress on the biosynthesis, accumulation, and transcriptional regulation of catechins in, with emphasis on the latest research progress in the function and transcriptional regulation of LAR, ANR and leucoanthocyanidin dioxygenase (LODX). Our views on the unsolved problems in catechin synthetic pathway were also proposed.
catechins, proanthocyanidinsleucoanthocyanidin reductase, anthocyanin reductase, leucoanthocyanidin dioxygenase
S571.1
A
1000-369X(2022)01-001-17
2021-08-11
2021-09-07
国家自然科学基金(31870677、32072621、31902070、31902069)
刘亚军,男,教授,主要从事植物酚类代谢研究,liuyajun1228@163.com。*通信作者:gaolp62@126.com;xiatao62@126.com
(责任编辑:黄晨)

