基因编辑技术及其在农作物中的应用进展
闫磊, 张金山, 朱健康,3*, 夏兰琴*
(1.中国农业科学院作物科学研究所,北京 100081; 2. 山东舜丰生物科技有限公司,济南 250399; 3.南方科技大学前沿生物技术研究院,广东 深圳 518055)
随着全球人口的不断增长、气候变暖、耕地和水资源不断减少,粮食安全将面临巨大挑战,即使保持现有耕地面积不变,到2030年粮食单产仍需提高1/3以上[1]。农作物的传统育种方法在很大程度上依赖于自然变异或人为通过化学和物理诱变产生的随机突变,育种周期长、效率较低、成本较高,因此仅仅依靠传统育种技术难以达到预期目标。近年来兴起的基因编辑技术能够对目标基因进行定点编辑,实现基因组DNA片段的敲除、插入和替换等修饰,该技术将颠覆农作物传统育种模式,实现精准化品种改良[2]。因此,利用基因编辑技术快速、定向创制农作物新种质,加快农作物新品种培育进程,是保障我国乃至世界粮食安全以及农业可持续发展的重要途径。
目前,应用较为广泛的基因编辑技术主要包括 3 种:锌指核酸酶(zinc finger nucleases, ZFNs)、类转录激活因子效应物核酸酶(transcription-like activator effector nucleases, TALENs)和 CRISPR/Cas (clustered regularly interspaced short palindromic repeats/CRISPR associated nuclease),这3类基因编辑技术均能对植物基因组进行精准的定点敲除、插入和替换[3-6]。其中,CRISPR/Cas系统作为第三代基因组编辑技术,能够进行基因敲除、单碱基编辑、基因定点替换和插入以及引导编辑(prime editing,PE),已成为农作物基因功能验证和遗传改良的重要工具和研究热点[2,7]。目前,CRISPR/Cas系统中研究和应用最为广泛的是Ⅱ型CRISPR/Cas9系统和Ⅴ型CRISPR/Cpf1系统,这些技术已广泛应用于农作物如水稻、小麦、玉米、大豆等基因功能研究和重要农艺性状(产量、品质、抗逆和抗病虫等)的遗传改良[8-12]。同时,科研人员也通过不断地研发新型基因编辑工具,进一步扩展CRISPR/Cas系统的基因组编辑范围,提高其定点编辑和修饰效率,并降低脱靶频率。
1 CRISPR/Cas介导的基因敲除及其在农作物中的应用
1.1 CRISPR/Cas系统的原理
CRISPR/Cas系统通过引起基因组DNA目标区域双链断裂,进而通过非同源末端连接修复(non-homologous end-joining,NHEJ)或同源重组修复 (homology directed repair, HDR)2种方式对基因组进行修复,基因敲除即是通过NHEJ修复方式使目标基因产生随机插入、缺失或碱基替换变异,导致基因移码突变或提前终止[13-16]。目前在农作物中已报道了多项利用CRISPR/Cas系统进行基因敲除的研究,这些研究创制了不同农作物多个重要基因的突变体,为农作物基因功能研究和育种应用提供了优异基因资源。
1.2 CRISPR/Cas系统在农作物产量改良中的应用
提高农作物产量是保障我国粮食安全的首要需求,产量性状是复杂的数量性状,一般由多个基因控制。目前,利用基因编辑技术已对多个控制农作物产量性状的基因进行了编辑,例如水稻穗粒数基因OsGn1a、穗型基因OsDEP1、穗粒数和分蘖数相关基因OsIPA1以及粒型和粒重相关基因OsGW2、OsGW5、OsTGW6和OsGS3等[9,17-20];OsPYLs基因家族的3个成员编辑突变后,水稻的生物量和产量都显著增加[21];通过编辑非编码基因OsmiRNA396S实现水稻高效氮肥利用,从而达到增加产量的目的[22]。小麦粒重相关基因TaGW2、TaGASR7等也被作为提高产量的靶标基因进行定点编辑[23-26]。Zhang等[27]对小麦氮素利用相关基因TaARE1进行敲除,创制了氮利用效率和产量提高的小麦新种质。
1.3 CRISPR/Cas系统在农作物品质改良中的应用
农作物的品质性状也是重要的育种改良目标之一,重要的品质性状包括淀粉和蛋白质含量、食味品质、营养成分含量等指标。通过定点编辑水稻和小麦淀粉分支酶基因OsSBEIIb和TaSBEIIa,成功创制了高抗性淀粉水稻和小麦新种质,对于降低糖尿病、高血脂等慢性疾病风险具有重要意义[7,12]。Hui等[8]对水稻控制香味性状的OsBADH2基因进行编辑,改良了水稻的香味品质。Dong等[28]对玉米淀粉合成相关基因ZmWX与ZmSH2同时敲除,创制了甜糯玉米新材料。Sanchez等[29]对小麦α醇溶蛋白基因进行敲除,能够显著降低小麦籽粒醇溶蛋白含量,提高小麦加工品质。Wang等[30]通过敲除大豆脂肪氧化酶基GmLOX,可以快速在主推大豆品种中叠加无豆腥性状,提升大豆加工和营养品质的同时不影响其他主要农艺性状。Chen等[31]对棉花种子油脂合成代谢过程中的关键基因FAD2进行编辑,创制了能够提高棉籽油品质的高油酸棉花新种质。
1.4 CRISPR/Cas系统在农作物抗逆性改良中的应用
生物或非生物胁迫会直接影响农作物的产量和品质,通过基因编辑技术可有效提高农作物的抗逆境和抗病能力。通过敲除小麦白粉病抗性基因TaMLO能够显著提高小麦白粉病抗性[32]。对小麦条锈病易感基因TaPsIPK1进行敲除,创制了具有条锈病广谱抗性的小麦材料[33]。对水稻糖转运蛋白家族基因OsSWEET的启动子区域进行编辑,破坏其感病效应蛋白结合元件,提高了水稻对白叶枯病的抗性[11]。对柑橘中的易感病基因CsLOB1启动子进行靶向修饰,提高了柑橘对溃疡病的抗性[34]。通过敲除水稻OsVQ25基因,显著提高了水稻对稻瘟菌及白叶枯菌的广谱抗性[35]。
1.5 CRISPR/Cas系统在农作物育种技术研发中的应用
农作物传统育种方法具有技术复杂、人力投入大、周期长等不足,新兴的基因编辑技术能够为农作物育种提供新的思路,在单倍体诱导、杂交制种、杂种优势固定以及多基因聚合育种的应用中取得了多项进展。通过编辑玉米单倍体诱导基因PLA1/MTL、DMP和PLD3,可以高效创制地玉米单倍体诱导系[36-39]。对小麦着丝粒特异组蛋白基因TaCENH3进行编辑,也能够创制高效的小麦单倍体诱导系[40]。同时,基因编辑技术也推动了杂交制种技术不断进步,通过操纵玉米育性基因ZmMs26,并利用荧光标记进行筛选,可以在一代获得雄性不育系和保持系,大大缩短了育种周期[41]。水稻杂种优势固定的研究也借助基因编辑技术取得了重大突破:①基因编辑技术敲除3个或减数分裂相关的基因(MiMe)以获得二倍体配子,并同时在卵细胞中异位表达BBM1使卵细胞直接发育成胚;②同时敲除PAIR1、REC8、OSD1和MTL4个内源基因,均成功创制了无融合生殖的杂种优势固定的水稻材料[42-43]。聚合多个优异基因也是农作物改良的重要手段,但是基因聚合育种的周期长且难度大。为了解决这一问题,Luo等[44]建立了小麦多基因编辑技术体系,成功实现了小麦穗发芽抗性相关基因TaQsd1、氮吸收利用基因TaARE1、株型相关基因TaNPT1、支链淀粉合成基因TaSBEIIa和磷转运相关基因TaSPDT共5个基因的同时编辑,在T1代即可获得无转基因、多个优异等位基因聚合的小麦新种质,为加速农作物多基因聚合育种提供了有力的技术支持。
1.6 CRISPR/Cas系统在农作物快速驯化中的应用
农作物在驯化和育种过程中,重要农艺性状改良的同时也会造成基因遗传多样性的大量丢失,基因编辑技术的出现使农作物野生种和祖先种实现快速从头驯化成为了可能。Li等[45]选用天然抗逆性的野生番茄品种醋栗番茄作为底盘材料,对调控开花光周期敏感性、株型和果实成熟的基因SP和SP5G的编码区、控制果实大小的基因CLV3和WUS以及维生素C合成酶基因GGP1的调控区进行了定点编辑,消除了野生番茄开花的光周期敏感性,驯化出株型紧凑、果实大小和维生素C含量显著提高的新型番茄,同时还保持了野生番茄的耐盐碱和抗病性状。Yu等[46]在对异源四倍体野生水稻进行高质量基因图谱和高效遗传转化体系构建的基础上,对四倍体野生稻的相关驯化基因包括落粒基因qSH-1、芒长基因An-1,绿色革命基因SD1、粒长基因GS3以及理想株型基因IPA1进行基因编辑,获得了表型为不落粒、芒长变短、株高降低、茎秆粗壮、开花期缩短等系列突变体。通过基因编辑实现野生近缘植物的快速驯化,为精准设计和快速创制农作物新种质提供了新的策略。
2 碱基编辑系统及其在农作物中的应用
2.1 碱基编辑系统的原理及类型
单核苷酸变异是农作物许多重要农艺性状改良的遗传基础,因此开发一种精准和高效的碱基替换技术对农作物精准育种具有重要意义。2016年,David R. Liu实验室开创性地研发了基于胞嘧啶脱氨酶的碱基编辑系统(base editing),首次将CRISPR/Cas系统从切割DNA的“分子剪刀”变为可以改写特定碱基的“修正器”,扩展了基因组精准编辑技术体系[47-48]。目前,已被广泛应用的单碱基编辑系统主要有3种:能够实现C:G到T:A转换的胞嘧啶单碱基编辑系统(cytosine base editor, CBE)、A:T到G:C转换的腺嘌呤碱基编辑器(adenine base editor,ABE)以及 C:G 到 G:C 转换的碱基编辑器(C-to-G base editor,CGBE)。碱基编辑系统(图1)使单个核苷酸的精准替换成为可能,能够使靶基因产生功能缺失或功能获得型突变,在作物基因功能研究、遗传改良和目标基因定向进化中具有重大的应用潜力[49-52]。
2.2 CBE碱基编辑系统及其在农作物改良中的应用
CBE系统主要由具有切口酶活性的nCas9、不具有核酸酶活性的dCas9或者dCas12a蛋白,以及胞嘧啶脱氨酶和尿嘧啶糖基化酶抑制剂(uracil glycosylase inhibitor,UGI)融合而成[53](图1B)。为了提高单碱基编辑效率、缩小编辑窗口、扩展编辑范围、降低脱靶频率等,先后建立了编辑效率提高的CBE4、BE4max、AncBE4max、FNLS-BE3、evorAP OBEC1、evoFERNY和evoCDA1系统[54],编辑窗口缩小的YE1-BE3、YE2-BE3和YEE-BE3系统[55],单碱基替换活性更高和indel发生频率更低的AID碱基编辑器[56],Indel发生频率降低的BE3-Gam、SaBE3-Gam、BE4-Gam、SaBE4-Gam 和 eBE-S3系统[57],以及编辑范围扩展的LbCas12a-BE0、xBE3、CBE-NG、CBE-SpG和CBE-SpRY碱基编辑器[58-61]。
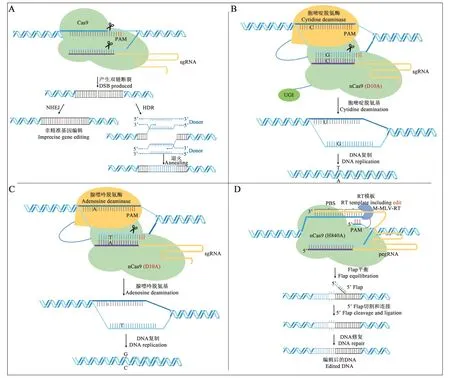
图1 CRISPR/Cas介导的基因编辑技术[12]Fig. 1 CRISPR/CAS-mediated gene editing technology[12]
近年来,CBE碱基编辑系统已被广泛应用于多种农作物性状改良研究[49]。植物CBE系统最初是由本实验室和多个研究团队同时建立[54,62-63],利用CBE3碱基编辑器成功在水稻八氢番茄红素脱氢酶基因OsPDS和淀粉分支酶Ⅱb基因OsSBEIIb的3个靶点实现了单碱基编辑,编辑效率高达20%左右。与此同时,利用CBE3碱基编辑器获得了稳定遗传的水稻硝转运蛋白基因OsNRT1.1B和水稻侧根发育基因OsSLR1的单碱基编辑植株,编辑效率分别为2.7%和13.3%[63]。利用碱基偏好性消除的单碱基编辑系统rBE5对水稻花发育基因OsPi-d2进行了精准编辑,获得了抗稻瘟病的水稻新种质[56]。此外,通过对番茄、马铃薯、西瓜和拟南芥等多种植物的乙酰乳酸合成酶基因ALS进行单碱基替换,获得了一系列抗除草剂的编辑植株[64-67]。在异源四倍体棉花中也建立了高效的单碱基编辑系统,实现了1-脱氧木酮糖-5-磷酸合成酶基因GhCLA和磷脂酰乙醇胺结合蛋白基因GhPEBP的定点突变[68]。在六倍体小麦中,利用CBE系统成功编辑了小麦抗除草剂基因TaALS和TaACC,创制了一系列抗除草剂小麦新种质,为麦田杂草防治提供了新材料[54,69]。
2.3 ABE碱基编辑系统及其在农作物改良中的应用
ABE系统主要由具有切口酶活性的nCas9、不具有核酸酶活性的dCas9或者dCas12a蛋白与腺嘌呤脱氨酶融合而成[50,56](图1C)。科研人员对ABE系统的优化主要集中在提高碱基编辑效率、扩大 PAM(protospacer adjacent motif)选择范围和降低可预测脱靶效应方面。通过优化腺嘌呤脱氨酶二聚体的位置、核定位信号肽(nuclear location signal,NLS)的位置和个数以及使用不同单链向导RNA(single-stranded guide RNA,sgRNA)的形式,将ABE系统的碱基编辑效率在小麦和水稻中提高了1.1倍[51]。通过优化密码子序列和增加NLS的个数构建了ABEmax,提高了碱基替换的效率[70]。为了提高ABE编辑系统的效率,进一步通过突变腺嘌呤脱氨酶TadA的8个氨基酸位点构建了TadA8e,大大提高了ABE碱基编辑的效率[71]。最近,Tan等[72]开发了一种更有效的腺嘌呤碱基编辑器 PhieABEs(plant high-efficiency ABEs),该 系 统利用高活性腺嘌呤脱氨酶TadA8e、单链DNA结合结构域(DNA binding domain,DBD)与广靶向性的SpCas9变体,相较于一般的ABE8e系统,它具有更高的碱基编辑活性和更宽的碱基编辑宽度。Yan等[73]通过对TadA8e引入2个突变V82S和Q154R,构建了更高效的腺嘌呤脱氨酶TadA9。与TadA8e相比,TadA9扩展了编辑窗口范围,同时对一些以往难以编辑的内源靶点,表现出更强的编辑能力。
目前,ABE系统已被广泛应用于多种农作物性状改良的研究和应用中。利用SaCas9的nCas变体与突变的TadA酶融合,构建了ABEP2,可在水稻基因组某些位点实现高达61.3%的碱基编辑效率[47,74]。Li等[48]利用nCas9融合大肠杆菌野生型腺嘌呤脱氨酶(ecTadA)和人工定向进化的腺嘌呤脱氨酶(ecTadA*)二聚体优化了ABE系统,在水稻中实现了高达59.1%的A→G编辑效率。Kang等[75]利用ABE系统在拟南芥及油菜中实现了对八氢番茄红素脱氢酶基因PDSmRNA的可变剪接。Wei等[76]利用ABE8e系统对水稻淀粉合成基因OsWaxy和除草剂抗性基因OsALS进行了定点编辑,编辑效率最高可达100%。
2.4 CGBE碱基编辑器
尽管CBE和ABE系统在已在不同植物中广泛应用,但只能诱导碱基发生转换而不能发生碱基颠换。CGBE是一种新型碱基编辑器,该系统由rAPOBEC1胞苷脱氨酶变体(R33A)、nCas9(D10A)和 尿 嘧 啶 DNA 糖 基 化 酶 (uracil DNA glycocylase,UNG)组成,能够在哺乳动物细胞和植物中进行C→G编辑。Tian等[77]对CGBE系统进一步优化,获得了可以在水稻中实现高效C>G编辑的OsCGBE03碱基编辑器,对5个水稻内源基因(OsIPA1、OsbZIP5、OsSLR1、OsALS1和OsNRT1.1 B)的C→G平均编辑效率可达21.3%。CGBE系统为植物育种中产生更多的碱基替换类型以及种质资源创新提供了有力工具。
2.5 碱基编辑系统在农作物定向进化中的应用
传统育种中的基因诱变方法往往存在靶向性不足、突变随机性较大等不足,利用碱基编辑系统可以对重要基因关键位点进行饱和氨基酸突变,构建目的基因片段突变文库,实现重要农艺性状基因的定向进化。Li等[78]构建了新型的饱和靶向内源基因突变碱基编辑器(saturated targeted endogenous mutagenesis editors,STEME),该系统在水稻原生质体中C→T诱导效率高达61.61%,C→T和A→G同时突变的效率高达15.50%。利用该系统对水稻乙酰辅酶A羧化酶基因OsACC进行饱和突变,并对再生植株进行除草剂筛选,鉴定到3个新的除草剂抗性突变位点:P1927F、S1866F和A1884P。其中,P1927F突变表现出强除草剂抗性,具有较高的生产应用价值。Kuang等[79]利用胞嘧啶单碱基编辑器rBE9以及腺嘌呤单碱基编辑器rBE14对水稻乙酰乳酸合酶基因OsALS1进行了饱和突变,通过抗除草剂鉴定,筛选到4个新的抗除草剂等位基因。Wang等[80]利用优化的CBE和ABE以及兼具腺嘌呤和胞嘧啶碱基编辑功能的双元编辑器ACE,对水稻乙酰辅酶A羧化酶基因OsACCase进行饱和突变及定向进化,最终获得了多个新的具有除草剂抗性的突变类型。
3 CRISPR/Cas介导的HDR及在农作物中的应用
3.1 CRISPR/Cas介导的HDR
CRISPR/Cas介导的NHEJ突变效率较高,并且已广泛应用于农作物遗传改良应用中,但是农作物改良中往往需要将优异等位基因精准引入植物中。这就需要利用HDR介导的基因组精准编辑技术,来实现优异等位基因的定点替换和插入(图1A)。目前,在许多农作物中已实现HDR介导的基因组精准编辑,但是鉴于HDR的低效率和植物细胞中供体模板递送的局限性,进行HDR介导的农作物精准基因编辑仍然存在较大的困难和挑战[81]。
3.2 在农作物改良中的应用
本实验室分别利用CRISPR/Cas9和CRISPR/LbCpf1系统成功实现了水稻抗除草剂基因OsALS的等位替换,通过同时改变其第548和第627位的2个氨基酸,赋予了水稻对磺酰脲类除草剂的抗性[8,82]。进一步利用RNA转录本作为修复模板,通过HDR方式实现了OsALS的等位基因替换,首次在植物细胞中建立了RNA转录本作为修复模板介导的同源重组修复体系[83]。同时,还将粳稻中的氮吸收相关基因NRT1.1B替换为籼稻品种的有利等位基因,以提高其氮素利用效率[84]。Li等[85]利用CRISPR/Cas9系统成功编辑大豆第4号染色体上的2个基因位点DD20和DD43,并在T1代检测到了DD43的HDR事件。该团队还使用CRISPR/Cas9将单链寡核苷酸(single-stranded oligonucleotides,ssODNs)或双链DNA作为修复模板,将ALS第165位的脯氨酸替换为丝氨酸,创制了抗除草剂玉米[86]。在木薯和亚麻中已实现5-烯醇丙酮莽草酸-3-磷酸合成酶(5-enolpyruvylshikimate-3-phosphatesynthase,EPSPS)中2个氨基酸的替换(T102I/P106S),创制了抗草甘膦除草剂的新材料[87-88]。Wang等[89]将CRISPR/Cas9 系统和小麦矮化病毒(wheat dwarf virus,WDV)双生载体系统整合,实现了水稻中外源基因的定点插入,其效率最高可达到19%。Shi等[90]利用CRISPR/Cas9系统在玉米中实现了乙烯负调控因子ARGOS8基因启动子的替换,以增加ARGOS8基因的表达,提高了干旱胁迫下的玉米产量。此外,在拟南芥和番茄等作物中,通过CRISPR/Cas9引起双链断裂实现了同源染色体靶位点间的同源重组修复,为作物精准育种提供了有效策略[91-92]。
3.3 CRISPR/Cas介导HDR的优化
为进一步提高CRISPR/Cas介导的HDR效率,多项研究采用不同策略进行了尝试。Gil-Humane等[93]采用携带CRISPR/Cas9和DRT的小麦矮化病毒(WDV)复制子的策略,将编辑内源性泛素基因的HDR效率较非病毒传递方法提高了12倍。Vu等[94]利用CRISPR/Cpf1系统在番茄中发现多复制子系统可以有效地将HDR效率提高约3倍,尤其是在温度较高的环境下对HDR效率的提升更为显著。Huang等[95]通过突变LbCas12a的氨基酸(D156R)获得了编辑效率更高的ttLbCas12a,在烟草中实现了更高效的基因敲入。Aird等[96]采用将ssODNs与Cas/sgRNA的核糖核蛋白复合体(ribonucleo protein,RNP)共价结合的方法使HDR效率提高了30倍。Miki等[97]使用卵细胞和早期胚胎特异性DD45基因启动子驱动SpCas9表达并结合二代转化策略,在拟南芥中提高了HDR介导的基因敲入和基因替换的效率。该团队将供体DNA片段进行硫代修饰和磷酸化修饰,极大地提高了CRISPR/Cas9介导的靶向敲入效率,最高可达47.3%。进一步开发了重复片段介导的同源重组(tandem repeats-HDR,TR-HDR)方法,通过将修饰的片段靶向敲入至目标位点制造的串联重复结构,能够诱导TR-HDR实现片段替换,该策略的基因替换效率最高可达11.4%,这项技术的突破将大大促进农作物定向遗传改良的进程[98]。此外,过表达DNA修复蛋白RAD51能够显著提高动物胚胎细胞中CRISPR/cas9介导的基因敲入效率,该策略为在植物中进一步提高HDR效率提供了重要参考[99]。
4 引导编辑技术
2019年,一种新型的基因组精确编辑技术——引导编辑(prime editing,PE)系统问世。该系统利用nSpCas9(H840A)和经过工程化改造的M-MLV RT逆转录酶融合构建了引导编辑器(prime editor),由引导编辑向导RNA(prime editing guide RNA,pegRNA)中向导RNA部分引导在基因组靶点位置附近形成编辑链上的单链切口,进而通过pegRNA中的引导结合位点(primer binding site,PBS)序列引导逆转录酶工作,以利用含有目标编辑序列的逆转录模板(RT template)将突变精确导入基因组中。该系统在不产生DSB或引入供体DNA的情况下,在哺乳动物细胞中实现小片段的插入、缺失和12种类型碱基-碱基的任意转换,相对于CRISPR介导、依赖于HDR的精确编辑,引导编辑的效率更高,副产物更少;而相对于碱基编辑,引导编辑也表现出明显的优势[100](图1D)。
4.1 引导编辑技术在农作物中的应用
在此基础上,多家实验室构建了适于植物表达的引导编辑工具PPE(plant prime editing)系统,并成功地在水稻、小麦、玉米等作物中实现了基因组精准编辑。Lin等[101]通过PPE系统在水稻和小麦原生质体中实现了多个内源基因的精准编辑,获得了单碱基突变、多碱基突变及精准删除的水稻植株。Li等[102]将水稻密码子优化的逆转录酶突变体融合在具有双核定位信号的nCas9蛋白的C末端,使用玉米增强型启动子Ubiquitin驱动该融合基因表达,并添加了具有增强mRNA稳定性的多聚腺苷酸(polyA)结构和豌豆Rubisco小亚基E9终止子终止转录和翻译;同时使用水稻组成型Actin启动子驱动pegRNA和sgRNA的共表达,采用体内可自我剪切的tRNA间隔,构建了高效的植物引导编辑系统。利用该系统分别对水稻外源潮霉素抗性基因hptII和内源草甘膦靶标蛋白基因OsEPSPS进行精准编辑,成功获得了突变基因精准修饰的纯合植株和杂合植株,精确编辑效率分别为9.38%和2.22%[102]。Tang等[103]在水稻中对PE2、PE3、PE3b系统进行测试,引导编辑效率为0.05%~0.15%,该研究通过增加核定位信号的数量等方法使引导编辑系统的效率提升至1.55%。Xu等[104]利用自切割多肽串联表达PE系统使筛选标记和逆转录酶共表达,并由玉米Ubi启动子驱动,同时使用增强型的 SgRNA(esgRNA scaffold)作为pegRNA的骨架结构,使得编辑效率平均提高8倍以上,最高可达26%。此外,Xu等[105]利用引导编辑系统在水稻中实现了1个外源靶点和3个内源靶点的精准编辑。Hua等[106]首次利用SaCas9介导的引导编辑系统在水稻细胞中实现了报告系统的精确修改。Jiang等[107]首次在玉米中利用引导编辑工具获得了乙酰乳酸合酶基因ZmALS1和ZmALS2的W542L和S621I双位点的纯合突变体,编辑效率最高可达72%~75%。
4.2 引导编辑技术的影响因素及优化
引导编辑系统中pegRNA的PBS、RT模板、nicking sgRNA的相对位置对引导编辑的效率有很大的影响。因此,多项研究针对pegRNA开发了设计工具,优化了pegRNA的设计流程,如pegFinder和PrimeDesign等软件,以方便研究人员更便捷地进行pegRNA 的设计[108-109]。Lin等[110]发现PBS的Tm值对引导系统的编辑效率有重要影响,当PBS的Tm值为30 ℃左右时,引导编辑效率在多数水稻内源位点中达到最高,并且随着温度的增高或降低均呈现递减的趋势。同时还发现对1个位点的正链和负链各设计1个pegRNA,利用DNA 2条链之间存在反向互补序列,可能同时“启动”细胞中微同源末端修复途径,共同提高引导编辑系统的编辑效率。Xu等[111]采用在Cas9的N端融合逆转录酶M-MLV同时在RT模板中引入同义错配碱基的策略,对水稻8个内源基因进行了精准编辑,平均编辑效率可以提高10倍以上。Zou等[112]通过在pegRNAs的3’末端添加结构性的RNA motifs来增强引导编辑系统的稳定性,建立了PPE3-evopreQ1引导编辑系统,对水稻内源基因OsCDC48、OsALS、OsDEP1、OsEPSPS等进行单个及多个碱基的精准替换,该系统的平均编辑效率达10.0%以上,最高可达47.5%。Li等[113]对PE3引导编辑系统进一步优化,分别建立了基于潮霉素抗性基因的代理引导基因编辑器PE3-HS和基于双草醚抗性基因的代理引导基因编辑器PE3-AS,以及基于这2个基因的双代理引导基因编辑器PE3-DS。对水稻内源基因OsSPL14、OsDHDPS和OsNR2进行编辑,PE3-HS 和PE3-AS编辑器的精准编辑效率提高了约2~14倍,双代理PE3-DS编辑器的效率最高可提高约50倍,同时率先实现了水稻多个内源基因同时精准编辑[113]。
5 我国农作物基因编辑研究展望
目前,CRISPR/Cas系统已成为定向改良农作物的重要技术工具,我国科研人员也建立了基因定点敲除、单碱基编辑、等位基因替换和引导编辑等多项基因编辑技术体系,并成功应用于水稻、小麦、玉米等主要农作物的遗传改良,研究水平处于国际领跑或并跑的地位,具有较强的竞争优势。其中,CRISPR/Cas介导的基因敲除和单碱基编辑技术应用最为广泛,但基因精准替换等基因精准编辑的效率仍然较低,大大限制了其在农作物改良中的应用。因此,建立高效的精准编辑和基因替换技术体系成为该领域的重要目标和研发方向。
理论上CRISPR/Cas系统介导的HDR可实现任意碱基之间的转变,但该途径多发生于细胞周期的S期和G2期,而转化所需要的外植体如胚性愈伤是体细胞,且大多数处在G1期。这些弊端导致HDR在植物细胞中的发生频率及应用范围均受到了一定的限制。针对HDR介导的基因替换效率较低的问题,可以通过增加修复模板数量、促进修复模板靠近DNA双链断裂部位、采用特异表达启动子驱动Cas蛋白表达等策略提高效率[2]。例如采用RNA适配体结合链霉亲和素的策略能够有效增加修复模板数量,从而提高HDR介导的精准替换效率[114];通过将Cas蛋白和修复模板结合并递送至细胞核内也能够有效富集修复模板的数量,比如采用ssODNs与Cas/sgRNA的核糖核蛋白复合体共价结合的策略可以有效提高HDR介导的精准编辑效率[96];利用卵细胞和早期胚胎特异启动子驱动Cas基因表达也能够有效提高HDR效率[97,115]。此外,如何将外源供体模板高效递送至细胞中也是一大难题,目前在植物中使用最广泛的递送方法主要有基因枪法和农杆菌转化法,这2种方法都存在一定的不足,迫切需要开发新的递送方法,提高递送效率。近年来,利用病毒载体系统,例如烟草脆裂病毒(tobacco rattle virus,TRV)、大麦条纹花叶病毒(barley stripe mosaic virus,BSMV)等将CRISPR/Cas系统递送到植物中,该策略在玉米、小麦、烟草等作物中已成功实现了可遗传的靶向基因编辑[116-119],且该转化过程不需要经历组织培养阶段,但其基因编辑效率以及适用的植物物种范围仍需进一步提高。此外,随着纳米技术的发展,已有多种纳米材料能够高效递送遗传物质或蛋白质,该方法具有高效率、无毒性和无转基因整合的优势。目前,纳米材料介导的CRISPR/Cas基因编辑系统已在动物细胞中成功应用,但在植物中利用该方法进行基因编辑尚存在障碍[120-121],因此,加大对该基因编辑系统在植物中的研究力度,开发可以高效携带瞬时表达载体、Cas/RNP复合体等的纳米颗粒递送体系,将为高效、便捷地创制基因编辑农作物提供新的途径。
我国科研人员利用基因编辑技术在农作物遗传改良中的研究虽然取得了一系列重要进展,但是前沿技术的短板依然突出,特别是缺乏核心技术专利。从ZFNs、TALEN到CRISPR/Cas9等主流基因编辑工具的底盘技术专利都掌握在美国等发达国家手中,这将使我国基因编辑育种产业面临“卡脖子”的风险,同时也会影响基因编辑农作物的产业化进程。因此,当前的首要任务是积极开发原始创新的基因编辑工具。在此基础上,研发一系列具有自主知识产权的基因编辑技术体系。国家需要进一步加强基因编辑领域基础研发和技术创新的投入,重视原创性工具和技术研发以及对原始创新技术的知识产权保护。此外,相关技术的研发与育种应用结合不够紧密,科技创新与市场需求脱节的现象也是目前存在的主要问题,亟需建立种业上下游衔接紧密,以市场为导向的高效育种创新体系,以保障我国的粮食安全。

