人工智能加速作物表观遗传设计育种
郭位军, 李东维, 谢上, 杨立文, 李聪, 田健, 普莉, 谷晓峰
(中国农业科学院生物技术研究所,北京 100081)
随着全球人口迅速增长、有效耕地面积持续减少和环境条件恶化,全球粮食生产面临巨大压力。目前,我国粮食作物平均单产增速变缓,亟需应用新育种策略和前沿技术解决作物增产、稳产的瓶颈问题。大数据和人工智能等信息技术在生物育种领域逐渐渗透和融合,驱动了作物智能设计技术的快速发展及其在作物关键育种性状定向改良上的应用。作物重要育种性状受到遗传、表观遗传和环境因子3个层次协同精准调控,其中表观遗传能够增强遗传因子和环境之间的联系,在调控植物生长发育、环境适应性等方面发挥关键作用。目前,以表观遗传为核心的表观智能设计育种已经成为改良作物关键育种性状的全新途径。本文系统介绍了表观遗传修饰在植物生长发育和环境适应性中的调控作用,以及针对重要育种性状的表观智能设计改良策略,旨在为利用表观智能设计育种技术创制优异作物新种质提供参考,以推动作物育种进入智能化时代。
1 表观遗传修饰为智能设计育种提供新方向
表观遗传是指是不依赖DNA序列改变调控基因表达的方式,主要包括DNA甲基化(DNA methylation)、RNA 甲基化(RNA methylation)、组蛋白 修 饰 (histone modification)、非 编 码 RNA(noncoding RNA)、染色质重塑(chromatin remodeling)等类型(图1),在调控植物生长发育和环境适应性等方面发挥重要功能[1-2]。表观遗传修饰被甲基转移酶(writer)、去甲基化酶(eraser)和识别蛋白(reader)共同调控,呈现动态可逆的变化特点。
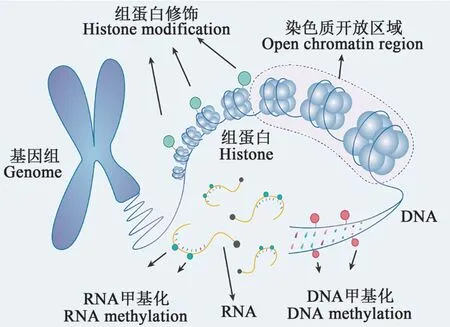
图1 表观基因组构成Fig. 1 Component of epigenome
1.1 表观修饰类型的鉴定
不同表观遗传修饰水平的变化能够影响染色质可及性,通过调节相关基因的表达从而调控生物多种生命过程[3-5]。表观遗传学方法的发展推进了遗传的研究,利用了包括高质量的抗体、染色质功能分析、成像工具、高通量测序技术和整合生物信息学流程等在内的多种策略(图2)。同时,测序技术的不断进步极大地推动了全基因组范围内的表观遗传修饰位点鉴定和功能机制研究。
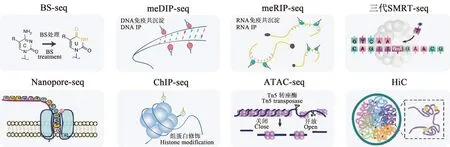
图2 表观遗传修饰检测方法Fig. 2 Technologies relevant to epigenome
DNA甲基化是现阶段功能解析最为深入的表观遗传修饰类型之一,主要有5-甲基胞嘧啶 (5-methylcytosine,5mC)和 N6-甲 基 腺 苷 (N6-methyladenine, 6mA)两 种 修 饰 类 型[2,6-7]。 DNA 5mC是真核生物中分布最广泛的甲基化修饰,同时也是研究最多的DNA修饰标记[2]。DNA 6mA在真核生物中被认为是一种全新的核酸修饰类型,在基因表达调控中发挥了重要的作用[2,7]。许多技术体系被开发用于DNA甲基化的定性和定量分析,其中重亚硫酸盐(bisulfite, BS)处理是大多数DNA 5mC分析的基础,它将单链DNA中的胞嘧啶(ctosine, C)转化为尿嘧啶(uracil, U),但不影响5mC修饰位点,结合测序技术可以获得全基因组范围内单碱基精度的5mC位点和修饰丰度[8]。其他检测技术主要基于酶切法,将不同甲基化敏感性的特异性内切酶消化基因组DNA,获得DNA甲基化的粗略定量[9]。DNA甲基化检测技术在过去的几十年里不断改进和提升,加速了DNA甲基化修饰在特定位点和总体水平上的研究。例如,近年来兴起的以Oxford Nanopore Technologies (ONT)和 Pacific Biosciences (PacBio)为代表的三代测序技术(third-generation sequencing-based technologies, TGS)可以不经过聚合酶链式反应(polymerase chain reaction, PCR)扩增,实现长片段DNA分子的测序,这使得在同一文库研究多种DNA甲基化修饰成为可能,并且可以摆脱BS处理对DNA甲基化带来的不利影响。
近年来,RNA甲基化修饰作为转录后修饰的调控方式,正引领全新的表观遗传学研究的浪潮。6-甲基腺嘌呤(N6-methyladenosine, m6A)和 5-甲基胞 嘧 啶 (5-methylcytosine, m5C)是 最 常 见 的 两 种RNA修饰类型。RNA甲基化免疫共沉淀测序(methylated RNA immunoprecipitation sequencing,MeRIP-Seq)借助RNA-蛋白免疫共沉淀和高通量测序技术,可实现表观转录组上RNA甲基化修饰位点鉴定[2]。此外,下一代测序(next generation sequencing,NGS)测序技术的开发和应用也进一步加速RNA甲基化在全基因组范围的精确定量研究,但其高昂的成本和检测修饰产生的假阳性仍需进一步改善。
组蛋白修饰是另一种重要的表观遗传修饰,研究人员已经开发了许多技术成功应用到其功能和动态变化特性的研究中。其中绝大部分方法都是基于染色质免疫共沉淀(chromatin immunoprecipitation, ChIP)试验开发的[10-11]。特异性抗体靶向ChIP是高效研究DNA-蛋白质互作的技术,可以研究特定DNA序列附近的染色质结构。同时,ChIP也可以和其他技术(ATAC-seq和Hi-C等)结合来阐释组蛋白修饰和其他染色质调控因子在特定位点的相互作用[12]。将ChIP和捕获染色质三维空间结构的3C(chromosome conformation capture,3C)技术结合,可以获得三维染色质空间结构上表观遗传修饰分布和染色质空间重排之间的潜在关系[13]。
1.2 表观遗传修饰影响植物生长发育和环境适应性
DNA 5mC是哺乳动物和植物中含量最丰富的表观遗传修饰类型,通过维持基因组稳定性、调控基因表达进而控制生物的生长发育和环境适应性。DNA 6mA作为一种新的表观修饰标记,广泛存在于原核和真核生物基因组中[6]。近年来,多种检测技术的成功应用使得DNA 6mA的精确定量成为可能,液相色谱-串联质谱技术(LC-MS/MS)、6mA IP结合TGS能够精确定量DNA 6mA修饰水平。目前,研究者已经在拟南芥(Arabidopsis thaliana)、水稻(Oryza sativa)、大豆(Glycine max)等物种中鉴定了DNA 6mA修饰丰度,并且揭示其行使的生物学功能[2]。拟南芥中DNA 6mA含量(6mA/A)约为0.048%、水稻中DNA 6mA修饰丰度约为0.55%、大豆中为DNA 6mA修饰丰度约为0.026 4%,这些植物的6mA含量低于真菌和细菌[2,6,14-16]。 在 水 稻 中 ,ALKBH(α-ketoglutaratedependent dioxygenase alkB homolog)家 族 中 的ALKBH1及其同源蛋白作为去甲基化酶主要行使去甲基化酶的功能——移除6mA上的甲基,但不会去除RNA上的甲基。目前,还未在植物中鉴定到DNA 6mA的识别蛋白[16]。已有研究表明,6mA与植物发育和胁迫响应密切相关。水稻deficient in dna methylation 1a/1b(ddm1a/1b)突变体中 6mA修饰丰度降低,导致植株呈现矮化、结实率降低的表型[6]。此外,还有研究表明,ALKBH1能够介导DNA 6mA修饰延迟水稻的抽穗期[16]。我们近期的研究结果表明,在水稻中DNA 6mA和3D染色质结构共同调控水稻对高温胁迫的应答[17]。
真核生物中包含170多种RNA甲基化修饰,其中分布最广泛的是m6A修饰[18]和m5C修饰。研究表明,RNA m6A和m5C修饰参与调控RNA加工、降解、翻译等转录后几乎所有代谢过程影响生物体发育[19]。RNA m6A作为RNA甲基化修饰含量最丰富的一种修饰,能够被甲基转移酶、去甲基转移酶和识别蛋白共同调控,呈现动态可逆的变化[20-23]。人类 ALKBH 家 族 成 员 FAT MASS AND OBESITYASSOCIATED PROTEIN (FTO)是第一个被鉴定到的RNA m6A去甲基化酶[24],由此掀开了RNA甲基化研究的热潮。但是,在植物中仍然没有鉴定到FTO的同源基因。在人类中还存在ALKBH家族的其他成员——HsALKBH5,在拟南芥中为AtALKBH9B和AtALKBH10B,这些成员可以行使去甲基化酶的功能,但其在不同物种间的保守性和特异性仍需进一步研究[25-27]。m6A甲基化转移酶复合体在人和植物中存在部分保守性,其中人类去甲基化转移酶复合体关键成员METTL3、METTL14和WTAP与水稻和拟南芥中的成员MTA、 MTB和FIP37高度保守[28-29]。人类和拟南芥包含YTH (YT521-B同源性)结构域的蛋白质(ECT2、 ECT3和ECT4)通过YTH结构域识别m6A,从而调节细胞核中的可变多聚腺苷酸化和3’UTR加工以及细胞质中RNA的降解[30-31]。m6A在胚胎发育、叶片形态发生、花转变、雄性减数分裂、绒毡层降解、硝酸盐代谢和根发育等过程中具有重要作用[20,23,25-26,28-33]。拟南芥甲基转移酶 FIP37 介导 RNA m6A调控WUSCHEL和SHOOT MERISTEMLESSmRNA的稳定性,最后控制茎尖干细胞的命运决定[28]。水稻中的FIP37同源蛋白和OsFIP37-ASSOCIATED PROTEIN 1调节小孢子的早期退化和雄性减数分裂[29,32]。已有研究表明,在拟南芥中人类METTL16同源蛋白FIONA1 (FIO1)作为一个新的 m6A 甲基转移酶,参与调控开花过程[23,33]。YTH结构域蛋白ECT2、ECT3和ECT4能够识别RNA m6A位点,冗余性地调节毛状体和叶片的发育[30],其中ECT2通过调控m6A修饰的毛状体分枝相关基因mRNA的稳定性行使功能[31]。在拟南芥 中 ,CLEAVAGE AND POLYADENYLATION SPECIFICITY FACTOR30 (CPSF30)读取m6A,调控可变多聚腺苷酸化调控开花和氮素吸收[20,22]。同时,m6A也能调节植物对多种胁迫的适应性。MTA、MTB、VIRILIZER (VIR)和 HAKAI介导 m6A修饰增强拟南芥的耐盐性[34],此外m6A甲基化也参与了水稻和植物病毒之间的相互作用[35]。RNA m5C作为另外一种主要的mRNA修饰类型,仅在水稻和拟南芥中鉴定到了一个甲基转移酶——HsNSUN2同源蛋白[36-38]。目前,在人类中只报道了1个去甲基酶——TET2,在植物中去甲基转移酶仍未被鉴定和报道[28]。ALYREF是人类m5C的识别蛋白,但并未在植物中鉴定到它的同源蛋白。m5C修饰能够调控拟南芥mRNA的翻译活性和稳定性,并且能够增强水稻蛋白质的合成。在根发育进程中,m5C参与mRNA到靶细胞的转运[36]。水稻m5C甲基转移酶OsNSUN2能够控制抗氧化应激能力并且增强耐热性。
染色体主要由折叠的DNA缠绕组蛋白八聚体组成,组蛋白修饰是指翻译后蛋白复合体氨基末端进行的共价修饰。近年来,组蛋白修饰相关研究已取得大量进展,其在植物基因表达、生长发育、逆境响应等过程中发挥着十分重要的功能[39]。截至目前,鉴定到的组蛋白修饰主要有甲基化、乙酰化、磷酸化、腺苷酸化、泛素化、SUMO化、ADP核糖基化等类型[36]。组蛋白甲基化由组蛋白甲基化转移酶(histone methyl transferase, HMT)介导,在氨基末端的不同位点、不同氨基酸残基上进行修饰,同时实现不同甲基化数目(me1、me2和me3)的修饰。组蛋白H3赖氨酸甲基化主要发生在4个位点上——K4、K9、K27和K36,已有研究表明,H3K4me3和H3K36me3主要富集在常染色质上,与基因的激活表达相关[40-41],而H3K27me3和H3K9me2则是抑制基因表达[42-43]。组蛋白修饰呈现时空特异性,利用甲基转移酶和去甲基转移酶和识别蛋白,可以动态调控生物体的生长发育进程[39]。
综上所述,由表观转录组和表观基因组调节因子共同调控的生长发育和胁迫响应过程与作物重要与育种性状紧密关联,表明其可作为作物育种性状和环境适应性改良的目标,具有极高的应用价值。
1.3 表观遗传修饰调控回路设计控制重要性状
表观遗传修饰设计调控回路在动物中已取得快速进展。利用合成的6mA识别蛋白/甲基转移酶在哺乳动物细胞中构建了人工表观遗传调控系统,促进了染色质的空间传播和标记扩散以及转录状态的表观遗传记忆[44]。在人类和小鼠中,通过m6A识别蛋白/去甲基化酶回路与有规则间隔的短回文重复(clustered regularly interspaced short palindromic repeats,CRISPR)系统 CRISPR-Cas9 或CRISPR-CasRx结合,通过甲基转移酶或者去甲基化酶调控RNA特定位点的修饰水平[45]。近期,研究人员在人类中设计了一种CRISPRoff系统[46],将DNA 5mC甲基化转移酶DNMT3A与DNMT3L蛋白结构域和锌指蛋白ZNF10 KRAB结构域融合到失活的Cas9蛋白上(dCas9)。DNMT3A和DNMT3L位于dCas9蛋白的N端,使得DNMT3A能够拥有接近DNA甲基化CpG岛的最优途径。该系统能够沉默大多数含有CpG岛的基因,调控不含CpG岛的基因,并通过细胞分裂和干细胞分化向神经元灌输表观遗传记忆。
相比在动物中的研究进展,目前植物表观遗传模块设计研究比较缓慢。在拟南芥中,花期基因FLOWERING WAGENINGEN(FWA)的5mC甲基化可被与锌指融合的蛋白SU(VAR)3-9 HOMOLOG 9(或TET1)特异控制,从而调控开花过程[47-48]。此外,研究人员开发了基于CRISPR/dCas9的靶向甲基化/去甲基化工具,该工具可以在基因组的靶向位点精准高效地调控5mC修饰水平[48-49]。这些系统通过重激活或沉默植物表观基因组特定位点的修饰,进而创制新表观等位基因。上述植物相关研究主要关注了5mC甲基化水平的调控,但是新型甲基化修饰(DNA 6mA,RNA m5C和m6A等)和组蛋白修饰的调控还未在植物中实现。这些研究为重要农艺性状的调控提供了全新的思路,为培育智能作物提供新的设计策略。
2 人工智能技术加速作物设计育种
近年来,随着科学技术和计算能力的不断发展,人工智能(artificial intelligence,AI)已广泛应用于大规模数据分析以解决计算机视觉(computer vision,CV)、自然语言处理(natural language processing,NLP)、自动驾驶(autonomous driving)、知识图谱(knowledge graph)、蛋白质结构(protein structure)等领域的复杂问题。而对于作物育种领域,人工智能与生物技术的融合将作物育种推进“育种4.0时代”[50]。
2.1 人工智能技术加速多组学图谱绘制和功能解析
得益于以高通量测序技术为代表的前沿技术迅速发展,针对不同物种、不同组织、不同生长发育时期、不同胁迫环境条件下的,包括基因组、表观遗传组、转录组、蛋白组、代谢组、环境组、微生物组、表型组等在内的生物多组学数据得以大量积累,它们的挖掘与利用可以加速作物的改良与育种。
受到表观组学存在动态变化特性的影响,试验数据的收集通常局限于局部的组织、基因型或环境条件,限制了针对多组学动态性调控的研究范围,同时阻碍了对整个生物生命周期中的基因在不同环境条件下表观遗传调控方式的研究。对于这种复杂系统,机器学习在加速多组学图谱绘制的领域中展现了出色的效果。在植物基因组研究中,机器学习已被用于提升基因组组装质量[51];表观基因组学研究中,机器学习已用于绘制杨树DNA 5mC图谱[52]和水稻DNA 6mA图谱[53-58];转录组研究中,机器学习也被用于进行胁迫响应的转录组差异网络分析[59]以及多聚腺苷酸化(polyadenylation, PolyA)位点的预测[60];表观转录组研究中,RNA上多种表观修饰位点都被建立了机器学习的模型学习其潜在的修饰规律[61];而在植物的表型组研究中,基于计算机视觉的机器学习方法在高光谱、X光、遥感等数据上实现了表型识别、测量、预测等任务[62]。我们整合课题组发表的粳稻品种‘日本晴’和籼稻品种‘93-11’的基因组、DNA 6mA和5mC全基因组图谱、公开的多种组蛋白修饰和转录组组学数据[6],利用人工智能开发了DNA甲基化智能预测模型,并进行全基因组预测,综合这些数据构建了世界上首个水稻表观智能数据库eRice[57]。数据库表观组和基因组数据的整合提升了数据利用和深度挖掘能力,实现了基因序列和注释信息、DNA甲基化、组蛋白修饰及DNA甲基化智能预测数据的系统查询和一体化展示。同时,我们基于深度学习算法构建了植物表观遗传修饰智能预测模型SMEP(smart model for epigenetics in plants),通 过 利 用 植 物DNA甲基化、RNA甲基化、组蛋白修饰等序列信息,实现了水稻、玉米等物种中表观修饰位点的预测[58]。可及性是染色质的重要特征,决定细胞核大分子和DNA的接触程度,这对于基于的转录调控起着至关重要的作用。因此,基于籼粳稻在正常条件和热处理条件下的染色质开放数据[17],利用深度学习算法,构建了高精度基于序列的染色质可及性智能预测模型,实现了植物表观组学数据可视化与人工智能的交叉融合[63]。这些模型的成功开发为作物功能基因组研究和表观智能设计育种提供工具和数据支撑。
随着对多组学及其调控网络的复杂性以及不同组学之间相互调控的认识不断提高,生命活动各个不同层级信息的多组学数据可以被整合起来,用于发现和挖掘复杂的生命活动,这也带来了更多对多组学整合工具的需求。机器学习作为高维数据的挖掘方法,被应用于多个不同组学的整合分析上。在杨树的转录组、蛋白组、代谢组的整合分析中,研究人员开发了OnPLS方法以探索PttSCAMP3基因在木质形成和次级细胞壁组成中的作用[64]。而玉米的研究中,MCIA方法被用于整合代谢组和蛋白组分析[65],GFLASSO工具则被用于整合代谢组和转录组分析[66]。除了加速多组学图谱的绘制、整合多组学数据的分析之外,机器学习也常被用于挖掘多组学数据中隐藏在高维的信息,找到整个复杂的调控网络中的关键节点,解析与推测基因的潜在功能。多变量降维判别分析方法DIABLO在整合早期和中期季节哈斯鳄梨的蛋白质组学和代谢组学数据的过程中,将热处理胁迫与成熟的均匀性联系起来,识别了导致明显差异的相关判别变量,推测了热胁迫下糖酵解和蛋白质降解的生物学途径[67]。
2.2 全基因组优异等位变异的发掘
解析遗传与表观遗传变异是如何影响复杂表型和挖掘全基因组优异等位变异是生物性状改良研究的核心内容。全基因组关联分析(genomewide association study, GWAS)已鉴定出数以万计的与特定性状相关联的遗传变异,但这些变异超过一半都定位于非编码区;同时由于连锁不平衡(linkage disequilibrium, LD)的存在,GWAS 很难区分出其中真正发挥作用的因果变异(causal variant)。相较于传统的统计学方法,机器学习方法能够从不同层面(包括转录因子结合位点、表观修饰位点、染色质可及性以及基因表达等)的信息学习分布规律构建模型,从而预测和挖掘变异位点的潜在功能。
在转录层面,ExPecot[68]和 Enformer[69]使用基因组上调控序列进行建模,利用深度卷积神经网络和Transformer结构拟合基因表达和调控序列之间的关联,从而从海量的表达数量性状位点(expression quantitative trait loci, eQTL)中挖掘真正影响基因表达的变异位点。DeepSEA[70]、DeepBind[71]、BPNet[72]等方法利用DNA元素百科全书计划(encyclopedia of DNA elements, ENCODE)[73]等数据集中几百种转录因子结合位点的信息,构建深度学习模型,预测输入序列的转录因子结合情况。将GWAS关联到的每个单核苷酸多态性(single-nucleotide polymorphism, SNP)位点替换到基因组的参考序列上,通过评估序列替换前后预测分值的改变,将其视为该SNP对调控序列影响的衡量标准。除了通过转录因子结合位点,基因组上的表观修饰和染色质开放性区域也常被用于挖掘基因组变异造成的影响。Basset[74]利用脱氧核糖核酸酶I超敏感位点测序(DNase I hypersensitive site sequencing, DNase-seq)获 得 基因 组 开 放 信 息 ,在 水 稻 中 RiceVarMap V2.0[75]、PlantDeepSEA和SMOC[63]也利用了染色质可及性测 序 (assay of transposase accessible chromatin sequencing, ATAC-seq)所产生的基因组开放区域;Basenji[76]则同时利用了组蛋白修饰和染色质开放情况作为建模来揭示组蛋白修饰和染色质开放区域对基因组变异的影响。INTERACT[77]专注于挖掘5mC修饰程度造成的影响,而Akita[78]和Orca[79]则利用染色质3D的互作情况,从大片段的结构变异(structure variant, SV)搜索对基因组 3D 构象造成改变的变异。
2.3 基因组设计助力种质资源创制
为克服分子标记辅助育种(marker-assisted breeding,MAS)在数量性状上的局限性,Meuwissen[80]在2001年提出的新型的分子辅助育种策略——基因组选择(genomic selection, GS)发挥了主要的作用。相较于分子标记辅助育种,基因组选择尽可能地在全基因组范围内考虑遗传变异的影响,大大提升了育种的效率,同时也对计算方法提出了新的挑战。
早期的基因组选择大多选用统计学方法,如实现岭回归最小无偏估计(ridge regression best linear unbiased prediction)的 rrBLUP[81],集 成 BayesA、BayesB、BayesC等贝叶斯相关算法的BGLR[82],以及可以实现稀疏偏最小二乘回归算法的spls(sparse partial least squares)。但随着机器学习方法在其他领域的成功应用,基于随机森林(random forest, RF)和支持向量机(support vector machine, SVM)的R语言e1071模块也被应用于基因型到表型的预测,而基于LightGBM的育种工具箱CropGBM最终在各项指标上都取得出了优异的结果[83]。以DeepGS[84]、DLGWAS[85]和DNNGP[86]为代表的深度学习方法也开始在基因组选择领域崭露头角。
基因组选择是自下而上,从基因组上聚合优良的变异位点从而实现基因组优化(genome optimization)[87]。从“最优表型”反推“最优基因组”所需要的片段组合方式,最终通过拼接重组片段的方式实现基因组设计。基因组优化首先需要通过大规模重组自交系基因型数据,利用计算出的重组热点将整个基因组切割为许多最小的高频重组片段,再通过机器学习算法将这些片段与表型进行关联建模,并推测出达到最优表型的片段组合方式,最后按片段在基因组上的位置拼接出一种虚拟的“理想基因组”[87-88]。基因组优化也是目前技术挑战最难、潜力最大的基因组设计方法,需要人工智能算法和育种家相互协助,才能完成复杂的基因组顶层设计。
3 表观遗传智能设计定向创制优良种质
表观遗传修饰位点预测和设计的准确性,以及表观遗传调控回路定向改造的高效性是实现表观智能设计和作物改良的关键。通过整合工程学理念和表观遗传修饰位点、智能设计和合成调控回路,精准改良作物的农艺性状,融合表观遗传和人工智能的“表观遗传智能设计技术”可为作物遗传改良提供新范式,开辟出新的作物育种方向[89]。
3.1 表观遗传编辑技术精准调控基因表达
以CRISPR/Cas9技术为代表的基因编辑技术可以在基因组水平对DNA序列进行精准、高效编辑,对基因功能研究有着至关重要的作用。在农业领域CRISPR/Cas9技术被广泛使用在农艺性状改良、新种质创制、作物遗传育种等多个方面。研究者在蕃茄上使用CRISPR技术对启动子序列进行精准的编辑,改变了果实大小、花序分枝和株型等重要的农艺性状,从而打破了产量的限制[90]。基于CRISPR的表观遗传编辑技术可以在不改变基因编码序列的情况下,通过表观修饰位点数量和修饰丰度的定向编辑,实现基因表达水平的定向调控,以改良作物产量、耐逆性等重要农艺性状,其在作物产量、耐逆性改良上具有重要应用潜力。表观遗传编辑技术无需对基因编码序列进行切割就能抑制靶基因的表达,并且可以逆转,能在多种表观遗传修饰的共同作用下产生更持久叠加的生物学效应,从而更加安全和灵活的精准调控基因表达。CRISPRoff技术是一种以CRISPR为基础的新型表观编辑技术,其在保持序列不变的基础上促进靶位点附近DNA甲基化,从而实现持久可遗传的基因转录抑制,同时CRISPRon技术可利用去甲基化酶逆转沉默效果[46]。在作物产量性状的研究方面,研究者将哺乳动物的RNA去甲基化转移酶FTO引入到水稻和土豆基因组中,通过对其RNA修饰m6A进行特异性去甲基化促进了分蘖的增加和根系的生长,大幅度地提高了生物量和产量,该技术显示了RNA表观遗传修饰在作物产量性状的改良上的具大应用潜力[91]。
3.2 表观遗传智能设计和合成创制优异新种质
植物表观遗传结合基因编辑、合成生物等技术的研究,不仅提升对表观调控机制更深入的了解,同时在结合人工智能决策体系下,形成了表观遗传智能设计育种技术,为定向创制智能适应性作物提供新的方向。智能模型可以实现全基因组水平上的表观遗传修饰位点的精准预测,突破了通过实验手段无法检测的潜在表观遗传修饰位点的限制,为作物功能基因组研究和智能设计育种提供了强有力的工具和数据支撑。同时,表观遗传智能设计技术在筛选影响关键农艺性状基因表达的表观遗传修饰位点时,通过模型在全基因组范围内鉴定顺式调控元件,进一步对靶基因启动子、编码区上的表观遗传修饰位点、染色质的开放性以及表观修饰酶酶活进行模拟改造,设计定向改良作物农艺性状的调控回路。另一方面,利用表观遗传智能设计技术鉴定作物株型、抗逆、产量等重要育种性状的关键基因及表观调控位点,重点聚焦目标基因启动子区域以及非编码区序列构成、表观修饰程度等因素,构建通用表观调控核心元件、基因特异表观调控元件等数据库,通过模型或试验确认表观遗传设计和合成对目标基因表达水平及表型间的对应关系,创建“位点鉴定-表观设计-合成重构-迭代优化”的表观遗传智能设计育种体系(图3)。
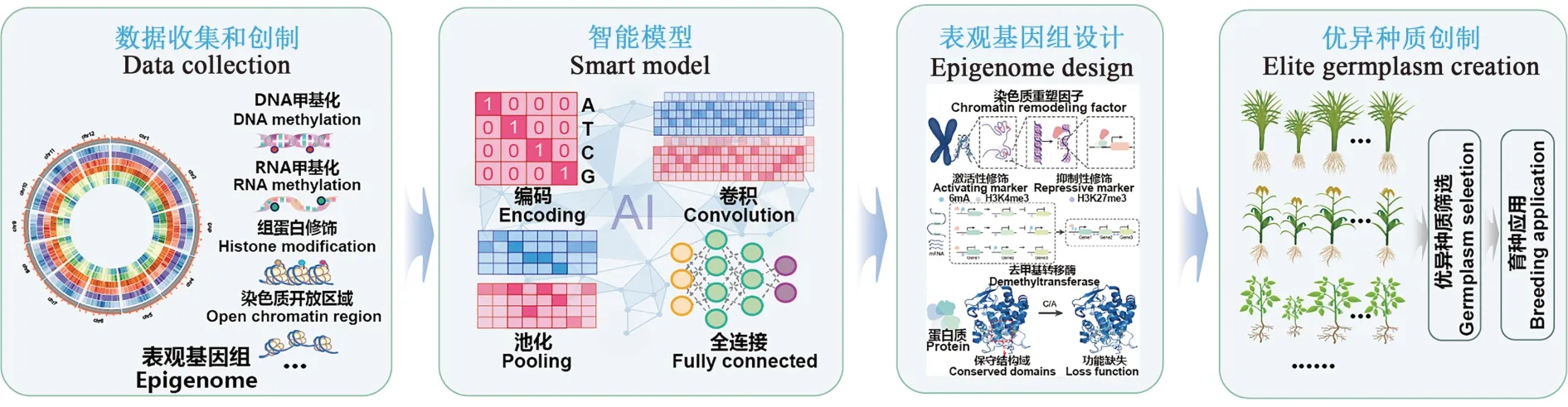
图3 表观遗传智能设计创制优异种质Fig. 3 Smart epigenetic design to culture elite germplasm
4 结语
得益于高通量测序、精准检测、生物技术的快速发展,表观遗传研究取得大量突破性进展,多种表观遗传调控网络相继被解析。新型表观遗传修饰类型也成为研究热点,为作物表观智能设计提供了大量的修饰位点信息和设计靶点。同时,人工智能技术在生命科学领域成功应用及多种表观遗传智能预测工具的开发和应用,极大地推动了表观遗传位点的鉴定和研究,突破了表观遗传修饰动态变化、时空特异性等限制因素,为作物智能设计带来了新机遇。表观遗传智能设计技术采用模型驱动的育种策略,可以设计、评估和量化表观遗传修饰对作物性状的影响。未来,表观遗传智能育种技术可精准设计具有高产、优质、多抗、高效等多种性状聚合的作物新种质和新品种,实现产量和抗性平衡、提高营养品质、增强水肥利用效率并优化植物与微生物间的互作。

