基于融合基因作用于横纹肌肉瘤的miRNA-mRNA 调控网络的生物信息学分析
赵志娟, 孟 莲, 刘春霞,2
(1.石河子大学医学院病理学系 石河子大学医学院第一附属医院病理科,新疆 石河子 832002;2.广州医科大学附属第二医院病理科,广东 广州 510260)
横纹肌肉瘤(rhabdomyosarcoma,RMS) 是儿童最常见的软组织恶性肿瘤,主要包括腺泡状RMS (alveolar RMS,ARMS)、 胚胎型RMS(embryonal RMS,ERMS) 和成人多形性RMS(polymorphic RMS,PRMS)。ARMS 和ERMS 分别占所有RMS 病例的20% 和60%,ARMS 经常出现t (2; 13)(q35; q14) 或t (1; 13)(p36;q14) 染色体易位,分别产生配对盒基因3(paired box gene 3,PAX3) - 叉头型转录因子(forkhead-type transcripition factor,FKHR) 或配对盒基因7 (paired box gene 7,PAX7)-FKHR融合基因[1]。在ARMS 中融合基因具有明确的诊断及预后价值,其代表一种具有明显侵袭性的亚群,该亚群通常对常规治疗无反应,且复发风险高[2]。
微小核糖核酸(microRNAs,miRNAs) 是小的核糖核酸分子,广泛参与肿瘤细胞增殖、分化和凋亡的过程[3]。本课题组前期研究[4]显示:miR-410-3p 高表达可以抑制RMS 细胞的增殖、侵袭和迁移,促进细胞凋亡,并影响上皮- 间质转化(epithelial-mesenchymal transition,EMT) 进程,miRNA 与RMS 的发展和预后有密切关联。miR-874 在RMS 中起抑癌作用,并通过调控鸟嘌呤核苷酸交换因子T (guanine nucleotide exchange factor T,GEFT) 抑制RMS 细胞的生长和转移[5]。但miRNA 在RMS 中参与融合基因形成的相关研究较少。
本研究通过生物信息学方法寻找融合基因阳性和阴性RMS 组织中差异表达基因(differentially expressed genes,DEGs) 和差异表达miRNA,构建miRNA-mRNA 分子调控网络,为RMS 的诊断、治疗和预后提供新的思路。
1 资料与方法
1.1 资料来源从基因表达图谱(Gene Expression Omnibus,GEO) 数据库(https://www.ncbi.nlm.nih.gov/geo/) 中下载GSE66533和GSE97553 数据集。其中GSE66533 为33 例融合基因阳性和25 例融合基因阴性RMS 组织的基因表达谱数据,GSE97553 为2 例融合基因阳性和7 例融合基因阴性RMS 异种移植模型组织的miRNA 微阵列数据。
1.2 筛选DEGs 和差异表达miRNA使用GEO2R 分析工具(https://www.ncbi.nlm.nih.gov/geo/geo2r/) 分别对GSE66533 和GSE97553数据集进行分析,以P<0.01 且|logFC|≥1 为标准筛选DEGs,以P<0.05 且|logFC|≥3 为标准筛选差异表达miRNA。使用R 软件ggplot2 和pheatmap程序包进行可视化分析,生成火山图和热图。
1.3 miRNA 靶基因预测通过在线数据库miRDB (http://mirdb.org/)、 TargetScan(http://www.targetscan.org/vert_71/) 和StarBase (http://starbase.sysu.edu.cn/) 3 个平台进行差异表达miRNA 靶基因预测,采用Bioinformatics Evolutionary Genomics (http://bioinformatics.psb.ugent.be/webtools/Venn/) 将3 个平台的预测结果与前面筛选的DEGs 取交集,得到目标基因。
1.4 基因本体(Gene Ontology,GO)功能和京都基因与基因组百科全书(Kyoto Encyclopedia of Genes and Genomes,KEGG)通路富集分析使用在线工具DAVID (https://david.ncifcrf.gov/)对筛选的目标基因进行GO 功能和KEGG 通路富集分析,GO 功能分析包括生物学过程、细胞组成和分子功能,以P<0.05 为有效。
1.5 蛋白-蛋白互作(protein-protein interaction,PPI)网络构建和核心基因(hub 基因)筛选通过在线分析工具STRING (https://string-db.org/) 预测并构建目标基因的PPI 网络,将文件导入Cytoscape 3.6.1 软件进行可视化,并使用插件cytoHubba 筛选Top10 hub 基因。
1.6 生存分析使用Kaplan-Meier Plotter 数据库(http://kmplot.com/analysis/) 评估Top10 hub基因与肉瘤患者总生存(overall survival,OS) 率的关系,绘制Kaplan-Meier 生存曲线。
1.7 统计学分析采用R Studio 软件制图并进行统计学分析。采用Kaplan-Meier 法(Log-rank 检验) 绘制生存曲线,进行生存分析。以P<0.05 为差异有统计学意义。
2 结 果
2.1 融合基因阳性和阴性RMS 组织中DEGs 和差异表达miRNA对融合基因阳性和阴性RMS 组织的基因和miRNA 表达数据进行分析,以P<0.01且|logFC|≥1 为标准 筛 出891 个DEGs ,其中512 个表达上调,379 个表达下调(图1A 和B)。以P<0.05 且|logFC|≥3 为标准筛出14 个差异表达miRNA (图1C 和D),表达上调和表达下调各7 个(表1)。

表1 本研究筛选的差异表达miRNATab.1 Differentially expressed miRNA screened in this study
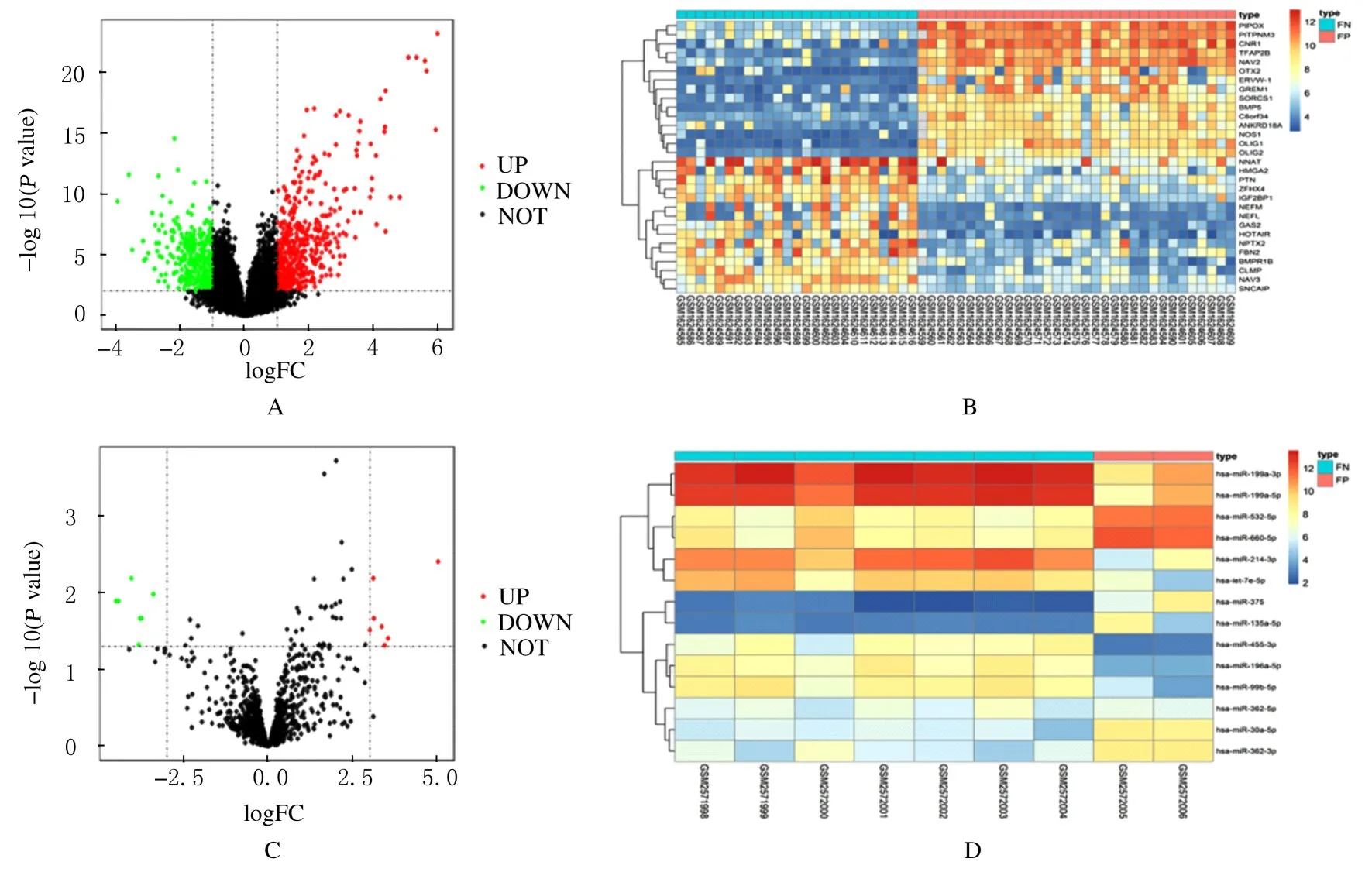
图1 GSE66533 数据集中DEGs 和GSE97553 数据集中差异表达miRNAFig.1 DEGs in GSE66533 dataset and differentially expressed miRNA in GSE97553 dataset
2.2 差异表达miRNA 靶基因的预测采用TargetScan、miRDB 和StarBase 数据库预测差异表达miRNA 的靶基因,并对3 个数据库的结果进行重叠,共预测出1 654 个靶基因。与891 个DEGs 取交集得到115 个目标基因(图2)。

图2 DEGs 与预测的靶基因交集的韦恩图Fig.2 Venn diagram of intersection of DEGs and predicted target genes
2.3 目标基因的GO 功能和KEGG 通路富集分析GO 功能富集分析显示:目标基因富集于RNA聚合酶Ⅱ启动子转录、细胞增殖的正向调节和多细胞生物的发展等生物学过程,富集于细胞质膜、细胞表面和转录因子复合体等细胞组成成分,富集于转录因子活性和DNA 序列特异性结合、蛋白结合和染色质结合等分子功能(图3A~C)。KEGG 通路富集分析显示:目标基因参与癌症中的蛋白聚糖、黑色素生成和细胞外基质-受体相互作用等通路(图3D)。

图3 目标基因的GO 功能(A-C)和KEGG 通路(D)富集分析Fig.3 GO function(A-C) and KEGG pathway enrichment analysis(D) of target genes
2.4 PPI 网络和hub 基因分析通过STRING 软件绘制115 个目标基因的PPI 网络,得到1 个包含74 个结点和81 条边的PPI 网络图(图4)。使用插件cytoHubba 筛选出Top10 hub 基因,分别为表皮生长因子受体4 (oncogene encoding human epidermal growth factor receptor 4,ERBB4)、受体型蛋白质酪氨酸磷酸酶D (protein tyrosine phosphate receptor type delta,PTPRD)、 胰岛素受体底物1 (insulin receptor substrate 1,IRS1)、整合素金属蛋白酶 10 (a disintegrin and metalloprotease 10,ADAM10)、 Yes 相关蛋白1(Yes associated protin1,YAP1)、转录因子AP-2A(transcription factor AP-2 alpha,TFAP2A)、细胞黏附分子1 (cell adhesion molecule 1,CADM1)、ELAV 类似RNA 结合蛋白2 (ELAV like RNA binding protein 2,ELAVL2)、 锌指转录因子1(zinc finger protein 1,SNAI1) 和ERBB 受体反馈抑制剂1 (ERBB receptor feedback inhibitor 1,ERRFI1)(图5A)。通过Cytoscape 软件对hub 基因进行miRNA-mRNA 分子调控网络的构建(图5B)。
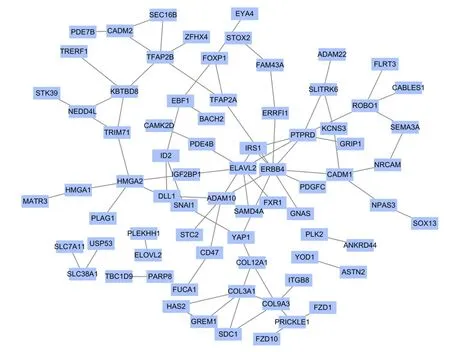
图4 目标基因的PPI 网络图Fig.4 PPI network diagram of target genes

图5 PPI 网络的Top10 hub 基因(A)和对应的miRNA-mRNA 分子调控网络(B)Fig.5 Top10 hub genes of PPI network(A)and corresponding miRNA-mRNA molecular regulation network(B)
2.5 Top10 hub 基因的生存分析因GEO 数据库里无RMS 患者相关临床信息,所以针对筛选出的Top10 hub 基因,在Kaplan-Meier Plotter 数据库对其进行生存分析,该数据库包括259 例肉瘤患者的数据,但是未对肉瘤进行具体分型,结果显示:PTPRD (HR=1.79,P=0.005)、ADAM10 (HR=1.94,P=0.010)、ELAVL2 (HR=1.56,P=0.031) 和ERRFI1 (HR=2.05,P=0.005) 表达水平升高与RMS 患者的不良预后有关,而上述4 个基因在融合基因阳性的肉瘤患者肿瘤组织中表达上调(图6)。其余各基因的表达差异对肉瘤患者的预后无明显影响,差异无统计学意义(P>0.05)。

图6 与肉瘤患者预后相关的hub 基因Fig.6 Hub genes associated with prognosis of sarcoma patients
3 讨 论
本研究采用生物信息学方法对融合基因阳性和阴性RMS 患者进行分析,共筛选出115 个目标基因。KEGG 富集分析显示:目标基因主要富集在细胞外基质-受体相互作用、癌症中蛋白聚糖等途径中。细胞外基质是调节细胞发育、正常功能和体内平衡的最重要的因素之一。研究[6]表明:细胞外基质和细胞外基质结合受体的异常组成和功能对癌症发生发展具有重要意义。miR-215-5p 通过调节细胞外基质-受体相互作用和局灶性黏附等分子途径,抑制结直肠癌细胞的转移[7]。在肿瘤组织中,肿瘤细胞分泌的可溶性因子可被蛋白聚糖激活。由于其作为共受体的功能,蛋白聚糖可促进肿瘤细胞的增殖[6]。蛋白聚糖不仅在肿瘤基质中发挥作用,还可以对癌细胞的表型、特性和耐药性的发展进行调节[8]。上述研究表明:细胞外基质-受体相互作用和癌症中蛋白聚糖等途径可能是融合基因在RMS中潜在的作用机制。
本研究利用cytoHubba 插件对115 个目标基因进行筛选,选出Top10 hub 基因,根据hub 基因查询预测靶基因的miRNA,将hub 基因联合miRNA得到miRNA-mRNA 分子调控网络,结合生存分析筛 选 出miRNA-214-3p (PTPRD、 ERRFI1)、miR-375 (ELAVL2) 和miR-199a-3p (ADAM10、ELAVL2),可为研究RMS 的融合基因提供参考。
miRNA 是一类保守的非编码核糖核酸,广泛参与肿瘤的增殖、 侵袭和血管生成等过程。在骨肉瘤组织中miR-214-3p 的高表达能通过靶向泛醌-细胞色素c 还原酶复合物核心蛋白1 (ubiquinol cytochrome c reductase core protein 1,UQCRC1)促进骨肉瘤细胞增殖,为预防和治疗骨肉瘤提供了潜在的治疗靶点[9]。在食管鳞状细胞癌中miR-214-3p为抑癌因子,其表达下调通过靶向生存素(survivin) 和CUG 结 合 蛋 白1 (CUG-binding protein 1,CUG-BP1) 促进食管癌细胞的化疗耐药性[10]。有研究[11]显示:miR-214 在人RMS 细胞系中明显下调,且miR-214 和神经母细胞瘤RAS病毒致癌基因(neuroblastoma RAS viral oncogene,
N-ras) 调控还能抑制RMS 细胞生长和异种移植瘤的发生。miR-214-3p 在融合基因阳性的RMS 中下调,且PTPRD 和ERRFI1 为miR-214-3p 的靶基因。PTPRD 是蛋白质酪氨酸磷酸酶家族的成员之一,在胶质母细胞瘤和肺癌中已有通过缺失、突变或表观遗传甲基化使PTPRD 失活的报道[12]。PTPRD 在神经母细胞瘤中通过Aurora 激酶A(Aurora kinase A,AURKA) 去磷酸化和去稳定化以及下游编码转录因子N-MYC (encoding N-MYC,MYCN) 蛋白的去稳定化发挥抑瘤作用[13]。外源性表达PTPRD 在人胶质母细胞瘤、黑色素瘤和结肠癌中可以抑制肿瘤细胞生长和迁移,还可以通过诱导细胞凋亡降低细胞活性[14-15]。胶质瘤衍生的miR-148a-3p 通过抑制ERRFI1 激活表皮生长因子受体(epidermal growth factor receptor,EGFR) /丝裂原活化蛋白激酶(mitogen-activated protein kinase,MAPK) 信号通路促进肿瘤血管生成[16]。长链非编码RNA (long non- coding RNA,LncRNA) 中细胞周期激酶抑制因子4 基因座中反义非编码RNA (antisense non-coding RNA in INK4 locus,ANRIL) 通过表观遗传抑制ERRFI1的表达促进胆管癌的恶性进展[17]。目前在RMS 中尚未发现miR-214-3p 与靶基因PTPRD 和ERRFI1关系的相关研究报道。
miR-375 在多种癌组织中表达下调,通过作用于重要的癌基因来调节癌症相关的过程,如细胞增殖、凋亡、侵袭、迁移和自噬[18-22]。在骨肉瘤患者中miR-375 表达下调,其可以成为骨肉瘤诊断、预后和化疗敏感性预测的标志物[23]。在胶质瘤中miR-375 表达下调且通过抑制结缔组织生长因子-表皮生长因子受体(connective tissue growth factorepidermal growth factor receptor,CTGF-EGFR)信号通路影响神经胶质瘤细胞的增殖、侵袭和迁移[24]。然而在某些类型癌组织中miR-375 表达上调,如前列腺癌,miR-375 的高表达影响前列腺癌的分期和淋巴结转移[25]。本研究结果显示:在融合基因阳性的RMS 中,miRNA-375 表达上调。ELAVL2 作为miRNA-375 的靶基因,在食管鳞状细胞癌组织中过表达,且通过抑制细胞凋亡提高对紫杉醇的耐药性[26]。在RMS 中上调的miRNA-375可结合ELAVL2 做进一步分析。
MiR-199a-3p 通过调节乙醇胺激酶1(ethanolamine kinase 1,ETNK1) 可以促进胃癌细胞的侵袭和迁移,影响胃癌患者的预后[27]。LncRNA 中DNA 损伤激活的非编码RNA (noncoding RNA-activated by DNA damage,NORAD)通过与miR-199a-3p 内源性竞争调节人骨肉瘤癌细胞的增殖和迁移[28]。本研究结果显示:ADAM10为miR-199a-3p 的靶基因,但目前尚未见相关研究报道。研究[29]表明: 在三阴性乳腺癌患者中miR-365 通过调节ADAM10 抑制肿瘤细胞的侵袭。促红细胞生成素产生肝细胞受体A8(erythropoietin-producing hepatocyte receptor A8,EphA8) 是一种致癌基因,在胃癌组织中通过调节ADAM10 导致胃癌患者预后不良[30]。ADAM10 可能在骨肉瘤的进展和成骨细胞骨肉瘤的病理发展过程中起重要作用[31]。
本研究通过生物信息学的方法对融合基因作用于RMS 的miRNA-mRNA 调控网络进行分析,可为研究融合基因在RMS 发生发展中的作用提供新思路,为后续的实验研究提供参考。

