阿尔茨海默病患者载脂蛋白E 3种分型方法的比较*
王 琪,李 颖,秦 伟,魏一平,曹淑曼,贾建平
首都医科大学宣武医院神经疾病高创中心,北京 100051
阿尔茨海默病(AD)是一种遗传度很高的疾病,而载脂蛋白E(APOE)是AD最为明确的风险基因[1-2]。APOE有3种主要亚型,分别为APOEε2、ε3 和ε4。这3种亚型由APOE基因两个邻近位点rs429358 (C > T)和rs7412 (C > T)的共同多态性导致,对AD的发生、发展产生不同影响。APOEε3是人群中最常见的类型,APOEε4是AD的主要遗传风险因素,而APOEε2是AD发病的保护因素[3-5]。因此,APOE分型成为AD研究中不可或缺的重要手段。目前受到普遍认可并被广泛使用的APOE分型方法主要包括Sanger测序法、Taqman探针法等,但分别存在通量低、时效差或价格高昂等缺点[6]。因此,建立高效、稳定、经济的中、高通量APOE分型方法具有极其重要的意义。竞争性等位基因特异性PCR(KASP)法和单碱基末端延伸(SNaPshot)法都是得到认可的中、高通量单核苷酸多态性(SNP)检测方法,尤其是近年来兴起的KASP法,由于通量高、成本低、更具SNP位点适用性,已经成为国际上SNP分析的主流方法之一,但初期主要应用于植物育种[7-8],在人类基因分型中尚鲜见应用。本研究旨在建立基于KASP、SNaPshot法,更为高效、稳定、经济的中、高通量APOE分型方法,为AD患者APOE分型研究提供更多方法选择。
1 材料与方法
1.1材料来源 48份已确定APOE分型的DNA样本来源于2017-2019年于首都医科大学宣武医院进行APOE分型检测的AD或轻度认知障碍(MCI)患者,该部分样本均采用普通PCR联合Sanger测序法确定APOE分型。为保证新建立检测方法的稳定性和可靠性,选取的48份样本覆盖6种常见的APOE分型,包括2/2型5份,2/3型6份,2/4型6份,3/3型11份,3/4型12份,4/4型8份。进一步收集2019年10-12月于首都医科大学宣武医院就诊的AD、MCI患者的全血样本共107份,对所建立的方法进行扩大样本量验证。本研究获得首都医科大学宣武医院伦理委员会批准,批号为临研审[2017]004号。
1.2方法
1.2.1外周血白细胞基因组DNA提取 所有全血样本均采用盐析法提取基因组DNA,并经紫外分光光度仪定量。
1.2.2基于KASP法的APOE分型检测 根据APOE变异位点序列信息,使用Primer5软件设计能够特异性识别APOE基因分型的2个关键SNP位点rs429358和rs7412的引物,见表1。PCR扩增目的片段,10 μL体系如下:HiGeno 2X Probe mix(北京嘉程基因技术有限公司)5 μL,Primer mix 1 μL(北京睿博兴科科技有限公司),DNA 1 μL,ddH2O 3 μL。反应条件:95 ℃ 10 min;95 ℃ 20 s,61 ℃→55 ℃(-0.6 ℃/cycles)45 s,10个循环;95 ℃ 20 s,55 ℃ 45 s,35个循环。ABI7500(ABI)上机采集FAM和VIC荧光信号,将检测得到的原始数据文件导入到TaqMan Genotyper Software Version 1.3分析软件中进行分析。上述引物合成及产物检测由北京睿博兴科科技有限公司完成。

表1 基于KASP法的APOE分型引物序列
1.2.3基于SNaPshot法的APOE分型检测 根据APOE变异位点序列信息,使用Primer5软件设计引物。预扩增程序为95 ℃ 5 min;95 ℃ 30 s,60 ℃ 30 s,72 ℃ 30 s×35个循环;72 ℃ 7 min,4 ℃ ∞。30 μL预扩增体系包括Ruibio 2×Taq 15 μL,Forward primer(10 μmol/L) 0.5 μL,Reverse primer(10 μmol/L) 0.5 μL,DNA 1 μL,ddH2O 13 μL,预扩增引物见表2。预扩增产物纯化回收后延伸。采用SNaPshot试剂盒(ABI)对样本延伸,5 μL延伸体系包括Ready Reaction Mix 0.4 μL,5×seq Buffer,纯化后PCR产物2.5 μL,检测引物(10 μmol/L)各0.1 μL,ddH2O 0.9 μL,检测引物见表2。延伸程序为96 ℃ 2 min;96 ℃ 10 s,50 ℃ 5 s,60 ℃ 30 s,共35个循环;14 ℃ ∞。Applied Biosystems 3730-XL测序仪(ABI)上机检测,将检测得到的原始数据文件导入到分析软件中进行分析。上述引物合成及产物检测由北京睿博兴科科技有限公司完成。

表2 基于SNaPshot法的APOE分型引物序列
1.2.4普通PCR结合Sanger测序法进行APOE分型验证 使用Primer5软件设计引物,上游引物为5′-AGA CGC GGG CAC GGC TGT CCA AGG A-3′,下游引物为5′-CCC TCG CGG GCC CCG GCC TGG TAC AC-3′。反应体系均为50 μL,含100 ng DNA模板,上下游引物浓度为0.5 μmol/L,dNTP(TAKARA)终浓度为200 μmol/L,10×PCR缓冲液(TAKARA)5 μL,rTaq酶(TAKARA)终浓度为50 000 U/L,DMSO 5 μL。扩增条件为:95 ℃预变性5 min,主循环30次(变性95 ℃ 30 s、退火60 ℃ 30 s、延伸72 ℃ 30 s),72 ℃再延伸7 min。反应产物Applied Biosystems 3730-XL测序仪(ABI)进行测序,Chromas2.23软件分析测序结果。上述引物合成及产物检测由北京睿博兴科科技有限公司完成。
1.3统计学处理 采用特异度和灵敏度评价KASP、SNaPshot法判断APOE分型的准确性,采用χ2检验检测3种APOE分型方法有无差异,采用SPSS22.0统计软件进行数据分析,统计学检验水准设为0.05,以P<0.05为差异有统计学意义。
2 结 果
2.1基于KASP法的APOE分型 KASP法通过对产物中荧光信号的识别,可以很好地进行APOE分型,见图1。图1A是位点rs429358的分型结果,左上蓝色圆形聚集点为FAM荧光信号,识别的是T碱基;右下红色圆形聚集点为VIC荧光信号,识别的是C碱基;图1B是位点rs7412的分型结果,FAM荧光信号识别C碱基,VIC荧光信号识别的是T碱基;两图中间绿色圆形聚集点为同时有FAM和VIC荧光信号,同时识别到T和C碱基,这些样本在该位点是杂合型;靠近坐标轴原点的浅蓝色正方形点是空白对照,无荧光信号,代表未识别到T或C碱基。对已确定APOE分型的全血核酸样本48份进行检测及打乱顺序重复检测,其结果与样本已知分型一致,其灵敏度和特异度均为100%。进一步扩大样本量对107例AD、MCI患者样本进行检测,检出2/3型4份,2/4型1份,3/3型59份,3/4型40份,4/4型3份。
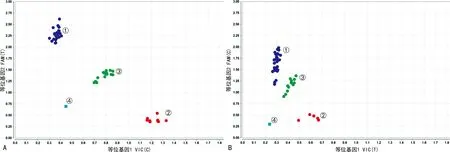
注:A为位点rs429358;B为位点rs7412;①为FAM荧光信号,②为VIC荧光信号,③为同时有FAM和VIC荧光信号,④为无荧光信号。
2.2基于SNaPshot法的APOE分型 SNaPshot法根据峰的移动位置确定产物对应的SNP位点,根据峰的颜色可得知掺入的碱基种类,从而确定该样本的APOE基因型,见图2。

注:A~F依次为2/2、3/2、3/3、4/2、4/3、4/4分型测序图。
图2为APOE基因6种分型的测序图,其中第1个峰为rs429358位点,第2个峰为rs7412位点。对已确定APOE分型的全血核酸样本48份进行检测及打乱顺序重复检测,其结果与样本已知分型一致,其灵敏度和特异度均为100%。对107例AD、MCI患者进行扩大样本检测,与KASP法结果一致,差异无统计学意义(P=1.00)。
2.3Sanger测序法进行APOE分型验证 将上述样本用普通PCR扩增并Sanger测序后分析,结合每个样本2个SNP位点上的碱基情况得出的APOE分型数据与KASP法分型结果及SnaPshot法分型结果完全一致,差异无统计学意义(P=1.00)。见图3。

图3 Sanger测序法APOE分型测序图
3 讨 论
1973年HAVEL等[9]发现了一种“富含精氨酸”的蛋白质,1975年UTERMANN等[10]将这种蛋白质被命名为载脂蛋E,1982年ZANNIS等[11]明确了它的3种主要亚型,并命名为APOEε2、ε3 和ε4。这3种亚型在112位和158位氨基酸的变化,驱动蛋白质中的结构域相互作用,影响其生物学功能。随着APOEε4等位基因的数量增加,AD的患病风险增加,发病年龄减小;随着APOEε2等位基因数量的增加,AD发病风险降低,发病年龄增大[3-5]。因此,APOE分型在AD的基础或临床研究领域都成为不可或缺的重要手段。
近年来,随着各种新技术的涌现,APOE分型方法有了越来越多的选择,较为经典的有聚合酶链反应限制性片段多态性(PCR-RFLP)法、Sanger测序法和TaqMan探针法等[6]。PCR-RFLP法因所需材料设备简单,在早期得到广泛应用,但步骤烦琐且需对PCR产物进行第2次操作,现应用减少;Sanger测序也称一代测序,常作为基因序列检测的“金标准”和APOE分型的验证方法,但通量低,成本略高,时效性较差;TaqMan探针法效率高,耗时短,但由于需合成特异性探针引物,成本高昂。因此,本研究通过建立基于KASP法和SNaPshot法的APOE分型方法,希望能在满足大样本、中高通量APOE分型需求的同时降低检测成本。
KASP法是近年来新兴的一种高通量SNP检测技术,是在等位基因特异性PCR基础上,再加入通用荧光探针,通过设计2个SNP PCR引物,其3′端各对应一个SNP的等位基因,5′端是一段特定的序列,与通用荧光探针序列一致,然后通过直接识别荧光信号来对SNP进行双等位基因判断。该技术初期在植物育种方面应用较多,随着技术的发展成熟,现已被应用于医学领域[12-13]。它与TaqMan探针法原理类似,但不需要每个SNP位点都合成特异荧光引物,所有位点检测最终都可以使用通用荧光引物进行扩增,大大降低了成本。KASP法对DNA样本的浓度和纯度要求不高,且每天可检测SNP及样本数均可达数万以上,具有精度高、灵敏度高、灵活性强、转换速度快、SNP位点适用性更好的特点,已经成为国际上SNP分析的主流方法之一。与APOE分型相关的SNP位点只有2个,因此,在本研究中KASP法的优势主要体现在大样本量分型检测时的检测效率和单位成本上。当一次需要检测上千甚至上万的样本时,KASP法可作为优先选择,比本研究中涉及的其他两种方法更加高效和节约成本。但目前利用KASP法进行APOE分型检测的研究尚不多见。
SNaPshot法是基于荧光标记单碱基延伸原理的分型技术,也称小测序,是中等通量SNP分型较为适用的一种方法[14-15]。它不受SNP位点多态特性限制,杂合、插入或缺失多态都可一个体系检测,一个反应可检测1~10个SNP位点,因此适用于检测两个SNP位点的APOE分型。该方法适合样本数目在几十到1 000的项目,相较于Sanger测序法可以极大地提高效率、降低成本,并且可以直接得到位点的碱基组成,结果更加直观,具有一定优势。
使用本研究建立的APOE分型的KASP法及SNaPshot法对已知分型的48份样本进行检测及重复试验,其结果完全相符。进一步对新收集的107份样本采用KASP法、SNaPshot法、Sanger测序法检测APOE分型,3种方法所得结果完全一致,共检出2/3型4份,2/4型1份,3/3型59份,3/4型40份,4/4型3份,其比例亦与中国大陆散发AD患者APOE分型比例相符[16]。因此,本研究所建立的方法,不仅被基因序列检测的“金标准”Sanger测序法验证,同时在KASP法和SNaPshot法结果之间也得到互相印证,再通过重复实验、分型比例比较等手段,进一步证实了其准确性、可靠性和可重复性。同时,本研究所使用的作为验证“金标准”的APOE分型的Sanger测序法,巧妙地将两个SNP位点设计在一个测序反应内,这也较常规针对两个位点分别扩增的方法简化了步骤,且节约了成本,在样本量不大,时效性要求不太高,或作为其他分型手段的验证时均可优先选择应用。
综上所述,本研究采用KASP法及SNaPshot法进行APOE分型检测,灵敏度和特异度高,与Sanger测序法相比,结果更直观和易判读,尤其在检测中、高通量样本时效率更高,成本更低,其中近年来新兴的KASP法更适合高通量检测,SNaPshot法更适合中等通量检测,这为不同的研究需求提供了更优方法选择。

