转基因玉米NK603转化体/zSSIIb内标基因二重微滴数字PCR方法的建立及应用
肖芳,李俊,王颢潜,翟杉杉,陈子言,高鸿飞,李允静,吴刚,张秀杰,武玉花
转基因玉米NK603转化体/内标基因二重微滴数字PCR方法的建立及应用
肖芳1,李俊1,王颢潜2,翟杉杉1,陈子言2,高鸿飞1,李允静1,吴刚1,张秀杰2,武玉花1
1中国农业科学院油料作物研究所/农业农村部油料作物生物学与遗传育种重点实验室,武汉 430062;2农业农村部科技发展中心,北京 100025
【目的】批准进口的转基因玉米NK603是中国转基因生物安全监管的主要对象。转基因安全监管需要标准物质和标准检测方法,建立转基因玉米NK603的数字PCR(dPCR)方法将为转基因玉米NK603的定量检测和标准物质研制提供精准测量技术。【方法】采用人工合成技术构建标准质粒分子pUC57-NK603;将NK603转化体与不同的玉米内标基因引物/探针一一组合,遴选与NK603转化体特异性PCR具有相同扩增能力的玉米内标基因PCR方法;设置二重微滴数字PCR(ddPCR)的退火温度梯度和引物/探针浓度梯度,优化二重ddPCR的反应体系和反应条件;用梯度稀释的标准质粒溶液作模板,考察二重ddPCR的检测极限、定量极限和动力学范围;将转基因玉米NK603种子粉末和非转基因玉米种子粉末混合,配制质量分数分别为100%、10%和6%的盲样,考察二重ddPCR的定量准确性。【结果】用标准质粒分子pUC57-NK603作为二重ddPCR的质控对照,通过考察二重ddPCR反应热图的微滴信号强度、阳性微滴与阴性微滴分辨率、雨滴数量、NK603转化体与内标基因拷贝数比值测量值与预期值的一致性,确定将NK603转化体特异性PCR方法与内标基因PCR方法组合,建立NK603/二重ddPCR方法。二重ddPCR反应体系中NK603转化体和内标基因的引物/探针浓度相同,均为400 nmol·L-1/200 nmol·L-1,在60℃退火延伸。NK603/二重ddPCR的检测极限是2 copies DNA模板,定量极限是48 copies DNA模板,动力学范围是10—60 000 copies DNA。应用NK603/二重ddPCR方法可准确定量玉米盲样中的NK603转化体含量,定值结果变异系数小于25%;dPCR定量结果与荧光定量PCR(qPCR)定量结果无显著差异,且具有更高的精确性。【结论】内标基因的选择会影响dPCR定量结果的准确性,在建立dPCR方法的过程中,要用具有准确量值的样品作为质控对照,评估内标准基因的适用性。以人工合成的标准质粒分子pUC57-NK603为质控对照,建立了NK603/二重ddPCR方法。应用建立的二重ddPCR定值方法进行标准物质的研制和定值,已成功研制出转基因玉米NK603有证标准物质。
转基因玉米NK603;二重数字PCR;标准质粒分子;内标基因;定量
0 引言
【研究意义】转基因检测是转基因生物安全监管和转基因标识制度实施的技术基础,需要标准化的检测方法和标准物质。目前,荧光定量PCR(qPCR)被认为是转基因定量检测的金标方法,国际标准化组织(international organization for standardization,ISO)及欧盟等发布的转基因产品定量检测标准都是基于qPCR技术[1]。研制转基因检测标准物质需要一种参考方法对标准物质进行定值,若用qPCR方法为标准物质进行定值,则会令定值过程陷入用依赖标准物质的方法为标准物质进行定值的悖论[2]。数字PCR(dPCR)是近几年发展起来的核酸精确定量技术,与实时荧光定量PCR技术相比,dPCR可不依赖标准物质和标准曲线对PCR反应中初始DNA模板进行绝对定量,是进行标准物质定值的潜在基准方法[3-4]。前期研究表明,在同一PCR管中同时进行外源基因和内标基因的二重PCR扩增,可消外源基因和内标基因的取样误差,与单重dPCR相比,二重dPCR的测量结果具有更高的准确性和重复性[5-6]。因此,建立和优化二重dPCR方法对转基因产品的定量检测和标准物质定值具有重要意义。【前人研究进展】dPCR技术已广泛应用于转基因检测、标准物质定值、临床诊断、法医科学、基因表达检测、拷贝数变异分析等领域[7-11]。为保证dPCR测量结果的准确性,需对dPCR操作进行严格的质量控制。2013年Huggett等[12]发表了dPCR MIQE指南,列出了发表dPCR研究应包含的最少信息,旨在促进dPCR试验程序的标准化。Yoo等[13]为了验证用dPCR定量核酸分子的准确性,同时使用流式细胞仪和dPCR仪测量质粒拷贝数浓度,2种方法的测量结果具有良好的一致性,表明dPCR可成为测量核酸拷贝数浓度的潜在参考方法。dPCR基于泊松分布原理对初始DNA模板进行定量,其定量结果主要受3个因素的影响,分别是阳性微滴与阴性微滴的准确识别、反应单元的体积和DNA模板的稀释倍数[14-15]。一些研究已经证实总反应单元数、阳性反应单元比率和反应单元的体积影响dPCR测量结果的准确性[16-17]。Corbisier等[16]用光学显微镜测定了QX100生成微滴的体积,发现测量体积比数据分析软件默认的体积小8%,反应单元体积的错估会直接影响测量结果。分析以上3个因素引入的测量不确定度并合成,有望将ddPCR结果溯源至国际单位(système international d'unités,SI)[14,18]。以dPCR作为定值技术,美国国家标准与技术研究所(national institute of standard and technology,NIST)研制出了用于巨细胞病毒检测(SRM 2366)、亨廷顿病检测(SRM 2393)等用途的核酸标准物质[19]。欧盟标准物质与测量研究所(institute of reference materials and measurement,IRMM)研制出了用于慢性骨髓性白血病检测的核酸标准物质(编号ERM-AD623)[20]。在转基因检测领域,dPCR已成功用于转基因产品定量检测和标准物质定值,如dPCR已用于转基因玉米DAS1507、NK603,转基因大豆MON87769、MON87708、MON87705、FG72、GTS40-3-2等的定量检测[21-23];在前期,中国也成功将dPCR技术用于转基因水稻TT51-1、G6H1、科丰6号、克螟稻[24-27],转基因玉米T25、MIR604,以及转基因大豆MON89788标准物质特性量值的测定[28-29],成功发布了多个有证标准物质。【本研究切入点】NIST研究认为,实现影响dPCR准确测量的3个要素的溯源,可将dPCR测量结果溯源到SI单位[14]。而准确识别阳性微滴和阴性微滴是影响定量结果准确性的关键因素,在读取反应单元信号时,若中等信号强度单元(下雨现象)太多会影响阳性反应单元和阴性反应单元的准确识别,进而影响定量结果的准确性。因此,在实际测量中,dPCR测量结果的准确性还依赖于引物/探针组合、反应体系、反应条件、操作程序等因素。转基因玉米NK603是中国批准进口的转基因品种,是中国生物安全监管的重要对象,急需研制NK603标准物质,为转基因玉米NK603的安全监管提供物质基础。虽然欧盟已经建立了NK603转化体特异性qPCR方法,但由于qPCR和dPCR的反应试剂不同,不能直接将qPCR方法平移到dPCR平台上,需对dPCR反应体系和反应条件等进行优化。【拟解决的关键问题】本研究拟以转基因玉米NK603为材料,将NK603转化体特异性引物/探针与不同的玉米内标基因引物/探针进行组合,比较测量结果,遴选出与NK603转化体特异性PCR方法组合的最适内标基因PCR方法,并对二重微滴数字PCR(ddPCR)的反应体系和反应条件进行优化,建立NK603转化体与内标基因的二重ddPCR,为转基因玉米NK603定量检测和标准物质定值提供准确的测量方法。
1 材料与方法
1.1 材料
转基因玉米NK603是美国孟山都公司研发的抗草铵膦转基因玉米,转基因玉米NK603及其受体对照种子由农业农村部科技发展中心提供。将转基因玉米NK603种子及其受体玉米种子研磨成粉末,用校准的天平按照质量比分别称量NK603玉米粉末和相应的受体粉末,混合均匀,配制成转基因玉米NK603质量分数为10%、6%的样品,用于二重ddPCR方法的测试。
1.2 引物及探针
NK603转化体特异性引物/探针来源于欧盟发布的标准[30],玉米内标基因、、、、的引物/探针来源于发表的文献或国家标准[31],其中,和是同一内标基因的2个方法。在NK603转化体特异性探针603-QP的5’端标记激发荧光染料6-FAM、3’端标记淬灭荧光染料BHQ1;在内标基因探针的5’端标记激发荧光染料HEX、3’端标记BHQ1。NK603转化体和玉米内标基因的引物/探针均由生工生物工程(上海)股份有限公司合成,序列信息详见表1。
1.3 基因组DNA的提取与纯化
称取质量分数分别为100%、10%和6%的NK603样品100—200 mg,用QIAGEN试剂盒DNeasy Plant Mini Kit(货号69106)提取纯化基因组DNA,用微量紫外可见分光光度计NanoDrop one测定DNA的浓度,用0.1×TE缓冲液将DNA浓度调整到约100 ng·μL-1备用。试验于2020年4月在转基因检测中心DNA提取室开展。
1.4 重组质粒的构建与酶切
将NK603转化体特异性片段和玉米内标基因(Accession No. X07535)、(Accession No. X07535)、(Accession No. AJ131373)、(Accession No. NC_024462)和(Accession No. X04050)的PCR扩增片段依次拼接,拼接出一个融合片段。委托北京擎科生物科技有限公司(武汉分公司)人工合成NK603转化体和玉米内标基因的融合序列,并克隆至载体pUC57中,构建标准质粒分子pUC57-NK603。用质粒提取试剂盒提取纯化重组质粒pUC57-NK603。用环形质粒分子作ddPCR的模板,会引发严重的下雨现象,导致难以准确区分阳性微滴和阴性微滴[15],需将质粒分子线性化。用限制性内切酶Ⅰ对质粒pUC57-NK603进行单酶切,酶切反应体系为Ⅰ 2 µL、质粒1 µg、10×buffer 5 µL,加ddH2O至50 µL;反应条件为37℃30 min;65℃20 min,反应终止。线性化的质粒pUC57-NK603保存于-20℃,用于二重ddPCR方法的建立。试验于2020年2月—3月在转基因检测中心分子生物学实验室开展。
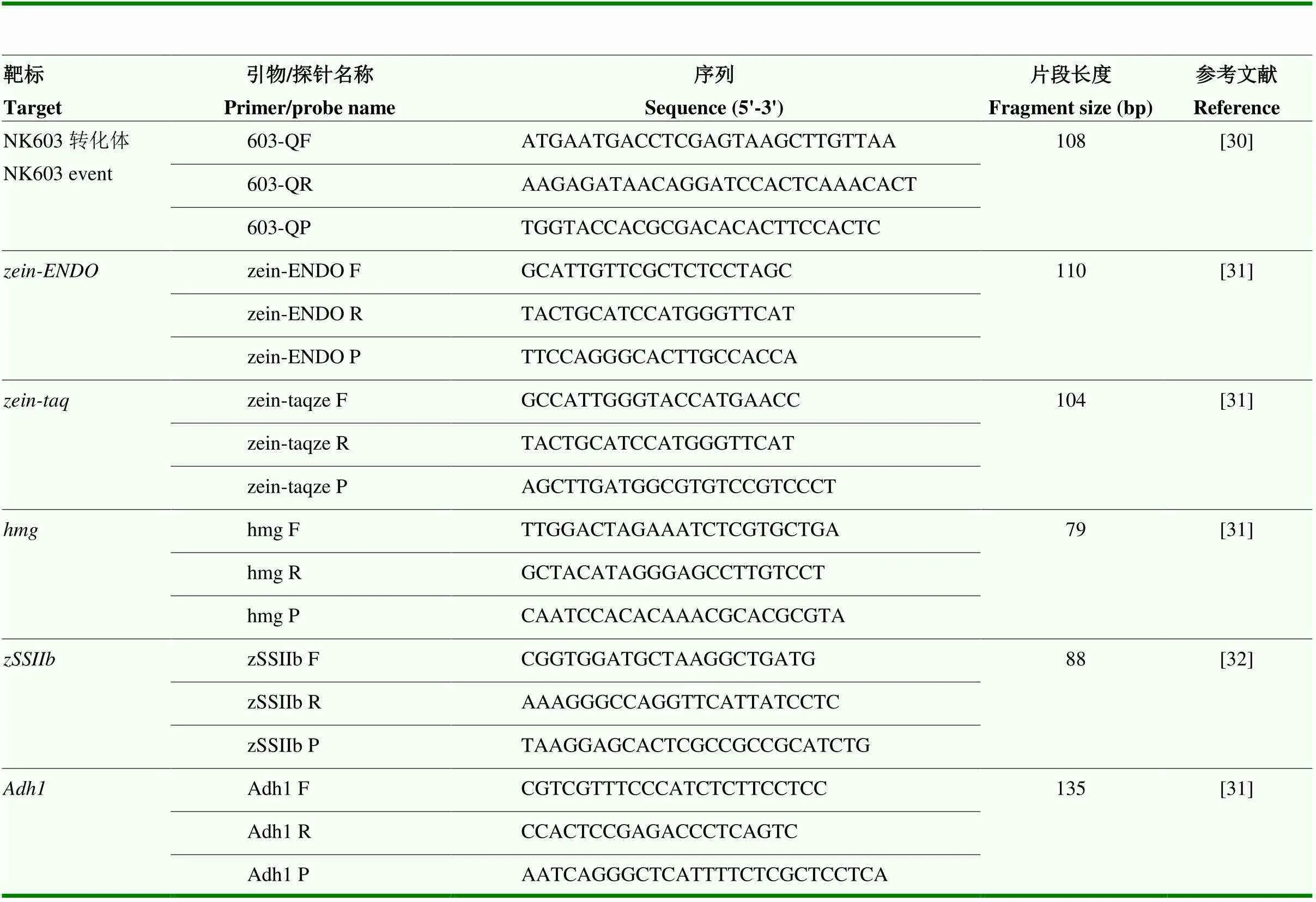
表1 PCR引物/探针信息
1.5 实时荧光定量PCR方法
在CFX96荧光定量PCR仪上进行qPCR,反应体系为1×Premix Ex Taq™、上、下游引物终浓度400 nmol·L-1、探针终浓度200 nmol·L-1、DNA模板浓度为100—1 000 000 copies·μL-1。反应程序为95℃1 min;95℃ 15 s,60℃60 s,40个循环。在退火延伸(60℃)时收集荧光信号。反应结束后用软件Bio-Rad CFX Manager 3.1分析数据。试验设置3个重复。以梯度稀释的标准质粒pUC57-NK603为校准样品进行NK603转化体和玉米内标基因的实时荧光PCR扩增,根据校准样品模板拷贝数的对数与Ct值之间的线性关系,分别绘制NK603转化体PCR和玉米内标基因PCR的标准曲线。将测试样品中NK603转化体和内标基因的Ct值分别代入标准曲线,计算测试样品中NK603转化体和内标基因的拷贝数。测试样品中转基因玉米NK603含量=NK603转化体拷贝数/内标基因拷贝数×100%。试验于2020年7月在转基因检测中心PCR准备室和PCR扩增室开展。
1.6 二重ddPCR方法
用QX200微滴数字PCR系统进行二重ddPCR反应,反应体系为20 µL,反应体系中各组分的浓度同qPCR。将配制的20 µL反应液转移至微滴生成卡中,然后加入70 µL微滴生成油,盖上胶垫,放入微滴生成仪(Bio-Rad,美国)中生成微滴,将40 µL微滴小心缓慢地转移至96孔板中,盖上铝膜,用热封仪封膜,在普通PCR仪C1000中进行扩增。反应结束后将96孔板放入微滴读取仪(Bio-Rad,美国)中读取微滴信号,用软件QuantaSoft Version 1.7.4.0917分析数据。试验设置3个重复。转基因玉米NK603含量=NK603转化体拷贝数/内标基因拷贝数×100%。
引物/探针浓度优化,反应体系包括10 µL 1× ddPCR Supermix、1 μL DNA模板、上下游引物、探针(NK603转化体和内标基因),加水至20 µL。设置4个引物/探针浓度梯度,终浓度依次为100/50、200/100、400/200和800 nmol·L-1/400 nmol·L-1。反应程序退火温度优化,为95℃10 min;94℃30 s,退火60 s(温度梯度设置为52℃、52.7℃、54℃、55.9℃、58.4℃、60.3℃、61.4℃和62℃),40个循环;98℃10 min,4℃保温。根据ddPCR反应热图确定二重ddPCR的最适引物/探针浓度和退火温度。
以测试样品的基因组DNA为模板进行二重ddPCR定量检测,通过软件QuantaSoft Version 1.7.4.0917直接读取测试样品单位体积DNA中NK603转化体和内标基因的拷贝数。测试样品中转基因玉米NK603含量的计算,同qPCR方法。试验于2020年4月—7月在转基因检测中心PCR准备室和PCR扩增室完成。
2 结果
2.1 构建标准质粒分子
将NK603转化体和玉米内标基因的PCR扩增序列依次拼接,拼接时在转化体特异性序列与内标基因之间插入一段20 bp的间隔序列,拼接出一个611 bp的核苷酸片段(图1-A),人工合成并克隆至载体pUC57中,构建了携带NK603转化体序列和4个玉米内标基因的标准质粒分子pUC57-NK603(图1-B)。经重测序验证,标准质粒分子pUC57-NK603携带一个拷贝的NK603转化体和各内标基因的靶标序列,NK603转化体和各玉米内标基因的拷贝数比值为1.0(转基因含量100%)。
2.2 玉米内标基因的比较
将NK603转化体引物/探针与不同的玉米内标基因、、、和引物/探针分别组合,以标准质粒分子pUC57-NK603为模板,进行二重ddPCR扩增。每个二重ddPCR组合设置了8个退火温度梯度(52℃、52.7℃、54℃、55.9℃、58.4℃、60.3℃、61.4℃和62℃)。二重ddPCR一维热图显示,NK603转化体引物/探针与不同的玉米内标基因引物/探针组合,NK603转化体阳性微滴的信号强度相似;在不同退火温度下NK603转化体阳性微滴的信号强度相近,但随着退火温度的降低,中等信号强度微滴数增加(下雨现象加重)(图2-A—E)。NK603/、NK603/二重ddPCR中,在任何退火温度下都不能清晰地区分和的阴性微滴和阳性微滴(图2-A和图2-B);NK603/二重ddPCR中,虽能区分的阳性微滴和阴性微滴,但“下雨”现象相对严重(图2-C);NK603/、NK603/二重ddPCR中,在所有温度下都能清晰地区分和的阳性微滴和阴性微滴,微滴的信号强度也比较稳定,但与相比,在8个不同退火温度下,的阳性微滴和阴性微滴的区分度更高、信号更强、中等信号强度的雨滴相对更少(图2-D和图2-E)。因此,初步选择作为二重ddPCR的候选内标基因。
在不同退火温度下,统计NK603/、NK603/和NK603/二重ddPCR测量的NK603转化体与内标基因的拷贝数比值。NK603/二重ddPCR测量的拷贝数比值为0.92—0.94,低于理论值1.0;NK603/二重ddPCR测量的拷贝数比值为1.00—1.01,接近理论值1.0;NK603/二重ddPCR测量的拷贝数比值为1.04—1.06,高于理论值1.0(图3)。比较3个二重ddPCR组合,NK603/二重ddPCR的拷贝数测量值最接近理论值1.0,表明玉米内标基因与NK603转化体在PCR反应中具有相似的扩增能力,因此,确定用作为NK603转化体定量检测的内标基因,并与NK603转化体组合,建立NK603/二重ddPCR方法。NK603/二重ddPCR的NK603转化体和内标基因引物/探针均对退火温度不敏感,在不同的退火温度下,拷贝数比值测量值接近,但NK603转化体引物/探针在退火温度低于55.9℃时,中等信号强度微滴明显增多(图2-D)。58.4℃—62℃均是NK603/二重ddPCR的适宜退火温度,选择60℃作为NK603/二重ddPCR的最适退火温度。在60.3℃退火温度下,NK603/二重ddPCR的二维热图中有4个清晰的微滴群,对应阴性微滴、FAM荧光微滴、HEX荧光微滴和双荧光微滴(图2-F)。通过考察NK603转化体和内标基因的微滴信号强度、阳性微滴与阴性微滴的分辨率、中等信号强度雨滴数量、以及NK603转化体与内标基因拷贝数的比值,确定能与NK603转化体组合进行二重ddPCR扩增的最适内标基因为,NK603/二重ddPCR的最适退火温度为60℃。
2.3 NK603/zSSIIb二重ddPCR引物探针浓度优化
引物/探针浓度会影响ddPCR微滴信号强度、雨滴数量及测量值的准确性,通过设置引物/探针浓度梯度,遴选出最佳的引物/探针浓度。设计了2种模式的引物/探针浓度,第一种模式中NK603转化体和内标基因的引物/探针浓度相同,设置了4个浓度梯度,分别为100/50、200/100、400/200和800 nmol·L-1/ 400 nmol·L-1;第二种模式中转化体和内标基因的引物/探针浓度不同,转化体和内标基因的引物/探针浓度分别为400/200、200 nmol·L-1/100 nmol·L-1,200/100、400 nmol·L-1/200 nmol·L-1,400/200、800 nmol·L-1/400 nmol·L-1,800/400、400 nmol·L-1/200 nmol·L-1。二重ddPCR一维热图如图4所示,2种模式都可以根据扩增热图清晰的区分NK603转化体和内标基因的阳性微滴和阴性微滴,NK603转化体和内标基因的微滴信号强度都随着引物/探针浓度的升高而升高,但当引物/探针浓度升至800 nmol·L-1/400 nmol·L-1,阳性微滴的信号强度波动区间变大,且雨滴增多(图5-A4、图5-A8、图5-B4和图5-B7)。统计不同的引物探针浓度下NK603转化体与内标基因的拷贝数比值,发现拷贝数比值的测量值在0.98—1.04范围内波动,均接近理论值(图4-C)。当NK603转化体和内标基因的引物/探针浓度均为400 nmol·L-1/200 nmol·L-1,或分别为200/100、400 nmol·L-1/200 nmol·L-1时,测得的NK603转化体与内标基因的拷贝数比值更接近理论值1.0(图4-C)。为方便反应体系配置,选择400 nmol·L-1/200 nmol·L-1作为NK603/双重ddPCR的引物探针浓度,NK603转化体和内标基因的引物/探针浓度相同,且与qPCR反应体系一致。
2.4 NK603/zSSIIb二重ddPCR方法检测限及线性范围
用ddH2O梯度稀释线性化的质粒pUC57-NK603到3×105、6×104、3×104、6×103、1.2×103、2.4×102、48、10和2 copies·μL-1,测试NK603/双重ddPCR的线性动力学范围及检测极限。结果显示,在空白对照中NK603转化体和均未产生阳性微滴;当模板拷贝数浓度为3×105copies·μL-1时,NK603转化体和全部微滴均为阳性,超过了ddPCR的最高检测极限;当模板拷贝数浓度低至2 copies·μL-1时,所有反应中均有NK603转化体和的阳性信号,但拷贝数浓度的测量值显著偏离预期值,变异系数(relative standard deviation,RSD)大于25%,测量偏差(Bias)超过±25%,确定NK603/双重ddPCR的检测限为2 copies DNA;当模板浓度为48 copies·μL-1时,测量值的变异系数小于25%,测量偏差在±25%范围内,确定NK603/双重ddPCR的定量限为48 copies DNA。根据质粒模板预期浓度和测量浓度间的对应关系绘制NK603转化体和的回归曲线,NK603转化体回归曲线的决定系数2为0.999,内标基因回归曲线的决定系数2为0.9989,在模板浓度为10—60 000 copies·μL-1的范围内NK603/二重ddPCR测量的模板拷贝数与理论拷贝数间具有良好的线性关系(图5)。
2.5 NK603/zSSIIb二重ddPCR方法应用
用NK603/二重ddPCR定量检测转基因玉米NK603粉末质量分数分别为100%(S1)、10%(S2)和6%(S3)的盲样。3个盲样转基因含量的测量平均值分别为51.05%、4.82%和3.07%,定量结果的变异系数分别为1.91%、2.28%和0.50%(表2),都小于25%,符合欧盟规定的转基因产品定量方法的性能要求。3个盲样的实测转基因DNA含量约为预期质量分数的一半,定量结果进一步表明转基因玉米NK603种子为杂合体。

表2 二重ddPCR和qPCR检测盲样的转基因含量
用梯度稀释的标准质粒pUC57-NK603为标准样品分别绘制NK603转化体和内标基因的标准曲线,采用qPCR定量盲样S1、S2和S3的转基因含量。3个盲样转基因含量的测量平均值分别为55.55%、5.09%和3.59%,定量结果的变异系数分别为6.83%、4.17%和10.68%,均小于25%(表2)。比较二重ddPCR和qPCR的定量结果,3个盲样2组测量结果的值均大于0.05,表明这两种定量方法的定量结果间无显著差异。定量同一个盲样,二重ddPCR定量结果的变异系数小于qPCR,表明二重ddPCR定量结果具有更好的准确性。
3 讨论
在转基因检测领域,dPCR已成功用于转基因产品的定量检测和标准物质定值[3],与qPCR相比,dPCR不需要用标准物质绘制标准曲线,但这并不意味着dPCR检测不需要设置质控对照[12]。转基因含量通常表征为转基因DNA量与总DNA量的比值,转基因DNA量通过测量外源基因或转化体特异性序列拷贝数确定,总DNA量通过测量内标基因拷贝数确定。若要准确的测量样品中的转基因含量,在dPCR方法的建立和应用过程中,必须确保外源基因和内标基因具有相同的扩增能力,因此,dPCR虽然不依赖标准物质进行定量,但在dPCR反应时,必须设置至少1个有准确量值的样品为质控对照,而理论转基因含量为100%的纯合体DNA是质控对照的最佳选择。
玉米种子由种皮、胚和胚乳组成,种皮为二倍体,DNA全部来源于母本;胚乳占种子重量的80%—85%,为三倍体,2/3 DNA来源于母本,1/3来源于父本;胚为二倍体,一半DNA来源于母本,一半DNA来源于父本[33]。转基因玉米种子的转基因DNA含量与其基因型相关,纯合种子的转基因DNA含量理论上为100%,杂合种子的转基因含量则受到转基因亲本来源、组织倍性等因素的影响,其转基因含量不确定,理论值为33.3%—66.6%[34]。鉴定转基因玉米NK603种子的基因型,发现研发者提供的NK603种子均为杂交种(数据未给出)。本研究在建立转基因玉米NK603二重ddPCR方法过程中,必须要保证NK603转化体具有与玉米内标基因相同的扩增能力,由于转基因玉米NK603种子均为杂交种,其基因组DNA没有确定的转基因含量理论值,致使方法建立过程中缺少有准确量值的质控对照。为了获取到有准确转基因DNA含量的质控样品,本研究通过人工合成的方式,构建了携带NK603转化体和4个玉米内标基因扩增序列的标准质粒分子pUC57-NK603,转化体序列与内标基因的拷贝数比值是1.0。试验结果证明,质粒分子pUC57-NK603是一个合格的质控对照样品,这意味着在后续建立dPCR方法过程中,若缺乏合适的质控对照,可通过人工合成的方式快速构建质粒分子作为质控对照。
采用dPCR技术进行转基因成分定量检测,内标基因的选择会影响定量结果的准确性,要优先选择单拷贝内标基因,应用多拷贝内标基因会导致转基因含量的低估[35]。目前用于转基因检测的玉米内标基因有4个,涉及5种方法,分别为、、、、[29-30]。为评估各内标基因PCR方法的适用性,将NK603转化体与各内标基因PCR方法一一组合,以标准质粒分子pUC57-NK603为DNA模板进行二重ddPCR扩增,根据扩增热图,发现难以区分和的阳性微滴和阴性微滴,和的PCR扩增能力与NK603转化体PCR不一致,NK603/二重ddPCR会低估样品中NK603转化体含量,而NK603/二重ddPCR则会高估样品中NK603转化体含量,最终选择与NK603组合,建立NK603/二重ddPCR方法。研究结果进一步证实,作物内标基因的选择会影响转基因定量结果的准确性。
4 结论
以人工合成的标准质粒分子pUC57-NK603为质控对照,成功建立了NK603/二重ddPCR方法,其定量结果比qPCR具有更高的精确性。应用NK603/二重ddPCR方法为标准物质定值,中国已成功研制出转基因玉米NK603有证标准物质(有证标准物质编号GBW(E)100684、GBW(E)100685、GBW(E)100686)。
[1] Laura B, Marc H V d B, Marco M, Enrico B, Alexandre P. GMOMETHODS: the European Union database of reference methods for GMO analysis. Journal of AOAC International, 2012, 95(6): 1713-1719.
[2] Wu Y H, Li J, Li X Y, Zhai S S, Gao H F, Li Y J, Zhang X J, Wu G. Development and strategy of reference materials for the DNA-based detection of genetically modified organisms. Analytical and Bioanalytical Chemistry, 2019, 411(9): 1729-1744.
[3] Quan P L, Sauzade M, Brouzes E. dPCR: A technology review. Sensors, 2018, 18(4): 1271.
[4] Bhat S, Curach N, Mostyn T, Bains G S, Griffiths K R, Emslie K R. Comparison of methods for accurate quantification of DNA mass concentration with traceability to the international system of units. Analytical chemistry, 2010, 82(17): 7185-7192.
[5] Bogožalec Košir A, Demšar T, Štebih D, Žel J, Milavec M. Digital PCR as an effective tool for GMO quantification in complex matrices. Food Chemistry, 2019, 294(1): 73-78.
[6] Whale A S, Cowen S, Foy C A, Huggett J F. Methods for applying accurate digital PCR analysis on low copy DNA samples. PLoS One, 2013, 8(3): e58177.
[7] Cottenet G, Blancpain C, Chuah P F. Performance assessment of digital PCR for the quantification of GM-maize and GM-soya events. Analytical and Bioanalytical Chemistry, 2019, 411: 2461-2469.
[8] Köppel R, Bucher T, Frei A, Waiblinger H U. Droplet digital PCR versus multiplex real-time PCR method for the detection and quantification of DNA from the four transgenic soy traits MON87769, MON87708, MON87705 and FG72, and lectin. European Food Research and Technology, 2015, 241(4): 521-527.
[9] Cao Y, Raith M R, Griffith J F. Droplet digital PCR for simultaneous quantification of general and human-associated fecal indicators for water quality assessment. Water research, 2015, 70: 337-349.
[10] Devonshire A S, Honeyborne I, Gutteridge A, Whale A S, Nixon G, Wilson P, Jones G, McHugh T D, Foy C A, Huggett J F. Highly reproducible absolute quantification of Mycobacterium tuberculosis complex by digital PCR. Analytical chemistry, 2015, 87(7): 3706-3713.
[11] White H, Deprez L, Corbisier P, Hall V, Lin F, Mazoua S, Trapmann S, Aggerholm A, Andrikovics H, Akiki S, Barbany G, Boeckx N, Bench A, Catherwood M, Cayuela J-M, Chudleigh S, Clench T, Colomer D, Daraio F, Dulucq S, Farrugia J, Fletcher L, Foroni L, Ganderton R, Gerrard G, Gineikienė E, Hayette S, El Housni H, Izzo B, Jansson M, Johnels P, Jurcek T, Kairisto V, Kizilors A, Kim D-W, Lange T, Lion T, Polakova K M, Martinelli G, McCarron S, Merle P A, Milner B, Mitterbauer-Hohendanner G, Nagar M, Nickless G. A certified plasmid reference material for the standardisation of BCR-ABL1 mRNA quantification by real-time quantitative PCR. Leukemia, 2015, 29(2): 369-376.
[12] Huggett J F, Foy C A, Benes V, Emslie K, Garson J A, Haynes R, Hellemans J, Kubista M, Mueller R D, Nolan T, Pfaffl M W, Shipley G L, Vandesompele J, Wittwer C T, Bustin S A. The digital MIQE guidelines: Minimum information for publication of quantitative digital PCR experiments. Clinical chemistry, 2013, 59(6): 892-902.
[13] Yoo H B, Park S R, Dong L H, Wang J, Sui Z W, Pavšič J, Milavec M, Akgoz M, Mozioğlu E, Corbisier P, Janka M, Cosme B, de V Cavalcante J J, Flatshart R B, Burke D, Forbes-Smith M, McLaughlin J, Emslie K, Whale A S, Huggett J F, Parkes H, Kline M C, Harenza J L, Vallone P M. International comparison of enumeration-based quantification of DNA copy-concentration using flow cytometric counting and digital polymerase chain reaction. Analytical chemistry, 2016, 88(24): 12169-12176.
[14] Kline M C, Duewer D L. Evaluating droplet digital polymerase chain reaction for the quantification of human genomic DNA: Lifting the traceability fog. Analytical chemistry, 2017, 89(8): 4648-4654.
[15] Demeke T, Dobnik D. Critical assessment of digital PCR for the detection and quantification of genetically modified organisms. Analytical and Bioanalytical Chemistry, 2018, 410(17): 4039-4050.
[16] Corbisier P, Pinheiro L, Mazoua S, Kortekaas A M, Chung P Y J, Gerganova T, Roebben G, Emons H, Emslie K. DNA copy number concentration measured by digital and droplet digital quantitative PCR using certified reference materials. Analytical and bioanalytical chemistry, 2015, 407(7): 1831-1840.
[17] Dong L H, Meng Y, Sui Z W, Wang J, Wu L Q, Fu B Q. Comparison of four digital PCR platforms for accurate quantification of DNA copy number of a certified plasmid DNA reference material. Scientific reports, 2015, 5: 13174.
[18] Duewer D L, Kline M C, Romsos E L, Toman B. Evaluating droplet digital PCR for the quantification of human genomic DNA: converting copies per nanoliter to nanograms nuclear DNA per microliter. Analytical and bioanalytical chemistry, 2018, 410(12): 2879-2887.
[19] Haynes R J, Kline M C, Toman B, Scott C, Wallace P, Butler J M, Holden M J. Standard reference material 2366 for measurement of human cytomegalovirus DNA. The Journal of Molecular Diagnostics, 2013, 15(2): 177-185.
[20] Deprez L, Mazoua S, Corbisier P, Trapmann S, Schimmel H, White H, Cross N, Emons H. The certification of the copy number concentration of solutions of plasmid DNA containing a BCR-ABL b3a2 transcript fragment, Certified Reference Materials: ERM®-AD623a, ERM®-AD623b, ERM®-AD623c, ERM®-AD623d, ERM®-AD623e, ERM®-AD623f. Publications Office of the European Union, 2012, ISSN: 1831-9424.
[21] Dobnik D, Spilsberg B, Bogožalec K A, Holst-Jensen A, Žel J. Multiplex quantification of 12 European Union authorized genetically modified maize lines with droplet digital polymerase chain reaction. Analytical chemistry, 2015, 87(16): 8218-8226.
[22] Wan J R, Song L, Wu, Y L, Brzoska P, Keys D, Chen C F, Valliyodan B B, Shannon J G, Nguyen H T. Application of digital PCR in the analysis of transgenic soybean plants. Advances in Bioscience and Biotechnology, 2016, 7(10): 403-417.
[23] Košir A B, Spilsberg B, Holst-Jensen A, Žel J, Dobnik D. Development and inter-laboratory assessment of droplet digital PCR assays for multiplex quantification of 15 genetically modified soybean lines. Scientific reports, 2017, 7(1): 8601.
[24] Jiang Y, Yang H, Quan S, Liu Y N, Shen P, Yang L T.Development of certified matrix-based reference material of genetically modified rice event TT51-1 for real-time PCR quantification. Analytical and bioanalytical chemistry, 2015, 407(22): 6731-6739.
[25] Yang Y, Li L, Yang H, Li X Y, Zhang X J, Xu J F, Zhang D B, Jin W J, Yang L T. Development of certified matrix-based reference material as a calibrator for genetically modified rice G6H1 analysis. Journal of agricultural and food chemistry, 2018, 66(14): 3708-3715.
[26] Li J, Li L, Zhang L, Zhang X J, Li X Y, Zhai S S, Gao H F, Li Y J, Wu G, Wu Y H. Development of a certified genomic DNA reference material for detection and quantification of genetically modified rice KMD. Analytical and bioanalytical chemistry, 2020, 412(25): 7007-7016.
[27] Li J, Zhang L, Li L, Li X Y, Zhang X J, Zhai S S, Gao H F, Li Y J, Wu G, Wu Y H. Development of genomic DNA certified reference materials for genetically modified rice Kefeng 6. ACS omega, 2020, 5(34): 21602-21609.
[28] 李俊, 李夏莹, 李亮, 宋贵文, 沈平, 张丽, 翟杉杉, 柳方方, 吴刚, 张秀杰, 武玉花. 转基因大豆MON89788 纯品粉末标准物质的研制及定值. 农业生物技术学报, 2020, 28(6): 1084-1095.
LI J, LI X Y, LI L, SONG G W, SHEN P, ZHANG L, ZHAI S S, LIU F F, WU G, ZHANG X J, WU Y H. Development and characterization of pure matrix reference materials for detection of transgenic soybean () MON89788. Journal of Agricultural Biotechnology, 2020, 28(6): 1084-1095. (in Chinese)
[29] 李俊, 李夏莹, 李亮, 宋贵文, 沈平, 张丽, 翟杉杉, 柳方方, 吴刚, 张秀杰, 武玉花. 转基因玉米MIR604 基体标准物质研制. 作物学报, 2020, 46(4): 473-483.
LI J, LI X Y, LI L, SONG G W, SHEN P, ZHANG L, ZHAI S S, LIU F F, WU G, ZHANG X J, WU Y H. Development of genetically modified maize MIR604 matrix reference materials. Acta Agronomica Sinica, 2020, 46(4): 473-483. (in Chinese)
[30] Nielsen C R, Berdal K G, Holst-Jensen A. Characterization of the integration site and development of an event-specific real-time PCR assay for NK603 maize from a low starting copy number. European Food Research and Technology, 2004, 219(4): 421-427.
[31] Papazova N, Zhang D, Gruden K, Vojvoda J, Yang L T, Gašparič M B, Blejec A, Fouilloux S, Loose M, Taverniers I. Evaluation of the reliability of maize reference assays for GMO quantification. Analytical and Bioanalytical Chemistry, 2010, 396(6): 2189-2201.
[32] 杨立桃, 刘信, 张大兵, 沈平, 郭金超, 金芜军. 农业部1861号公告-3-2012 转基因植物及其产品成分检测玉米内标准基因定性PCR方法.北京: 中国农业出版社, 2012.
YANG L T, LIU X, ZHANG D B, SHEN P, GUO J C, JIN W J. Announcement by the Ministry of Agriculture No.1861-3-2012 Detection of genetically modified plants and derived products Target-taxon-specific qualitative PCR method for Maize. Beijing: China Agriculture Press, 2012. (in Chinese)
[33] Chaouachi M, Bérard A, Saïd K. Relative quantification in seed GMO analysis: state of art and bottlenecks. Transgenic Research, 2013, 22(3): 461-476.
[34] Holst-Jensen A, De Loose M, Van den Eede G. Coherence between legal requirements and approaches for detection of genetically modified organisms (GMO) and their derived products. Journal of agricultural and food chemistry, 2006, 54(8): 2799-2809.
[35] Demeke T, Eng M. Effect of endogenous reference genes on digital PCR assessment of genetically engineered canola events. Biomolecular Detection and Quantification, 2018, 15: 24-29.
Establishment and application of a duplex ddPCR method to quantify the NK603/copy number ratio in transgenic maize NK603
XIAO Fang1, LI Jun1, WANG Haoqian2, ZHAI Shanshan1, CHEN Ziyan2, GAO Hongfei1, LI Yunjing1, WU Gang1, ZHANG Xiujie2, WU Yuhua1
1Oil Crops Research Institute, Chinese Academy of Agricultural Sciences/Key Laboratory of Biology and Genetic Improvement Oil Crops Ministry of Agriculture and Rural Affairs, Wuhan 430062;2Development Center of Science and Technology, Ministry of Agriculture and Rural Affairs of People’s Republic of China, Beijing 100025
【Objective】Transgenic maize NK603 approved for import is an important target of genetically modified organism (GMO) regulation in China. The implementation of GMO regulation requires reference materials (RMs) and standardized detection methods. Establishment of a duplex droplet digital PCR (ddPCR) would provide accurate measurement technology for quantification of NK603 event and development of NK603 RMs. 【Method】 A standard plasmid molecule pUC57-NK603 was constructed by DNA synthetic technique; the primer/probe set of NK603 event was combined with different maize reference genes one by one to select reference gene PCR assay with identical amplification ability to NK603 event-specific PCR assay; main reaction parameters, such as annealing temperature and primer/probe concentration,were optimized in the course of establishing duplex ddPCR; the standard plasmid solution was serially diluted to investigate the limit of detection (LOD), limit of quantification (LOQ) and dynamic range of the duplex ddPCR assay; blinded samples with mass fractions of 100%, 10% and 6% were prepared by mixing NK603 powder with non-GM counterpart, to evaluate the accuracy of quantitative results of the duplex ddPCR. 【Result】The reference genewas determined to combine with NK603 event to establish the NK603/duplex ddPCR method with the standard plasmid molecule pUC57-NK603 as a quality control after analyzing the fluorescence amplitude of positive droplets, separation between positive and negative droplets, raindrop number, and consistency between measured copy number ratio and expected copy number ratio of NK603 event to reference gene. The primer/probe concentration was optimized to be 400 nmol·L-1/200 nmol·L-1for both NK603 event andgene, and the annealing temperature was determined to be 60°C. The LOD of NK603/duplex ddPCR was estimated to be 2 copies of DNA template, the LOQ was 48 copies of DNA template, both NK603 assay andassay showed good linearity between measured values and theoretical values over the dynamic range from 10 to 60 000 copies of DNA template. The NK603/duplex ddPCR achieved accurate quantitative results of NK603 content in blind maize samples with less than 25% of coefficient of variation; the quantitative results of ddPCR were not significantly different from those of real-time quantitative PCR (qPCR), moreover, the duplex ddPCR showed an advantage over qPCR in term of precision. 【Conclusion】 The selection of reference genes affects the accuracy of quantitative results by ddPCR. Establishment of ddPCR methods should use samples with accurate GMO content as quality controls to evaluate the applicability of reference genes. The NK603/duplex ddPCR method was successfully established using the synthetic standard plasmid molecule pUC57-NK603 as a quality control. NK603 certified reference materials (CRMs) have been successfully developed in China by applying the established NK603/duplex ddPCR assay to characterize the property values.
transgenic maize NK603; duplex droplet digital PCR; reference plasmid molecule; reference gene; quantification
2021-04-28;
2021-06-16
转基因生物新品种培育专项(2016ZX08012003)
肖芳,E-mail:xf_hzau@163.com。通信作者张秀杰,E-mail:zhxj7410@sina.com。通信作者武玉花,E-mail:wuyuhua@oilcrops.cn
(责任编辑 李莉)

