OsRhoGDI2过表达转基因水稻的筛选鉴定及外源基因拷贝数的初步分析
杜京尧 尚飞 王高华
摘要:水稻Rho GDP解离抑制基因OsRhoGDI2是从幼穗中分离出的功能未知基因。为鉴定该基因的功能,笔者所在实验室前期构建了植物过表达载体pCAMBIA1302-OsRhoGDI2-GFP,并对水稻进行了遗传转化。对OsRhoGDI2过表达转基因水稻T2代进行筛选和鉴定,采用PCR技术鉴定转基因植株,采用半定量RT-PCR和实时荧光定量PCR检测OsRhoGDI2在转基因水稻中的表达水平,结果显示,其中6个株系为过表达转基因植株,OsRhoGDI2表达水平上调1.69~13.35倍。为检测外源基因在转基因水稻中的拷贝数,分别以蔗糖磷酸合成酶基因SPS和潮霉素抗性基因HYG为内参基因和标记基因,采用实时荧光定量PCR(qPCR)技术结合内参基因和标记基因的标准曲线进行分析,结果显示在所检测的6个转基因株系中,外源基因的拷贝数均为1,提示已经获得稳定遗传的OsRhoGDI2过表达转基因水稻,为后续OsRhoGDI2基因的功能研究奠定基础。
关键词:OsRhoGDI2;转基因水稻;表达分析;拷贝数;外源基因
中图分类号: S511.03 文献标志码: A 文章编号:1002-1302(2019)14-0050-04
植物Rho/Rac也称为Rop(Rho of plant),是植物中唯一与信号转导有关的小GTP结合蛋白,功能涉及细胞极性生长[1-3]、肌动蛋白细胞骨架重组[4-5]、次生壁形成[6]、分生组织信号传递[7]以及抗逆和激素应答[8-11]等。作为信号通路的分子开关,Rho蛋白通过与靶蛋白的相互作用,实现对细胞生理活动的调控和影响,已明确的一类相互作用蛋白是Rho GDP解离抑制因子(Rho GDP dissociation inhibitors,RhoGDIs),功能是调节Rho的胞内定位,通过阻止GTP与Rho的结合,抑制Rho的活化,因而是Rho的负调控因子[12-14]。但是目前对植物RhoGDIs研究的报道较少,虽有从拟南芥、烟草、水稻等植物中筛选到RhoGDIs的报道[15-17],但是对其在植物生长发育等过程中生物学功能的研究非常有限。Carol等对拟南芥突变体scn1的研究发现,一种拟南芥的RhoGDI基因SCN1/AtrhoGDI1通过控制局部RHD2/AtrbohC NADPH氧化酶的激活,调控根毛细胞的生长[18];Wu等的研究发现,拟南芥AtRhoGDI1通过ROP信号通路,介导对幼苗和叶表皮细胞形态建成的调节,而且AtRhoGDI1被CPK3磷酸化是执行其功能的前提[19],但是至今尚未见水稻RhoGDIs基因功能的报道。
水稻Rho GDP解离抑制基因OsRhoGDI2是通过酵母双杂交筛选,从雌雄蕊形成期水稻幼穗中分离的功能未知基因。對该基因编码蛋白的生物信息学分析和亚细胞定位检测发现,该基因编码蛋白在活细胞中广泛分布[20]。对OsRhoGDI2基因启动子转基因功能鉴定的研究表明,该基因2 100 bp的上游调控序列即可以启动GUS在颖花、花药和花粉中的特异表达,该基因的表达受到生长素(IAA)等多种激素的胁迫和诱导[21]。为了进一步鉴定OsRhoGDI2的功能,本研究基于实验室的前期工作,对OsRhoGDI2过表达转基因水稻T2代进行筛选和鉴定,分别采用PCR、RT-PCR和qRT-PCR技术在DNA和RNA水平上进行检测,并通过qPCR技术鉴定OsRhoGDI2在转基因株系中的拷贝数,旨在为后续分析OsRhoGDI2基因的生物学功能奠定基础。
1 材料与方法
1.1 试验材料
1.1.1 植物材料 水稻粳稻品种日本晴(Oryza sativa L.japonica.cv.Nipponbare),简称WT,由山东省农业科学院作物与分子育种实验室提供;OsRhoGDI2过表达转基因水稻,简称OE-OsRhoGDI2,由笔者所在实验室前期构建,已筛选获得T1代种子,遗传背景为日本晴。
1.1.2 载体 植物过表达载体pCAMBIA1302-OsRhoGDI2-GFP由笔者所在实验室前期构建。
1.1.3 主要试剂 总RNA提取试剂RNAiso Plus、PrimeSTAR HS DNA Polymerase、PrimeScriptTM RT reagent Kit with gDNA Eraser (Perfect Real Time)、SYBR Premix Ex TaqTM Ⅱ购自宝生物工程(大连)有限公司,2×Es Taq MasterMix (Dye)购自北京康为世纪生物科技有限公司。
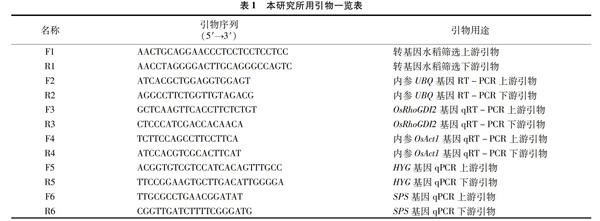
1.1.4 引物设计 本试验所用引物(表1)均由苏州金维智生物科技有限公司合成。
1.2 试验方法
1.2.1 水稻取材 选取适量的对照(WT)水稻种子和OE-OsRhoGDI2水稻T1代种子,2016年5月1日播种于河南师范大学试验园地(113.90°E、35.32°N)。水稻生长至 20 d (幼苗期)、90 d(抽穗期)时,分别取其植株叶片,液氮冷冻处理,-80 ℃ 保存备用。
1.2.2 OsRhoGDI2转基因植株的筛选 取幼苗期水稻嫩叶,采用十六烷基三甲基三甲基溴化铵(CTAB)法提取基因组DNA。以F1/R1为引物,基因组DNA为模板进行PCR反应,按照2×Es Taq MasterMix说明书配制反应体系(10 μL):2×Taq Master Mix 5.0 μL、F1(10 μmol/L)0.4 μL、R1(10 μmol/L)0.4 μL、基因组DNA 0.2 μL 和无菌水4.0 μL。PCR反应条件为94 ℃预变性 2 min;94 ℃变性30 s,58 ℃退火30 s,72 ℃延伸1 min,30个循环;72 ℃延伸2 min。扩增产物采用1%琼脂糖凝胶电泳检测。
1.2.3 OsRhoGDI2基因在转基因水稻中表达水平的检测
1.2.3.1 总RNA的提取和cDNA的制备 取抽穗期水稻叶片,根据宝生物工程(大连)有限公司RNAiso Plus试剂说明书,分别提取对照(WT)水稻和OE-OsRhoGDI2水稻T2代幼苗叶片总RNA,然后按照PrimeScriptTM RT reagent Kit with gDNA Eraser (Perfect Real Time)试剂盒说明书进行反转录,制备cDNA。
1.2.3.2 半定量RT-PCR 以“1.2.3.1”节获得的cDNA为模板,按照PrimeSTAR HS DNA Polymerase说明书配制半定量RT-PCR反应体系(50 μL),其中内参基因UBQ的PCR扩增体系为5×PrimeSTAR Buffer(Mg2+ Plus)10.0 μL、dNTP(2.5 mmol/L)4.0 μL、UBQ引物F2(10 μmol/L)1.0 μL、UBQ引物R2(10 μmol/L)1.0 μL、PrimeSTAR HS DNA Polymerase 0.5 μL、cDNA模板2.0 μL和无菌水31.5 μL。PCR反应条件为98 ℃变性30 s,55 ℃退火15 s,72 ℃延伸1 min,30个循环。OsRhoGDI2的扩增体系引物采用F1/R1,其余同UBQ的PCR扩增体系。PCR反应退火温度为58 ℃,其余参数同UBQ的PCR反应条件。扩增产物经1%琼脂糖凝胶电泳检测。
1.2.3.3 实时荧光定量PCR 取“1.2.3.1”节制备的cDNA为模板,以F3/R3为引物进行OsRhoGDI2的qRT-PCR反应,以水稻OsAct1基因为内参,按照SYBR Premix Ex TaqTM Ⅱ说明书配制反应体系(20 μL):2×SYBR Premix Ex Taq 10.0 μL、PCR Forward primer(10 μmol/L)0.8 μL、PCR Reverse primer(10 μmol/L)0.8 μL、cDNA 2.0 μL、滅菌水 6.4 μL。采用2步法进行PCR扩增:第1步95 ℃预变性 30 s;第2步PCR反应95 ℃变性5 s,60 ℃延伸30 s,40个循环。
每组试验做3次重复,利用Roche LightCycler 96自带软件LightCycler 96 v1.1进行数据分析,以2-ΔΔCT法[22]分析OsRhoGDI2基因表达水平。
1.2.4 OsRhoGDI2基因在转基因水稻中拷贝数的鉴定 HYG基因标准曲线的制作参考杨立桃等的方法[23],以含有HYG基因的pCAMBIA1302-OsRhoGDI2-GFP质粒DNA溶液(107、106、105、104、103拷贝/μL)为模板,以F5/R5为引物进行qPCR反应,按照SYBR Premix Ex TaqTM Ⅱ说明书进行配制,反应体系同“1.2.3.3”节。采用2步法进行PCR扩增,进行45个循环,其余参数同“1.2.3.3”节。得到等梯度拷贝数对应的CT值。依据CT值与起始模板数的对数值之间的一定的线性关系来制作标准曲线。水稻内参基因SPS标准曲线的制作参考Ding等的方法[24]。
以6个阳性OE-OsRhoGDI2水稻基因组DNA为模板,分别以F5/R5和F6/R6为引物进行qPCR反应,按照SYBR Premix Ex TaqTM Ⅱ说明书进行配制,反应体系同“1.2.3.3”节。采用2步法进行PCR扩增,进行45个循环,其余参数同“1.2.3.3”节。分别获得6个植株HYG与SPS基因的CT值,并由HYG与SPS基因的相关性方程计算其起始模板数。用纯合二倍体水稻SPS作为内参基因,将H0与S0的比值乘以2得到OsRhoGDI2基因在转基因水稻中的拷贝数。
2 结果与分析
2.1 OsRhoGDI2转基因水稻的筛选
以水稻基因组DNA为模板,用引物F1/R1进行PCR扩增,扩增产物经琼脂糖凝胶电泳检测,确定阳性转基因植株。由于OsRhoGDI2转基因表达载体内插入的目的基因长度为795 bp,所以从阳性转基因水稻基因组DNA中能扩增出 795 bp 特异片段,而对照(WT)植株中不能扩增出该大小的条带。电泳结果显示,在检测的15个样品中,除了3、8、11号之外,都可以扩增得到预期大小的片段,且与阳性对照条带位置一致(图1),说明OsRhoGDI2基因已经整合到水稻基因组中。
2.2 OsRhoGDI2在转基因水稻中表达水平的检测
选取PCR检测为阳性的转基因水稻,提取叶片总RNA,采用半定量RT-PCR检测该基因的表达水平,内参采用水稻泛素(UBQ)基因,扩增产物经琼脂糖凝胶电泳检测,结果显示,在对照(WT)水稻叶片中,OsRhoGDI2基因的表达水平较低,而在所检测的7株转基因水稻中,OsRhoGDI2基因的表达水平均有不同程度的提高,尤其是7、9、10、12、16号这5株的表达量有显著的增高(图2),说明这5株水稻是OE-OsRhoGDI2阳性植株。
进一步采用实时定量PCR方法检测OsRhoGDI2基因在转基因水稻中的表达水平。由图3可知,除2号外,在其余6株转基因水稻中OsRhoGDI2基因的表达水平均比对照(WT)高,基因表达量极显著上调的有6号(1.69倍)、16号(2.69倍)、10号(4.02倍)、12号(5.98倍)、9号(9.72倍)和7号(13.35倍),且与半定量RT-PCR检测结果一致,证明OsRhoGDI2基因在这些转基因水稻中实现了过量表达。
2.3 HYG基因标准曲线的制作
以含有HYG基因的pCAMBIA1302-OsRhoGDI2-GFP质粒DNA溶液(107、106、105、104、103拷贝/μL)为模板,基于qPCR数据获得了HYG基因的标准曲线(图4),其相关系数 r=0.999 7,说明相关性高。其CT值与起始模板数(H0)之间的相关性方程为:H0=10(-0.297CT+12.356)。
2.4 OsRhoGDI2在转基因水稻中拷贝数的测定
以上述鉴定的6株阳性OE-OsRhoGDI2水稻基因组DNA为模板,分别以F5/R5和F6/R6引物组合,对HYG基因与SPS基因进行qPCR,获得扩增曲线与待测样品的CT值(表2),并由HYG与SPS基因的相关性方程计算其起始模板数。
OsRhoGDI2基因在转基因水稻中的拷贝数的计算结果显示,对照水稻拷贝数为0,其余有6个OE-OsRhoGDI2水稻拷贝数均为1(表3)。
3 结论与讨论
植物遗传转化是农作物改良和基因功能研究的常用试验方法。本研究在实验室前期工作的基础上,对OsRhoGDI2转基因水稻T2代进行了筛选和鉴定,获得6个阳性转基因水稻株系。现有的研究认为,过表达后代中外源基因拷贝数是影响目的基因表达水平和遗传稳定性的主要因素。据统计分析显示,外源基因拷贝数为1或2时能够稳定遗传高表达,而外源基因拷贝数过多(达4~5)往往使外源基因不表达甚至导致基因沉默[25-26]。因此在获得阳性过表达转基因植株后,对其外源基因拷贝数进行鉴定分析是必要的。
本研究对外源基因拷贝数的检测所采用的内参基因为水稻特有的单拷贝基因SPS[24],转基因植株的标志基因是植物表达载体的标志基因HYG,也是转基因植物筛选常用的标志。检测结果显示,OsRhoGDI2在过表达转基因水稻中均为单拷贝,提示基因能够稳定遗传和表达,这与笔者所在实验室对其T1代和T2代的连续筛选结果是一致的,阳性率均在85%以上。
本研究采用基因组DNA扩增的方法,首先检测了OsRhoGDI2过表达植物载体是否稳定整合到水稻基因组中,进而采用半定量RT-PCR、实时荧光定量PCR技术在转录水平检测了OsRhoGDI2在转基因水稻中的表达水平,并通过外源基因拷贝数的测算,证实外源OsRhoGDI2基因整合到水稻基因組中的拷贝数均为1。本研究筛选和鉴定了6个独立的OsRhoGDI2过表达转基因株系,为后续以这些材料开展对OsRhoGDI2的功能鉴定奠定了良好的基础。
参考文献:
[1]Kost B,Lemichez E,Spielhofer P,et al. Rac homologues and compartmentalized phosphatidylinositol 4,5-bisphosphate act in a common pathway to regulate polar pollen tube growth[J]. Journal of Cell Biology,1999,145(2):317-330.
[2]Molendijk A J,Bischoff F,Rajendrakumar C S,et al. Arabidopsis thaliana Rop GTPases are localized to tips of root hairs and control polar growth[J]. EMBO Journal,2001,20(11):2779-2788.
[3]Jones M A,Shen J J,Fu Y,et al. The Arabidopsis Rop2 GTPase is a positive regulator of both root hair initiation and tip growth[J]. The Plant Cell,2002,14(4):763-776.
[4]Fu Y,Wu G,Yang Z B. Rop GTPase-dependent dynamics of tip-localized F-actin controls tip growth in pollen tubes[J]. Journal of Cell Biology,2001,152(5):1019-1032.
[5]Fu Y,Li H,Yang Z B. The ROP2 GTPase controls the formation of cortical fine F-actin and the early phase of directional cell expansion during Arabidopsis organogenesis[J]. The Plant Cell,2002,14(4):777-794.
[6]Delmer D P,Pear J R,Andrawis A,et al. Genes encoding small GTP-binding proteins analogous to mammalian rac are preferentially expressed in developing cotton fibers[J]. Molecular & General Genetics,1995,248(1):43-51.
[7]Trotochaud A E,Hao T,Wu G,et al. The CLAVATA1 receptor-like kinase requires CLAVATA3 for its assembly into a signaling complex that includes KAPP and a Rho-related protein[J]. The Plant Cell,1999,11(3):393-406.
[8]Kawasaki T,Henmi K,Ono E,et al. The small GTP-binding protein rac is a regulator of cell death in plants[J]. Proceedings of the National Academy of Sciences of the United States of America,1999,96(19):10922-10926.
[9]Kawasaki T,Koita H,Nakatsubo T,et al. Cinnamoyl-CoA reductase,a key enzyme in lignin biosynthesis,is an effector of small GTPase Rac in defense signaling in rice[J]. Proceedings of the National Academy of Sciences of the United States of America,2006,103(1):230-235.
[10]Fujiwara M,Umemura K,Kawasaki T,et al. Proteomics of Rac GTPase signaling reveals its predominant role in elicitor-induced defense response of cultured rice cells[J]. Plant Physiology,2006,140(2):734-745.
[11]Schultheiss H,Hensel G,Imani J,et al. Ectopic expression of constitutively activated RACB in barley enhances susceptibility to powdery mildew and abiotic stress[J]. Plant Physiology,2005,139(1):353-362.
[12]Dermardirossian C,Bokoch G M. GDIs:central regulatory molecules in Rho GTPase activation[J]. Trends in Cell Biology,2005,15(7):356-363.
[13]Dovas A,Couchman J R. RhoGDI:multiple functions in the regulation of Rho family GTPase activities[J]. The Biochemical Journal,2005,390:1-9.
[14]Klahre U,Becker C,Schmitt A C,et al. Nt-RhoGDI2 regulates Rac/Rop signaling and polar cell growth in tobacco pollen tubes[J]. Plant Journal,2006,46(6):1018-1031.
[15]Bischoff F,Vahlkamp L,Molendijk A,et al. Localization of AtROP4 and AtROP6 and interaction with the guanine nucleotide dissociation inhibitor AtRhoGDI1 from Arabidopsis[J]. Plant Molecular Biology,2000,42(3):515-530.
[16]Kieffer F,Elmayan T,Rubier S,et al. Cloning of Rac and Rho-GDI from tobacco using an heterologous two-hybrid screen[J]. Biochimie,2000,82(12):1099-1105.
[17]梁卫红,唐朝荣,吴乃虎. 两种水稻GDP解离抑制蛋白基因的分离及特征分析[J]. 中国生物化学与分子生物学报,2004,20(6):785-791.
[18]Carol R J,Takeda S,Linstead P,et al. A RhoGDP dissociation inhibitor spatially regulates growth in root hair cells[J]. Nature,2005,438(770):1013-1016.
[19]Wu Y X,Zhao S J,Tian H,et al. CPK3-phosphorylated RhoGDI1 is essential in the development of Arabidopsis seedlings and leaf epidermal cells[J]. Journal of Experimental Botany,2013,64(11):3327-3338.
[20]彭威风,梁卫红. 水稻OsRhoGDI2蛋白生物信息学分析及亚细胞定位研究[J]. 生物技术通报,2010(5):82-86,92.
[21]Huang J J,Zhang J,Hao Y F,et al. Distinct expression patterns of the GDP dissociation inhibitor protein gene (OsRhoGDI2) from Oryza sativa during development and abiotic stresses[J]. Biologia,2016,71(11):1230-1239.
[22]Livak K J,Schmittgen T D. Analysis of relative gene expression data using real-time quantitative PCR and the 2-ΔΔCT method[J]. Methods,2001,25(4):402-408.
[23]楊立桃,赵志辉,丁嘉羽,等. 利用实时荧光定量PCR方法分析转基因水稻外源基因拷贝数[J]. 中国食品卫生杂志,2005,17(2):140-144.
[24]Ding J Y,Jia J W,Yang L T,et al. Validation of a rice specific gene,sucrose phosphate synthase,used as the endogenous reference gene for qualitative and real-time quantitative PCR detection of transgenes[J]. Journal of Agricultural&Food Chemistry,2004,52(11):3372-3377.
[25]Flavell R B. Inactivation of gene expression in plants as a consequence of specific sequence duplication[J]. Proceedings of the National Academy of Sciences of the United States of America,1994,91(9):3490-3496.
[26]Vaucheret H,Béclin C,Elmayan T,et al. Transgene-induced gene silencing in plants[J]. The Plant Journal,1998,16(6):651-659.

