人肠源植物乳杆菌PUM1785全基因组测序分析与功能挖掘
孙 杰, 沈 敏, 吴恬菲,伦永志1,*
(1.莆田学院药学与医学技术学院 医学微生态学福建省高校重点实验室,福建 莆田 351100;2.莆田学院药学与医学技术学院 医学检验系,福建 莆田 351100)
益生菌只有达到一定的摄入量时,才能够对机体产生益生作用,不同种类的益生菌具有相似或特有的生理特性[1]。乳杆菌作为益生菌的主要类别,具有无毒、调节肠道菌群平衡、抑菌、抗癌等优良特性,常作为微生态制剂的生产菌种而被广泛开发及应用[2],其作为抗生素替代品能够抑制细菌生长,同时降低细菌耐药性,提高机体免疫力,其中植物乳杆菌(Lactobacillusplantarum)耐受性较强,某些特定的植物乳杆菌还能够预防和抵抗疾病发生[3]。植物乳杆菌PUM1785更有别于一般来自健康人体或植物的植物乳杆菌,其分离自长期接受抗生素或激素治疗患者的粪便[4]。此类患者免疫力较低,且由于长期服用抗生素或激素,导致益生菌难以在肠道中定植[5],因此较难从上述患者的肠道微生态环境中分离得到益生菌。若益生菌不能定植则其益生作用便无法发挥,而能否稳定定植的关键取决于益生菌是否具有强耐受性和黏附能力,这表明植物乳杆菌PUM1785可能具备足够强的耐受性和抗性。随着高通量检测技术和生物信息学的高速发展,对益生菌研究也上升到了基因组学层次。通过对植物乳杆菌PUM1785进行基因组测序,全面分析该菌株的基因组功能,从而预测和发现其潜在的益生作用,为其成为微生态制剂或功能性食品的生产菌株提供参考。
1 材料与方法
1.1 材料
1.1.1 试验菌株 植物乳杆菌(Lactobacillusplantarum)PUM1785由莆田学院医学微生态学福建省高校重点实验室分离并保存。
1.1.2 培养基 MRS肉汤培养基购自杭州滨和微生物试剂有限公司。
1.1.3 仪器与设备 超低温冰箱(Forma-990,美国Thermo Fisher Scientific公司);手提式压力蒸汽灭菌器(DSX-280B,上海申安医疗器械厂);生物安全柜(HFsafe-1200LC,上海力申科学仪器有限公司);电热恒温培养箱(DRP-9082,上海森信实验仪器有限公司);全温培养摇床(QYC-200,上海新苗医疗器械制造有限公司);医用离心机(L420,湖南湘仪实验室仪器开发有限公司)。
1.2 方法
1.2.1 菌株活化与培养 将瓷珠法保存的菌种接种于MRS肉汤培养基,37 ℃、150 r/min摇床培养过夜,转接种至MRS平板,37 ℃厌氧培养48 h,分离出单个菌落,再转接至MRS肉汤培养基于30 ℃、180 r/min培养2 d,收集菌体。
1.2.2 细菌基因组提取与测序 按细菌DNA提取试剂盒(E.Z.N.A.Bacterial DNA Kit)说明书提取细菌基因组,进行质量鉴定,在浓度和纯度达到测序要求后,构建DNA文库。将DNA样品随机打断成长度约为350 bp或6 kb的片段,后者再进行环化扩增,产物随机打断为350 bp左右的片段,并用探针捕获环化引物两侧的序列。处理后的DNA片段,经末端修复、加A尾、加测序接头、纯化、PCR扩增等步骤完成DNA文库制备,插入片段检测符合预期后,准确定量文库的有效浓度,以保证文库质量。随后利用Illumina HiSeq高通量测序平台完成全基因组测序。
1.2.3 基因组装 测序得到的原始数据(Raw Data)经过滤处理后得到Clean Data,利用SOAP denovo 2.04[6]、SPAdes[7]和ABySS 2.0[8]软件进行组装,并利用CISA软件进行整合;利用GapClose 1.12[6]软件对初步组装结果进行优化和补洞,从而得到最终的组装结果。
1.2.4 序列分析 利用GeneMarkS[9]软件对植物乳杆菌PUM1785全基因组测序结果进行编码基因预测。基于获得的氨基酸序列,利用Diamond[10]软件与非冗余蛋白数据库(Non-Redundant Protein Database,NR)、京都基因和基因组百科全书数据库(Kyoto Encyclopedia of Genes and Genomes,KEGG)、碳水化合物活性酶数据库(Carbohydrate-Active EnZymes Database,CAZy)、直系同源基因数据库(Cluster of Orthologous Groups of proteins,COG)、转运蛋白分类数据库(Transporter Classification Database,TCDB)、毒力因子数据库(Virulence Factors of Pathogenic Bacteria,VFDB)、抗生素抗性基因数据库(Antibiotic Resistance Genes Database,ARDB)进行比对分析;利用IPRscan[11]软件与基因本体数据库(Gene Ontology,GO)进行比对分析;利用Resistance Gene Identifier[12]软件与综合抗生素研究数据库(Comprehensive Antibiotic Research Database,CARD)进行比对分析,逐一得到基因功能注释结果。利用antiSMASH-4.0.2[13]软件预测全基因组中的已知次级代谢产物合成基因簇。从NCBI GenBank数据库下载20株植物乳杆菌参考基因组序列(表1),与PUM1785株基因组序列进行分子进化比较分析。利用在线工具MUSCLE(http://www.ebi.ac.uk/Tools/msa/muscle)进行直系同源比对,利用MEGA X[14]软件构建全基因组系统发育树(自展一致树)。

表1 植物乳杆菌参考基因组信息
2 结果与分析
2.1 基因组概况
植物乳杆菌PUM1785与模式菌株WCSF1基因组特征高度相似。前者基因组大小3 128 032 bp,GC含量44.56%,共计注释得到2 995个编码基因,其序列长度达到2 642 256 bp,占全基因组总长84.47%;后者基因组大小3 308 273 bp;GC含量44.5%,共计注释得到3 042个编码基因[15]。
2.2 功能注释
2.2.1 NR注释 NR注释结果表明,物种分类高度集中于植物乳杆菌类别中,从而确定了植物乳杆菌PUM1785的分类归属。
2.2.2 GO注释 共有2 014个基因在GO数据库中得到功能注释(图1)。在分子功能中,基因数量位列榜首的注释功能为催化,其次是结合和转运;与细胞组分有关的基因集中注释于细胞、细胞组分和细胞器;在生物过程乃至整个GO注释结果中,基因注释量最高的是新陈代谢,达到1 140,细胞过程的基因注释量为1 139,与定位有关的基因注释量共计816。细胞组分注释结果表明,植物乳杆菌PUM1785具有较强的生物膜形成能力,生物膜能够保护菌体抵抗外界环境。定位和建立定位的基因主要参与细菌黏附,有利于该菌株定殖在宿主细胞表面,并降低病原菌对宿主细胞的侵袭[16]。
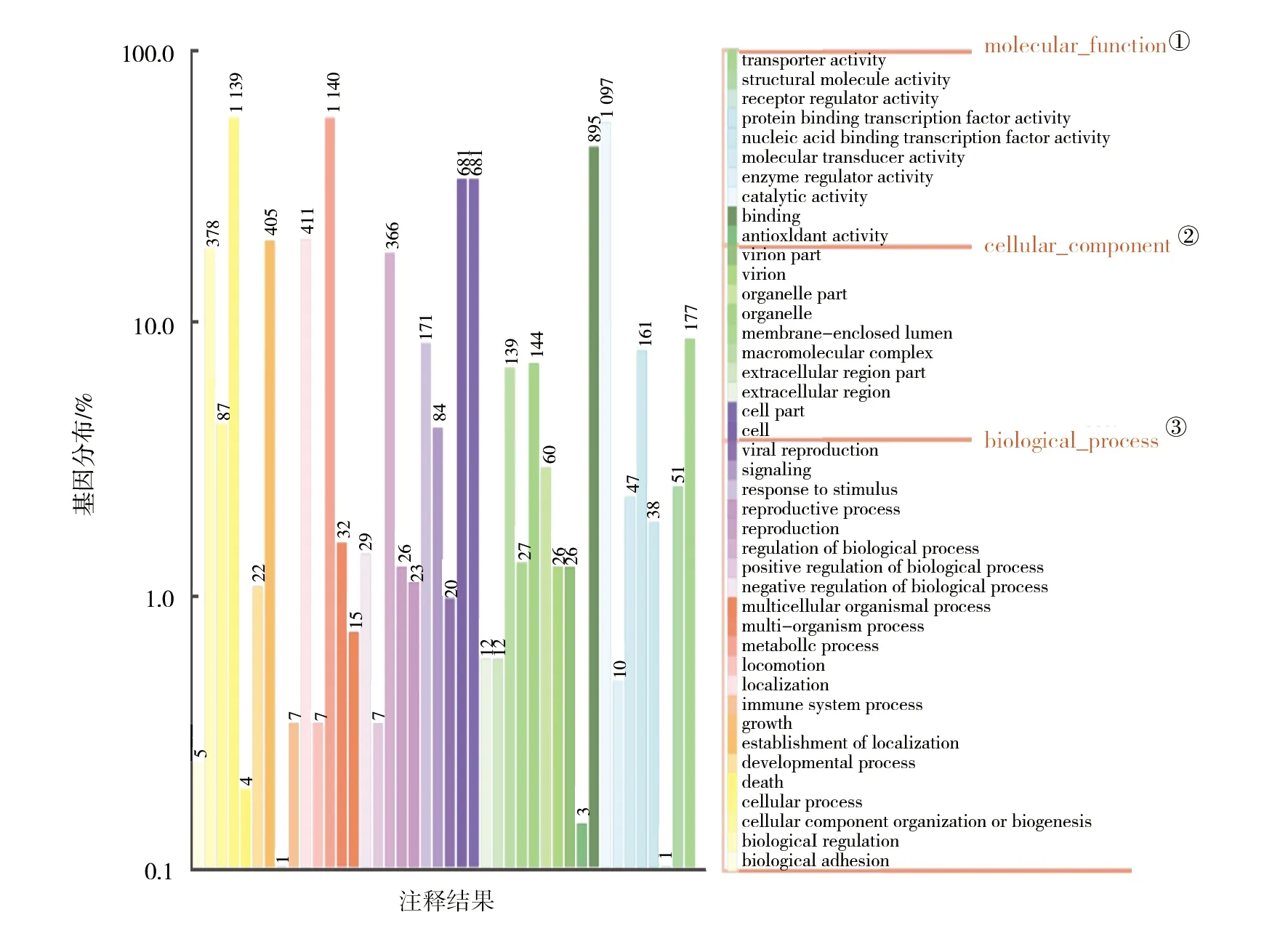
图1 植物乳杆菌PUM1785 GO数据库注释结果
2.2.3 KEGG注释 共有2 870个基因在KEGG数据库中得到功能注释,其中1 299个基因被注释到各个代谢通路(图2),主要分布于代谢通路和环境信息处理通路中。代谢通路中基因数量最多的是碳水化合物代谢,氨基酸代谢次之。而环境信息处理中得到最多注释的是膜运输。碳水化合物代谢通路包括各种糖类的代谢通路,高达206个基因的注释量意味着植物乳杆菌PUM1785具有较强的糖类分解和代谢功能。膜运输通路主要集中于ABC转运蛋白、磷酸转移酶系统以及细菌分泌系统,表明该菌株具有较强的适应外界环境的能力。

图2 植物乳杆菌PUM1785 KEGG数据库通路注释结果
2.2.4 COG注释 COG注释结果表明,转录功能得到最多基因注释量(图3),达到262个。糖类物质和氨基酸转运及代谢相关功能蛋白也得到了较高的基因注释量,意味着该菌株具有较强的碳水化合物代谢能力。该菌株还在细胞壁、生物膜和包膜生物发生中获得141个基因注释量,可以辅证该菌株具有较强的生物膜形成能力,并在防御机制中得到69个基因注释量,提示该菌株能够抵抗消化道环境,为稳定定殖于肠道上皮细胞提供必要条件。

图3 植物乳杆菌PUM1785COG数据库注释结果
2.2.5 TCDB注释 在TCDB注释结果中,以主动转运蛋白、电化学势驱动转运子为主(图4)。根据结合膜运输通路基因的高注释结果,可以推测植物乳杆菌PUM1785的运输方式以主动转运为主、离子通道运输为辅。
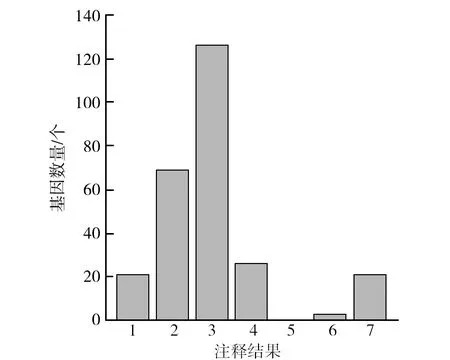
图4 植物乳杆菌PUM1785 TCDB数据库注释结果
2.2.6 CAZy注释 在CAZy注释结果中(图5),得到最高注释量的是糖苷水解酶,其次是糖基转移酶。糖苷水解酶作用于含有2个及以上1, 4-α-D-葡萄糖基的多糖中的1, 4-α-D-葡萄糖苷键,参与糖代谢通路中的多糖水解过程。糖代谢过程中释放的大量能量,为菌体活动提供充足能量。糖基转移酶催化糖基与非糖物质或者其他糖类物质的基团进行转移或交换。单糖通过糖苷键聚合起来形成聚糖,聚糖再与蛋白质共价结合生成蛋白聚糖或糖蛋白,从而参与各种生理过程[17]。

图5 植物乳杆菌PUM1785CAZy数据库注释结果
2.3 分子进化分析
用于比较分析的植物乳杆菌基因组均为单一拷贝,经多序列比对后构建全基因组进化树(图6)。结果表明,植物乳杆菌PUM1785在进化上与CGMCC 1.557株的亲缘关系最近,尤其与植物乳杆菌模式菌株WCSF1株亲缘关系相当近[18],而与ST-III株和CAUH2株的亲缘关系最远。

图6 植物乳杆菌PUM1785全基因组系统发育树
2.4 次级代谢合成产物基因簇分析
植物乳杆菌PUM1785基因组具有1个细菌素类基因簇,共计15个基因,与其他乳杆菌同源基因簇的比对结果见图7A。相似度≥50%的细菌共有10个,其中日本清酒乳杆菌I151sakacinP基因簇、日本清酒乳杆菌spiA-sppA-sppE-sppK-sppR-sppT基因簇、鼠李糖乳杆菌Lc705基因簇一致性为66%。日本清酒乳杆菌I151sakacin P由spp基因簇编码,具有抑制单核李斯特菌的功能[19]。spiA为免疫基因,sppA为结构基因,sppE和sppT负责细菌素sakacin P的转运,sppK包含突变和/或插入元件[20]。鼠李糖乳杆菌Lc705能够下调β-葡萄糖醛酸酶和脲酶活性,从而抑制结肠癌的发生[21]。具有50%一致性的细菌分别为日本清酒乳杆菌sakacinA基因簇[22]、干酪乳杆菌LOCK919基因簇[23]、鼠李糖乳杆菌CBZU00000000序列[24]、鼠李糖乳杆菌ATCC8530基因簇[25],均具有抑菌活性。此外,干酪乳杆菌张氏株能够预防和抵抗结肠癌发生[26]。综合细菌素基因簇序列对比结果,可以预见植物乳杆菌PUM1785具有以上类似功能。
植物乳杆菌PUM1785具有1个萜类基因簇,共24个基因,与其他乳杆菌同源基因簇的比对结果见图7B。萜类物质为异戊二烯整数倍的烯烃类化合物,具有独特芳香。由于其独特的化学结构,能够在抑菌、抗炎、抗肿瘤、抗抑郁、治疗糖尿病及神经保护等方面发挥作用[27-28],故作为新药开发的重要来源而得到重视。

图7 植物乳杆菌PUM1785次级代谢产物合成基因簇的序列比较
2.5 致病性分析
2.5.1 毒力因子注释 在VFDB注释结果中,得到107个基因注释量,主要涉及细菌黏附与定殖、细菌逃避宿主免疫防御系统和细菌毒素(表2)。一般来说,细菌进入宿主体内后会被吞噬细胞捕捉并消灭,但具有逃逸宿主免疫防御系统的毒力因子能够帮助细菌免于清除。细菌的黏附功能与其特殊结构及某些蛋白有关。植物乳杆菌PUM1785的一部分毒力因子能够介导侵袭过程,如抑制补体介导的吞噬作用、激发炎症、使细菌黏附到宿主靶细胞;还包括溶血素和细胞溶素,能够刺激IL-1β和TNF的释放,并溶解多种革兰阳性菌。此类毒力因子数量极少,因此可以忽略其致病性。

表2 细菌黏附素和细菌毒素的类型、数量及作用机制
2.5.2 耐药基因注释 ARDB结果中仅注释到十一碳二烯基焦磷酸磷酸酶,表现为杆菌肽耐药。CARD得到18个注释结果(表3),共包括14种抗生素抗性基因,包括林可霉素、喹诺酮类抗生素、磷霉素、伊霉素、利福平、多肽类抗生素、莫匹罗星等。虽然林可霉素和喹诺酮类的抗性蛋白注释量较高,但并不意味植物乳杆菌PUM1785一定表达该抗性。细菌可以通过外排抗性蛋白从而达到保护自身的目的,如细菌外排丝氨酸蛋白酶抑制剂使得自身能够免受肠道胆汁盐水解及其他外在环境的影响,有利于在肠道中生存[37]。已有研究表明,植物乳杆菌WCSF1株对人体消化道环境有很强的耐受性[38],在相同给药浓度和方式下,WCSF1株有更高的肠道存活率[39]。值得注意的是,由于PUM1785株与WCSF1株进化距离很近,这意味着PUM1785株具有同样的肠道耐受性。外排泵赋予细菌抗生素耐药性,通过抑制外分泌过程阻断耐药蛋白分泌到细菌体外,从而解除耐药蛋白对抗生素的抑制功能,使菌株不再对该抗生素产生耐药[40]。

表3 抗生素耐药基因的频率和种类
3 讨 论
植物乳杆菌PUM1785分离自潜在多重耐药肿瘤患者肠道,若能证明其同时具有高耐受性和益生功能,且对宿主无致病性,则有望成为后续的微生态制剂或功能性食品开发的生产菌株。本研究从基因组层面对该菌株的基因功能进行了全面分析:①该菌株具有较强的生物膜形成能力和识别宿主细胞的能力,而生物膜能够帮助细菌抵抗外界不利的因素;②该菌株含有与碳水化合物代谢过程相关的酶类和所需的蛋白,能够充分利用碳水化合物,并在肠道内具有较强的环境适应及生存能力;③当该菌株进入肠道后,通过对宿主细胞识别能力的调控和表达荚膜、鞭毛、纤连蛋白结合蛋白等黏附因子,使菌株能够牢固黏附于宿主细胞表面,细菌黏附作用既有利于细菌对宿主防御系统的逃逸和在宿主体内的定植作用,也有利于激活宿主免疫防御系统;④由于该菌株具有多种抗生素抗性并仅对杆菌肽耐药,因此可将其直接应用于临床治疗,在恢复肠道菌群平衡、预防因肠道菌群紊乱引起二重感染的同时,避免患者由于前期大量使用药物导致后补充的益生菌大量死亡而降低疗效;⑤该菌株对外界环境适应性强的特性有助于其在患者体内定植,促进代谢以提供肠内营养,有利于患者机体功能恢复,增强治疗效果,并能够解释该菌株在潜在多重耐药患者或接受抗生素治疗患者的肠道中得以生存的原因。
结合上述分析结果,植物乳杆菌PUM1785一旦黏附于宿主细胞后,利用糖苷水解酶和糖基转移酶将多糖水解为单糖,有助于宿主细胞摄取能量。糖苷水解酶不仅可以水解食物中的糖苷键,还可以破坏某些细菌的生物膜,从而发挥抑菌作用,对于某些耐药菌而言,也是很好的生物膜破坏剂[41]。由糖基转移酶参与调控形成的蛋白聚糖和糖蛋白因具有不同的核心蛋白而发挥不同的作用,如用于参与细胞识别、黏附和免疫识别及信号转导、微生物致病过程和肿瘤转移过程等[42]。糖苷水解酶和糖基转移酶的任一失活或缺失,都可能导致原有的聚糖结构发生变化,导致功能缺失或细胞恶变,糖苷水解酶和糖基转移酶能够影响细菌识别宿主细胞、细菌黏附功能、致病菌致病过程和肿瘤转移过程[43]。当疾病发生时,可定位蛋白聚糖所对应的糖基转移酶位点,并成为药物靶点。
由于植物乳杆菌PUM1785的基因突变类型主要为毒力减弱型,细菌毒素基因数量很低且仅对杆菌肽耐药,故认为该菌株无致病性。该菌株表达的细菌素类物质作为抗生素替代药物,不仅能够帮助宿主抵抗病原菌侵袭、调节肠道菌群平衡,而且可以用于结肠癌防治;产生的萜类物质具有抗炎、抗肿瘤、抗抑郁、治疗糖尿病及神经保护等作用。综上,植物乳杆菌PUM1785适合作为研发微生态制剂的候选益生菌株。

