黄曲霉功能基因组学研究
王 鹏, 张颖超, 管轶男, 孔 青*, 郁东兴
(1.中国海洋大学 食品科学与工程学院,山东 青岛 266003;2.青岛蔚蓝生物股份有限公司,山东 青岛 266101;3.青岛尚好科技有限公司,山东 青岛 266700)
黄曲霉是一种常见的腐生菌真菌和条件致病菌,多见于发霉的粮食、粮制品及其他霉腐的有机物上。黄曲霉产生的黄曲霉毒素是毒性极强的剧毒物质,具有明显的“三致”作用,对人和动物肝脏组织具有严重的破坏性,黄曲霉及黄曲霉毒素对多个国家和地区造成了无法估量的经济损失[1]。黄曲霉是仅次于烟曲霉的第二大致病菌,可在免疫抑制的患者中引起侵袭性曲霉病[2]。黄曲霉基因组中存在丰富的次级代谢产物基因簇,具备产生许多有益的次级代谢产物的能力。预计将有56个次级代谢产物生物合成基因簇位于黄曲霉基因组中,其中27个基因簇包含至少一种聚酮化合物合酶(PKS),而26个簇包含至少一种非核糖体肽合成酶(NRPS)[3-4]。在丝状真菌中,源自NRPS的天然产物包括抗生素、霉菌毒素、铁载体和生物碱[5]等。这些次级代谢产物的功能有待进一步探讨。米曲霉是一种常见的工业菌株,在传统发酵工业中已经被广泛使用了1 000多年。米曲霉发酵技术已经成熟,其安全性已获得美国食品药品监督管理局(FDA)的认可[6]。Payne等[7]研究表明,黄曲霉和米曲霉之间具有源自同一生态型的序列高度相似性,由此推测米曲霉是由黄曲霉驯化而来。此外,黄曲霉具有生产纤维素酶和生物乙醇的潜力[8]。目前,大多数研究中,常用的黄曲霉菌株的全基因组测序已经完成。选择标记是分子生物学中必不可少的组成部分,由于黄曲霉可用的选择标记较少、情况复杂,因此,黄曲霉的功能基因组学研究仍受到诸多限制。本文描述了包括黄曲霉选择标记和遗传转化方法等方面的最新研究进展。此外,还讨论了黄曲霉功能基因组学的应用前景及改善黄曲霉功能基因组学研究策略的潜在技术。
1 黄曲霉的功能基因组学策略
1.1 黄曲霉次生代谢生物合成基因簇(BGCs)的预测
真菌是具有生物活性化合物的天然产物的重要来源之一,并应用于从农业到医学等的各个领域。丝状真菌的一些次生代谢物(SM)如抗生素(青霉素和头孢菌素)、抗真菌药物(灰黄霉素)和降低胆固醇的药物(洛伐他汀),在食品、医疗、农业和其他领域均得到了广泛应用[9]。次生代谢物由低分子量物质构成,通常由大型多模块PKS和NRPS或异戊二烯基转移酶和二甲基烯丙基色氨酸合酶组成[5]。Khaldi等[10]设计和开发了基于Web软件的SMURF(www.jcvi.org/smurf/),根据基因组背景和范围系统预测次生代谢物基因簇。SMURF依靠隐马尔可夫模型(HMM)检测真菌基因组中的骨架基因,从而可以准确地恢复所有基因簇并预测其他潜在基因簇。最近,Blin等[11]设计了次生代谢生物合成基因簇快速识别工具antiSMASH(已更新为antiSMASH 5.0),以便更好地应用于次生代谢生物合成基因簇挖掘。PRISM3是一种预测非核糖体肽以及I型和II型聚酮化合物化学结构的工具,在预测次生代谢生物合成基因簇方面具有很大作用[12]。基于SMURF的分析表明,黄曲霉基因组至少包含70个编码酶的基因,这些酶催化多种次级代谢物生物合成途径的第一步[10]。值得注意的是,次生代谢生物合成基因簇的预测并不完全准确,如存在低估或高估次生代谢生物合成基因簇中的基因功能。因此,要准确了解与次生代谢生物合成基因簇真正关联的基因,只能通过基因敲除来验证。
1.2 黄曲霉基因研究方法
1.2.1 筛选标记 筛选标记是黄曲霉功能基因研究的关键问题之一。有效的筛选标记可极大提高基因操作效率。目前,黄曲霉的筛选标记主要分为两类:耐药标记和营养缺陷型标记(表1)。Seip等[13]利用β-微管蛋白基因作为筛选标记基因,发现阳性转化子对苯并咪唑具有抗性。Ramesh等[14]利用潮霉素抗性基因(hph)作为筛选标记基因对黄曲霉丝氨酸蛋白酶基因(sep)进行敲除。Chang等[15]利用米曲霉中的巯乙胺抗性基因(ptr)作为筛选标记基因,构建了Ku70缺陷型黄曲霉菌株。同时,黄曲霉对博来霉素敏感,ble作为稳定的转化标记物的基因,可使黄曲霉产生博来霉素抗性[16]。最近,Tao等[17]开发了一种新的羧甲基纤维素作为筛选标记的转化系统,转化子可以有效地整合到黄曲霉基因组中。但曲霉属真菌本身具有极强的抗生素抗性;同时,抗生素抗性标记在实际生产中存在安全隐患,微生物中耐药基因的基因流动会增加病原微生物的耐药性,并对环境和人类健康构成严重威胁。

表1 黄曲霉的筛选标记
粗糙脉孢菌(Neurosporacrassa)中的Pyr-4是用于黄曲霉转化的最常见筛选标记基因之一[18]。Chang等[15]利用寄生曲霉(Aspergillusparasitica)的pyrG基因构建一种营养缺陷型黄曲霉尿嘧啶菌株,用于基因敲除研究。pyrG基因编码的5′-单磷酸胞苷脱羧酶是真菌尿嘧啶生物合成的关键酶,当补充尿苷或尿嘧啶时,pyrG突变体才能够将乳清酸核苷转化为尿嘧啶,以维持真菌正常生长[6]。niaD基因编码的NADPH依赖性硝酸盐还原酶,可将硝酸盐还原为亚硝酸盐,而niaD突变体无法在以硝酸盐为唯一氮源的培养基上生长[19]。与其他曲霉属真菌相比,黄曲霉中营养缺陷型筛选标记的种类较少。Fiedller等[20]利用hisB基因作为筛选标记基因,构建了用于黑曲霉基因靶向研究的组氨酸营养缺陷型菌株,在其他类似的病原性丝状真菌如黄曲霉应用中极具开发潜力。Lenouvel等[21]利用argB基因(编码鸟氨酸转氨甲酰酶)的缺失,在黑曲霉中构建了一株精氨酸营养缺陷型菌株,并将其应用于黑曲霉转化研究。sC基因编码的ATP 硫酸盐酶可催化无机硫酸盐的ATP激活,sC突变体不能利用硫酸盐作为硫的来源。sC作为筛选标记基因已成功地应用于各种曲霉中,例如构巢曲霉、烟曲霉、黑曲霉、米曲霉[22]等。目前,黄曲霉营养缺陷型筛选标记基因开发较少,hisB、argB和sC等筛选标记基因在黄曲霉遗传学研究中极具发展前景。因此,开发丰富多样的筛选标记基因对于黄曲霉基因组学的研究至关重要,也将很大程度上提高黄曲霉的转化效率。
1.2.2 黄曲霉转化策略 高效的转化策略是黄曲霉遗传学研究的基础。目前,黄曲霉的转化策略主要分为原生质体介导的转化(PMT)和根瘤农杆菌介导的转化(AMT)两种。其中,PEG介导的原生质体转化方法是黄曲霉遗传学研究中最常用的转化方法。利用多种细胞壁裂解酶,从真菌细胞中制备原生质体仍然是制备真菌感受态细胞的最常见方法。黄曲霉的PMT方法具有以下步骤[15](图1):①将10个营养缺陷型菌株孢子(ΔpyrG)接种到100 mL的Czepak-Dox肉汤(含酪蛋白0.5%、硫酸铵10 mmol/L、尿嘧啶0.2%和葡萄糖1%)中,30 ℃、150 r/min恒温生化培养振荡器中培养11~12 h,100 μm尼龙细胞滤器收集菌丝体[8]。②将收集的菌丝体放入新鲜制备的酶解液中,50~70 r/min、30 ℃水解2~3 h。在酶解过程中,观察原生质体的状态以确保原生质体的质量。酶解液主要有以下两种:哈茨木霉、裂解酶和细胞壁消化酶,以含有0.55 mol/L KCl、0.05 mol/L柠檬酸溶液(pH 5.8)为溶剂;崩溃酶、β-葡萄糖苷酶、裂解酶,以含有1.2 mol/L NaCl、10 mmol/L NaPO3溶液(pH 7.0)为溶剂。③将收集的原生质体用0.6 mol/L KCl、50 mmol/L CaCl2和10 mmol/L Tris-HCl,pH 8.0的溶液洗涤2次,然后加入新鲜配制的50%PEG-4000溶液(10 mmol/L CaCl2、10 mmol/L Tris pH 7.5、0.6 mol/L KCl)。④将每种转化混合物添加到100~130 mL预热的再生/选择培养基(Czapek-box中加入0.6 mol/L KCl和10 mmol/L硫酸铵)中。
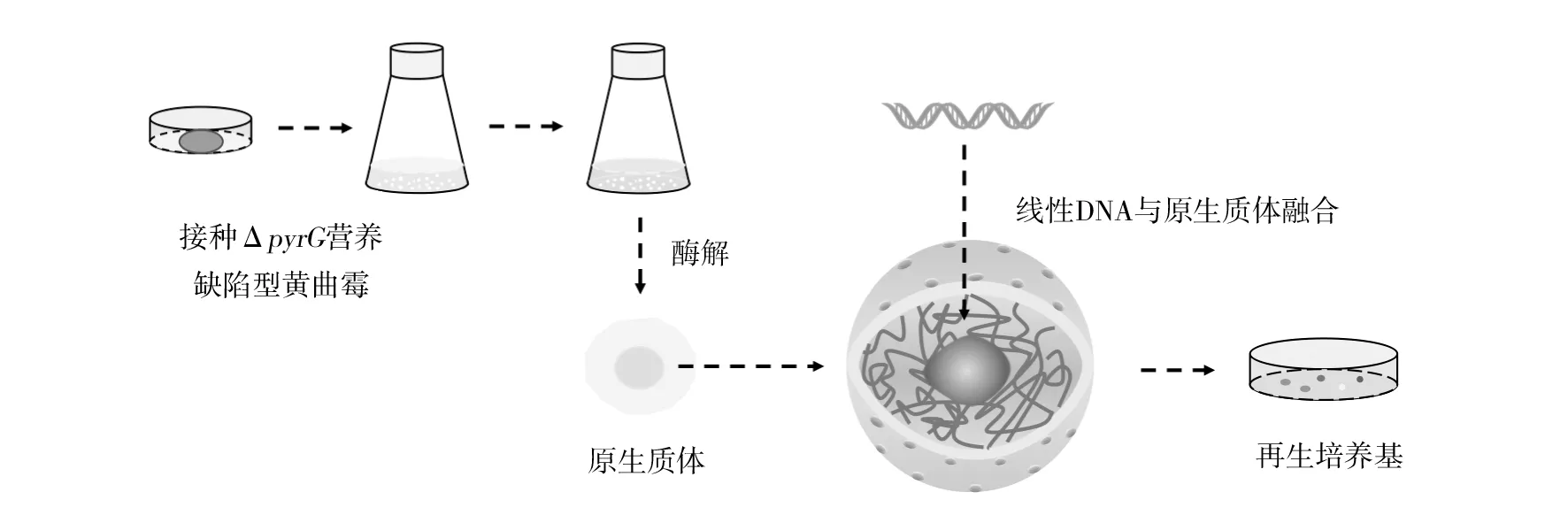
图1 黄曲霉原生质体介导的转化(PMT)流程图
根瘤农杆菌是一种植物性病原体,可通过IV型分泌系统将Ti质粒上的一部分T-DNA(8个左右的基因在植物细胞内表达)转移到宿主中,从而诱导宿主产生冠状胆囊肿瘤[23]。若将T-DNA转移到酵母和丝状真菌中,根瘤农杆菌必须具有完整的毒力系统。用于转化的DNA与DNA结合蛋白VirD2和VirE2一起作为单链DNA分子转运到宿主[24]。与原生质体介导的转化相比,根瘤农杆菌介导的转化是一种更有效的基因靶向方法,不需要制备可转化的原生质体。通常使用乙酰丁香酮作为诱导剂激活细胞表面的VirA和VirG蛋白,这些蛋白可以将其T-DNA随机转移至受体基因组中的随机位点[25]。黄曲霉的根瘤农杆菌介导的转化方法具有以下步骤(图2):①通过电穿孔将载体引入AGL-1感受态细胞;②将黄曲霉孢子悬浮液和诱导的根瘤农杆菌悬浮液的混合物涂布在Co-IM培养基上(IM用5 mmol/L葡萄糖代替10 mmol/L葡萄糖,加1.8%(w/v)琼脂)[26];③将硝酸纤维素膜转移到含有抗生素的选择培养基中。目前,报道的转化黄曲霉的方法仅有PMT和AMT两种。PMT和AMT方法的优缺点如表2所示。在其他丝状真菌中,除了这两种常规的方法外,还有两种常用的转化方法电穿孔转化法(ET)和基因枪转化法(BT)。每种方法独具优势且被应用相应研究中,适当的转化方法,可以显著提高基因工程的成功率。

图2 根瘤农杆菌介导的黄曲霉转化(AMT)流程图

表2 黄曲霉两种常用转化方法的机制和优劣势
2 提高黄曲霉基因操作效率的策略
2.1 缺陷型菌株非同源末端连接(NHEJ)的基因工程研究
当外源DNA作为目标基因整合到受体染色体时,会涉及到DNA双链断裂(DSB),而DNA双链断裂会导致细胞死亡或多种遗传变化的损害,可通过非同源末端连接途径和同源重组(HR)进行修复[27]。基于同源重组的基因敲除是真菌遗传学研究中最常用的方法,但大多数丝状真菌的同源重组频率较低(<5%)。因此,在丝状真菌中,需要通过NHEJ途径来增加HR途径以实现高效的基因靶向技术。
NHEJ过程由DNA依赖性蛋白激酶催化亚基(DNA-PKcs)、Ku70-Ku80异二聚体和DNA连接酶IV-Xrcc4复合体介导[28]。在真核细胞中,Ku70-Ku80异二聚体识别断裂的DNA双链,形成桥接复合体并集合其他在NHEJ途径中起到至关重要作用的NHEJ蛋白[29]。目前,通过敲除ku70/ku80基因的基因靶向技术已成功地应用于包括黑曲霉、米曲霉、酱油曲霉和黄曲霉在内的许多曲霉属物种[15,30-31]。在红曲霉M7中,通过敲除ku70和ku80,基因置换的频率从15%分别增加到42.2%和61.5%[29]。在黑曲霉中,ku70和ku80双缺陷突变体的基因靶向频率增加到100%[30]。在指状青霉中,ku70的敲除极大地增加了同源重组的发生频率,但同时发现Δku70菌株的生长和分生孢子的发育也受到影响。因此,Δku70/ku80被用作基因靶向技术的受体菌株,必须确认ku70/ku80等基因对分生孢子产生以及菌体生长发育功能无影响[32]。
目前,在黄曲霉中用于遗传学研究的受体菌株主要是TJES19.1(Δku70,pyrG-)和CA14(Δku70,pyrG-)[15]。为了提高基因靶向技术的成功率,应尽可能消除NHEJ途径。ligD基因编码DNA连接酶IV,参与NHEJ途径的最后一步。在米曲霉中,ligD敲除菌株的基因靶向频率高达100%,在青曲霉中,通过破坏ligD,目标基因替换的频率从低于2%增加到85%[34]。在黄曲霉中,可以分别或同时建立Δku70、Δku80和ΔligD菌株,以测试所构建菌株的靶基因置换频率,从而进一步提高基因组学研究的成功率。
2.2 使用Cre-loxP重组系统拯救标记基因
在丝状真菌中,由于筛选标记种类较少,其实用性受到限制,而标记拯救方法的出现可以实现连续基因敲除,从而使基因操作更加方便。噬菌体P1 Cre-loxP重组系统包含重组酶(Cre),重组酶可特异性识别34 bp的靶DNA序列(loxP)并催化它们之间发生重组[35]。其原理如图3所示。首先,构建具有上下游同源臂,筛选标记基因(M)和loxP位点的载体;然后将具有loxP位点的载体通过同源重组与目标基因组置换;最后,Cre重组酶消化loxP位点,以切除整个消化区域,包括位于loxP侧翼的目标基因,并重组原始目标基因组。

图3 Cre-loxP重组原理
通过使用Cre-loxP重组系统,能够比较容易地在米曲霉中构建ΔligD标记基因拯救菌株,从而提高了同源重组效率[36]。但是,由于在标记物拯救过程中,Cre重组酶介导的切除在基因组上留下了loxP位点,因此使用Cre-loxP系统进行的几轮基因敲除或整合导致许多loxP位点在基因组上积累。为了避免由剩余的loxP位点引起的相邻基因座同源重组而发生染色体DNA区域的不必要切除,在米曲霉中,成功地构建了具有自灭标记回收盒的质粒载体[37]。Cre-loxP重组系统同时已成功用于黑曲霉中,可以在有效的细胞工厂中敲除和/或插入多个基因结构,从而用于草酸和苹果酸的生产[38]。在黄曲霉中,可以通过Cre-loxP重组系统,快速构建连续的基因敲除菌株,可用于研究多个基因对真菌生长发育功能的相互作用,也可用于快速构建NHEJ途径缺陷的基因研究受体菌株。
2.3 利用CRISPR-Cas9系统开发基因组编辑方法
CRISPR(Clustered Regularly Interspaced Short Palindromic Repeats)是成簇的、规律间隔的短回文重复序列,是生物体(存在于大多数细菌和所有古细菌)为应对病毒和质粒不断攻击而演化来的获得性免疫防御机制[39]。Cas9蛋白是一种双链DNA核酸酶,能在介导RNA引导下对靶位点进行切割。CRISPR-Cas系统主要有3种类型,其中II型系统的发现,使RNA介导的基因编辑技术成为简单、方便、快速的功能基因组学研究工具。
CRISPA-Cas9系统仅需要Cas9核酸酶即可破坏与由crRNA-tracrRNA(反式激活crRNA)杂交体组成的双介导RNA所匹配的DNA[40]。crRNA-tracrRNA被设计为具有两个关键特征的单介导RNA(sgRNA):5′端的序列通过Watson-Crick碱基配对原则确定DNA靶位点,3′端(双链RNA)是Cas9的结合端(图4)[41]。sgRNA原始间隔区序列的核苷酸(包含20个碱基配对识别的靶位点)可以使RNA介导的Cas9核酸酶准确地定位到靶向DNA序列,通过改变sgRNA的前20个核苷酸以对应目标DNA的核苷酸序列,且Cas9核酸酶活性可以针对任何带有N20-NGG形式的DNA序列[42]。Cas9核酸酶对靶位点进行切割后,目标基因座通常经历两次主要的DNA损伤修复(图4):通过NHEJ途径的易错修复或高保真的同源介导的双链DNA修复(HDR)。在没有修复模板的情况下,DNA双链断裂通过NHEJ过程重新连接以实现突变形式的基因敲除。HDR通过外源加入修复模板,并使用它在目标位置产生精确的修复。

图4 CRISPR-Cas9介导的基因敲除
目前,CRISPR-Cas9系统已被用于许多曲霉属的遗传学研究中。在烟曲霉中,CRISPR-Cas9系统已成功用于靶向聚酮化合物合酶基因(pksP)的敲除。Cas9核酸酶本身表达对烟曲霉的生长或毒力无影响,因此CRISPR系统可用于烟曲霉相关的发病机制研究[43]。在黑曲霉中,使用CRISPR-Cas9系统产生营养缺陷型受体菌株并整合基因组,整合效率提高到100%[44]。在米曲霉中,使用CRISPR-Cas9系统开发基因组编辑方法。这项技术为广泛使用的工业菌株米曲霉的基因工程改造提供便利性[45]。由于使用固定真菌启动子表达在不同真菌中实际效果存在差异,Cas9和sgRNA基因在不同物种中的表达可能不同。如果Cas9和sgRNA基因表达受限,则RNA定向诱变可能无效。在真核生物中,寻找和优化用于sgRNA表达的合适启动子是一项主要的技术挑战。在体内表达gRNA的最常见方法是使用两个位于gRNA两侧的核酶序列,即5′末端锤头(HH)和3′末端丁型肝炎病毒(HDV)序列。在黑曲霉中,使用U6启动子建立了一个简单的CRISPR-Cas9系统。sgRNA的转录由两个异源(PhU6和PyU6)和一个内源性U6启动子驱动。虽然简化了黑曲霉的遗传操作,但基因编辑效率仅为15%~36%[46]。真核生物中高度保守和有效表达的5S rRNA基因可用作sgRNA启动子。在米曲霉中,通过构建包含基于AMA1带有抗性标记ptrA的自主复制质粒,改进了CRISPR-Cas9方法。由于该质粒带有抗药性标记,供体DNA进行有效的基因编辑无需插入任何选择标记,因此可以重复进行基因工程研究[47]。CRISPR-Cas9系统在曲霉中显示出强大的应用潜力,极大地提高了基因编辑的便利性。在黄曲霉中尚未报道CRISPR-Cas9系统,但其极具潜在应用价值。CRISPR-Cas9系统无毒,无需标记即可连续进行基因编辑。在研究黄曲霉与植物之间的相互作用时,具有很大的优势。
2.4 RNA干扰(RNAi)技术
RNA干扰(RNAi)是一种保守的真核基因调控机制,该机制使用小的非编码RNA(sRNA)介导转录后/转录基因沉默。在已鉴定的sRNA中,RNAi主要分为三类:小干扰RNA(siRNA),microRNA(miRNA)和PIWI相互作用RNA(piRNA)[48]。RNAi最初被描述为一种宿主防御机制,通过产生源自这些外源序列的小干扰RNA或短发夹RNA(shRNA),可以保护基因组免受侵入性核酸(例如病毒和转座子)的侵害[49]。RNAi也是在真核生物中诱导基因沉默或敲低的独特技术。sRNA由依赖性Dicer酶或非依赖性生物途径产生,并整合到效应蛋白Argonaute(AGO)家族中,通过与靶RNA碱基配对来指导特异性序列的基因沉默[50]。
真核生物中的RNAi由RNAse III酶(Dicer)引发,该酶将长双链RNA(dsRNA)切割成siRNA,并且siRNA与AGO和其他蛋白质结合形成RNA诱导的沉默复合物(RISC)。然后,siRNA/RISC复合物结合靶mRNA的互补序列,从而阻止靶转录本的翻译[49]。RNAi与CRISPR-Cas9系统具有明显的相似性,例如,两者均使用靶标特异性sRNA。表3显示了RNAi和CRISPR-Cas9技术的比较。

表3 RNAi和CRISPR-Cas9技术的比较
基于RNAi技术的宿主诱导基因沉默(HIGS)是研究真菌与宿主之间相互作用的重要手段之一。真菌sRNA与宿主RNAi机制相互作用,从而下调宿主的防御基因并增强致病性[51]。宿主诱导的基因沉默在发展对霉菌毒素具有抗性的农作物中具有潜力。例如,用目标aflR基因778 bp的RNAi构建体转化玉米基因组序列。该构建体在宿主植物中产生初级siRNA。当植物被黄曲霉菌感染时,初级siRNA进入真菌并切割互补的aflRmRNA序列,从而阻碍aflR基因的表达并导致黄曲霉毒素生物合成的下调[52]。在玉米种子中,通过构建α-淀粉酶基因RNAi构建体,抑制了黄曲霉α-淀粉酶(amy1)基因的表达,减少了黄曲霉对宿主的感染,从而导致黄曲霉毒素产量降低[53]。由于具有沉默特性的RNA可以从植物宿主转移到各种入侵病原体, RNAi在研究预防黄曲霉毒素污染方面具有巨大潜力。
3 黄曲霉功能基因组学的潜在应用
3.1 黄曲霉基因组学用于阐明主要真菌毒素的合成途径和调控
随着基因组测序技术的飞速进步,越来越多的黄曲霉菌株完成全基因组测序,其中完成基因组测序最早的是黄曲霉菌株NRRL3357。Faustinelli等[54]报道了8种黄曲霉菌株的基因组序列,其中3种是不产黄曲霉毒素菌株。不产黄曲霉毒素菌株被用作减少黄曲霉毒素污染,是一种用来防治黄曲霉毒素污染的重要生物学策略。Weaver等[55]报道了3株被用于黄曲霉毒素的生物防治的黄曲霉菌株(NRRL21882、AF36和K49)的全基因组序列。Weaver等[56]还报道了从田间土壤和玉米中分离的20株具有地理参考价值菌株的全基因组序列。Chang等[57]报道了黄曲霉菌株CA14的基因组,一株被广泛用于黄曲霉基因功能研究的菌株。黄曲霉基因组的大小约为36.5~38.8 Mb。序列数据保存在GenBank数据库中(http://www.ncbi.nlm.nih.gov/)。
3.1.1 通过功能基因组学研究黄曲霉毒素的生物合成 黄曲霉基因组编码超过13,000个功能基因,生物信息学分析表明,这些酶由多种基因编码。通过对黄曲霉表达的序列标签(EST)进行大规模测序,确定了与黄曲霉毒素生物合成、信号转导、总体调控、真菌致病性和应激反应相关的基因[58]。目前,黄曲霉毒素的生物合成基因簇是研究最为广泛的次级代谢途径之一(图5)。调控基因aflR编码AflR蛋白以促进黄曲霉毒素的合成。当aflR过表达时,黄曲霉毒素合成中的基因转录被上调,黄曲霉毒素的产生量增加[59]。alfS基因与aflR相互作用并促进黄曲霉毒素的生物合成。

图5 黄曲霉中的黄曲霉毒素途径基因簇
黄曲霉毒素的生物合成不仅受黄曲霉毒素合成基因簇上基因的影响,还受到多种因素的影响。laeA是黄曲霉二级代谢的全局调控因子。敲除laeA后,黄曲霉的疏水性丧失,黄曲霉毒素和其他次生代谢受到显著影响[60]。同时,laeA、VelB和VeA形成异源三聚体天鹅绒复合体,将光响应的发育调控与次级代谢的控制联系起来[61]。研究表明,分解代谢抑制基因creA调节黄曲霉毒素的形态,定殖和黄曲霉毒素的合成[62]。黄曲霉毒素的生物合成也受到外界环境因素的影响。与37 ℃相比,黄曲霉毒素合成簇基因aflR和aflS在28 ℃显著上调,促进黄曲霉毒素的产生[63]。温度和水活度的相互作用还可以通过调节黄曲霉毒素合成簇基因的转录来调节黄曲霉毒素的合成。与液体培养基相比,固体培养基中黄曲霉通过G蛋白信号传导途径、氨基酸代谢、菌核发育和氧化应激等途径蛋白的表达,从而促进黄曲霉毒素的生物合成[64]。
3.1.2 通过功能基因组学研究环匹阿尼酸(CPA)生物合成 环匹阿尼酸(CPA)是由青霉和曲霉真菌的二次代谢产生的吲哚-四甲酸霉菌毒素,可通过抑制肌浆网中钙依赖性ATP酶来增加肌肉收缩[65]。由于曲霉产生的CPA含量低,很少报道发生CPA真菌毒素事件,但黄曲霉毒素中毒可能会使CPA的作用变得更加复杂化[66]。CPA具有五环吲哚四羧酸支架,该支架由色氨酸、乙酰辅酶A、丙二酰辅酶A和焦磷酸二甲基烯丙基焦磷酸酯分子通过三种酶(CpaS、CpaD和CpaO)的连续作用而组成[67]。在黄曲霉中,CpaS催化了CPA生物合成中的第一个稳定中间体,即环乙酰乙酰基L-色氨酸(cAATrp)。cAATrp通过dmaT编码的环乙酰乙酰基乙酰甲基苯甲基二甲基烯丙基转移酶(CpaD)转化为β-CPA。maoA编码单胺氧化酶(CpaO)催化了α-CPA生物合成的最后一步[68]。通过敲除与CPA产生相关基因簇基因(maoA、dmaT、pks-nrps和ctfR1,发现除ctfR1敲除菌株外,所有类型的敲除菌株均丧失了产生CPA的能力[69]。在黄曲霉ΔveA突变体中,黄曲霉震颤毒素被完全阻断,CPA的产量同时大大降低[70]。laeA是真菌次级代谢途径的调节因子,其在烟曲霉中的过表达增加了CPA的产生[71]。CPA 还受到非生物因素(如温度和水活度)的影响。在25 ℃条件下,黄曲霉的CPA产量最高,CPA作为侵染农作物的有毒污染物的重要性被黄曲霉毒素污染带来的担忧所掩盖。需要更多的研究来阐明低水平的CPA对人类和动物健康的影响。
3.2 利用功能基因组学研究黄曲霉与植物的相互作用
真菌生长和黄曲霉毒素污染是宿主、真菌和环境之间相互作用的结果。花生作为最具重要商业价值的农作物之一,一直饱受黄曲霉毒素污染的严重困扰。RNA-seq是一种使用深度测序技术进行转录组学分析的方法,改变了人们对真核转录组范围和复杂性的认识。通过RNA-seq进行的初步分析可以表明蛋白质组学或代谢组学等其他组学技术的适用性或实用性[72]。在通过RNA-seq研究黄曲霉菌和花生(S和R基因型)之间的相互作用时,发现与R基因型花生相比,S基因型花生的黄曲霉毒素含量增加,差异表达基因(DEG)与菌丝体生长和穿透、分生孢子形成和发育有关[73]。在玉米和黄曲霉的相互作用中,涉及与囊泡、内核体和糖代谢基因相互作用的中心枢纽基因Ubi4与pH调节剂PalF和氮调节剂AreA的相互作用[74]。通过RNA-seq研究微生物对体外种子定殖(IVSC)的抗性,了解宿主与病原体之间的相互作用。通过RNA-seq鉴定与宿主-病原体相互干扰和标记有关的基因;通过基因工程(例如RNAi)培育抗性品种;通过多组学方法(如代谢组学、基因组学、转录组学和蛋白质组学)研究病原真菌的相互作用,获得前后的鉴定、分离和描述真菌毒素产生的能力;通过多组学方法,可以更准确地发现影响真菌发育、毒素产生以及真菌与植物相互作用的关键因素。找到相关基因后,可以通过本文中提及的基因编辑技术敲低或敲除相关基因来进行准确验证。
目前,大多数关于真菌与农作物之间相互作用的研究都停留在实验室阶段。基于实验室的理论研究可以被视为具有指导价值的理想模型。受多因素影响,大多数实验还旨在单个产毒素黄曲霉菌株的研究及其与宿主的相互作用。然而,田间真实的情况是复杂多样的,黄曲霉毒素的污染与多种真菌或细菌相关。通过功能基因组学研究黄曲霉不同菌株的相互作用以及黄曲霉与其他曲霉菌甚至其他病原菌的相互作用是未来的发展趋势。
4 展 望
近年来,功能基因组学研究迅速崛起,在真菌生长发育、挖掘真菌次级代谢产物以及研究包括黄曲霉毒素在内的真菌毒素等方面得到了广泛应用。然而,黄曲霉功能基因组学受到其细胞壁难以破除、耐药性高、选择标记少、缺陷型菌株构建费力耗时等因素的影响而发展缓慢。目前,在黄曲霉中,主要通过构建NHEJ缺陷型菌株来提高菌株的同源重组效率,从而提高其基因编辑的效率。但这种构建方式,不能解决选择标记少等问题,不利于连续的遗传学研究。正是由于缺乏选择标记,使得大多黄曲霉研究停留在单个基因的研究。而Cre-loxP重组系统具有可以删除选择标记的特征,可以连续构建多基因敲除菌株。同时,CRISPR-Cas9等基因编辑技术的出现,积极推动了功能基因组学的发展。目前,CRISPR-Cas9被广泛应用于米曲霉、黑曲霉、烟曲霉等曲霉属菌株构建中,在黄曲霉菌株中还未见相关报道。同时,功能基因组学在研究真菌与其他病原微生物的互作方面具有良好的发展前景。通过多组学方法,可以更准确地寻找影响真菌发育、毒素产生以及真菌与植物相互作用的相关基因。而影响真菌发育、毒素产生以及真菌在植物中的定殖往往并不是由单一基因调控的。找到这些相关基因后,可以通过本文中提及的基因编辑技术敲低或敲除相关基因来进行准确验证。同时,通过这些基因编辑技术可以更好地研究完整的代谢通路,为黄曲霉的遗传学研究提供参考。

