同源蛋白序列比对探讨AmPR-10核酸酶活性机理
王 珏,胡丽丽,李桂兰,吴 娜,张育敏
(山西中医药大学 基础医学院,晋中 030600)
蒙古黄芪病程相关蛋白(Astragalusmembranaceuspathogenesis-related protein-10,AmPR-10)是在其遭遇环境压力和病原体侵袭时表达的一种抵抗外部不利条件的蛋白。天然提取的AmPR-10具有核酸酶活性[1],且该活性很大程度是与植物防御病原体感染降解RNA类病毒有关[2]。而无论是天然黄芪中提取的AmPR-10还是研究中得到的外源重组蛋白,核酸酶活性都比较低[3]。因此进一步提高AmPR-10核酸酶活性,探究相关机理,对深入了解黄芪生长发育抗病机制具有重要意义。目前,已知PR-10类蛋白的核酸酶活性可能与一段保守的P-loop环(GxGGxGxxK)相关[4],然而也有很多具有该结构的PR-10类蛋白被证明并没有核酸酶活性,这一特征可能不具备必然性[5]。因此需探索可能影响AmPR-10核酸酶活性的新的保守区及特定氨基酸位点,为目的基因进化改造提供参考。
我们对AmPR-10及其同源蛋白进行了序列比对,并将AmPR-10空腔处(该区域已被证实具有配体结合活性[6])与核苷酸单体进行分子对接,由对接结果设计氨基酸定点突变,并根据突变后AmPR-10与配体对接结合自由能变化判断可能对酶结合底物有影响的氨基酸位点。首次对影响AmPR-10核酸酶活性的蛋白保守区及特定氨基酸做出深入研究,为其他PR类基因外源表达及活性分析提供理论依据。
1 材料与方法
1.1 材料
1.1.1 菌种材料
2019年夏季6月采摘蒙古黄芪嫩叶(山西中医药大学种植),长约10 mm,宽约6 mm,将叶片剪碎称取80 mg左右,用于RNA的提取;感受态E.coliBL21购于生工生物工程(上海)有限公司用于重组子转化。
1.1.2 工具酶及载体
PCR聚合酶、限制性内切酶EcoR I、XhoI、T4DNA快速连接酶购于生工生物工程(上海)有限公司;克隆表达载体pET-30a由山西中医药大学基础医学院实验中心提供。
1.1.3 试剂盒及相关试剂
细菌基因组提取试剂盒、RT-PCR试剂盒、质粒提取试剂盒、凝胶纯化试剂盒、Ni2+纯化蛋白试剂盒、IPTG、Trizol试剂、酵母tRNA均购于生工生物工程(上海)有限公司。
1.2 方法
1.2.1 重组子pET30a-AmPR-10的构建和表达
从蒙古黄芪叶片中提取总RNA,通过RT-PCR进行目的基因的扩增,扩增引物以GenBank 公布的AmPR-10编码序列为模板进行设计(Gene ID:ASA69247.1),上游引物:5′-CGCGAATTCATGGGTGTTA-TAAGTTTCA-3′(划线部分为EcoR I酶切位点);下游引物:5′-CGCCTCGAGCTTGTATTCAGGATTGGCC-3′(划线部分为XhoI酶切位点)。扩增产物与载体pET30a进行连接、转化、筛选得到目标重组子,经测序确定基因序列正确。随后使用IPTG诱导目标蛋白表达,通过SDS-PAGE检测目标蛋白表达条带。
1.2.2 AmPR-10外源蛋白的纯化及核酸酶活性测定
使用Ni2+纯化蛋白试剂盒纯化1.2.1表达的外源蛋白AmPR-10,首先对IPTG诱导后的重组菌进行超声破碎获得胞内粗蛋白液,然后利用商业购买的Ni-IDA树脂,对目标蛋白进行提取纯化:将粗蛋白液与缓冲液按1∶1比例混合后过柱,利用组氨酸标签与树脂上Ni+的螯合作用,使AmPR-10吸附于树脂柱上,最后通过洗脱液将目标蛋白洗脱,获得AmPR-10纯蛋白(以上实验过程需在冰上操作,以保证维持目标蛋白活性)。
检测纯化后AmPR-10的核酸酶活性,测定方法如下:称取2 mg酵母tRNA溶于 1 mL缓冲液(100 mmol/L MES,pH 6.0)中,加入100 μL蛋白纯化液,50 ℃保温30 min。反应完成后立即加入1 mL预冷的氯化锂终止反应,冰浴3 h,12 000 r/min,4 ℃离心15 min,取上清液260 nm 处测定其吸光度,以未加酶液的样品为对照[3]。酶活定义:在50 ℃,pH 6.0反应条件,2 mL反应体系中,在30 min内使A260吸收值变化1.0的酶量定义为一个活性单位U(参照商品酶Benzonase核酸酶活性定义),通过考马斯亮蓝法检测纯蛋白的浓度,换算得AmPR-10的比活(U/mg)。
1.2.3 AmPR-10同源蛋白序列比对
(1)通过Protein BLAST获得AmPR-10的同源蛋白序列,结合文献报道,选择已证实具有核酸酶活性的蛋白,使用Clustal X软件进行序列比对,找出AmPR-10在进化过程中的保守区域。(2)选择PSIPRED,对AmPR-10进行二级结构分析,分析(1)中的保守区域在二级结构上的分布特征。
1.2.4 AmPR-10与核酸单体分子对接
对AmPR-10进行SWISS-MODE建模(http://swissmodel.expasy.org/),获得PDB文件。利用AutoDock分子对接软件将AmPR-10蛋白三级结构与核酸单体小分子(PubChem:6030)进行对接。然后通过所提供的半经验自由能结果来评价对接过程的能量及稳定性[7]。根据野生型AmPR-10与核苷酸单体的对接结果,找出配体结合位点,随后对这些点的氨基酸进行定点突变,并将突变型AmPR-10与核苷酸单体进行对接,通过比较蛋白突变前后与同一种小分子对接的自由能变化,分析AmPR-10展现核酸酶活性的关键氨基酸位点,并寻找出可能提高AmPR-10核酸酶活性的突变点。
2 结果与分析
2.1 重组子pET30a-AmPR-10的表达及核酸酶活性测定
蛋白纯化结果(图1)显示,AmPR-10由158个氨基酸组成,蛋白分子质量为16.8 ku[8]。外源基因连接在载体pET30a上,其中用于纯化的融合标签His-tag为6个氨基酸。通过SDS-PAGE检测纯化后AmPR-10大小,在约17 ku处有目标条带。采用1.2.2所述方法,测得外源表达的AmPR-10对酵母tRNA的水解活性(核酸酶活性)为1.1 U/mg。
2.2 AmPR-10同源序列比对及结构分析
通过NCBI BLAST找到与AmPR-10氨基酸排列顺序同源性较高的一系列蛋白,结合相关文献[9-11]确定每一条同源蛋白是否具有核酸酶活性,筛选出7条同源蛋白信息,具体见表1。

表1 与AmPR-10同源且具有核酸酶活性的病程相关蛋白Table 1 The proteins with nuclease activity and homologous to AmPR-10
其中基因标识为蛋白在NCBI数据库中的信息识别序列。将表1中的8条蛋白信息进行氨基酸排列顺序的同源比对,具体结果见图2。
如图2所示,比对结果分为(a)~(f)6个部分,以AmPR-10(NCBI ID:ASA69247.1)为研究对象(图中红色标注),该病程相关蛋白与其他蛋白保守性较高的区间主要体现在14~24位、44~55位、64~70位、83~88位。在此基础上,研究对AmPR-10的二级结构进行分析,结果如图3所示。由图3可以看出:14~24位氨基酸位于第一个α螺旋处,44~55位氨基酸包括2个连续的β折叠部分序列及中间的β-转角,P-loop环则集中在46~51位(GNGGXG);64~70、83~88位氨基酸分别分布于2个β折叠结构上。而较长的α螺旋由130~153位氨基酸构成,保守性不强,相对灵活。

图2 AmPR-10同源蛋白氨基酸序列比对Figure 2 Amino acid sequences alignment of AmPR-10 homologous proteins

图3 AmPR-10二级结构分析结果(黄色标识代表β折叠结构,粉色标识代表α螺旋结构)Figure 3 The result for the 2D structure of AmPR-10
2.3 AmPR-10与核苷酸单体分子对接
为进一步探讨2.2中得到的保守序列氨基酸在AmPR-10水解核酸过程中的角色及作用,研究将AmPR-10与核苷酸单体进行分子对接,结果如图4所示。其中图4(a)表示对接整体示意图,核苷酸单体与AmPR-10的空间结构疏水腔进行对接;图4(b)表示对接结果形成氢键情况,分别在83位Tyr及140位Gly形成氢键(图中绿色短线标识)。
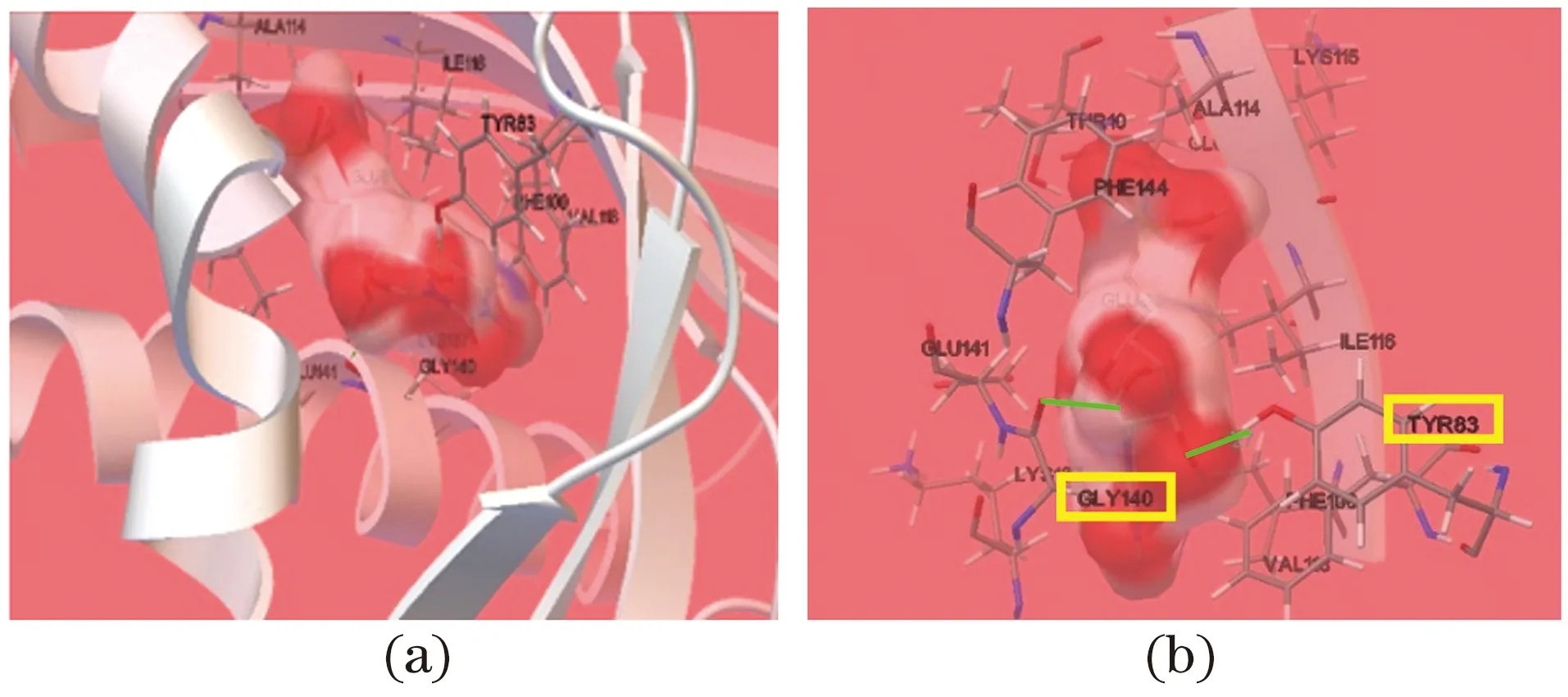
图4 野生型AmPR-10与核苷酸单体分子对接结果Figure 4 Docking results of wild type AmPR-10 with nucleotide monomer
结合2.2中的分析,83位Tyr是位于AmPR-10第4段保守区上的氨基酸位点,研究将其分别突变为其余19种氨基酸,再将突变体蛋白与核苷酸单体分子对接,发现蛋白与配体的结合自由能均增大;而140位氨基酸位于C-末端α螺旋上,该区域保守性差,同理将140位Gly突变为其余19种氨基酸,并进行突变体蛋白与配体的分子对接,发现当突变为Asn、Trp及Val后,蛋白与配体的结合自由能会明显降低,具体见表2。
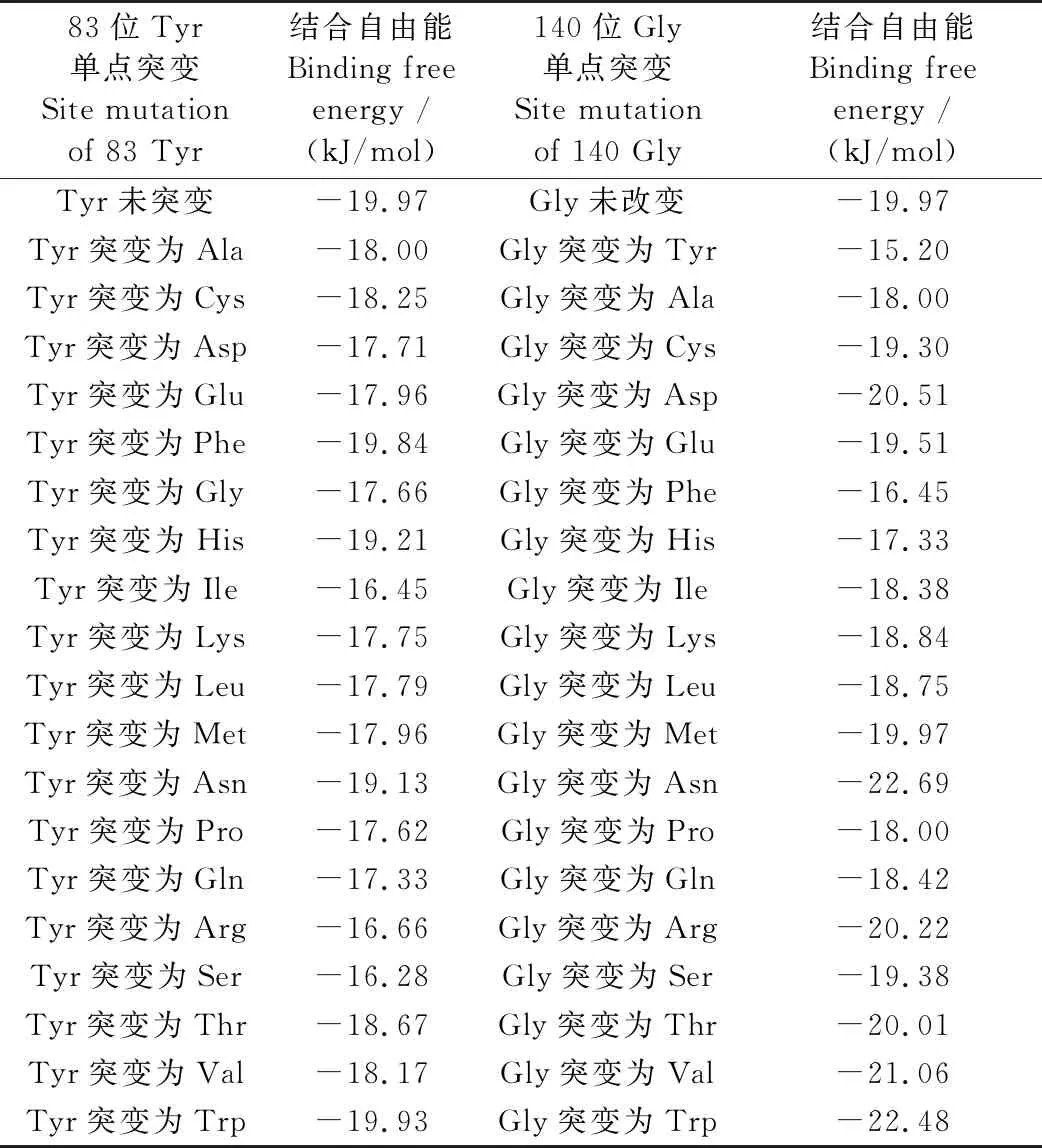
表2 AmPR-10野生型及突变型与核苷酸单体对接结合自由能结果Table 2 The results of docking energy for wild type and mutants AmPR-10 with nucleotide monome
3 讨论
3.1 AmPR-10 N末端保守α螺旋在三级结构中的意义
由2.2中的基因序列比对结果可知,AmPR-10有4段最明显的保守区,集中于前100位氨基酸,而C末端的α螺旋保守性不强,该部分结构犹如手柄状,对AmPR-10的空腔形成遮蔽的作用,参与PR-10类蛋白与配体的结合,进而影响目标蛋白的生物学功能[6],这说明若对AmPR-10核酸酶活性进行改造,C端α螺旋的可操作性更强。而从AmPR-10的三级结构可以看出[图5(a)],该区段与严格保守区14~24位氨基酸构成的第一个α螺旋在空间上距离很近。因此,研究通过chimera软件对14~24位氨基酸逐一分析后发现:14位Val与151位Val、152位Leu的距离在空间上<5Å[图5(b)];19位Leu与144位Phe的距离在空间上<5Å[图5(c)];22位Ala与147位Leu、151位Val的距离在空间上<5Å[图5(d)];23位Leu与144位Phe、147位Leu的距离在空间上<5Å[图5(e)]。这些近距离氨基酸全部为疏水性氨基酸,并且均朝向蛋白空腔一侧,这是利于蛋白稳定的,因此考虑这些氨基酸可能通过形成局部的一个较强疏水区而有助于维持AmPR-10空腔的结构,进而利于目标蛋白的生物学活性。

图5 N端α螺旋空间构象及特定氨基酸位点分析Figure 5 Analysis of spatial conformation and specific amino acid sites for α-helix in N-terminal
3.2 AmPR-10 特定氨基酸位点对核酸酶活性的意义
由2.3中的分子对接结果发现,参与空腔形成的保守位点83位Tyr突变为其余19种氨基酸后,结合自由能均提高,这说明蛋白与配体的结合稳定性下降[12],而空腔结构的轻微改变可能会致使PR-10蛋白在植物防御过程中发挥多种功能[13],故83位Tyr可能是AmPR-10发挥核酸酶活性抵御病毒侵袭的重要氨基酸位点,而这也与Tyr本身具有共轭π电子体系,可在分子识别过程中起重要作用的观点相一致[14];另一个与核苷酸单体形成氢键的氨基酸是位于非保守区的140位Gly,将其突变为其余19种氨基酸后,发现当突变氨基酸是Asn、Val、和Trp时,目标蛋白与配体的结合自由能明显下降,表明整体稳定性更高,具体如图6所示。图6(a)显示140位氨基酸为Asn时,配体与Asn形成一条氢键;图6(b)显示140位氨基酸为Val时,配体与81位Tyr、23位Leu、140位Val形成3条氢键;图6(c)显示140位氨基酸为Trp时,配体与81位Tyr,83位Tyr,27位Ser形成3条氢键。这一结果显示可能由于单点氨基酸的改变,使得AmPR-10空腔构象有不同变化,从而与配体形成不同的结合方式。
以上分析指出对提升AmPR-10的核酸酶活性,C末端α螺旋的基因进化空间较大,这一结论也与基因序列比对结果相一致:保守区域氨基酸可能是AmPR-10具有核酸酶活性的关键位点(如83位),非保守区域氨基酸灵活性更高,可作为进一步提升目标蛋白性能的参考位点(如140位)。
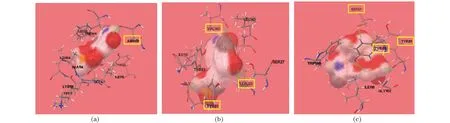
(a)140位Gly突变为Asn,突变蛋白与核苷酸单体对接示意图;(b)140位Gly突变为Val,突变蛋白与核苷酸单体对接示意图;(c)140位Gly突变为Trp,突变蛋白与核苷酸单体对接示意图 (黄色标注为目标蛋白与配体分子对接后,与配体形成氢键的氨基酸;绿色标注为所形成的氢键位置)。图6 140位氨基酸突变后蛋白分子对接示意图 Figure 6 Schematic diagram of protein molecular docking after 140-amino acid mutation
4 结论
研究检测外源表达的AmPR-10核酸酶活性为1.1 U/mg,考虑通过基因进化进一步提高此活性。目前国内外对黄芪病程相关蛋白的活性研究非常少,而核酸酶也因种类较多,蛋白催化机制没有明确统一的解释[15]。现已知PR-10蛋白具有两个特征:(1)具有P-loop环结构;(2)具有结合配体活性的空腔结构。在此基础上,研究通过对同源蛋白基因序列比对及分子对接试验对AmPR-10的核酸酶活性机理进行了更为深入的探讨。
研究发现:(1)AmPR-10除了P-loop环结构以外,还存在3段保守的区域,并且第一段保守区N端α螺旋结构与C末端的α螺旋在空间上距离很近,经过氨基酸位点逐一分析,在空间上距离小于5Å的氨基酸均是疏水性氨基酸,全部朝向空腔侧,通过形成较强的疏水区而稳定AmPR-10的空间结构,有利于蛋白在抵抗防御过程中发挥作用。(2)通过分子对接及氨基酸定点突变,发现位于第4段保守区域的83位Tyr可能是AmPR-10具有核酸酶活性的关键氨基酸位点,其改变会引起蛋白与核苷酸单体对接整体的不稳定;而位于保守性较差的C-末端α螺旋上的140位Gly经突变为Asn、Val和Trp后,分子对接的结合自由能下降,因此考虑该区域结构的氨基酸可作为基因进化的参考位点。据此,研究找出了AmPR-10具有核酸酶活性的独立特征、关键氨基酸位点以及可进行定向进化的可操作区域,这一结果为后续目标蛋白活性提升的研究提供理论依据;另一方面也为类似催化活性机制不明确的蛋白提供一种新的研究思路和策略。

