双片段连接法克隆融合基因及其突变体
王 媛,程盈盈,马梦琪,梁明星,陈华波
(湖北文理学院 基础医学院,襄阳 441053)
将两个或多个基因的编码区首尾相连,置于同一套调控序列控制之下,即为融合基因。克隆融合基因是研究工作中的常用手段。一则自然融合基因广泛存在于诸多肿瘤细胞中[1-2],它本身就是重要的研究对象;二则人工融合基因可为研究工作提供诸多便利。如在基因两端分别连接不同生色团,辅以荧光共振能量转移等技术可研究目标蛋白在细胞内的动态变化[3-4];或者将两个功能基因融合在一起,以研究其特殊功能或调节机制[5-6]。将功能基因连接GFP、GST等标签简单易行,不在本文讨论之列。
构建融合基因的方法主要有基于II型限制性内切酶的酶切-连接法,非典型酶切-连接技术,重叠延伸PCR(overlap extension PCR,OE-PCR)融合法,Gibson组装等,这些方法各有利弊,须结合实际案例选择具体方案。其中常规双酶切-连接法操作简单,技术成熟。由于体外连接的低效性,往往需要将一个基因片段连接插入载体后进行筛选,然后再进行第二次连接。每次连接反应都需要两个合适的内切酶,因此两个基因片段顺序连接,依次插入载体常受制于严格的酶切位点要求而难以实行。我们发现双片段连接法可突破上述僵化的酶切位点限制,极大拓宽酶切-连接法拼接基因片段的适用范围。
周期蛋白E(cyclinE)是周期蛋白家族重要成员,主要通过激活周期蛋白依赖性激酶2(cyclin dependent kinase 2,CDK2)促进细胞周期G1/S转换[7],此外还可能通过非典型方式在细胞质内发挥作用[8-9]。为研究cyclinE/CDK2复合物在中心体复制中的作用机制,需克隆cyclinE-CDK2融合基因及其突变体。通过PCR在CDK2 序列5′端接入连接序列(linker),将cyclinE与linker-CDK2序列一起连接插入质粒载体构建融合基因;采用相似策略,以cyclinE或CDK2突变体替换融合基因中对应野生型序列,构建融合基因突变体。本研究对常规酶切-连接法作适当改进,以期提供一种克隆融合基因及其突变体的简便方法。
1 材料与方法
1.1 菌种与质粒
感受态大肠杆菌DH5α(CB101)购自天根生化科技(北京)有限公司;pEGFP_C2质粒(V800-20)购自Thermo Fisher。
1.2 试剂
PrimeSTAR Max DNA Polymerase (R450A),DNA Ligation Kit Ver.2.1 (6002)购自Takara;BamHⅠ(R0136V),EcoRⅠ(R0101V),MluⅠ(R0198V),SalⅠ(R3138V)购自NEB;DNA回收试剂盒(BSC02M1/BSC03M1);质粒小量抽提试剂盒(BSC01M1)购自杭州博日科技有限公司。
1.3 引物设计与合成
引物采用Primer Premier 5.0软件设计,由北京天润奥科生物科技有限公司合成,经PAGE纯化。引物序列见表1。

表1 引物序列Table 1 Primer sequences
1.4 基因克隆
通过PCR将linker接入CDK2需合成一条约70 nt的引物。考虑到化学合成长引物易出错且价格昂贵,故设计两条略有重叠的嵌套引物。PS1匹配CDK2基因5′端;PS2匹配PS1补齐linker序列并添加SalⅠ位点。PA匹配CDK2基因3′端并添加BamHⅠ位点。PCR方案:先将常规浓度的PS1引物1∶1 000稀释,采用PrimeSTAR Max DNA Polymerase高保真DNA聚合酶。25 μL 2×Premix,0.5 μL PS2/0.5 μL PS1/0.5 μL PA,1 ng pEGFP_C2-CDK2质粒,补水至50 μL。运行程序:94 ℃预变性5 min;94 ℃变性30 s,58 ℃ 退火30 s,72 ℃延伸1 min,30个循环;72 ℃再延伸10 min。预期产物为SalⅠ-linker-CDK2-BamHⅠ。酶切方案:取PCR回收物17 μL(或相应质粒1 μg稀释到17 μL ddH2O中),加2 μL 10×buffer,0.5 μL酶1/0.5 μL酶2,37 ℃孵育4 h。PCR及酶切产物经电泳检测后回收至50 μL ddH2O中。连接方案:采用DNA Ligation Kit Ver.2.1试剂盒。载体0.5 μL,两片段各3.5 μL,solution I 7.5 μL;16 ℃孵育30 min。连接产物直接转化感受态大肠杆菌DH5α,涂布于卡那霉素-LB固体培养基平板上,37 ℃培养过夜。
1.5 阳性克隆的筛选与鉴定
从过夜平板上随机挑取数个菌落,置于4 mL卡那霉素-LB培养基中继续培养8 h。取3 mL菌液小量提取质粒进行酶切:7.5 μL质粒DNA,2 μL 10×buffer,0.5 μL内切酶,补水至20 μL,37 ℃孵育3 h后电泳检测。阳性质粒测序由北京天润奥科生物科技有限公司完成。采用DNAMAN 4.0软件进行序列比对与限制性酶切分析。
2 结果与分析
2.1 融合基因克隆方案
在前期工作中已构建了pEGFP_C2-cyclinE和pEGFP_C2-CDK2质粒(图1)及其激酶活性缺失突变体cyclinEM(188-192A)和CDK2M(T160A)[10-11]。现欲克隆cyclinE-CDK2融合基因,在两基因之间还需插入一段编码Gly-Gly-Thr 5次重复的连接序列(linker)以减少蛋白质空间结构的相互影响[6]。linker为一段45 bp的重复性序列,以之为桥梁,通过OE-PCR融合cyclinE与CDK2基因易因错配而产生缺失突变。
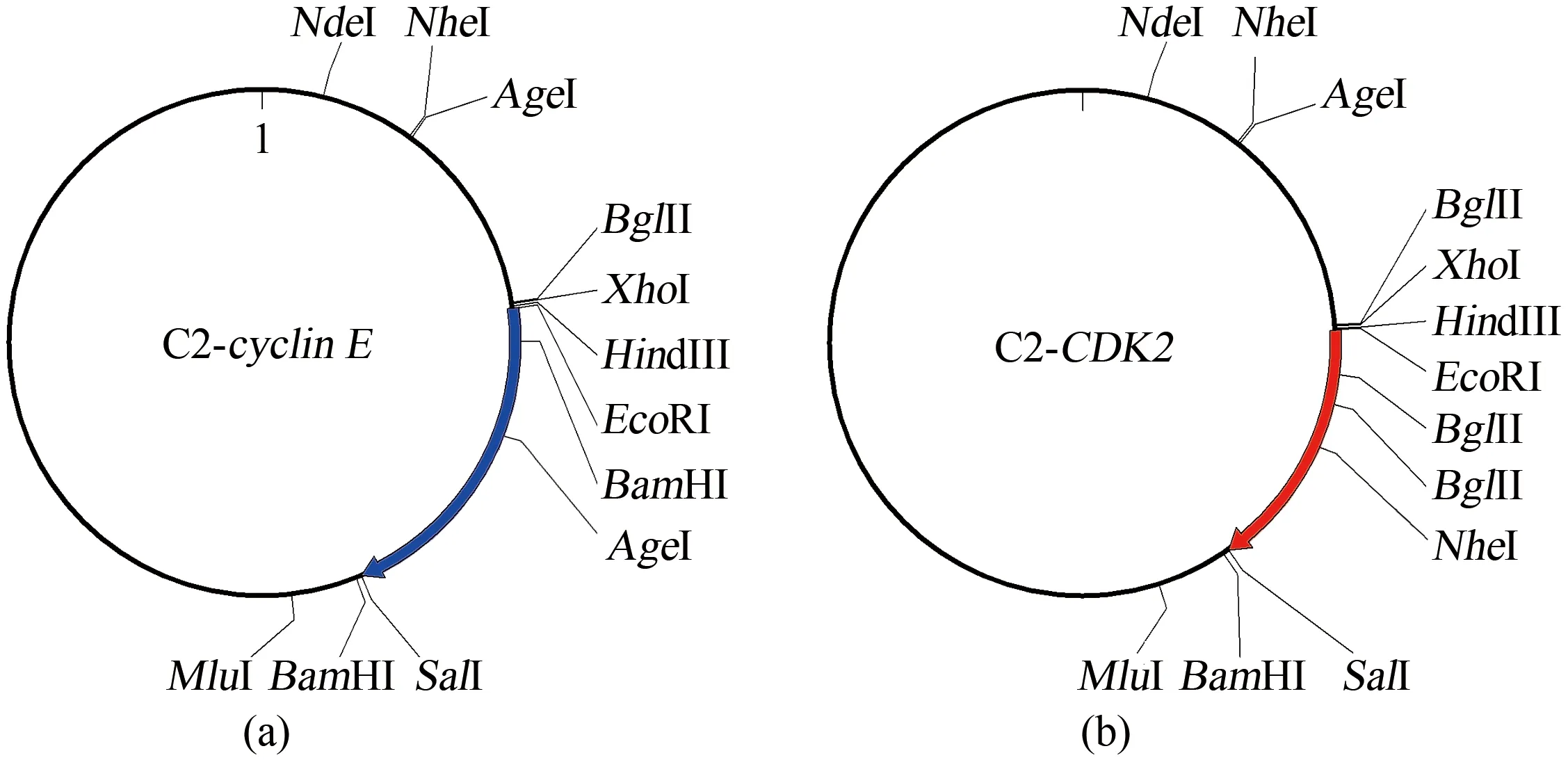
图1 原始质粒酶切图谱Figure 1 Restriction map of original plasmid
设计含linker序列的引物,通过PCR将其接入CDK2序列5′端,并在两端分别添加SalⅠ与BamHⅠ位点以便基因拼接。由于cyclinE序列内还有另一BamHⅠ位点,linker-CDK2序列无法直接插入载体。故先用EcoRⅠ/SalⅠ将cyclinE序列酶切出来,连同linker-CDK2序列一起连接插入载体,可以绕过双BamHⅠ位点对顺序连接的限制。若克隆含cyclinEM(188-192A)突变体的融合基因,仍可按上述方案进行,且linker-CDK2序列无需再次PCR扩增,直接从经序列鉴定的融合基因重组质粒中酶切出来即可。

图2 cyclin E-CDK2融合基因克隆过程示意图Figure 2 Schematic diagram of cloning process of cyclin E-CDK2 fusion gene
2.2 融合基因的克隆与筛选
以pEGFP_C2-CDK2为模板,PCR扩增得到linker-CDK2片段[图3(a)],再以SalⅠ/BamHⅠ双酶切得到一个基因片段[图3(b),1泳道]。pEGFP_C2-cyclinE质粒以EcoRⅠ/SalⅠ双酶切得到cyclinE基因片段[图3(b),2泳道],pEGFP_C2质粒以EcoRⅠ/BamHⅠ双酶切作为载体[图3(b),3泳道]。双片段连接产物转化感受态大肠杆菌,经筛选得到十余个菌落[图3(c)]。任意选取8个克隆小量提取质粒,分别以NheⅠ或BamHⅠ单酶切检测,得到3个阳性克隆[图3(d)和(e)]。经序列测定与比对,3个克隆都完全符合预期融合基因序列[图3(f)]。

(a)PCR扩增linker-CDK2序列;(b)酶切获取基因片段及载体,箭头指示需回收的DNA片段;(c)过夜平板上的成型菌落;(d)和(e)平板菌落酶切检测结果;(f)3个阳性克隆与目标序列比对结果。图3 克隆cyclin E-CDK2融合基因Figure 3 Clone cyclinE-CDK2 fusion gene
2.3 融合基因突变体克隆方案
欲克隆pEGFP_C2-cyclinE-CDK2M(T160A)突变体,用OE-PCR或滚换扩增等方法都可能会引入随机突变。从pEGFP_C2-CDK2M质粒中切取突变序列置换融合基因中对应序列可避免上述风险。由于两种质粒中CDK2序列3′端酶切位点不同,故利用EcoRⅠ/MluⅠ将CDK2M连同其后的一段序列(F2)一起酶切。重组质粒中cyclinE5′端尚有另一个EcoRⅠ位点,故CDK2M--序列无法直接插入载体。利用BamHⅠ/EcoRⅠ从融合质粒中将cyclinE大部分连同linker序列(F1)酶切,两片段一起连接载体部分可重构完整融合质粒。此融合质粒CDK2末端为SalⅠ位点,不同于原融合质粒,但CDK2末尾含TGA终止密码,不会改变融合蛋白的C端序列。

图4 cyclin E-CDK2M融合基因克隆过程示意图Figure 4 Diagram of cloning process of cyclin E-CDK2M fusion gene
2.4 融合基因突变的克隆与筛选
将pEGFP_C2-cyclinE-CDK2质粒以BamHⅠ/MluⅠ双酶切得到251、2 065及4 578 bp 3个片段,其中最大的片段即所需载体部分[图5(a),1泳道];将pEGFP_C2-cyclinE-CDK2质粒以BamHⅠ/EcoRⅠ双酶切得到125、903、1 162 bp及4 704 bp 4个片段,其中1 162 bp即为所需的F1片段[图5(a),2泳道]。它与903 bp片段相距不远,需谨慎分离回收;将pEGFP_C2-CDK2M质粒以EcoRⅠ/MluⅠ双酶切得到1 175 bp与4 453 bp 2个片段,其中1 175 bp即为所需的F2片段[图5(a),3泳道]。F1与F2片段一起连接载体部分,再转化感受态大肠杆菌,筛选得到数十个克隆[图5(b)]。任选8个克隆,对其质粒分别以NheⅠ或SalⅠ单酶切检测,结果一致显示皆为阳性[图5(c)和(d)]。由于新、旧融合质粒CDK2序列末端酶切位点不同,SalⅠ单酶切阳性可确保目标质粒含CDK2M突变体。新融合质粒中各部分都未经PCR扩增,一般无需测序鉴定。
3 讨论
采用基于Ⅱ型限制性内切酶的酶切-连接技术克隆融合基因有着天然优势,该方法可将基因片段视作独立的DNA元件进行灵活替换,该方法最大的障碍是不易找到合适的酶切位点以实现基因片段的顺序连接。我们对常规酶切-连接法作适当改进,采用了两种不同的双片段连接方案,成功克隆了cyclinE-CDK2融合基因及其突变体。为避免受酶切位点限制,研究者也尝试用某些非典型酶切-连接技术拼接DNA,如依赖于同尾酶的BioBrick技术和依赖于IIs型限制性内切酶的Golden gate克隆技术[12-14]。两者的共同弊端是会将拼接片段“焊死”,无法进行后续DNA元件的灵活替换。OE-PCR融合法不受酶切位点限制,但在此有3点缺陷:OE-PCR受上、下游DNA片段长度等诸多因素影响,一般融合长基因片段成功率较低;涉及体外DNA扩增的PCR可能引入随机突变,须全基因测序加以验证;融合基因的linker序列具有重复性,以之为重叠序列的OE-PCR易产生缺失突变。Gibson组装也涉及体外DNA合成[15],遇到的问题与OE-PCR法相似。多片段顺序连接对酶切位点及其顺序有严格要求,须3个酶切位点在基因片段与载体MCS上顺序一致才能保证两个DNA片段依次连接插入载体[图6(a)]。双片段连接法极大拓宽了酶切-连接法的适用范围,具体表现在以下两种情况:其一是载体MCS上切点b在a、c外侧或没有b切点[图6(b)],如本例中克隆融合基因突变体;其二是待拼接的DNA片段上有多余酶切位点会干扰顺序连接[图6(c)],如本例中克隆融合基因。上述两种情况都无法顺序连接,但双片段连接法则不受影响。双片段连接法还可用于克隆一些序列较长而不易直接获取的基因,如多片段拼接法克隆长基因,OE-PCR法克隆长基因定点突变,序列置换法克隆系列截短基因突变体等。相对常规单片段连接,双片段连接时得到的转化克隆数较少,且阳性率偏低[16]。在筛选目标克隆时,可适当增加检测菌落数,并以多种方案相互佐证检测结果。在尝试双片段连接法之前应掌握娴熟的单片段、双酶切连接实验操作技巧。

(a)酶切获取载体及基因片段(其中1泳道251 bp及2泳道125 bp条带弱,不易见,箭头指示需回收的DNA片段);(b)过夜平板上的成型菌落;(c)和(d)平板菌落酶切检测结果。图5 克隆cyclin E-CDK2M融合基因Figure 5 Clone cyclinE-CDK2M fusion gene
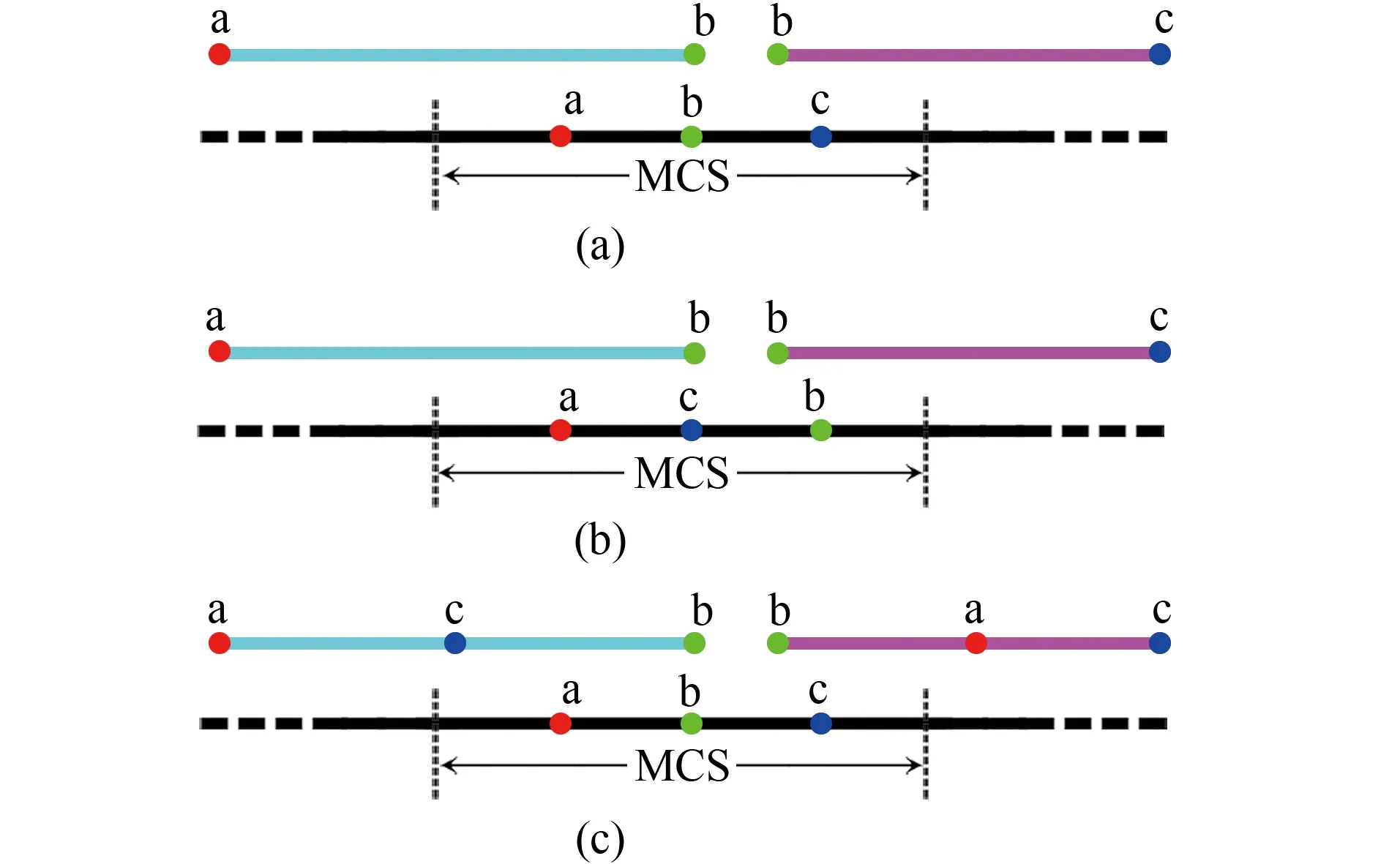
图6 双片段连接法拓宽酶切-连接法在DNA拼接中的适用范围Figure 6 Double fragments ligation expanded the application of enzyme cleave-ligation in DNA assembly

