基于加权基因共表达网络分析筛选全身炎症反应综合征相关急性肾损伤基因*
刘新园, 阳 光, 程 青△
1泰康同济(武汉)医院急诊科,武汉 430050 2中部战区总医院老年病科,武汉 430070
全身炎症反应综合征相关急性肾损伤(SIRS-associated acute kidney injury,S-AKI)是危重患者常见的并发症,有着极高的死亡率[1-4]。全身炎症反应综合征和急性肾损伤(acute kidney injury,AKI)虽然为独立的疾病,但二者常互为因果[5]。由于全身炎症反应综合征本身复杂而独特的病理生理机制,使得S-AKI与其他原因诱导的AKI不同。我们常难以通过临床常见指标来判断患者在发生全身炎症反应综合征期间何时出现S-AKI,导致难以及时干预预防肾损伤。本研究拟通过对全身炎症反应综合征发生和未发生急性肾损伤患者的基因芯片数据进行再分析整合寻找出与S-AKI发生和严重程度相关的分子标记物,从而为今后的研究和临床治疗提供线索。
1 研究方法
1.1 GEO数据集获取
本研究从Gene Expression Omnibus(GEO)数据库(https://www.ncbi.nlm.nih.gov/geo/)中筛选样本数大于100,且为全身炎症反应综合征合并AKI和不合并AKI的临床结果数据集,筛选到了数据集GSE67401(下载时间:2020.12.28)。其中包括发生全身炎症反应综合征不合并AKI患者58例,全身炎症反应综合征合并1级AKI 36例,全身炎症反应综合征合并2/3级AKI 17例,需进行血液透析治疗患者22例。依据GEO数据库中提供的注释信息,对上述数据集进行注释、数据集标准化处理后获得可以进一步分析的数据。
1.2 基因共表达网络构建(weighted gene co-expression network analysis,WGCNA)和模块筛选
WGCNA是一种常用的基因共表达网络筛选技术,已被广泛应用于识别和筛选各种疾病生物标记物[6]。我们对其步骤进行简要描述,利用R软件中的“WGCNA”分析包构建基因共表达网络[7-8],据此获得每个基因之间的关联性,并构建基因之间相关矩阵和拓扑重叠矩阵,用于度量基因的网络连通性[9-10]和确定软阈值大小。将表达量相似基因归为一个基因模块中,并进行连锁层次聚类[7-8]。同时计算每个模块在数据集中权重大小,筛选权重最大数据集,用于后续研究。
1.3 核心基因筛选
我们对上述筛选得到权重最大的GREEN模块进一步分析以便寻找模块中核心基因。首先利用STRING网站对模块中所有基因进行蛋白与蛋白之间的互作分析(PPI),并利用Cytoscape软件中的MCODE插件和MCC评分对模块中基因进一步评分筛选(筛选MOCDE评分>10分,MCC评分排序前50位)。同时利用“STEM(Short Time-series Expression Miner,基因表达趋势分析)(版本:1.3.12)”软件对数据集中所有基因进行趋势表达分析,并聚类。进一步对数据集中发生和未发生AKI的患者进行差异基因分析,综合上述4个结果筛选出有效的生物标记物,CANT1和RAB37。
1.4 GO(Gene Ontology)功能学、KEGG(Kyoto Encyclopedia of Genes and Genomes)通路分析和GSEA分析
为了解GREEN模块主要涉及的功能学和通路。采用标准富集计算方法进行GO功能分析和KEGG通路分析用以筛选与其相关的功能和通路。为了解CANT1和RAB37在全身炎症反应综合征相关急性肾损伤患者中的潜在功能,利用本研究所纳入的数据集进行GSEA分析。其注释基因集c2.cp.kegg.v7.2,选择GMT通路数据库作为参考基因集。将P<0.05和|富集评分(ES)|>0.3作为截止标准,并选择两个基因集中一致的通路作为其可能影响通路。
1.5 统计学方法
统计分析采用SPSS 24.0软件、GraphPad Prism 7.0和R 3.4.1,同时用上述软件进行图像生成处理。t检验用来分析两个组别之间均数的差异。绘制受试者工作特征ROC曲线,评估生物标记物的预测能力,利用曲线下面积AUC评估敏感性和特异性。以P<0.05为差异具有统计学意义。
2 结果
2.1 构建共表达模块
基于18619个基因在133例全身炎症反应综合征或者进行了血液透析患者中的表达数据,利用基于无序列网络的WGCNA方法,首先对软阈值进行筛选,结果如图1所示,当软阈值选择为9时即可获得较好的连接关系。将基因进行模块化富集分析,结果如图2。本研究一共筛选相应的基因模块并对其进行权重累计分析(图3),最后筛选获取了GREEN模块,GREEN模块共含有1193个基因。
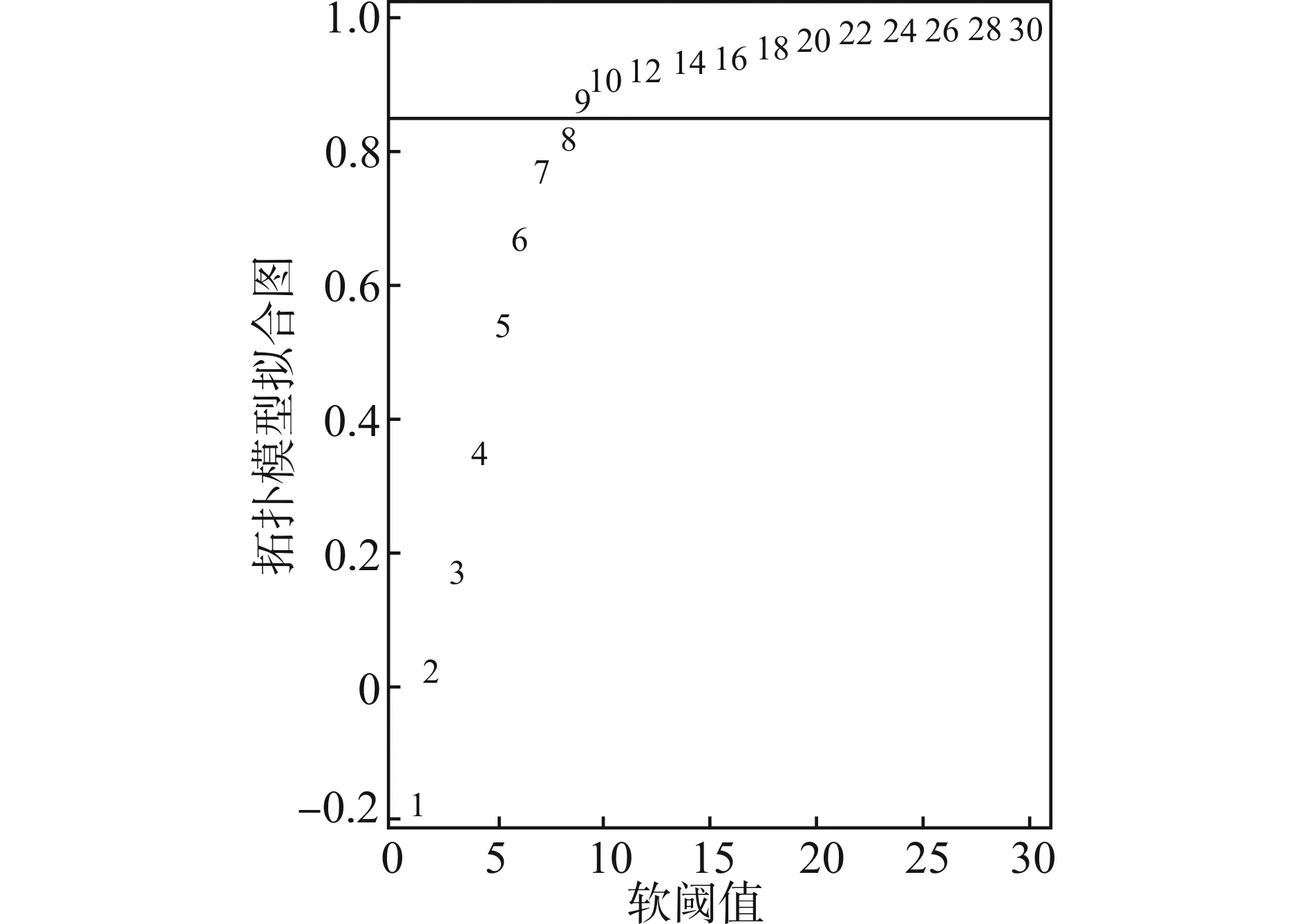
图1 软阈值图

图2 WGCNA模式图

图3 模块相关热图
2.2 GREEN模块相关功能分析以及核心分子的筛选
为寻找GREEN模块中主要富集的细胞功能和通路,基于GO和KEGG富集分析,本研究依据GREEN模块中所含有的1193个相关基因筛选表达响应基因参与的信号通路的关联图(图4A、4B)。结果提示:自噬、细胞因子释放和细胞形态调控等功能和信号通路与GREEN模块相关。为了解GREEN模块中基因之间的内部关联性,我们依据STRING网站数据,对GREEN模块中的基因进行蛋白互作分析,并利用Cystacape软件中MOCDE插件和MCC评分对蛋白互作进行评分,筛选位于调控核心位置的基因(图5)。
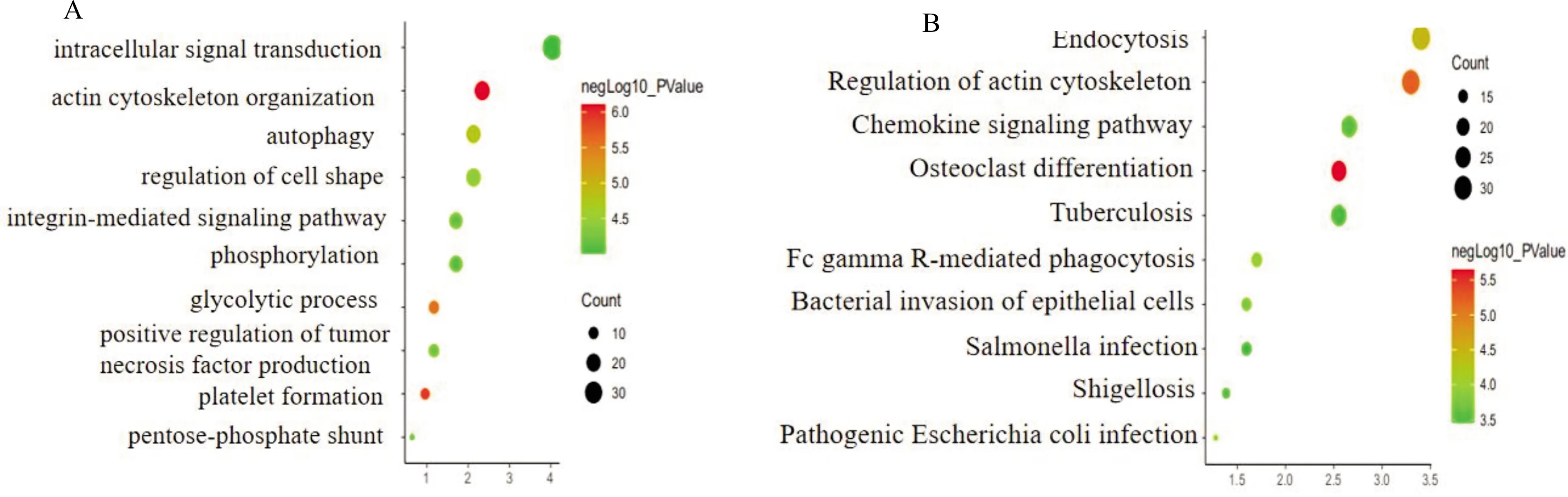
A:GO分析;B:KEGG分析

图5 GREEN模块中核心基因蛋白互作分析
2.3 依据STEM软件对数据集基因进行聚类分析
在急性炎症反应综合征患者中,部分合并有急性肾损伤,发生急性肾损伤患者中部分患者损伤较轻,部分患者甚至需要行血液透析治疗,因此我们不仅需要寻找发生肾损伤的分子标记物,还需要寻找到与肾损伤发生严重程度相关的分子标记物。利用STEM软件和数据集GSE67401中的临床信息,将患者依据AKI发生程度分为0(无损伤)、1(AKI=1)、2(AKI=2/3)、3(血液透析患者)四级。将数据纳入进行分析,共获得了50个聚类(图6),并依据临床信息结合基因表达聚类的结果,我们将第“1”、“9”和“11”基因聚类结果纳入分析(图7)。
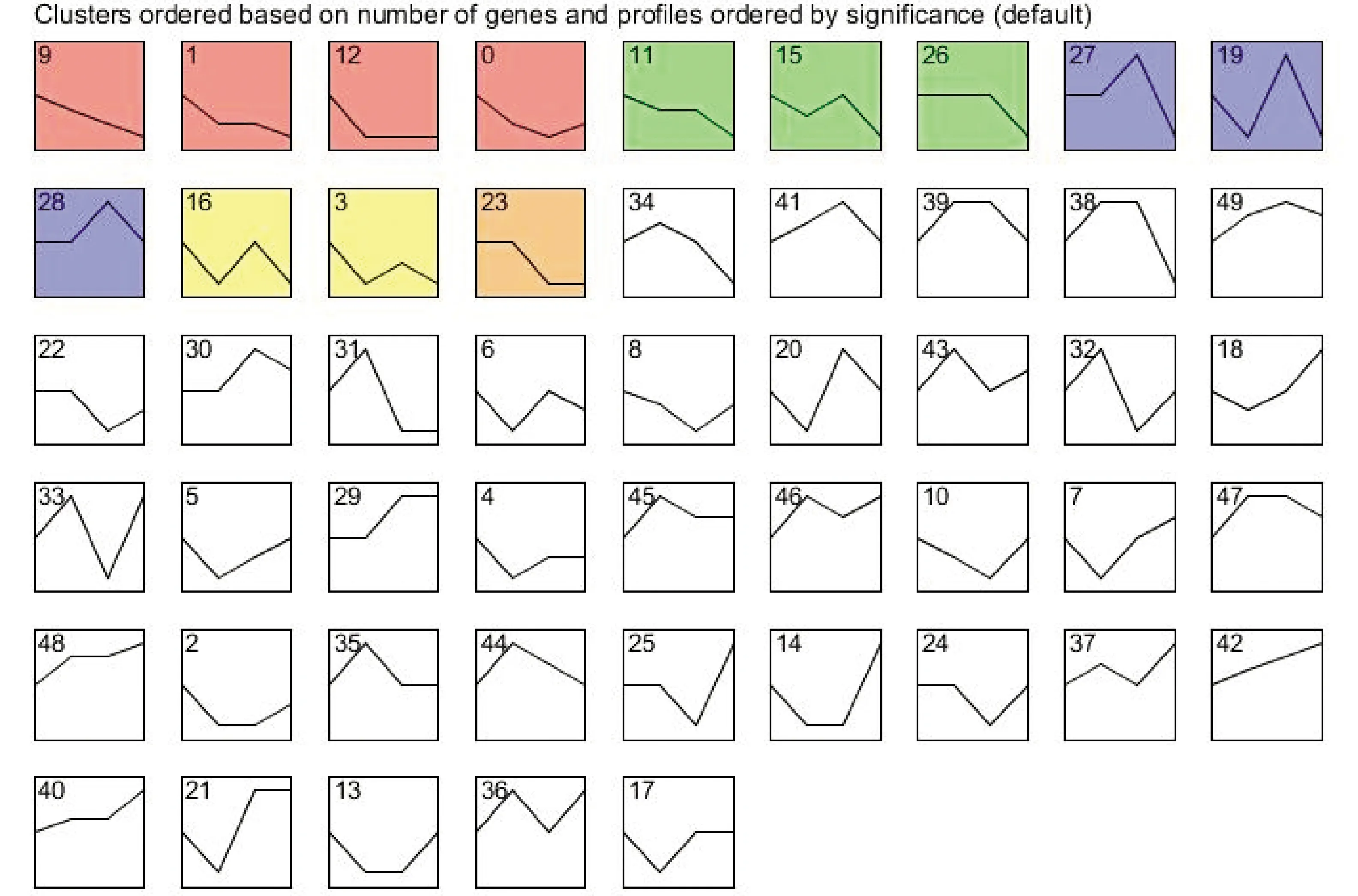
图6 STEM聚类图

A:“聚类1”;B:“聚类9”;C:“聚类11”
2.4 有效的生物标记物筛选和验证
基于上述3个评分手段,为使我们筛选的核心分子更为准确,我们对数据集中合并和未合并AKI的患者筛选差异基因,并将上述4种方法所筛选的结果综合分析绘制VENN图(图8),最后筛选出钙激活核苷酸酶1(calcium-activated nucleotidase1,CANT1)和RAS癌基因家族37(member RAS oncogene family 37,RAB37)为有效的生物标记物。
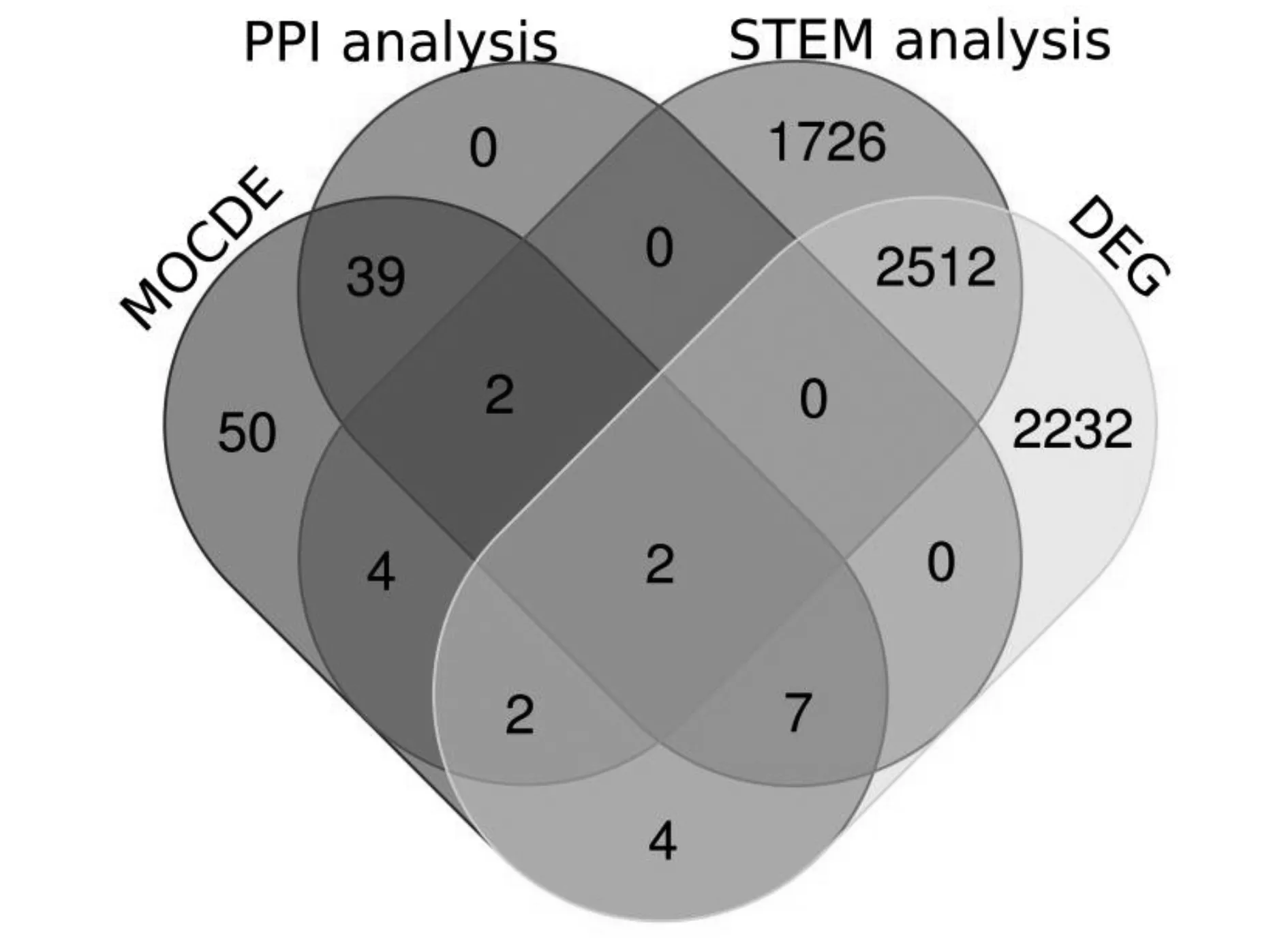
图8 VENN图展示筛选结果
验证所筛选的基因在全身炎症反应综合征合并和未合并AKI患者中表达是否存在差异性,结果如图9所示,CANT1和RAB37基因在未发生AKI组中的表达较发生AKI患者高表达(CANT1:非AKI组,117.3±7.3vs.AKI组,90.6±6.1;P<0.01。RAB37:非AKI组,9.036±0.032vs.AKI组,8.857±0.067;P=0.02)。同时在受试者工作曲线(ROC)分析中,发现CANT1和RAB37基因对于全身炎症反应综合征患者是否发生AKI具有较好的预测能力(图10)。其在预测全身炎症反应综合征患者发生AKI的ROC曲线下面积(AUC)分别为(CANT1:AUC=0.65,P<0.01;RAB37:AUC=0.61,P=0.04)。进一步分析其在预测S-AKI患者是否需要血液透析治疗中的价值,结果如图11所示(CANT1:AUC=0.69,P<0.01;RAB37:AUC=0.65,P=0.03)。相关性分析提示,CANT1和RAB37基因表达和AKI级别显著相关(CANT1:R2=0.72,P<0.01;RAB37:R2=0.78,P<0.01,图12)。

A:CANT1基因表达;B:RAB37基因表达

A:CANT1预测AKI发生;B:RAB37预测AKI发生

A:CANT1预测S-AKI患者是否需血液透析;B:RAB37预测S-AKI患者是否需血液透析

A:CANT1基因表达与AKI分级相关性分析;B:RAB37基因表达与AKI分级相关性分析
2.5 GSEA功能学分析
为了解CANT1和RAB37基因主要涉及何种功能调控,本研究进一步分析了GSE67401数据集并采用GSEA分析其所涉及的下游通路。结果如图13所示,CANT1和RAB37基因主要涉及溶酶体代谢通路。

A:CANT1基因;B:RAB37基因
3 讨论
全身炎症反应综合征相关急性肾损伤是宿主对感染反应失调的结果。全身炎症反应综合征患者发生急性肾损伤时,如果临床干预处置得当,其肾脏功能是可能恢复的。因此,如何快速确定患者是否发生了急性肾损伤或者可能即将要发生急性肾损伤是目前临床上亟待解决的问题。因此发现合理可靠的生物学标记物将对临床诊治提供较大的帮助。本研究基于近年来快速发展的高通量测序技术,筛选获得数据库中全身炎症反应综合征相关急性肾损伤患者的数据,利用近年来被广泛应用的基因共表达网络分析(weighted gene co-expression network analysis,WGCNA)方法,避免了基于传统差异基因筛选方法只对数据集局部研究,极易遗漏调控过程中的核心分子和难以对生物系统整体探索研究的缺点。通过系统地绘制个体生物互作网络图可以精准地寻找出与预后相关的核心分子[8,10]。本研究基于此方法,并结合了基因表达趋势分析筛选获得核心基因CANT1和RAB37。并利用多角度分析验证了其可靠性。
CANT1是一种cDNA与三磷酸腺苷二磷酸酶cDNA同源的糖基化蛋白,主要通过水解UDP发挥生物学效应[11]。CANT1在人体组织中广泛表达,大量研究表明,CANT1的突变会引起一种常染色体隐性遗传疾病Desbuquois dysplasia(DBQD),主要表现为生长迟滞、关节松弛、四肢短小、进行性脊柱侧突等症状,体现了CANT1对正常生长发育的重要作用[12-15]。RAB37是Ras小GTP酶超家族成员,负责细胞内信号转导和囊泡运输。近期研究发现,RAB37直接与自噬相关基因5(ATG5)结合[16],以GTP依赖的方式促进自噬小体的形成,从而诱导自噬,抑制肿瘤生长[17]。RAB37的表达和活性受多种途径的调控。在鼻咽癌和肺癌中,RAB37被证实是通过其启动区的高甲基化来下调表达[18-19]。同时,RAB37作为一种GTPase蛋白,与GDP结合时是不活跃的,与GTP结合后变为活跃的形式[20]。Kim等[21]报道,蛋白激酶C-α(PKC-α)磷酸化RAB37的苏氨酸172位点,解除RAB37的GTP结合位点,从而抑制RAB37的活性。目前尚无研究报道CANT1和RAB37在全身炎症反应综合征发生中的作用和与全身炎症反应综合征相关急性肾损伤的关联性。
本研究通过对GEO数据库中S-AKI患者数据集GSE67401进行分析,通过WGCNA算法和STEM软件获得趋势基因集,识别出核心分子CANT1和RAB37可能与全身炎症反应综合征相关急性肾损伤发生和急性肾损伤程度相关。筛选获得的CANT1和RAB37基因可能具有较强的可靠性。为验证我们所筛选的基因是否真实可靠,进一步利用GSE67401分析,发现CANT1和RAB37基因在未发生AKI患者中较发生AKI患者中显著增高。同时ROC分析结果也证实了上述两个基因能预测全身炎症反应综合征患者是否发生AKI。AKI患者中需行血液透析患者常为分级严重的患者,因此若能早期识别S-AKI患者是否会进展到需要行血透治疗,并予以早期干预将会获得较好的临床效果。本研究进一步验证了上述的CANT1和RAB37基因在预测患者是否需接受血液透析治疗上具有较强的可靠性,这个结果与我们前期结果相符。本研究应用多种网络互作识别和基因趋势分析筛选获得CANT1和RAB37,为进一步了解二者涉及的信号通路和功能之间是否存在关联性,我们利用GSEA分析获得了二者均可能涉及溶酶体通路。溶酶体是细胞内重要的信号转导中心,它可以感知细胞内的营养及能量状态,并调节细胞的生长和代谢。因此,溶酶体作为细胞内的降解、循环以及信号转导中心,维持着细胞的稳态。本研究结果提示S-AKI患者可能由于炎症风暴等因素导致内环境紊乱,引起全身炎症反应综合征和S-AKI也可能涉及了溶酶体功能。这也为临床治疗提供了一个新的思路。
总之,本研究采用WGCNA和STEM聚类等生物信息学方法,利用GEO数据库研究了全身炎症反应综合征相关急性肾损伤患者的相关基因,并发现了CANT1和RAB37的低表达与全身炎症反应综合征相关急性肾损伤发生相关,以及CANT1可以较好地预测全身炎症反应综合征相关急性肾损伤是否需行血液透析治疗。上述的CANT1和RAB37有可能成为新的分子标记物,有助于全身炎症反应综合征患者个性化治疗及临床干预判断。

