DNA数字信息存储的研究进展
董一名,孙法家,武瑞君,钱珑
(1北京大学定量生物学中心,北京 100871;2中国生物技术发展中心战略与政策处,北京 100039)

随着人类对世界的观测向着更高精度和更大广度发展,多样化、微型化、动态化传感器的发明和普及,人类数据量保持指数甚至超指数形式增长,“天文数字”这一概念被不断颠覆[1]。如今,在科研领域,观测太空的阿塔卡玛大型毫米阵列每天会增加2 TB的观测数据;在健康领域,数字人体和数字医疗涵盖了个人健康数据、临床大数据和运营数据各种类型,全球医疗保健数据已达到2.26 ZB;此外,金融、工业生产、安防等领域的网络化、实时化已成为现代社会的标配,这些领域的数据以人口为基数、以秒为时间单位不断积累。依据国际数据公司(International Data Corporation,IDC)的估计,2025年全球数据产出量将会达到175 ZB(1 ZB≈1.18×1021B)[2],而当前主流存储介质的生产已经不堪重负[3]。海量数据的拷贝和传输也面临挑战。按民用光纤传输速率1 Gbps估计,PB(1PB≈106GB)量级的数据交流花费的时间远长于物理运输,而后者产生了大量非必要成本。除此之外,现有存储介质不可避免地随着读写次数和自然时间发生损耗,导致每年数以亿计的信息维护费用。因此,实用的新型数据存储介质亟待开发,以应对信息爆炸式增长的挑战。
脱氧核糖核酸(DNA)是生物体用于存储遗传信息的载体。通过A、T、C、G四个碱基,DNA存储了物种的全部遗传信息并且稳定遗传给后代,我们的身高、肤色、虹膜等信息都被记录在小小的细胞中,基因组和中心法则称得上是自然界最精妙绝伦的信息存储与传递算法[4-5]。DNA同样具有存储数字信息的潜力。数据可转化为碱基的线性顺序,编码在DNA这种新型信息存储介质中。最引人注目的是DNA的信息存储容量和存储密度,研究表明,DNA信息存储密度可以达到1019bit/cm3,是硬盘的106倍[6-7]。此外,DNA稳定性强,存储时间长,并且无需频繁维护。化石中的DNA平均半衰期估计为521年[8];利用一些特殊的材料如合成二氧化硅或者凝胶则可以保存更久的时间[9-10]。利用生物化学手段可以便利地对信息进行复制(PCR方法)、切割(限制性内切核酸酶)和粘贴(DNA连接酶)等。这些特性使得DNA分子成为一种理想的新型数据存储介质。
1 DNA数据存储的研究进展
1.1 DNA信息存储流程简述
使用DNA分子进行信息存储,可以分为信息编码、DNA合成(写入)、DNA测序(读取)和信息解码四个步骤,如图1所示。
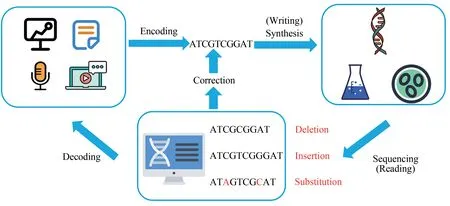
图1 DNA信息存储流程[11]Fig.1 The process of DNA-based information storage[11]
首先必须将信息转换为DNA分子中4种碱基的序列。在信息科学领域,不同的数据类型有不同的编码和压缩算法,常用的算法有霍夫曼编码、算术编码、字典编码等[12-13]。此外,对于DNA分子而言,在合成、复制、测序的过程中都可能发生错误[14],物理冗余和逻辑冗余可以在信息失真的情况下恢复原始数据,也就是纠错码[15-16]。图2分别展示了信息直接转换、线性分组码[17-20]、喷泉码[21-22]和卷积码[23-27]的原理。
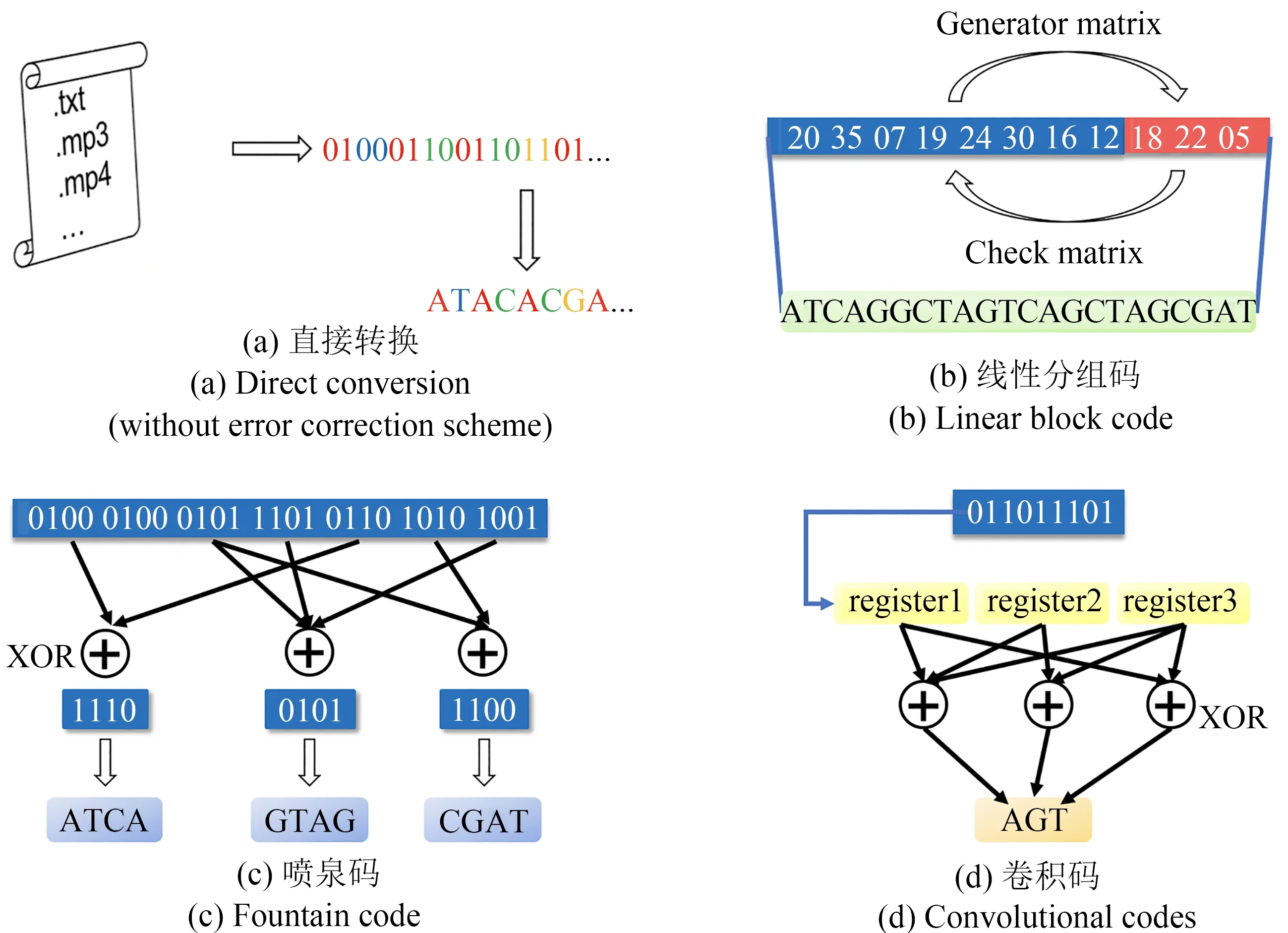
图2 DNA存储研究中使用的信息编码方法(前向纠错体系)[(a)直接转换,不包含纠错方案。在这种方案中,数据被读取为数字流,然后转换为DNA序列。例如,Church等[28]和Goldman等[29]分别将二进制数字流和三进制数字流中的每一位转换为一个DNA碱基。(b)线性分组码,即通过线性运算,从原始信息(信息码元)产生用于纠错的冗余(称为“校验码元”或“监督码元”)。在解码时,与生成矩阵相对应的校验矩阵可以用于校验接收到的信息中是否包含错误,并进行纠正。(c)喷泉码,即将原始信息转换为大量较短的信息,这些较短的信息并非原始信息的一部分,而是将原始信息中的符号通过特定的分布进行异或运算得到的。在解码时,只要获得了足够数量的短信息,就可以恢复原始信息。(d)卷积码,即“有记忆”的编码方案。在编码用于传输的符号时,不仅需要处理当前的信息符号,还要对当前位置之前的数个信息符号进行运算]Fig.2 Information encoding method(forward error correction system)used in DNA storage research[(a)Direct conversion without error correction scheme.In thismethod,thedata isread asadigital stream and then converted into DNA sequences.For example,Church et al.[28]and Goldman et al.[29]converted each bit in a binary number stream and aternary number stream into a DNA base,respectively.(b)Linear block code,namely,generating redundancy for error correction(called"check symbols"or"supervision symbols")from the original information(information symbols)through linear operations.In thedecoding process,thecheck matrix corresponding to thegenerator matrix can beused to check whether thereceived information containserrorsand then correct them.(c)Fountain code,which convertstheoriginal information into alargenumber of shorter sequences.Theseshorter sequencesarenot part of the original information,but obtained by performing XORoperationson thesymbolsin the original information according to aspecific distribution.In thedecoding process,aslong asasufficient number of shorter sequencesareobtained,theoriginal information can be restored.(d)Convolutional codes,that is,coding schemes"with memory".Both thecurrent information symbol and several information symbolsbeforethecurrent position areused to generatetheencoding symbols]
在编码之后,进行DNA合成,即写入。三代DNA合成技术——化学合成法(固相亚磷酰胺化学法)[30-31]、微阵列DNA合成法[32]和酶合成法[33]的演化大大减少了DNA合成的时间和成本。另外,基因组装和编辑技术的发展让我们可以灵活而准确地改变遗传信息,并在活细胞中进行信息的处理和储存[11],为DNA信息存储的发展提供了有利的条件。
信息的读取依靠基因测序技术。自1977年第一代DNA测序技术(Sanger法)出现以来[34],测序技术已获得了巨大的发展。相比于最初,其成本下降了十万倍[35]。通过测序恢复碱基序列,根据编码原则可以预判信息恢复能力。在得到DNA序列信息之后,将碱基序列重新转换为二进制序列,此后,再利用编码的纠错原理将序列自动纠错,就可以得到原本的数字信息。
1.2 DNA信息存储发展史
关于DNA分子的认知始于19世纪70年代Miescher和Kossel等的研究[36-37],然而直到1953年Watson和Crick在Nature上发表了“Molecular Structures of Nucleic Acids”一文,人们才对DNA分子的结构有了清晰的认识[4]。同一时期Avery等[38]和Hershey等[39]的研究证实了DNA分子是生物体存储遗传信息的载体。后续的一些研究使人们认识到,生物体的遗传信息就存储在组成DNA分子的4种核苷酸的线性排列中。4种碱基的特定排列蕴藏了生物的遗传信息。
这些研究成果自然而然引发了使用DNA分子存储人工数据的猜想和尝试。然而,受限于当时尚不成熟的DNA合成和测序技术,这些尝试未能获得成功。直到1996年,Davis才将包含35个像素点的黑白图像信息编码到DNA分子,导入到大肠杆菌中并成功读取出来[40]。到了2001年,Bancroft等将《双城记》开篇的两句名言编码到了DNA分子中,使用的方法与DNA编码蛋白质序列的“密码子”方法类似[41]。在2012年和2013年,Nature和Science分别刊发了哈佛医学院Church等[28]和欧洲生物信息研究所Goldman等[29]在DNA数据存储领域的研究成果。与早期研究不同,两组研究都存储了可观的数据量。Church等的研究在DNA分子中存储了659 KB的数据,而Goldman等存储了739 KB。这两项研究的成功有赖于DNA合成和测序技术的巨大进步,使得合成与读取数以万计的DNA分子成为可能。
在这两项研究之后,DNA数据存储领域的新进展如雨后春笋般涌现出来。在2015年和2016年,Grass等[42]和Blawat等[43]的两项研究把信息科学领域的“前向纠错码”引入DNA数据存储领域,使在合成和测序过程中发生错误时,信息依然可以被恢复出来,从而提升了使用DNA分子进行数据存储的可靠性。2016年,Bornholt等[44]设计实现了DNA存储体系中数据的“随机访问”(random access)。2017年,Erlich等[45]将“喷泉码”引入了DNA编码体系中,称为“DNA喷泉”,实现了较高的数据存储密度。同年,Shipman等[46]将一部电影信息通过CRISPR技术编码到了活细胞中。2018年,Organick等[47]在DNA分子中存储了多达200 MB的数据,实现了大规模体系中的随机访问,并尝试使用单分子测序(single molecule sequencing,SMS)进行数据的读取和恢复。
2020年,Erlich和Grass将喷泉码运用于信息存储[48],他们提出了一个“万物皆可存储DNA信息”概念(DNA-of-things,DoT)。作者将3D打印的兔子——斯坦福兔子的设计蓝本信息转换为DNA序列,合成寡核苷酸片段,然后将这些短片段封装在大小为160 nm的二氧化硅纳米颗粒中,与可降解热塑性聚酯混合用于3D打印。信息的读取和复制也非常简便,从兔子耳朵处剪下一小块进行溶解,就可以得到其中的DNA,进而进行测序和扩增,得到的信息还可以进行下一代兔子的3D打印。最终,研究人员完美地复制和打印了五代兔子,展示了DNA作为信息存储介质的稳定性和保真性。此外,他们还将1.4 MB大小的视频编码存储到眼镜的树脂玻璃中。在这项研究中,他们同样使用了“DNA喷泉”,即使用LT码应对错误[49-50]。
2020年,Press等[51]开发出了一种能够处理DNA合成和测序错误中出现的增删(indel)错误的DNA编码算法,称为“HEDGES”。这种算法使用了RS码和卷积码进行编码,并使用树结构进行解码。基于HEDGES编码,他们合成了5865条长度为300 bp的寡核苷酸,这些DNA分子之后被人工引入了突变和增删错误并在Illumina平台上测序。解码结果表明,在牺牲一定编码密度的情况下,HEDGES能够处理总计约1.2%的增删错误。这种算法为应对更复杂的DNA错误类型提供了借鉴,从而保障DNA分子存储信息的稳健性。与传统的信息存储方式利用磁性存储介质(磁盘)、光学存储介质(光盘)和电子存储介质(内存、U盘)相比,DNA读写速度慢并且过程烦琐。很多研究人员致力于实现全自动DNA信息存储。微软公司和华盛顿大学搭建了一台基于柱式合成和三代测序的全自动DNA存储和读取设备,存储与读取“hello”的整个过程需要21 h[52]。尽管还有很长的路要走,但信息存储和读取的自动化对于DNA存储的产业化意义巨大。
可以看出,研究人员将DNA分子存储领域与DNA合成与测序技术、细胞生物学与分子生物学技术、信息科学与通信技术等领域不断交叉融合,为这一领域的未来描绘出更多的可能性,不断提高DNA分子的存储潜力,使得DNA数据存储越来越接近于生产和生活实际。
2 DNA信息存储的优势
2.1 存储密度
磁性存储介质利用磁性介质的电磁效应进行信息存储。光学存储介质将信息刻录在光盘表面的凹槽中,再通过激光读取,数据量越大要求激光的精度也越高。物理设备的工作分辨率决定了这些传统介质的极限密度。而碳基生物分子的存储密度在分子尺度,与传统介质相比,具备天然的优势。
理想情况下,DNA分子的存储密度可达约460 EB/g,这意味着仅需要数克的DNA分子即可存储全世界一年所产生的信息。DNA具有双螺旋立体结构,单位空间的数据密度非常高。由于不能无限地紧密堆积,体积密度更能够代表DNA分子实际数据存储能力。据估算,每立方厘米的DNA分子可以存储大约1 EB的信息,这一密度是当前存储密度最高的介质(闪存)的1000倍,是硬盘数据存储密度的百万倍[6]。即便因为封装、冗余等实际因素无法实现最大存储潜力,其可用的存储密度依然远远高于当前主流的数据存储介质。
天然DNA分子包含四种碱基,因此每一个碱基最多可以存储2 bit的信息。然而,也有一部分研究工作试图扩展碱基系统,即使用DNA分子中的四种天然碱基之外的“人工碱基”或“非天然碱基”来存储信息,从而提高DNA分子的信息存储密度。非天然碱基的工作起源于20世纪80年代[53],而在近几年有了较大的突破,目前已经实现了8个碱基的系统[54-56]。
除了使用额外的非天然碱基,也有一些研究使用“简并碱基”来扩展DNA分子的存储密度。在2019年,有几项不同的研究成功使用简并碱基进行数据存储,并且提升了存储密度。具体而言,简并碱基将DNA序列中每个位置的序列空间连续化,即表示为四种碱基的混合体系。例如,Anavy等[57]在其研究中定义了两个新的碱基符号:M,是等量A和T的混合体;K,是等量G和T的混合体。加入这两个符号之后,DNA分子中的每一位就包含了6个“碱基”,因而可以容纳2.58 bit的信息。这一碱基体系可以继续扩充,以包含更多的“简并碱基”符号,从而进一步提升DNA分子的存储潜力。在Anavy等的研究中,他们尝试使用更大的碱基空间存储较小规模的信息(22.5 B),并实现了每合成轮4.29 bit的存储密度。Choi等[58]也提出了类似的思路,并使用包含15个“碱基”的系统存储了854 B的信息,实现了每个DNA 3.37 bit的存储密度。
除了DNA之外,其他碳基存储介质也展现了信息存储能力。中国科学院上海微系统与信息技术研究所的陶虎教授课题组发明了基于蚕丝蛋白的生物存储器[59],每平方英寸可以存储64 GB数据信息(1平方英寸=6.4516×10-4m2),并且可重复擦写。蚕丝蛋白和DNA相似,可耐受异常湿度、辐射和磁场等环境。蚕丝蛋白也可以用于存储生物体DNA等生物样品,有望未来和DNA介质结合,用于数字存储。尽管其存储密度依旧受限于光学写入设备的分辨率,但展现了学术界对于碳基介质用作信息存储的认可。而代谢分子(糖类、氨基酸等)更小,也可以用作信息存储。布朗大学Kennedy等[60]受DNA存储的启发,利用代谢分子液滴在金属板点阵列存储图片等信息。与简并碱基的思想类似,他们利用对代谢组分分布的测量实现了更高维度空间中的信息编码。
尽管碳基存储尤其DNA在密度上有很大优势,考虑到随机访问所需的稀溶液条件和分子扩散速率,一个1 L的DNA存储池中可容纳的信息量被限制在TB~ZB量级[11]。因此,一个值得关注的概念是“Storage-on-Chip”。存储硬件体系的设计需要适配这些实际考量,超大规模的数据存储离不开存储体系的创新。
2.2 数据维护
传统的数据存储介质总会自发地发生损耗,导致信息损坏或丢失。硬盘和闪存能够存留信息的年限不超过十几年。在传统数据存储介质中维护大量数据需要极其高昂的成本。例如,如果一个数据中心要在磁带上存储109GB数据,需要高达十亿美元和十年以上的时间来建造和维护,以及上亿度电的耗费。
而DNA分子在适当的条件下具有极高的稳定性,可以保障存储在其中的信息不会受损。地质学家手中的化石为DNA分子的数据存留能力提供了有力的证明——有时可以获取甚至数十万年前化石中的DNA分子并读取其序列信息。如果将DNA分子保存在合适的环境中,其序列甚至可以存留更长的时间。例如,Grass等[42]将固态DNA分子封装在二氧化硅中,表现出了比纯固态DNA粉末和其他存储介质更好的存留特性。他们推算出了封装在二氧化硅小球中的DNA分子的一级降解动力学活化能,并由此推测在相同条件下其可在9.4℃下存留2000年,或在-18℃下存留200万年。
同时,相比传统介质,使用DNA分子进行数据存储几乎不需要维护成本。使用DNA分子存储109GB数据用电量不足0.1 W。如此之低的维护成本使得DNA分子尤其适用于存储大规模不需要经常访问的“冷数据”。
2.3 体内信息存储潜力
迄今为止,大多数DNA存储的尝试都是在体外进行的,例如DNA寡核苷酸池(oligo pool),或者对DNA片段进行物理封装以进一步增强存储稳定性(图3)。在当前的技术水平下,体外存储在存储成本(短片段存储、无需连接成长片段,也无需导入质粒或者基因组中)、DNA刻写(活细胞DNA在刻写时需要避开功能基因及其相关序列等)、DNA读取(二代测序技术比较成熟)和稳定性(活细胞DNA突变)等方面有较强的优势。

图3 DNA信息存储的载体Fig.3 The carrier of DNA-based information storage
尽管如此,越来越多科学家将目光投向了DNA体内存储。活细胞的基因组DNA由于其耐久性和生物功能相容性,已成为信息存储的另一潜在形式。与体外DNA存储相比,体内存储利用了细胞自身DNA复制和校对的机制,也提供了微尺度随机数据访问的实用手段[47]。除此之外,极端环境微生物在信息存储的能耗等方面有很大的发展空间。
对于DNA体内存储,研究人员首先将视线投向质粒(图3),因其操作简便、编辑和写入较简单。质粒DNA存储可以追溯到1996年,Davis[40]在大肠杆菌质粒中存储了小维纳斯女神“Microvenus”的图片。此后,很多研究人员将文本、音乐、图片信息存储到了质粒上。
但是存储量和遗传稳定性问题限制了质粒作为信息存储载体的应用,基因组作为替代选择成为了新型的体内存储方式。2010年的一项合成生物学里程碑式研究中,Venter团队[61]通过化学合成法合成了整个支原体的基因组,并证实其具有生物活性和复制能力。此外,他们在该合成基因组中加入了很多“水印信息”,包括作者名字、研究所信息和诗句等。这也是基因组存储信息的首次尝试。2017年,Shipman等[46]通过CRISPR技术将“奔跑的马”五帧视频存储到了群体细胞的基因组中,利用大肠杆菌传代进行数据的复制,证明视频可以在传代中比较稳定地保存下来。
基于体内DNA存储的信息保真和信息传代潜力,研究人员尝试利用DNA序列信息作为标签,来跟踪实验结果、信息流动,甚至进行物流追踪,该技术统称为“DNA条形码”(DNA barcoding)[62]。美国Springer教授提出了“BMS”技术,通过设计DNA条形码进行组合,并且将其整合到枯草芽孢杆菌和酿酒酵母孢子的基因组中,通过喷洒转移到接触的物体上实现痕迹追踪。DNA条形码的识别,可以利用SHERLOCK、RPA、Cas13a和测序等方法实现,从而进行食品等的物源追踪[63],还可以结合CRISPR技术追踪序列,研究肿瘤生长和癌症演化等动态过程[64]。这些概念展示性工作提示了体内DNA存储与细胞传感、细胞处理器等新型生物技术的可能接口。除了纳米物联网和疾病检测,DNA存储在不加干预的情况下,具有不可随意改变和擦写的性质,这使其天然适用于构建防篡改、防伪造和可追溯的“区块链”数据结构。但从信息操作的实用角度来讲,不可擦写的存储系统在应用领域将受到很大限制。在下文中,我们总结了人们针对DNA存储体系中数据擦写功能所做出的一些尝试。
尽管迄今DNA体内存储均以短片段的形式呈现,酵母人造染色体、大片段基因组操作等合成生物学最新进展完全可以应用于DNA存储。长片段DNA体内存储适配于第三代单分子测序,可能实现DNA信息实时读取。
3 DNA数据存储的挑战
3.1 数据安全
数据安全是信息储存和传输领域的重要问题,它包括信息的完整性、可靠性和机密性等指标。虽然存储于DNA分子上的信息具有动态稳定性,但其擦写、防伪等操作受限于生化反应的精确度而无法达到100%确定,这对于具体的应用具有两面性,将在一段时间内促进相关技术的迭代进步。
目前,合成生物学手段和基因编辑技术的发展和应用,使DNA分子的改写成为可能。这既有利于DNA存储走向更广阔的应用场景,也对数据安全的保障提出了更高的要求。在细胞内DNA存储体系中,我们可以利用一些工具酶实现信息的擦除和重写,例如位点特异性重组酶可以识别特定的DNA位点,进而翻转、插入或者切除位点之间的一段DNA[65-66]。此外,在体外DNA存储体系中,通过精心设计的生化反应,也可以实现信息“擦除”。2020年,Baym和Zhang课题组将真假两种信息编码在DNA溶液中,通过设计标记链并与溶液中的信息进行杂交来区分信息的真伪——真实信息可与“真实标记”寡核苷酸进行杂交,而错误信息的标记链可以阻止DNA链的延伸和扩增,这样保证只读取真实信息。基于DNA杂交分子的温度敏感性,作者发现在25°C下,DNA信息在存储65天后可以稳定地进行读取,并且推测DNA在25°C下的半衰期超过15年,可以进行长期稳定的信息存储;但是在95°C下DNA杂交分子很快解离,仅加热5 min,消息就会被永久擦除[67]。虽然目前受限于操作手段,人们对DNA存储的信息擦写研究并不深入,但是随着技术的发展和进步,可能出现适用于几大类存储体系的较为通用的擦写工具。
此外,信息科学中的加密编码原则同样适用于DNA存储。Grass等[68]从人类DNA中生成了80 bit的强密匙,对存储在DNA分子中的17 KB数据进行加密,并成功读取和恢复了原始信息。DNA折纸也具备三维加密信息的潜能。上海交通大学左小磊课题组和中国科学院上海应用物理研究所樊春海课题组先后利用DNA折纸的精确定位与组装能力,在存储方面做出了初步尝试[69-70]。在未来,DNA折纸的图样多样性或可用于信息加密等信息安全领域。
3.2 读写速度和成本
随着DNA合成技术的迅猛发展,人工合成DNA分子的成本持续下降。然而,如果要存储大量的信息,需要合成的DNA分子数量也是庞大的,成为DNA分子信息存储的主要开支。当前,使用阵列(高通量)合成DNA的成本约为每碱基0.0001美元。如果每个碱基存储1 bit的信息,那么存储1 TB的信息至少需要8亿美元。相比之下,使用磁带存储同等规模数据的成本仅为16美元[49]。显然,合成DNA的高昂成本削弱了DNA分子相比于传统存储介质的竞争力,限制着DNA数据存储进入大规模实用阶段。
微阵列DNA合成技术更高效、快速,具有更高的成本效用,合成的速度可以达到每秒几千碱基。第三代DNA合成技术以酶合成为基础,虽然还处于发展初期,但有望大大减少DNA合成的时间和成本。Lee等[33]给出酶促合成法时间估计为每周期40 s,是化学合成法速度的6倍。化学合成法使用的亚磷酰胺试剂每周期的成本为0.626美元;而酶促合成法每周期的成本将比亚磷酰胺便宜1000倍以上。一旦酶反应系统被微型化,预计成本将再减少几个数量级[31]。
自从1977年第一代DNA测序技术(Sanger法)出现以来,测序技术已获得了巨大的发展,相比于最初的测序成本下降了100 000倍[35]。目前DNA存储的主流方式是短片段信息存储(oligo pool),最合适的读取方式是二代测序。二代测序的核心思想是大规模平行测序,一次上样可并行几十万到几百万条DNA分子的序列测定,这足够满足当前的DNA存储规模的需求。但随着信息量的不断增加,二代测序的运行速度(含建库、读取等流程,一轮数天时间)仅能勉强满足冷数据读取的需求。
Helicos公司的Heliscope单分子测序仪、Pacific Biosciences公司的SMRT单分子测序技术和Oxford Nanopore Technologies公司的纳米孔单分子技术和单细胞基因组测序技术[71-78],被统称为三代测序技术,也被称为“单分子测序技术”。在DNA信息存储的应用范畴中,三代测序技术对于数据存储量的扩大和实时读取等目标的实现存在巨大的帮助。此外,三代测序除了消除对PCR扩增的依赖性外,更显著地增加了读取长度并提高了读取速度,在长片段数据存储上优势更大,有着广泛的应用前景。其中的纳米孔单分子技术,尽管目前错误率比其他生化测序平台高,但是在测序通量、读取长度、便携性等方面独具优势和发展潜力。例如Oxford Nanopore Technologies公司开发的三代测序系列产品,其DNA平均过孔速率为450 bp/s,袖珍便携三代测序MinION有多达512个纳米孔通道进行同时测序,而高通量台式产品PromethION 48的数据通量为7.6 TB(72 h)量级,相当于29 MB/s的数据读取速率。
随着技术更迭和算法升级,三代测序或可用于体内或体外稳定化的长片段DNA存储的信息读取,并与当前传统介质的读取速度(KB/s~GB/s)比肩。目前,已经有一些DNA存储工作尝试使用三代测序进行数据读取[47,79]。
4 总结和展望
DNA因其普遍存在的耐久性和生物功能兼容性成为人工信息储存的理想介质。从数据稳定性、传输、更迭、维护、保存等实用角度来讲,它具备得天独厚的优势,在如档案文件存储等特定的数据存储领域有可能替代传统存储介质。
在存储形式上,体外存储仍然是目前最常用的存储形式,体外存储利用短片段池(oligo pool)进行信息存储,主要的读取方式是二代测序技术。二代测序的核心思想是大规模平行测序,其特点是能一次并行几十万到几百万条DNA分子的序列测定,且一般读取长度较短,适合体外短片段存储的信息读取。但是随着信息量的不断增加,二代测序不能满足和适应其要求。三代测序技术尽管错误率更高,但是对于更大的数据量和实时读取等目标有着巨大的应用潜力。相对应读的速度更快,所以在长片段数据存储上优势更大。此外,三代测序除了消除对PCR扩增的依赖性外,显著地增加了读取长度并提高了读取速度,在DNA信息存储领域有着广泛的应用前景。
尽管如此,目前仍然存在一些问题影响DNA存储的使用和推广。首先是写和读的成本高,但随着DNA合成和测序技术的改善,其成本和准确性有望得到进一步优化,使其更好地适用于DNA存储领域。反之,DNA存储的快速发展也将带动合成和测序技术的二次飞跃。
其次,在信息编码和硬件体系上,DNA存储也将提供持续的技术发展动能。编码算法和DNA生化反应体系的联合发展,将主要攻克随机读取、擦写、信息加密等关键问题。例如随机读取问题,如何高效地从存储池中读取某一指定位置的文件是一个挑战。目前研究者们正通过在特定位置加入特定的标记或是优化检索算法,以攻克这个难题。对于擦写问题,新的工具和技术应用将使改写信息成为可能,尤其是合成生物学和基因组编辑技术的最新进展已经展示了在活细胞中灵活准确地改变遗传或人工信息的可能性[80]。天然和工程DNA靶向酶和修饰酶,包括重组酶[81]、逆转录酶[82]等多功能变体,可以用作DNA存储系统中的编写模块。而多样的信息编码方法和利用DNA三维结构等方法加密信息,可以保障DNA存储的信息安全。这些研究有望把DNA存储从冷数据档案文件存储的领域中释放出来,使其触及更广泛的数据操作领域,例如动态数据存储、新型加密、区块链等。
最后,活细胞DNA存储技术搭配先进的细胞微处理器技术,可以在小尺度范围整合数据的存储与决策,即数据“存”与“算”的一体化和边缘化,这个愿景的实现将依赖于DNA存储技术和细胞计算领域的巨大突破。在未来的超大数据时代,活细胞DNA存储或能以医疗健康为中心进行广泛的应用辐射,具备颠覆性技术的潜能。

