DNA信息存储:生命系统与信息系统的桥梁
韩明哲,陈为刚,宋理富,李炳志,元英进
(1天津大学,合成生物学前沿科学中心,系统生物工程教育部重点实验室,天津 300072;2天津大学化工学院,天津 300072;3天津大学微电子学院,天津 300072)
信息存储是文明传承的基础。人类是地球上最具智慧的生命体,从结绳记事开始,生命体外的数据存储就成为了人类思想的延续,记录了灿烂文明。造纸与印刷术的发明,使得人类能够存储的数据量在几百年内获得了大约5个数量级的提升[1];在计算机时代,尤其是近年来随着信息技术的快速发展,人类生活的方方面面都逐渐实现数字化转变,人类产生的数据爆发式增长。基于磁、光及集成电路的现代数据存储介质历经发展,存储体积密度已经可达到1010~1012bit/cm3[2]。与之相比,DNA存储具有更高密度存储潜力,如大肠杆菌染色体DNA的存储体积密度据估算达约1019bit/cm3[3]。近年来,随着合成生物学的快速发展,以高通量DNA合成技术[4]和人工合成染色体的工作为代表[5-6],标志着人类对DNA的设计[7]、合成[8]、编辑[9]和读取[10]能力已经进入到一个崭新的时代。在此背景下,利用合成DNA进行高密度信息存储成为一个非常有前景的研究方向[11],得到了相关领域研究者、信息技术企业与生物科技企业的广泛关注。2020年11月,微软、西部数据等传统信息技术企业与Twist Bioscience、Illumina等新兴生物技术公司一道,共同宣布成立了第一个DNA数据存储联盟,将制定全面的行业路线图,为经济高效的商业档案存储奠定基础[12]。
1 DNA存储数字信息
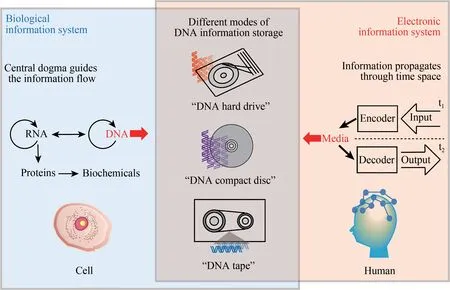
利用人工合成的脱氧核糖核酸(DNA)存储数字信息,简称DNA信息存储[13]。DNA用作信息存储载体,具有存储高密度、不受电磁干扰、长期高可靠和维护低成本等优势[13-16]。DNA作为天然的信息载体,以“A/T/C/G”数字信号的表示形式,存储了亿万年来无数生物的遗传信息,依托中心法则造就生命繁衍、进化演化及生物多样性。人类产生的海量信息,记录在各类数字存储介质,保存并得以延续,支撑了文明的传承与繁荣。利用DNA存储数字信息连通了生物系统与信息系统,发展了多种应用模式,成为近年重要的研究热点。
利用DNA存储数字信息的原理和技术流程如图1所示。其原理是:数字化信息在二进制码流、四进制碱基序列和实际DNA片段之间的转化与流动[3,13-14]。目前,基于此原理的技术流程主要包含两个方面:①信息写入,首先对文本、图片或视频等信息的二进制码流进行编码,得到A/T/C/G组成的碱基序列,随后利用DNA合成技术将信息写入对应的DNA片段,并对其进行多模式保存[17-18];②信息读取,首先对制造的数据DNA片段进行测序,随后进行识别、组装、纠错与解码等,将存储在DNA介质中的数据还原为原始数字化信息,得到原始文本、图片、声音和视频等。

图1 DNA存储的原理模型、技术流程和应用模式Fig.1 The basic principle,technical work flow and storage modes of DNA information storage
2 DNA信息存储的若干模式
依据DNA片段读写技术的特点,类似传统数据存储,也可划分为“硬盘”“光盘”“磁带”等应用模式。“DNA硬盘”具有高通量读写特征,面向海量数据的高密度存储;“DNA光盘”具有低成本快速复制特征,支持单写多读,面向数据的海量分发;“DNA磁带”具有体内串行刻写特征,面向数据或状态的顺时间记录。以下将对各个存储模式的特点和相关研究进展进行详细介绍。
2.1 “DNA硬盘”模式
2012年哈佛大学George Church等在《科学》杂志发表研究成果[19],成功存储和读取了5.27 Mb包含文字、图像和JavaScript程序的数字化信息,出错率仅为百万分之二。随后在Johns Hopkins Magazine上首次提出“DNA硬盘”(DNA hard drive)[20]。该模式依托高通量DNA芯片合成技术和高通量二代测序技术来写入和读出数据。与传统的硬盘类似,具有面向海量数据的高密度存储潜质。由此衍生的类似研究,可归纳为“DNA硬盘”。
“DNA硬盘”的数据端到端可靠性远不及传统硬盘,需要解决DNA作为载体的数据可靠性问题[21]。目前商业硬盘的读写错误率低至10-15以下,而高通量合成寡核苷酸的错误率一般在1/2000到1/200[22-23],二代测序的错误率在1/1000到1/100[24]。为了解决这些错误对信息可靠性的影响,多个信息领域的信息编码方法被引入到了“DNA硬盘”框架。欧洲分子生物学实验室的Goldman教授[25]通过添加四倍冗余和简单的校验机制实现了数据的可靠恢复,但是由于四倍冗余的设计,该方法实现的逻辑密度(bit/nt)和成本控制都不理想。苏黎世联邦理工大学Grass团队[26]引入了里德-所罗门(RS)纠删码,解决了寡核苷酸链池中部分片段丢失以及片段内碱基替代错误,在保证数据可靠恢复的同时使数据部分的逻辑密度超过了1 bit/nt。Erlich等[27]引入了喷泉码,更好地适配海量片段化的存储模式,将数据部分的逻辑密度进一步提升到1.57 bit/nt。另一思路,Anavy等[28]和Choi等[29]分别使用了简并碱基来拓展DNA的多进制表示方法,将“硬盘”模式下的逻辑密度推升到了2 bit/nt以上,但是此方法也面临需要更高测序覆盖度(覆盖度>150×)的问题。除此之外,在未来引入非天然碱基拓展存储单元,可进一步提升逻辑密度[30]。总而言之,在确保数据可靠性的前提下,逼近数据承载能力的极限是DNA信息存储发展的趋势[31]。
值得关注的是,“DNA硬盘”中合成与测序会引入碱基的插入和缺失错误(insertion/deletion,简称Indel),这有别于传统存储介质,处理较为困难[3]。针对该问题,Press等[32]提出了基于哈希编码和贪婪穷举解码的编码方案,该方案能够在单分子拷贝的情况下纠正插入和缺失错误,但是需要较高的冗余度来实现纠错,且解码复杂度较高。Sabary等[33]提出了几种动态的DNA重构算法,可直接用于较高错误率下的DNA序列重建。天津大学Song等[34]设计了一个基于德布莱英图(de Bruijn Graph)的DNA序列高鲁棒重建算法,如图2所示,可以从包含大量插入缺失和替代错误的多序列快速重建无错误的DNA片段序列。该方法可以从低质量的PCR产物(序列长度完全错误)中可靠地读取数据,实现高鲁棒读取。

图2 基于de Bruijn图论的DNA序列重建算法[34]Fig.2 Algorithm of de Bruijn graph-based reconstruction of DNA strands[34]
为降低“DNA硬盘”写入成本,提高写入速度,2019年,Lee等[35]采用非阻断型的末端脱氧核酸转移酶(TdT)合成DNA,实现了一种专用于信息存储的DNA酶法合成技术。2020年,Lee等[36]进一步利用图案化紫外光快速解离Co2+激活TdT,成功编码了110位的数据信息,初步验证了在阵列表面实现大规模DNA并行合成的可行性。
为解决“DNA硬盘”多轮PCR造成的偏好性累积和部分DNA片段丢失的问题,Lin等[37]通过对原始文库修饰并引入RNA逆转录过程,构建了始终以原始文库为模板的扩增方法,在一定程度上降低了多次访问对原始文库的影响。Choi等[38]将原始文库固定在具有二维码编号的微盘上,实现了对文库的原位(in situ)扩增,经过20轮扩增未发现产物片段分布的明显变化,显著降低了扩增带来的偏好性,同时还通过二维码实现了数据库管理。天津大学Gao等[39]将原始文库固定在磁珠上,通过等温链置换扩增技术,实现了对文库低偏好性、稳定重复的扩增。
“DNA硬盘”的应用模式已实现了一定规模的存储验证[40-47]。2018年,华盛顿大学和微软公司的研究团队实现了200 MB的数据存储和部分数据文件的随机访问[40],并于2019年开发了原型设备,实现了“HELLO”的自动读写[41],同时还设计了DNA保存和访问的微流控平台[42];2019年,美国Catalog公司[43]利用独创的DNA写入技术,存储了16 GB的维基百科数据,是目前最大规模的“DNA硬盘”。在国内,天津大学陈为刚等[44]采用LDPC码与RS码的乘积码保证可靠性,采用27万条的寡核苷酸池存储超过3 MB数据,存储了两段有历史价值的音视频片段以及13 000多汉字,实现了低样本浓度、低测序覆盖度的可靠读出(图3)。深圳华大生命科学研究院Ping等[45]设计的“阴-阳”编码策略可调整均聚物长度或GC含量等以满足不同用户需求,实现了2.02 MB数据的存储。

图3 “DNA硬盘”模式示意图[44]Fig.3 Schematic diagram of"DNA hard drive"[44]
2.2 “DNA光盘”模式
与“DNA硬盘”的体外存储方式不同,一种生命体内的DNA信息存储模式也被提出,其特征类似光盘,本文归纳为“DNA光盘”[48]。该模式的主要特征是采用较长DNA片段,通过细胞体内组装完成写入、借助细胞自身的快速低成本的DNA复制能力,快速且均一拷贝数据。虽然“CD母版”的制作成本较高,即合成与组装成本较高,但是其类似CD的低成本大量拷贝,使得“母版”成本得以分摊。受益于常用模式生物较低的突变率[49-50],“DNA光盘”亦可高保真拷贝,支持数据长期传代复制[51]。利用小型纳米孔测序器件,有望实现数据快速读出,便携式“DNA光驱”呼之欲出。值得注意的是,纳米孔测序错误率高达10%,并且包含难以处理的插入与缺失错误[52]。因此如何保证数据在纳米孔测序下的可靠读出,是一个值得研究的方向。
“DNA光盘”开始于早期细胞体内存储数字信息的概念验证,探索单个细胞内存储的数据量是个有价值的问题。概念验证多使用质粒在大肠杆菌内存储数据,编码的DNA长度通常不超过1 kbp[53-59]。2010年,Venter等[60]在化学合成蕈状支原体时,第一次在原核生物基因组中嵌入了超过4 kbp的编码DNA存储外部信息。本文作者[48]从头设计合成了一条254 886 bp的存储专用染色体,其中数据编码部分占95.27%,将单菌内数据存储DNA数量提升到了百kbp级,存储了37.8 KB图片、视频以及文字,利用叠加编码方案,有效克服三代测序的高错误率问题,实现了数据的可靠恢复。这项工作突破性地将单菌内数据存储DNA数量提升到百kbp级,初步打通了单细胞数据存储容量这个限制“DNA光盘”模式存储通量提升的关键因素(图4)。

图4 “DNA光盘”模式示意图[48]Fig.4 Schematic diagram of"DNA CD"[48]
“DNA光盘”模式除了提高单细胞数据容量外,增加并行通量也是提升数据存储容量的关键。Shipman等[61]通过CRISPR/CAS1-CAS2系统捕捉DNA小片段整合进大肠杆菌群体的CRISPR序列中,分别编码了494字节的21色图片和2.6 KB的动画短片。天津大学Hao等[62]构建了携带不同短信息片段质粒的大肠杆菌分布式混菌存储系统,在维持低成本的同时实现较大的体内存储通量,将445 KB的数字文件存储在11 520个115 bp的合成DNA中。
2.3 “DNA磁带”及其他模式
运用动态基因组工程(dynamic genome engineering)[63]在生命体内“书写”DNA来记录信息的新模式,一定程度上类似磁带,本文称之为“DNA磁带”。“书写”包括对特定DNA靶向插入、删除、倒位和单碱基突变等操作,类似于在磁带上磁化刻录以记录信息[64]。目前已经验证的模型中,“书写”过程的开启信号可以是对抗生素或病毒的暴露、营养底物的改变和对光及特定诱导剂的响应等[65-69]。起初“DNA磁带”主要记录细胞内的特定事件或状态,Harries Wang团队[70]首次构建了基于电刺激的“人-胞”输入接口,利用电压控制胞内的氧化还原对状态,从而诱导CRISPR/Cas1-Cas2系统在特定位点插入不同的DNA序列,实现信息写入。这使得未来半导体-生物接口的发展成为了可能。进一步,得益于基因线路设计的发展,生物“逻辑门”可与“DNA磁带”相结合,为生物细胞计算提供记录。然而,“DNA磁带”依然存在逻辑密度低、数据响应延迟和精准性较低等问题。此外,目前通常是基于菌群进行记录,通过加标签(barcode)对不同菌群进行区分[70],随机访问的难度较大。
与“DNA磁带”模式类似,为避免人工合成DNA产生的高昂成本,美国UIUC的Tabatabaei等[71]模仿古老的打孔卡存储方式,以天然的DNA分子链(例如基因组DNA、克隆或PCR扩增产物)为“卡纸”,以特定的酶为“打孔机”,建立了一种“打孔卡”DNA存储方法。该方法通过在DNA磷酸骨架上预设位置“打孔”来表示二进制数据中的“0”和“1”,从而避免了昂贵的DNA合成。与之相似,以天然M13噬菌体单链DNA为骨架,Chen等[72-73]在骨架上间隔插入带有生物素标记的支链DNA用以记录信息,并通过纳米孔测序检测是否带有标记物来读取数据的“0”和“1”。然而,这种基于天然DNA分子链的存储技术没有发挥DNA存储密度大的优势。
除此之外,华盛顿大学和微软公司的研究团队[74]也尝试了对组装后的寡核苷酸池进行纳米孔测序。上海交通大学Zhang等[75]利用DNA折纸技术实现信息的加解密,这种基于结构的信息表示和加密方法,为保证重要信息的安全性提供了新的方案。
3 挑战与展望
当前DNA信息存储的主要挑战为单位信息存储成本高,信息读写速度慢,无法高效对接现有信息系统。因此,DNA信息存储当前发展的重点是进一步降低成本,提高读写速度,实现与现有信息系统的融合。
3.1 更低成本的信息写入
目前,寡核苷酸池的商业合成价格大约为0.002美元/base,折合0.001美元/bit(约8.6×106美元/GB)[23,76],写入成本较高,是硬盘的108倍[77],如图5所示。美国情报高级研究计划局(IARPA)分子信息存储技术(MIST)项目的目标是到2023年DNA信息写入成本将降低至10-10美元/bit(约0.86美元/GB)[78]。

图5 DNA信息存储成本比较与预测Fig.5 Comparison and forecast of cost by DNA information storage
DNA信息存储成本在未来有很大下降的潜力。首先,Twist Bioscience的首席技术官在2016年声称其合成成本已经低于10-12美元/base[79]。但是,运行维护、合成芯片、试剂耗材、质量控制以及人工等其他成本造成了现有DNA信息写入成本较高的现状。可以从优化合成反应、改良芯片结构、替换廉价耗材、优化试剂分配量等多方面着手,有望大幅降低合成成本。其次,传统上DNA合成主要用于生命科学研究,其技术指标与DNA信息存储的需求不匹配。面向DNA信息存储的合成,可容忍合成步骤产生的更多错误,降低精度与纯度要求,减少质量控制成本,在保证数据准确性而不是序列准确性的基础上提升合成的长度和通量,从而有望大幅降低合成成本[80]。再者,由于信息存储领域市场规模巨大,随着半导体器件、微纳加工在DNA信息存储领域的应用,该领域的巨大投入将对DNA合成技术产生重大影响,DNA合成技术与装备快速迭代升级,合成通量快速提升,成本有望快速下降。
3.2 更快速的数据读取
DNA信息存储的读取依赖测序技术,与磁、光、电等存储相比,读取速度较慢,如图6所示。进一步提升读取速度,是DNA信息存储发展的一个需求。DNA的测序技术与现有电、磁存储技术的串行读取不同,具有高并行读取特点,以Illumina为代表的二代测序技术可以同时读取0.04亿~11亿个位点[81]。然而,每轮测序反应和信号采集时间长达2.2~19 min[82],所有反应所耗时间约占运行时间的90%。通过高通量(也即空间并行度)弥补反应时间较慢的缺陷,读取速度可达5~500 KB/s[81](最大数据产出/最长运行时间),但是需测序完全结束后才能获取原始数据。三代纳米孔测序已经做到便携化和低延迟数据生成,单通道测序速度约为450 bp/s(约112 B/s)[83],基 于MinION测序芯片(最多支持512通道同时读取)的最高读取速度约为56 KB/s(不包含电信号到碱基转换时间)。而现有电、磁存储技术通常每秒可读取几十到几百兆字节数据。基于二代测序的数据读取受化学反应限制,较难突破性地降低反应时间,可以通过进一步增大通量满足未来大规模冷数据读取需求;基于三代纳米孔测序的数据读取,依然有较大潜力提升单孔读取速度,如固相纳米孔的发展有望在保证分辨率的前提下继续提升读取速度1~3个数量级[84],甚至在未来超越现有存储的读取速度。此外,提高并行化读取的集成程度,构建一体化、自动化的读取专用设备也面临很大挑战,需要机械、生化、信息、控制等的多学科协同解决。

图6 DNA信息存储读取速度对比Fig.6 Comparison of reading rate for DNA information storage
3.3 DNA信息存储与现代存储系统的融合
依据DNA合成与读取的技术发展现状和特点,DNA信息存储有望率先在冷数据存储方面获得应用[85]。图7为DNA信息存储在开放系统互联(OSI)、模型中的映射关系以及存储系统分等级架构。DNA作为新介质,融入现代存储系统的过程,也是信息存储系统不断演化完善的过程。

图7 DNA信息存储与现代存储系统的融合Fig.7 Fusion of DNA information storage and information storage system
在物理层,造成DNA数据存储不可靠的因素主要包括:合成、扩增以及测序处理过程的非理想,体现在碱基的插入、缺失、替代(IDS)错误以及DNA分子或片段丢失等[86];按照信息理论研究范式,一旦建立了准确的碱基错误模型,就可以设计匹配的信息编码方法与数据恢复方法[31],设计有效的数据链路层。但是,由于DNA信息存储信道的一些新特点,例如包含Indel错误、信道容量尚无法准确计算[87],值得深入研究[13,32,88]。中间各层是DNA信息存储融入现代存储系统的桥梁。传统数据存储领域的关键技术,需要结合DNA介质与DNA存储的新特点进行优化设计。例如,目前纠删码已经在基于寡核苷酸池的信息存储模式得到了很好的应用[27,40]。同时,纠删码也广泛应用于存储系统的中间各层,如何协调设计是一个非常有价值的问题。在应用层,提供的用户服务需要与DNA存储特点相适配[89]。例如,数据检索、聚类分析、数据挖掘、特征识别等,需要方便地读取数据,而现阶段DNA信息存储将大块数据封装于无法实时读取的DNA介质。因此,探索结合DNA信息存储特点的“存算一体化”的处理引擎,设计跨层的直达DNA介质的机制就显得极为重要。
存储系统的分等级架构是存储系统充分发挥作用的基础,DNA作为新的存储介质,短期内其技术特性与大容量冷数据归档存储最为匹配。据预测,归档的冷数据比例高达60%[90],冷数据的DNA存储展现出了巨大的发展潜力,有望平稳融入现代数据存储体系。
值得一提的是,DNA信息存储也可能给传统信息系统带来安全方面的隐患。研究者可将计算机病毒信息存储于DNA,通过DNA测序以及处理过程,访问并进入非合作方的计算机系统,造成信息安全风险[91-92]。而DNA分子极小的物理尺度、特定条件下稳定的物理性质和无金属特征的非电/磁存储,为隐蔽数据传递提供了新途径。将携带信息的DNA封装为可打印材料,存储到常见的生活物品中并隐蔽传递[26,93],可能造成敏感数据泄露。
3.4 总结
近年来,DNA信息存储的基本原理、技术流程和应用模式引起了研究者的广泛关注。DNA信息存储连接了生命系统与信息系统,推动相关研究与应用的发展。以“DNA硬盘”为主的体外存储与电子信息系统耦合更多,拓展了现有基于磁、光、电的电子信息存储系统;以“DNA光盘”和“DNA磁带”为主的体内存储与生命信息系统耦合度比较大,提供了细胞内的信息存储器或记录器,为未来细胞计算或细胞通信的发展提供了更广阔的空间。DNA信息存储是一个新兴的、多学科深度交叉融合的研究方向。进一步推动其走向实用化,仍面临很多挑战。为应对挑战,美欧的相关企业、大学与研究机构已经组成了DNA数据存储联盟,通过广泛合作共同制定全面的行业路线图,以推动DNA信息存储的产业化发展。据高德纳咨询公司预测,到2024年,将有30%的数字业务进行DNA存储试验[94],以应对指数级增长的数据存储需求。面对未来的存储需求,国内也亟需布局和发展DNA信息存储研究与应用。本文从合成生物学与信息科学交叉融合的视角,对近年来DNA信息存储的研究进行了综述与展望,希望能吸引更多研究者在该交叉框架下提出有价值的研究问题,推动DNA信息存储的发展与应用。

