DNA微阵列原位化学合成
闫汉,肖鹏峰,刘全俊,陆祖宏
(东南大学生物科学与生物医学工程学院,生物电子学国家重点实验室,江苏 南京 210096)
随着生命科学的飞速发展,全球每年需要合成数以百万计的DNA片段,用于生命科学相关的研究[1-5]。在大多数应用中,主要采用基于DNA合成柱的固相化学合成方法来合成所需的DNA片段[6-10],然而该方法已经无法满足当下的合成要求。目前,合成生物学的高速发展引领第三次生物技术革命[11-19]。DNA作为生命遗传物质的载体,是绘制各种人工生命系统蓝图的基础,高通量低成本快速DNA合成已成为合成生物学的关键支撑性技术。近十年来,人类获取信息能力一直在快速增强。如图1(a)所示[20],每年全球获取信息的总量超过100 ZB量级。以硅为介质的信息存储器件存在信息保存时间短(一般十年左右)、信息保存容量低、耗能高等问题,不能适应大量“冷数据”(需要长期保存的信息)的存储要求。生物系统中DNA分子具有信息容量大、稳定性好、读取过程能耗低等特点,其作为未来新的信息存储介质正受到越来越多的关注[21-31],并正在成为半导体合成生物学这一新兴领域的重要研究方向。正是上述新兴研究领域的巨大需求加速了DNA合成技术的发展[32]。DNA微阵列原位化学合成作为一项DNA合成技术,为DNA存储、大基因组合成提供所需的目的序列。下游的DNA存储、大基因组合成等新兴应用要求合成更长序列、更大容量的DNA样本,这些是对DNA合成的要求和挑战。同时DNA合成技术能否合成出更长序列和更大容量的DNA样本将会制约着下游技术的发展。目前基于合成柱的DNA合成通量低,最大合成柱仪器的合成通量只有1536种DNA序列,成本在0.05~0.17美元/碱基,无法满足未来DNA合成的需求;基于酶等DNA合成技术尚未成熟,其发展还存在一定的不确定性;而基于固相化学合成的微阵列技术能够很好地兼顾合成通量和成本,同时该技术比较成熟。因此,发展基于微阵列芯片的固相化学合成技术,仍然是高通量低成本DNA合成和为下游产业技术提供DNA合成产物的一条重要技术路径。
基于微阵列的DNA合成方法可实现DNA序列的并行高通量合成,其合成密度可达到每平方厘米数百万。2014年基于寡核苷酸微阵列原位合成方法,DNA合成成本已低于10-3美元/碱基,2020年基于电致酸合成的DNA成本估计达到10-6美元/碱基,且具有进一步大规模降低成本的潜力[33]。图1(b)给出了美国半导体研究会2018年预测的DNA合成成本和速度的发展趋势。根据该预测,到2023年DNA合成成本要降低到10-10美元/碱基以下。然而受到合成产率的限制最近几年DNA合成发展比较缓慢,在合成长度上同时高通量、高产率地合成超过200 mer的DNA链仍然存在着挑战。基于微阵列的DNA合成方法在成本和合成通量方面具有明显的发展优势[34-38]。近年来该领域的进展和突破偏少,基于固相化学合成原理技术的发展遇到了瓶颈,包括:单条寡核苷酸的产量过低,通常比柱式合成低2~4个数量级;合成长度受限、产物过于复杂,干扰后续的DNA组装过程;合成准确性受限,引入更多合成错误,提高验证和错误矫正成本等。在此基础上更进一步地提高合成产率和合成长度,是目前该领域的科研工作者的目标。本文梳理了现有固相合成技术,对它们的原理及其特点加以介绍,从而给读者和该方向的研究者一些启发和思路。
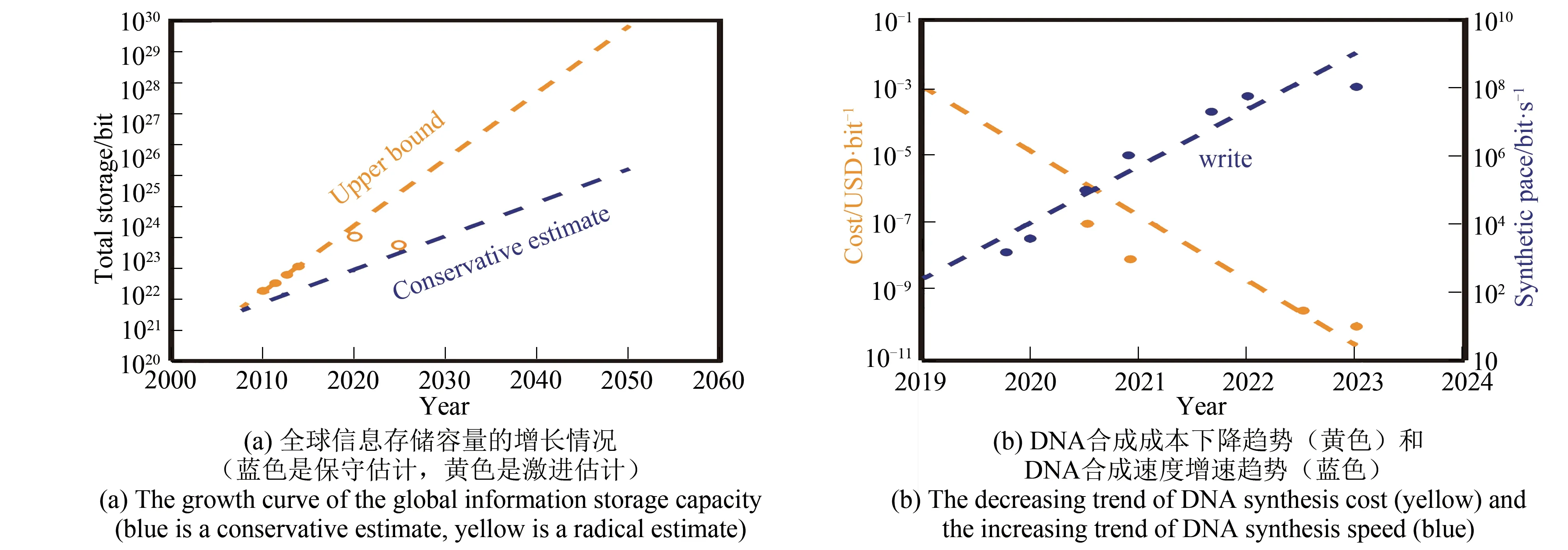
图1 美国半导体研究会预测的存储发展趋势[20]Fig.1 American Semiconductor Research Association predicts storage development trends[20]
1 DNA固相化学合成原理及技术路径
DNA微阵列(DNA芯片)原位合成是指直接在固体基质(芯片)上用四种单核苷酸实现所需的DNA片段的合成。其原理基于亚磷酸酰胺固相化学合成法(图2)及改良后的亚磷酸酰胺固相化学合成法[7,39-43]。芯片上每个阵点均由四个反应步骤构成一个循环,每循环一次完成一个碱基的合成。如图2所示,亚磷酸酰胺固相化学合成法每个循环的四个反应步骤为:①去封闭,待合成体系中加入三氯乙酸(TCA)以去除连接在固相载体上核苷酸5′-羟基端的二甲氧基三苯甲基(DMT)保护基团,从而获得游离的5′-羟基端,以供下一步合成反应。②活化偶联,3′端的亚磷酸酰胺单体与四氮唑活化剂混合形成亚磷酸酰胺四唑活性中间体,并在序列中与先前连接在固相载体上核苷的5′-羟基发生偶联反应,形成亚磷酸三酯,使合成序列链向前延长一个碱基。③盖帽,任何未与亚磷酸酰胺单体发生反应的5′-羟基通过乙酸酐酰基化反应被封闭,此外在第二步偶联反应中所有未向前延长一个碱基的序列在后续的合成反应中将是惰性的,始终维持其长度不变。④氧化,采用碘的四氢呋喃溶液将不稳定的、易被酸或碱水解的亚磷酸酯键转化为稳定的五价磷酸酯键。经过上述步骤的循环反应,即可得到特定长度的DNA片段。此后对所合成的DNA片段用浓氨水等试剂进行切割,使其从固相载体上脱落下来,同时脱去碱基上的保护基团,最后经过纯化得到目的长度的DNA序列。
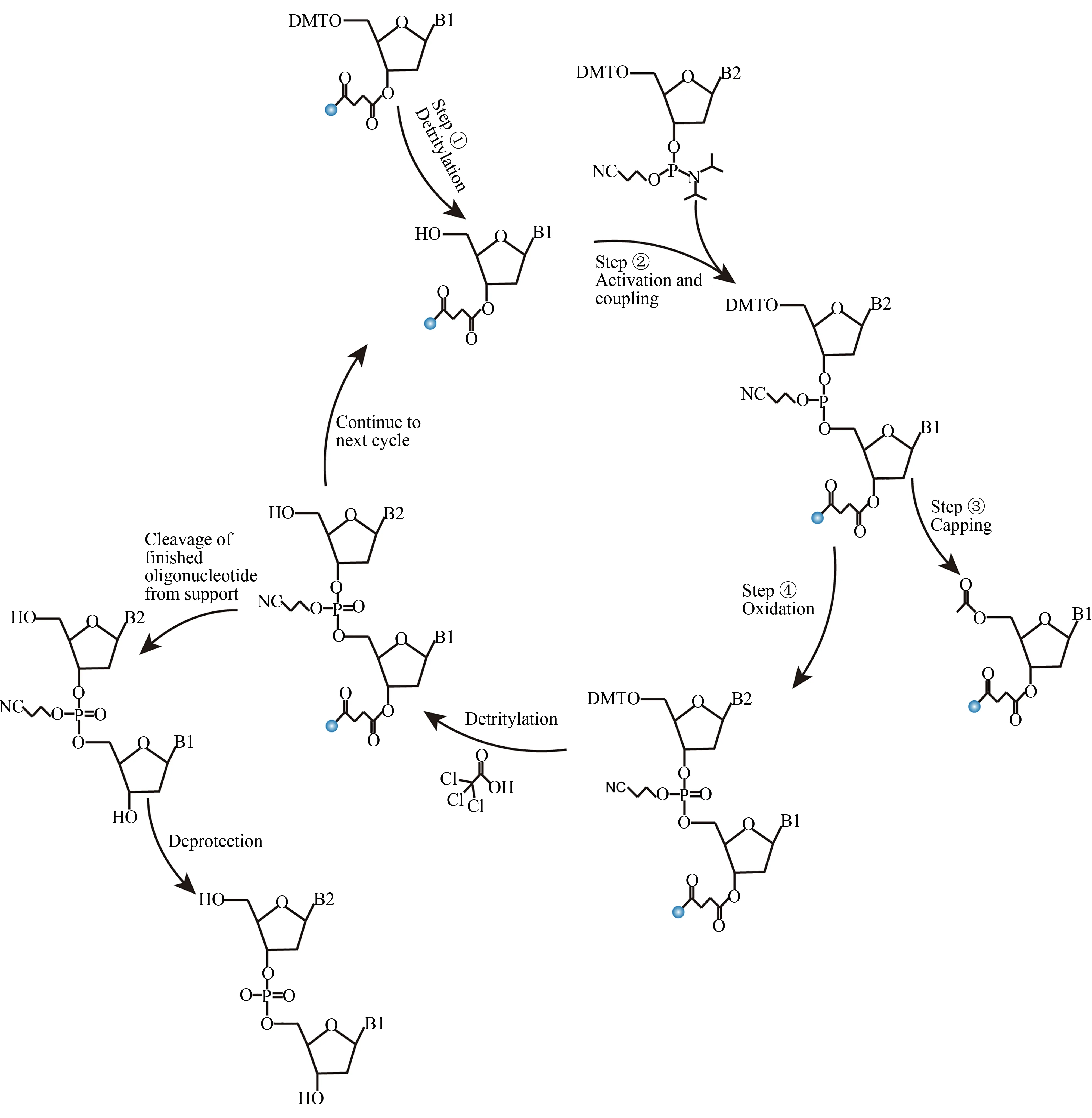
图2 亚磷酸酰胺DNA固相化学合成法Fig.2 Solid phase chemical synthesis cycle of DNA by phosphite amide method
DNA微阵列原位合成的最大优势是可以同时进行不同序列DNA的并行合成。其特征是芯片上所有阵点按照预设计的目标序列,将需要合成相同碱基的不同阵点上的碱基合成反应合并在一起进行特定的反应,从而并行完成一个碱基的合成[44-47];而预合成不同碱基的其他阵点则不参与本次反应,留给后续的反应以完成碱基的合成。Southern首次采用更换不同的微流体通道(相当于物理掩模板),将单体试剂通过微流体通道与合成区域接触,发明了微流通道原位合成技术[48-50]。之后,人们提出了不同DNA微阵列原位合成方法[34,51-56]。到目前为止,基于亚磷酸酰胺固相化学合成法原理的DNA微阵列合成策略根据脱保护方式不同,大致可以分为光化学选择性脱保护、酸选择性脱保护和单体选择性转移偶联三种类型(图3)。光化学选择性脱保护法是将亚磷酸酰胺法中所用的核苷酸单体的5'-羟基保护基团替换为光敏基团[图3(a)],在光照下该保护基团可被脱除。酸选择性脱保护法[包括光(或者电)致酸法]的特点是通过光照射(或者电极通电)使阵点上的物质发生光(或者电)化学反应从而产生酸将5′端的保护基团脱除,曝露出羟基以供后续的偶联反应[图3(b)]。单体选择性转移偶联法是直接采用亚磷酸酰胺合成方法,通过物理作用(点样、喷印、压印或者与微流体接触等方式),先后分次将相应的核苷酸单体反应液转移到芯片的不同阵点上实施偶联反应[图3(c)],经过多次循环后完成不同阵点上不同DNA序列的合成,这一类型还包括微流体通道载片合成、喷印合成法和压印(软光刻)法。下面将对不同DNA芯片原位合成方法分别进行具体的介绍。

图3 三类DNA微阵列原位化学合成策略Fig.3 Three types of DNA microarray in situ chemical synthesis strategies
2 DNA芯片原位合成法
2.1 原位光刻合成技术(原位光刻法)
Affymetrix公司的GeneChipTM是高密度DNA微阵列原位合成的代表,制造工艺采用原位光刻法。20世纪90年代初,Fodor等提出了大规模固定化多肽阵列和DNA阵列的光导原位合成技术[57-60]。DNA光导原位合成试剂是利用其碱基单体5'-羟基端的光敏保护基团,如图4所示,这些光敏基团可以是α-甲基(-2-硝基胡椒基)氧羰基或2(-2-硝基苯基)丙氧羰基。通过光掩模板(简称光掩模)可以精准地对特定位置的DNA基团进行脱保护(图5)[61],感光区域部分在光作用下保护基团被激活并脱去,从而暴露出活性羟基基团,进而发生后续的偶联反应,完成反应后洗去未有效结合的单体,最后将未偶联的活性羟基用乙酸酐试剂封闭、阻断其参与后续的反应。单体的合成只在脱保护基的区域进行,光照区域就是要合成的区域,该过程通过操作光掩模来控制。一般而言,每层的四个不同碱基需要相应的四个掩模板(简称掩模)来完成其偶联反应。当然,不同层的相同碱基可使用同一掩模来合成以减少合成的次数。通过交替使用不同的光掩模,重复上述步骤,直到目的探针序列被完全的合成。光去保护并行合成技术的关键在于合成出含有光敏保护基的核酸单体试剂,这种试剂要有高效的偶联和光脱保护基团效率同时不应对合成序列造成损伤。
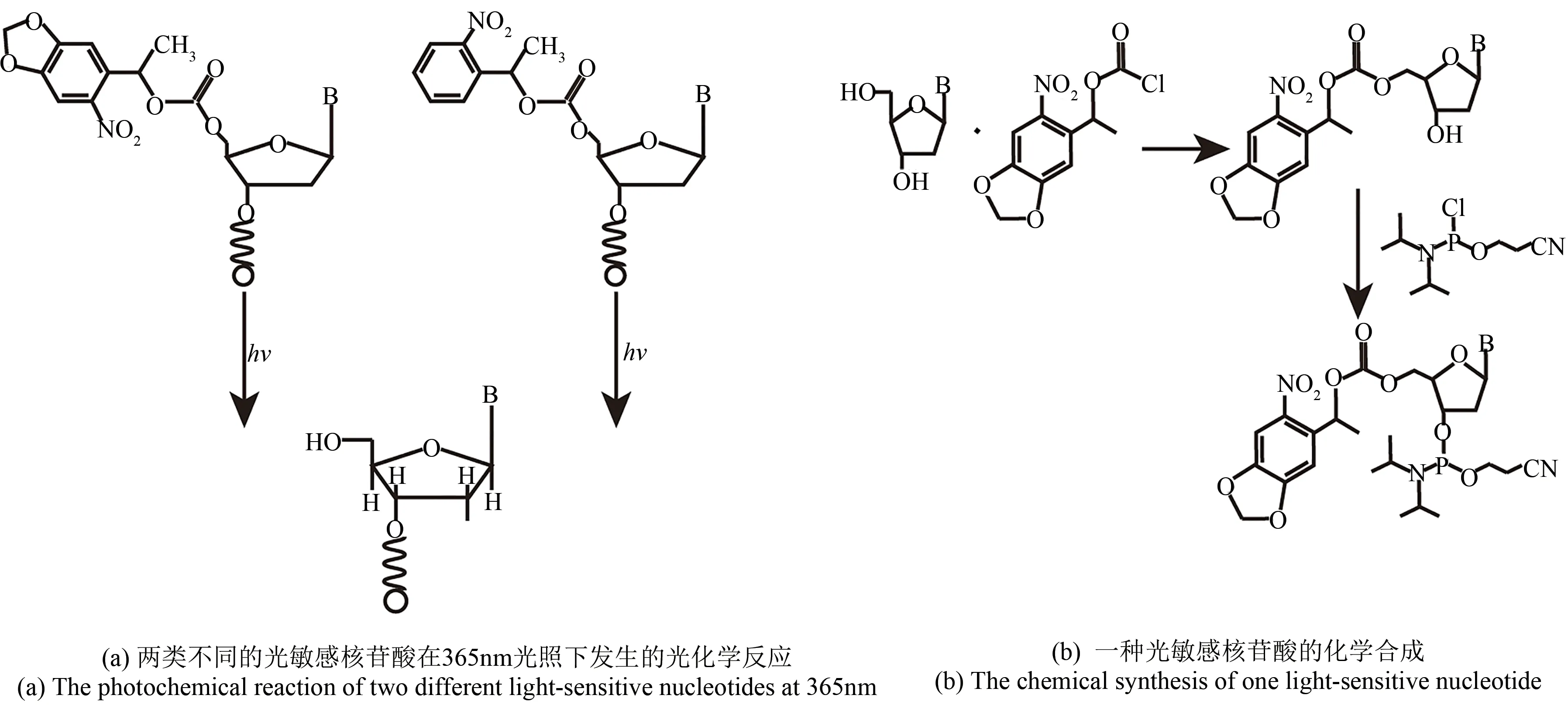
图4 不同类型的核苷酸Fig.4 Different types of nucleotides
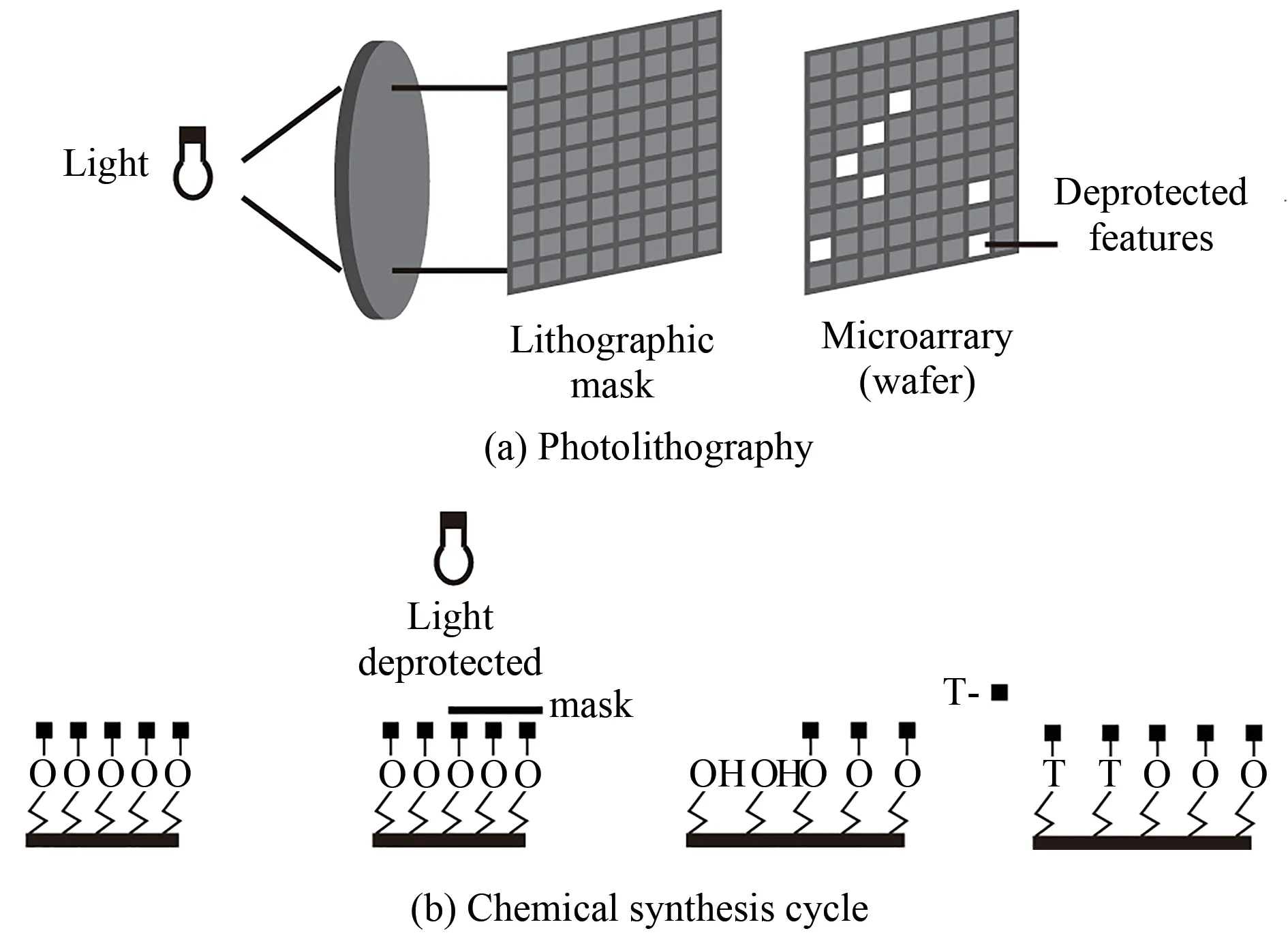
图5 GeneChip探针阵列的制备[61]Fig.5 Preparation of GeneChip probe array[61]
在利用光掩模进行DNA芯片制备时,每个碱基需要4块光掩模,50个碱基长度的核酸阵列需要200块光掩模。虽然通过碱基合成流程的优化,实际所需掩模可以适当减少,但光掩模数量仍然需要很多。对于大量研究用客户定制型芯片来说,芯片定制量很小,单一芯片制备的成本高、时间长。Gassgeet等[62]提出了一种虚拟掩模(无掩模)技术。该技术直接利用投影仪中常用的光学数字微转镜阵列芯片来进行位置选择性曝光,通过计算机控制实现核酸微阵列的光导原位合成。这种基于虚拟掩模的DNA芯片合成技术避免了大量昂贵光掩模的使用,是一种高效、灵活的寡核苷酸芯片原位合成方法[63-64]。数字微转镜芯片由数十万个微小铝镜阵列组成,每个微镜相当于一个像素点,把一束平行光分成数十万个光点,在计算机指令的控制下精确实时地分别控制其反射方向,在基片上的特定区域选择性地进行光脱保护处理。Gassgeet等在8 h内成功地在16 mm2的芯片表面上制备出了76 000种DNA探针。光脱保护并行原位合成法优点在于光刻的空间分辨率高,单位面积上可以合成更多的不同DNA序列,寡核苷酸合成序列密度可达106/cm2以上。
光脱保护并行原位合成法的化学合成循环与传统的固相合成寡核苷酸相似,但由于微阵列阵点合成的复杂性和玻璃基底上合成动力学的独特性,需要对合成参数和循环进行仔细的探索和独特的调整,以提升脱保护和偶联反应效率[55,65-69]。传统的固相合成中,偶联效率介于98%~99%,偶联效率低于100%的原因在于偶联反应的不完全以及嘌呤累积暴露于酸性条件下产生脱嘌呤反应。偶联反应和后续封闭反应的失败结果与脱保护的失败结果类似,将会导致合成序列中单个碱基的“删除”,这是人工基因构建的主要错误。因此合成反应后的样品库中存在着一些包含错误的非目的序列,当要获取目的链时可通过封闭反应以及在碱基脱保护步骤中将脱嘌呤反应的位点链断裂并纯化的手段来获得正确的全长序列。在光脱保护并行原位合成法中,偶联效率相对略低导致更多的“删除”错误;除此之外,“插入”错误对整体错误率有很大贡献,这是因为光学成像系统不可避免的缺陷导致微阵列在意外曝光时,就会发生序列“插入”误差。像漂移、散射、衍射和光晕都会引起去保护,继而对合成序列引入插入误差,这也是值得深入考虑的问题。以5'-NPPOC保护的亚磷酸酰胺单体为例,目前探索给出的最佳合成参数为:氧化反应60 s,偶联反应60 s[0.025 mol/L单体+0.25 mol/L催化剂4,5-二羟基咪唑(DCI)],曝光剂量12 J/cm2(脱保护),乙酸酐封闭120 s后氦气干燥30 s。测得的耦合效率分别为:99.8%(dA)、98.0%(dC)、98.6%(dG)、99.4%(dT),四个碱基的平均偶联效率为99.0%,远大于之前报道的95%~98%[64,70-71]。对化学合成和光敏保护基团的研究表明[67],使用DCI催化剂将偶联时间减少到几秒亦可以保持芯片的高质量。在对比了5种催化剂合成芯片的杂交信号后发现,使用偶联催化剂的杂交信号强度顺序为5-乙硫基四氮唑(ETT)>5(-二-3,5三氟甲基苯基)1-H-四唑(活化剂42)>DCI≥5-苄巯基-1-H-四氮唑(BTT)≥氯化吡啶,但只有DCI产生高强度和高稳定性的特征信号。而用噻吩基团取代NPPOC光敏基团后,光脱保护时间也缩短到几秒。通过优化,高复杂性DNA阵列合成中的偶联反应只需15 s,脱保护时间只需9 s,单循环的总时间(偶联反应-偶联反应)约50 s,整个合成时间被减少了三倍。在此基础上,非线性半导体光抗技术(non-linear semicondutor photoresist technology)的运用,使得阵点的间距更加微小,增加了单位面积上合成不同寡核苷酸序列的数量。
2.2 光敏抗蚀层合成法
IBM公司开发的“光敏抗蚀层合成法”类似于原位光刻法。该DNA合成方法基于酸脱保护的固相化学合成方法(亚磷酸酰胺法、氢磷酸酯法和磷酸三酯法),在每次选择性脱保护之前在芯片表面涂覆光敏抗蚀层(光刻胶),通过曝光显影把需要连接碱基区域的光刻胶层洗脱,在特定区域的核酸进行脱保护和偶联反应操作后,再把该层光刻胶层全部清除。该技术在合成一个碱基时要在芯片表面涂覆四次光刻胶,也需要采用四块不同的光掩模[70,72]。具体的制备方法如图6所示,首先对合成所用的玻璃基片进行硅烷化处理以便连接末端含羟基的连接手臂分子,手臂分子上端为羟基,并在其上连接一个带有保护基团的核苷酸(图6中的黑点);随后在其上均匀制备一层光敏抗蚀层,通过对应的光掩模把需要连接碱基的区域进行曝光显影后使待合成区域暴露出来,随后脱去核苷酸上的保护基团(黑点),并暴露出5′-羟基,在5′端发生偶联反应连接一个碱基。该方法运用成熟的酸脱保护合成试剂,有较高的合成产率。但其制备过程中需要大量的涂胶、显影、去胶等复杂光刻工艺,操作过程复杂,表面容易污染。特别是去胶过程要用化学活性较强的试剂和强力的物理条件,对表面上所合成的寡核苷酸会造成一定的损失。
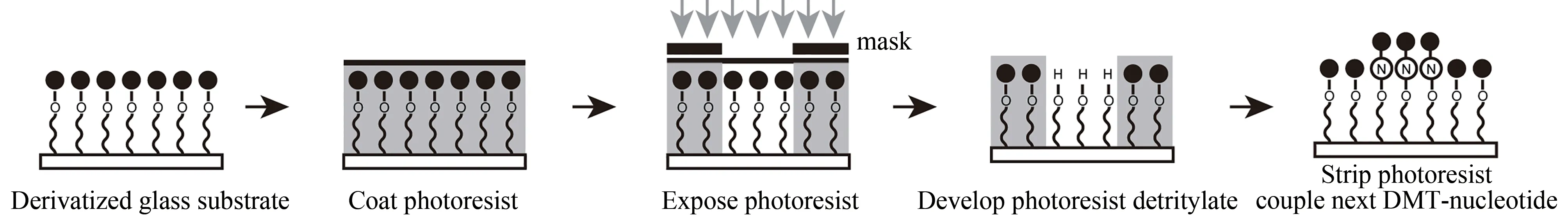
图6 光敏抗蚀层合成法原理(单层抗蚀剂工艺)Fig.6 Principle of parallel chemical synthesis of photoresist(Single layer resist process)
2.3 光致酸法
为了兼容光敏抗蚀层与寡核苷酸层之间的化学性质,人们开发了一种含有光致酸分子的光敏保护层[73-74],用其进行碱基选择性脱保护反应进而来制备基因芯片。在光致酸法中运用能够在光照下产生H+离子的化合物作为脱保护试剂。当施加光照时,光照区域的脱保护试剂产生大量的酸,使该区域核苷酸5′端的保护基团脱离,暴露出被激活的5′-羟基端,以进行下一步碱基的偶联合成反应,这样可简化制备基因芯片的工艺步骤。光致酸原位合成法除了用光致酸溶液替代传统的三氯乙酸外,其他的步骤与柱合成DNA相同。在光致酸脱保护这一步骤中,Gao等[75-76]引入了三芳基锍六氟锑酸盐(triarylsulfonium hexafluoroantimonate)溶液,在光的照射下,三芳基锍六氟锑酸盐光解生成活性较强的H+SbF-6,更容易解离出H+,从而使H+扩散区域的5′端保护基团DMT脱除,活化出5′-羟基,以进行下一步碱基合成(图7);而未光照区域周围的三芳基锍六氟锑酸盐,则不会发生光解反应而离解出H+,5'端的保护基团DMT仍然保留,不会发生偶联反应。光致酸DNA微阵列原位合成中合成反应需要与光致酸反应在溶液中并行运行。已有研究表明,光致酸的前体化合物如重氮酮磺酸盐、磺化钠或碘盐在二氯甲烷等有机溶液中,可以产生质子酸,并脱除5′端保护基团DMT[77-78]。除了光照条件外,还可以加入硫氰酮等增敏剂,这样10-3mol/L量级的光致酸前体溶液就能有效去除DMT基团,使5′-OH基团的核苷酸或载体可以用于下一个核苷酸的合成。实际上,某些光敏感的5′-OH保护基团,也就是核苷5′端的光活化基团,本身也可用作光致酸的前体。例如,在多肽微阵列合成中使用光致酸试剂2(-2-硝基苯基)丙氧羰基哌啶或2(-2-硝基苯基)丙氧羰基肼,可以提高光解反应效率[51]。
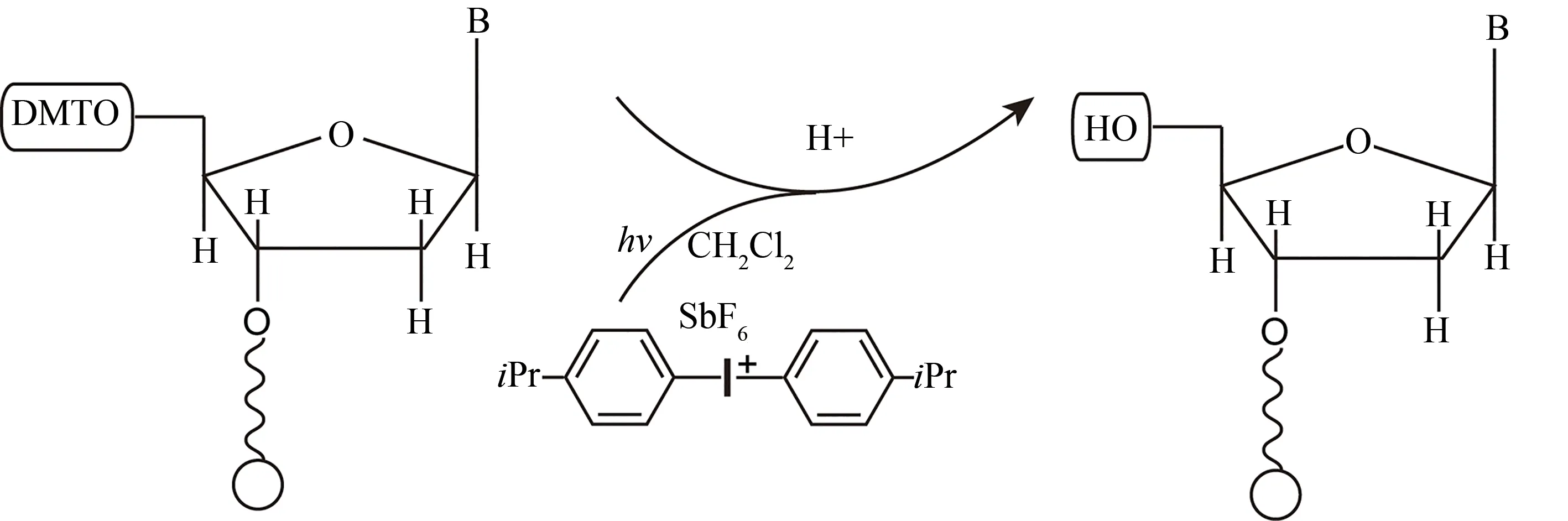
图7 光致酸合成法原理Fig.7 Chemical principles of photoacid DNA synthesis
2.4 喷印合成法
喷印合成法原理类似于在纸上喷射墨水所用的喷墨打印机。“墨盒”中装的是四种碱基的液体,喷印头可在整个芯片上移动并根据芯片上不同位点探针的序列需要将特定的碱基喷印到芯片上特定的位置[79-81]。与亚磷酸酰胺法DNA固相合成相比,主要的变化是用一种更加黏稠和不易挥发的溶剂,如己二腈,代替了常用的溶剂乙腈。己二腈代替乙腈的优势是减缓溶剂挥发,避免反应物固相化:延长液体与载体充分接触的反应时间,保障偶联效率。在玻璃基片上合成DNA阵列时,首先在基片上修饰一层一端为羟基的连接臂分子,计算机控制的微喷头可在整个芯片上移动,并根据芯片上不同位点DNA序列的需要将四种不同碱基的DNA合成单体试剂喷印在芯片上特定位置。微喷头在基片上方移动,分别喷印在基片表面的不同位置上(图8)。每次喷印即为在该点阵上进行一次偶联反应。当基片表面所有点阵全部喷印偶联完成后,再统一进行其他反应操作流程,包括冲洗、封闭,氧化和脱保护等化学处理步骤。然后,再进行下一个碱基的逐点喷印合成,循环各个步骤,直至完成目的DNA探针合成。喷印合成法单步合成只需输送很少量的偶联反应试剂,合成产率高,具有成本上的优势。喷印合成法可进行较长碱基序列的合成,可达到120个碱基以上,一般是低通量DNA合成的首选技术。喷印合成法也可以通过对所需脱保护的区域喷射三氯乙酸脱保护试剂的方法进行碱基的选择性合成。喷射脱保护试剂区域的保护基团被活化从而暴露出5′-羟基端以进行下一步的偶联反应。美国Agilent公司利用喷墨的原位合成技术合成了阵点大小为270μm,60个碱基的寡核苷酸芯片[82]。但该方法由于喷头的机械定位精度有限,喷印的位置不可能完全重叠。因此,喷印中心部位的产率比平均产率要高得多,限制了实现更高密度的DNA阵列合成。Twist Biosciences公司开发了一种基于半导体的合成DNA制造工艺,该工艺具有高通量硅平台,通过更加高效的反应效率使DNA合成所需的化学成分微量化。该技术将反应体所需的化学试剂体积减少100万倍,同时将合成通量提高了1000倍。实现了在单个硅芯片上高通量并行合成9600个基因。与传统的96孔板合成相比较,相同合成空间下96孔板只能合成单个基因。
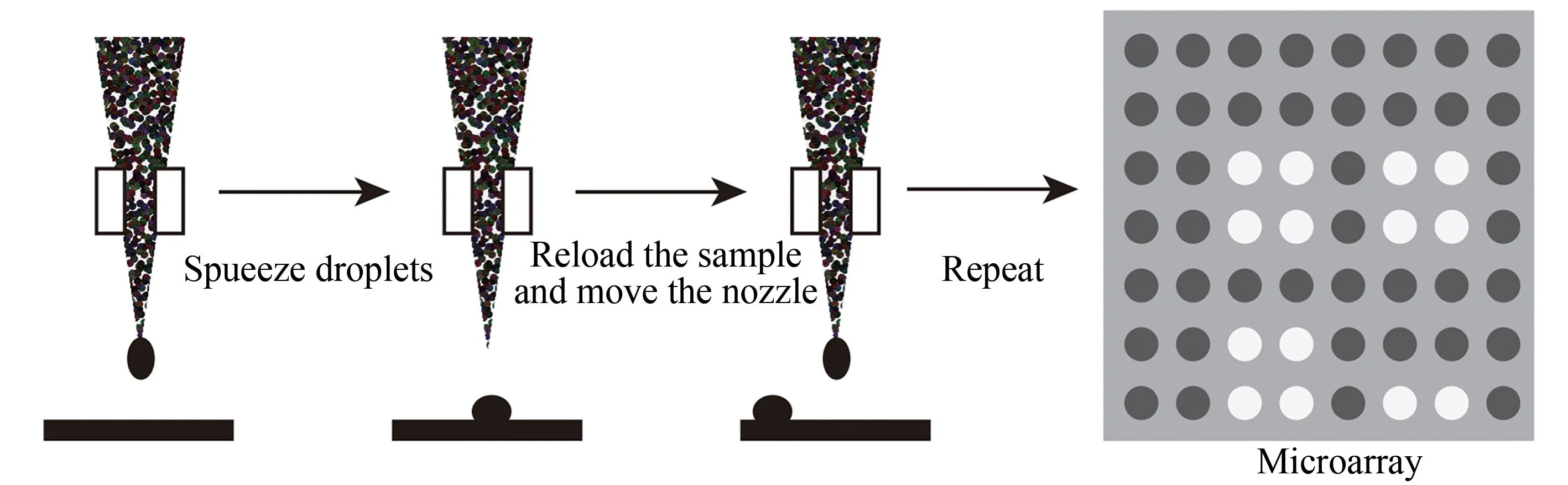
图8 喷印合成法示意图(通过机械手将“墨盒”中装的四种碱基合成试剂分别点样到基片预定设计的位置上进行碱基的合成,循环该步骤就可以在基片上不同位置合成出预设的DNA序列)Fig.8 Schematic diagram of the in-situ printing synthesis method(The four base synthesis reagents in the"cartridge"are respectively spotted to the predetermined position of the substrate to synthesize the base,and this step can be cycled to synthesize a preset DNA sequence in different positions of the substrate)
2.5 软光刻合成法
基于微阵列的软光刻合成法,是指模板与基质接触,将模板阵点上携带纳升级溶液通过印刷等方式转移到芯片表面进行反应的DNA微阵列制备技术。高质量DNA微阵列合成的关键技术是高效准确地将生物分子转移到基质指定的区域上,以及实现高效率的(偶联)反应[83-88]。软光刻原位合成法主要包括下列步骤[89]:①模板的制备;②模板携带的反应分子“墨水”向接触芯片转移;③反应分子在芯片上的原位合成。分子印章法原位合成DNA芯片是软光刻原位合成技术的一种,该方法采用涂有单核苷酸的印章多次压印在同一位置上,合成原理与印章盖印类似,将四种不同种类的核苷酸用四个不同印章压印在芯片上预先设定的部位[90-94](图9)。每层四种不同碱基的合成需要四个不同的印章携带相应的核苷酸液体(N个核苷酸长的芯片一般需要4N个压印步骤),通过“优化合成探针”把不同“层”相同碱基序列一同合成(比如第一个序列的第一个碱基C合成后,它的第二个碱基T可以和第二个序列的第一个碱基T一起合成)的设计可以减少16%的压印数[95]。该技术采用传统DNA固相合成的化学步骤,因此不需特殊制备的化学试剂即可以获得高的偶联效率。该方法成功合成出每立方厘米65 536种不同DNA序列的DNA微阵列。然而,分子印章法需要保持印章与芯片大面积间的平行微接触,否则将影响反应试剂有效传输,进而影响后续的合成效率。因此,在制备更高密度DNA芯片时该技术需要研制能够保持印章和芯片的大面积平行精密接触的压印机械装备。
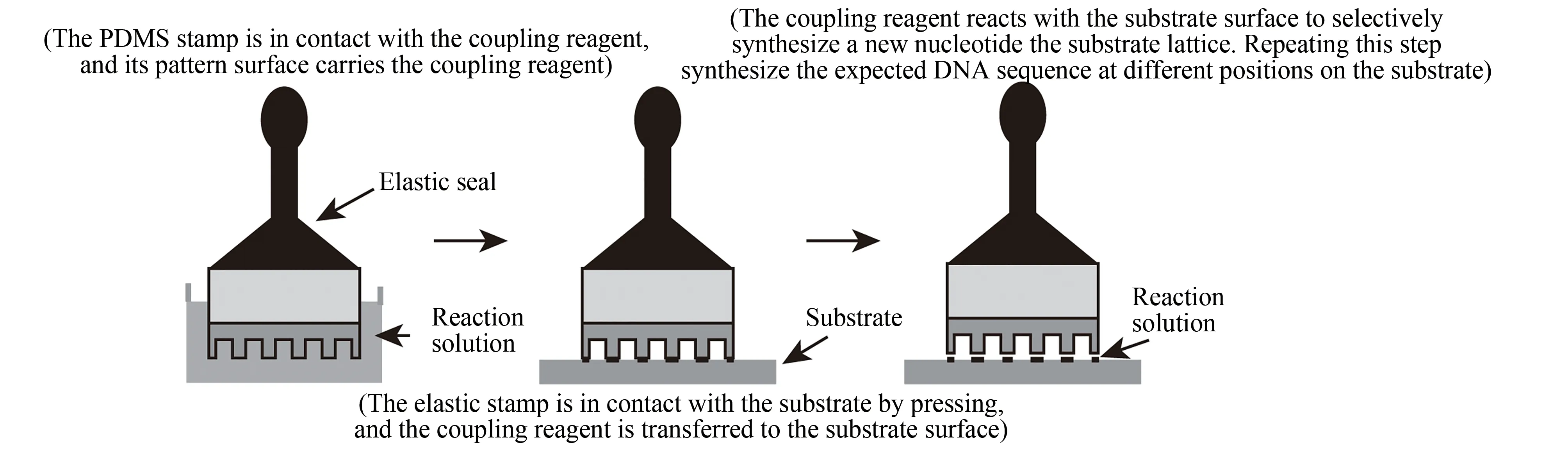
图9 分子印章原位合成法Fig.9 In-situ synthesis of soft-lithography
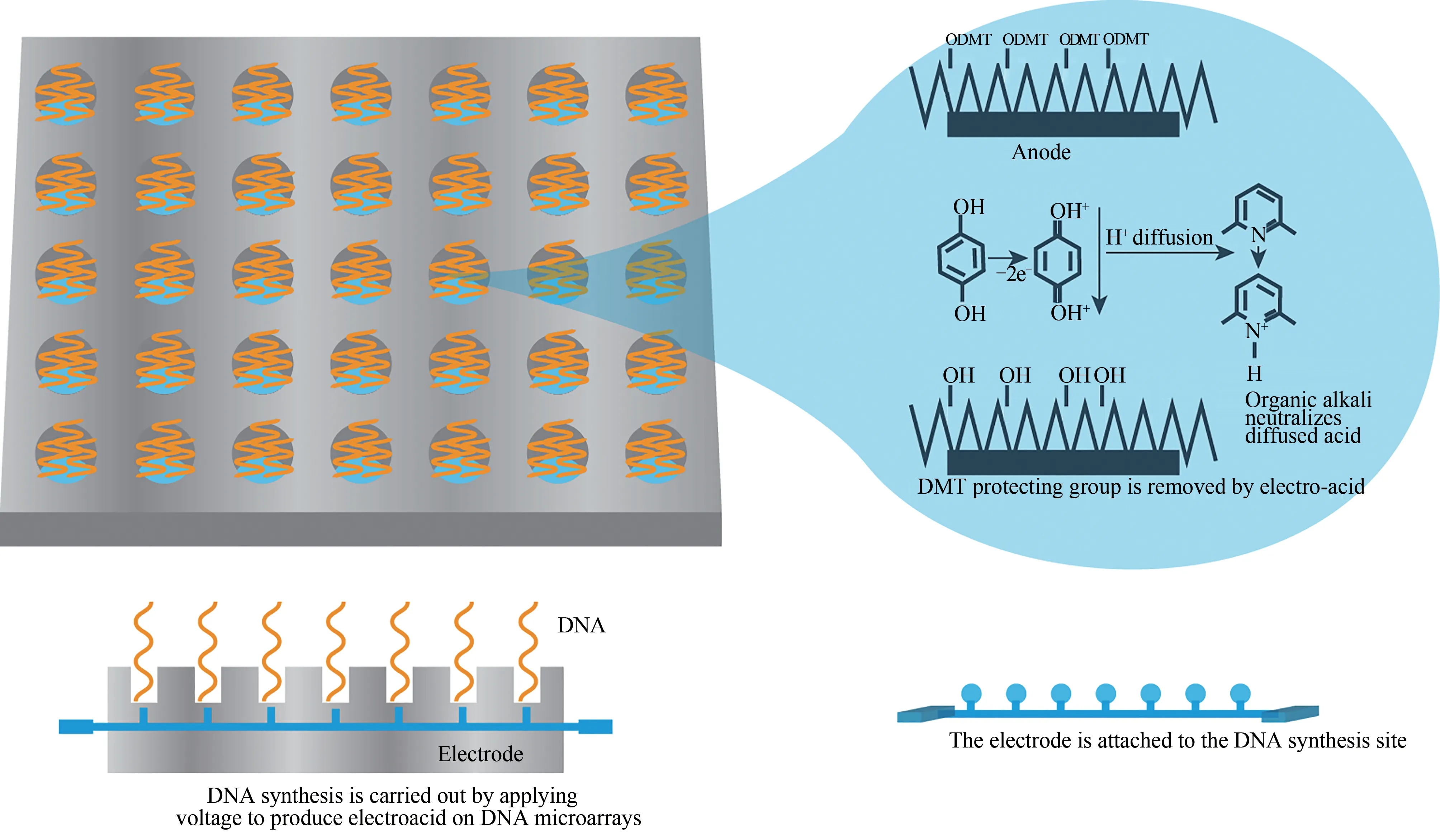
2.6 电致酸法
最早报导的电解致酸脱保护DNA芯片合成研究[96],在2.0 V电解电位下电解水,释放氢离子富集于阳极极板附近使得局部酸度增强,进而导致核酸单体分子发生脱保护反应。该研究发现,电解电位为2 V时,在0.1 mol/L高氯酸四乙基胺浓度条件下可获得较理想的实验结果。电解时间越长脱保护效率越高,两者基本呈线性关系,电解质浓度对脱保护效率基本无影响。
2005年Egeland和Southern报道了DNA微阵列的电化学脱保护合成法[97]。他们将正负电极做在同一块板上构成微电极阵列,与基片构成反应池,DNA合成反应试剂填充在反应池中。在含有氢醌(HQ)和苯醌(BQ)的乙腈溶液中,电解条件下,阳极发生氧化反应产生酸(阴影区域:HQ———→ H++Q),苯醌在阴极发生还原反应被再生成氢醌(H++Q———→ HQ)以减少阳极氢醌的消耗。酸(H+)从阳极迁移,向下迁移的酸将会到达基片表面并与DNA链末端DMT发生脱保护基团反应,从而有选择性地确定下一个核苷酸偶联反应发生的位置。
CombiMatrix公司(前身为Customarray)在早期从事微电极生物芯片制备的基础上[98-100],报道了电化学原位合成DNA阵列的方法[98,101]。他们利用以金属氧化物半导体(MOS)场效应晶体管为元件的集成电路芯片构成可寻址微电极阵列,单个可寻址的铂电极表面吸附、共价、共聚连接含有诸如蔗糖的高分子涂层,其表面带有大量羟基,以便衍生出足量用于DNA合成的连接分子。DNA合成时,计算机控制某些微电极通电,用可寻址电极阵列电解产生足够浓度的酸,选择性地将载体某些区域的DMT保护基团脱除,在有机碱存在的情况下生成的酸被控制在直径100μm的活性电极上(图10)。反应中较大的电流或电压将导致产生更多的酸,较长的反应时间将提供更多的时间去除DMT基团。但H+的扩散也随着浓度和时间增加而很难控制在直径100μm的活性电极上,进而导致污染。实验表明,合成20 mer探针的最佳窗口期为0.7~1.4μA/E。在电流常数保持在1μA/E条件下,电解1 min可以得到最佳的斑点形状和杂交强度,过长的时间将会导致斑点形状的退化,过短的时间将导致杂交强度的减弱。在上述电解条件下,5 mmol/L的2,6-二甲基吡啶提供了良好的酸扩散抑制环境,只在激活电极上进行了DNA合成;在低于1 mmol/L的2,6-二甲基吡啶条件下,非激活电极上也进行了DNA合成;当达到20 mmol/L时,激活电极上不能进行DNA合成。在不同的电压下,碱基浓度保持恒定在5 mmol/L,反应时间为60 s,这个过程中电压从1.2 V增加到2.6 V(改变间隔为0.2 V)。结果表明,在1.8 V的最佳电压下产生了良好的强杂交区域(代表高质量的DNA合成)。较低的电压产生的DNA质量较差,这可能是源于不完全脱保护;而在2.0 V以上的情况下可能破坏固定DNA的载体,从而降低DNA的合成质量。
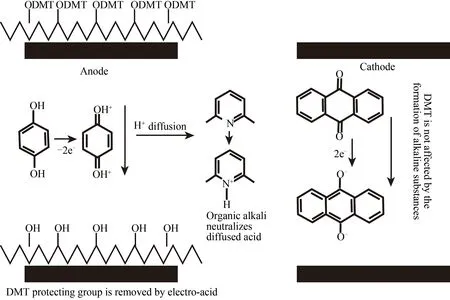
图10 电致酸法原理Fig.10 Principle of electrochemical deprotection microarray synthesis method
2009年麻省理工学院通过光电化学在虚拟电极附近产生氢离子进而合成DNA[102]。半导体电极通过光寻址应用于氧化还原反应的并行处理,即在光照射虚拟电极的附近产生氢离子,使周围的DMT保护基团脱除,曝露出羟基,供下一个核苷酸单体进行偶联反应;而其他未被光照射区域的DMT保护基团由于未被脱除而无法参与此次的偶联反应。依次循环制备单个阵点100μm的DNA微阵列。然而,该方法需要掩模层层间的精准对齐且不同的DNA序列需要不同的掩模。
目前基于集成电路芯片进行寻址合成DNA的技术已经商用化。该技术可以按照自己的设计快速合成所需的DNA片段,CombiMatrix公司可以为客户定制96K、12K、4×2K等不同类型的电化学合成DNA芯片,合成的DNA序列开始用于DNA存储研究[103]。
3 总结与展望
DNA微阵列原位化学合成具有高通量的特点,在降低成本和提高合成速度方面是满足合成生物学和DNA信息存储等快速发展需求的一种现实技术策略。我们把目前常用的基于DNA微阵列化学合成方法的技术特征和优缺点汇总在表1中。由表1可以看出,利用机械接触的DNA化学合成技术,其主要优势是可以提高每个阵点DNA片段的合成总量,但在芯片上合成不同DNA种类的通量方面提高空间十分有限。基于光学方法的原位合成技术,每个芯片一次可以获得百万级的寡核苷酸片段,通量虽高,DNA片段长度较短,需要提高光敏核酸基团的偶联效率,电致酸技术能够获得较长的核酸片段,但当芯片的通量大幅度提高时,游离H+会在电极间扩散,H+离子在电极间的串扰将成为DNA序列合成中重要的制约因素。
该模拟试验系统可进行掘进机机身位姿测量试验、不同形状断面自动截割轨迹规划模拟试验、掘进机智能控制试验等。

表1 基于微阵列DNA原位合成方法及其发展潜力Tab.1 Characteristicsof in situ synthesis of microarray DNA
化学合成法目前只能合成200 nt以内的DNA序列,长序列DNA片段合成仍存在着技术上的挑战。在长序列DNA合成方面目前主要通过后续拼接方法进行。从芯片合成的不同产物中挑选和鉴定出正确序列,结合多重PCR,选择性扩增所需的目的片段,通过DNA连接酶把短序列连接成长序列、结合合成序列纠错等实现超长DNA序列的制备。
近几年来,TdT聚合酶等DNA酶合成方法的合成准确率有所提高[104-105]。该方法在合成过程无需使用有害化学试剂,合成过程产生的副产物少;与目前化学合成法相比DNA酶合成法的合成条件更加温和,对DNA产物的损伤较小,具有长片段DNA合成的潜力。因此,基于DNA酶合成方法开始展示出巨大的发展潜力[106]。但目前TdT等聚合酶的合成方法还不能进行高通量平行地DNA合成,其低通量方法也尚未进入实际应用[11,107]。
英国创业公司Evonetix已经开发出一种独特的硅芯片,可以并行进行大规模的DNA合成。Evonetix并不是在物理分离井中创建不同的链,而是使用热工程来独立控制每个反应位点合成链的顺序。通过控制每个反应位点的温度,DNA的生长链被选择性地脱保护,为DNA序列添加新碱基做准备。由于四种碱基(A、C、G、T)的试剂依次引入,控制热脱保护的时间使得在每个反应位点合成不同的序列。由于硅芯片能够精确和独立地控制各个反应位点的温度,可以在将任何有错误的链并入双链DNA之前检测并移除它们。可以通过了解退火DNA链的熔化温度和序列错误引起的温度变化来实现这一点。这节省了搜索和组装无错DNA所需的工作。通过从短链组装长的、双
链的DNA,能够在组装过程的每一步测试准确性,从而防止错误污染最终产品。独特的二元组装过程是革命性DNA制造方法的核心。目前集成电路工艺技术已经突破10 nm,这为进一步大幅度提高微阵列DNA合成的通量提供了技术基础。对于DNA微阵列的电致酸合成方法,主要瓶颈是每个微小电极之间H+相互串扰,通过优化适用于DNA芯片的微电极以及相关微流控通道材料、工艺和结构设计,在微纳尺度上高效准确地调控微小电极上的氢离子输运。考虑到目前半导体集成电路芯片结构和材料等改进,人们正在克服电极之间酸性液体的串扰效应,电致酸技术的合成通量预计将能够大幅度提高。可以预测,电致酸合成方法有可能在现有基础上把DNA的合成通量进一步提高103量级。通过计算机自动化微流控技术,优化合成过程中相关化学反应过程,阻止过量的液相试剂进入芯片,减少每一步反应中试剂纯化步骤等[14,62,65,108-111]。目前我国在高通量DNA合成方面也有突破,泓迅科技Syno®3.0平台采用精准的电化学技术,可在一张半导体芯片上一次性合成上万甚至十万条引物。通过创新性的合成流程设计及质量管理确保可以提供高质量的DNA片段(包括引物、元件以及基因),实现全基因合成、通路合成、基因组合成、核苷酸池以及各种突变库的合成。对于CMOS芯片的电致酸DNA原位化学合成技术而言,目前仍然存在着“边缘效应”引起的离子串扰等问题,包括反应液滴未对准、试剂封闭不力和光控系统中光束偏移导致不精确的脱保护反应,使邻近区域的寡核苷酸序列产生碱基错误。通过对芯片设计的改进可以提高微阵列寡核苷酸的保真度,此外新型微电极材料和结构设计的运用,也能够很好地解决芯片上微电极间氢离子串扰问题,从而进一步提高DNA合成通量和速度。可以预计通过采用100 nm以下的集成电路工艺,单片DNA合成通量有望达到1012碱基,实现TB级的DNA快速低成本合成。

