成人与儿童急性髓系白血病患者肿瘤免疫相关的差异表达基因分析
李玲玲,李倩,李明玉,刘峥,沈倩诚
1.上海市儿童医院,上海交通大学附属儿童医院中心实验室,上海200062;2.上海交通大学基础医学院医药生物信息学中心,上海200025;3.上海工程技术大学管理学院,上海201620
急性髓系白血病(acute myeloid leukemia,AML)是一类发病机制具有高度异质性的造血干细胞异常的克隆性疾病,由于患者骨髓中不同分化阶段的髓系细胞分化阻滞与异常增殖,常出现造血功能衰竭、髓外器官浸润、代谢功能异常等临床表现[1]。AML可发生于任何年龄,在儿童急性白血病中的占比约为30%,在成人急性白血病中的占比约为80%。患有AML的儿童长期生存率为40%~60%,60岁以下的中青年患者长期生存率为35%~40%,60岁以上的老年患者则为5%~15%[2]。AML的预后不良多与复发和治疗相关的并发症有关。尽管AML的细胞分型和基因分型是预测患者生存率的重要指标,但年龄仍然是重要的预后独立预测因素之一,除了可能与老年人的身体条件较差有关以外,还可能与不同年龄阶段的骨髓免疫基因表达差异有关。AML表现为造血干细胞的分化异常,这与免疫基因的表达密不可分。肿瘤免疫相关基因的表达是免疫细胞、肿瘤干细胞、基质细胞与细胞因子之间相互作用的结果,它们共同进化,最终形成了对肿瘤有支持作用的肿瘤微环境,从而促进了白血病的发生、发展[3]。
为了探讨AML患者骨髓中肿瘤免疫相关基因的表达是否影响患者的长期生存率,本研究对GSE134589数据集中不同年龄段的AML患者骨髓中的肿瘤免疫相关基因进行生物信息学分析。根据不同年龄段的生存率差异[2],将AML患者分为儿童组、中青年组和老年组,筛选差异表达基因(differentially expressed genes,DEGs),探究骨髓中肿瘤免疫相关的DEGs与成人AML预后较差的相关性,并寻找对AML发展有关键影响的免疫基因。
1 资料与方法
1.1 数据筛选
在GEO(Gene Expression Omnibus)[4]数据库(http://www.ncbi.nlm.nih.gov/geo/)搜索并下载GSE134589数据集。此数据集是基于GPL26936平台,检测了442例AML患者骨髓穿刺样本的730个免疫相关基因的芯片数据[5],包含基因表达矩阵和样本信息,如取样时的患者状态、年龄、取样部位、RNA的处理方法、芯片信息等。另一部分数据来源于UCSC Genome Browser网站[6](https://xenabrowser.net/datapages/)的TCGA(The Cancer Genome Atlas)数据库(https://portal.gdc.cancer.gov/)AML数据集和GTEx(Genotype-Tissue Expression)数据库(https://gtexportal.org/home/)正常组织的RNA-Seq数据集。
1.2 DEGs筛选
根据长期生存率的差异[2],将数据分为儿童组(0~16岁)、中青年组(17~59岁)、老年组(60~80岁),选取初次确诊与复发的样本,其中儿童组34例,中青年组62例,老年组62例。基因表达的差异用FDR(false discovery rate)校 正 后 的P值(q-value)和 差 异 倍 数(fold change,FC)的对数(log2FC)表示。本研究以qvalue<0.01且|log2FC|>2为筛选条件,使用R语言(版本4.0.2)limma、pheatmap、ggplot2等程序包[7]分析中青年组与儿童组、老年组与儿童组、中青年组与老年组的DEGs。选择取样时患者状态为完全缓解的样本,以同样的条件筛选儿童组(5例)和成人组(17~80岁,23例)的DEGs,并与初次确诊/复发样本的DEGs进行比较。
1.3 富集分析
取中青年与儿童AML患者的DEGs和老年与儿童AML患者的DEGs的交集,绘制韦恩图,取共同上调或下调的DEGs,使用Metascape.org[8]映射到GO(Gene Ontology)、Reactome和KEGG(Kytoto Encyclopedia of Genes and Genomes)的生物过程和信号通路,将P<0.05作为其具有统计学意义的指标。
1.4 DEGs的mRNA表达及其与总体生存期的相关性分析
GEPIA(Gene Expression Profiling Interactive Analysis,GEPIA)是一个交互式网站服务器[9],包含TCGA和GTEx项目的33种癌症和正常样品的RNA测序表达数据。根据TPM(transcripts per million)归一化方法,将AML患者分为DEG高表达组(53例)和DEG低表达组(53例),采用Kaplan-Meier法分析以上2组组织中DEGs对AML总体生存期的影响,筛选与预后显著相关的基因,将其作为关键基因。
1.5 构建DEGs的蛋白质-蛋白质相互作用网络
使用在线数据库STRING(https://string-db.org/)分析蛋白质-蛋白质相互作用(protein-protein interaction,PPI)[10],构建DEGs的PPI网络;采用Cytoscape 3.8.0软件 进 行 可 视 化[11],并 使 用Cytoscape的CytoHubba插件[12]的EPC(edge percolated component)拓扑分析方法筛选连接度高的为有主要作用的蛋白分子。分别用Cytoscape的MCODE[13]与Metascape.org在线工具对PPI网络进行密集度分析,进一步筛选PPI网络最显著模块的中心节点基因,也将其作为关键基因。
1.6 关键基因验证
利用GEPIA网站对关键基因在173例AML患者白细胞样本与70例正常人白细胞样本的表达数据进行验证;同时横向对比关键基因在47例弥漫大B细胞淋巴瘤(diffuse large B cell lymphoma,DLBCL)肿瘤组织与337例正常人全血样本、118例胸腺癌(thymoma,THYM)肿瘤组织与337例正常人全血样本以及2例正常胸腺组织样本的表达数据,预测关键基因在肿瘤发展过程中的功能。从Gene Expression Commons(https://gexc.riken.jp/models/7/genes)下载数据[14],分析关键基因在AML患者肿瘤干细胞和健康造血细胞中mRNA的表达情况,筛选具有肿瘤干细胞特异性的基因。
2 结果
2.1 不同年龄段AML患者肿瘤免疫相关的DEGs聚类分析
本研究用R语言分析来源于数据集GSE134589的DEGs。将初次确诊与复发的样本分为儿童组34例、中青年组62例、老年组62例。从热图中,可以看出中青年组与儿童组(图1A)、老年组与儿童组(图1B)免疫相关基因的差异区分明显,组内基因表达情况具有高度一致性。
2.2 不同年龄段AML患者肿瘤免疫相关DEGs的筛选
将 参 数 符 合q-value<0.01且|log2FC|>2的 基 因 视 为DEGs,使用R语言绘制AML患者骨髓中肿瘤免疫相关基因的火山图。中青年组与儿童组比,结果显示上调的DEGs有51个,下调的有21个(图2A);老年组与儿童组比,上调的DEGs有47个,下调的有20个(图2B);而中青年组与老年组比,没有发现DEG(图2C),提示儿童与成人(包括老年人)AML患者骨髓中肿瘤免疫相关基因的表达具有显著差异,而成年后患者间的差异不明显。
2.3 DEGs的韦恩图和富集分析
从DEGs的韦恩图可知,相比儿童组,中青年组和老年组共同上调的基因有39个(图3A);共同下调的基因有18个(图3C)。为了探索DEGs相关的信号通路,用Metascape.org网站对39个共同上调的基因和18个共同下调的基因进行富集分析,结果显示共同上调的DEGs主要富集在白细胞迁移、细胞因子介导的信号通路、细胞因子产生的调节、细胞-细胞黏附的调节、造血细胞谱系、髓 系 白 细 胞 激 活、核 因 子-κB(nuclear factor-κB,NF-κB)信号通路和白介素介导的信号转导等(图3B);共同下调的DEGs主要富集在细胞对脂多糖的响应、白细胞增殖、炎症反应的正向调控、细胞因子产生的调节、白细胞迁移的调控和细胞因子介导的信号通路等(图3D)。
分析完全缓解状态下的成人与儿童AML患者肿瘤免疫相关基因的差异,与发病期内筛选出的共同DEGs比对后发现,仅白细胞表面抗原CD53(leukocyte surface antigen CD53,CD53)、原癌基因c-Rel(proto-oncogene c-Rel,REL)、趋化因子配体2(C-X-C motif chemokine ligand 2,CXCL2)这3个基因相同,发病期间成人与儿童之间的DEGs绝大部分在病情完全缓解后表达不再呈现差异(表1)。说明成人与儿童骨髓中的肿瘤免疫相关基因在AML发病过程中表达的差异多数与疾病有关,以成人和儿童分组来分析AML患者的DEGs具有生物学意义。
2.4 影响AML患者总体生存期的基因筛选
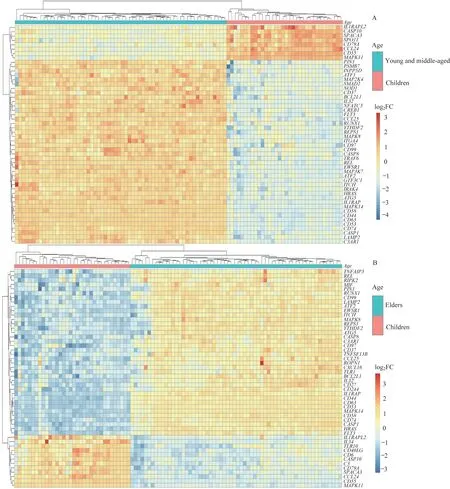
图1不同年龄分组的AML患者肿瘤免疫相关基因表达的热图分析Fig 1 Heatmap analysis of tumor immune-related genes expression in the AML patients of different age groups
为了探索DEGs与患者预后的相关性,使用Kaplan-Meier法分析DEGs的mRNA表达量对AML患者总体生存期的影响。结果显示在成人AML患者骨髓中上调的DEGs中,IL2RA和FLT3基因高表达组患者的总体生存期显著缩短(图4);下调的DEGs均对患者总体生存期无显著影响。
2.5 PPI网络与关键模块筛选
使用在线数据库STRING分析39个共同上调DEGs对应的蛋白之间的相互作用,通过Cytoscape 3.8.0软件进行可视化分析(图5A),采用Cytoscape的CytoHubba插件筛选连接度最高的18个主要蛋白分子,包含IL2RA、FLT3、C3AR1、趋化因子和黏附因子等(图5B)。分别用Cytoscape的MCODE与Metascape.org在线工具对PPI网络进行密集度分析,得出最显著模块均是以C3AR1为中心的调控网络(图5C)。

图2不同年龄分组的AML患者肿瘤免疫相关基因的火山图分析Fig 2 Volcano map analysis of tumor immune-related genes in the AML patients of different age groups
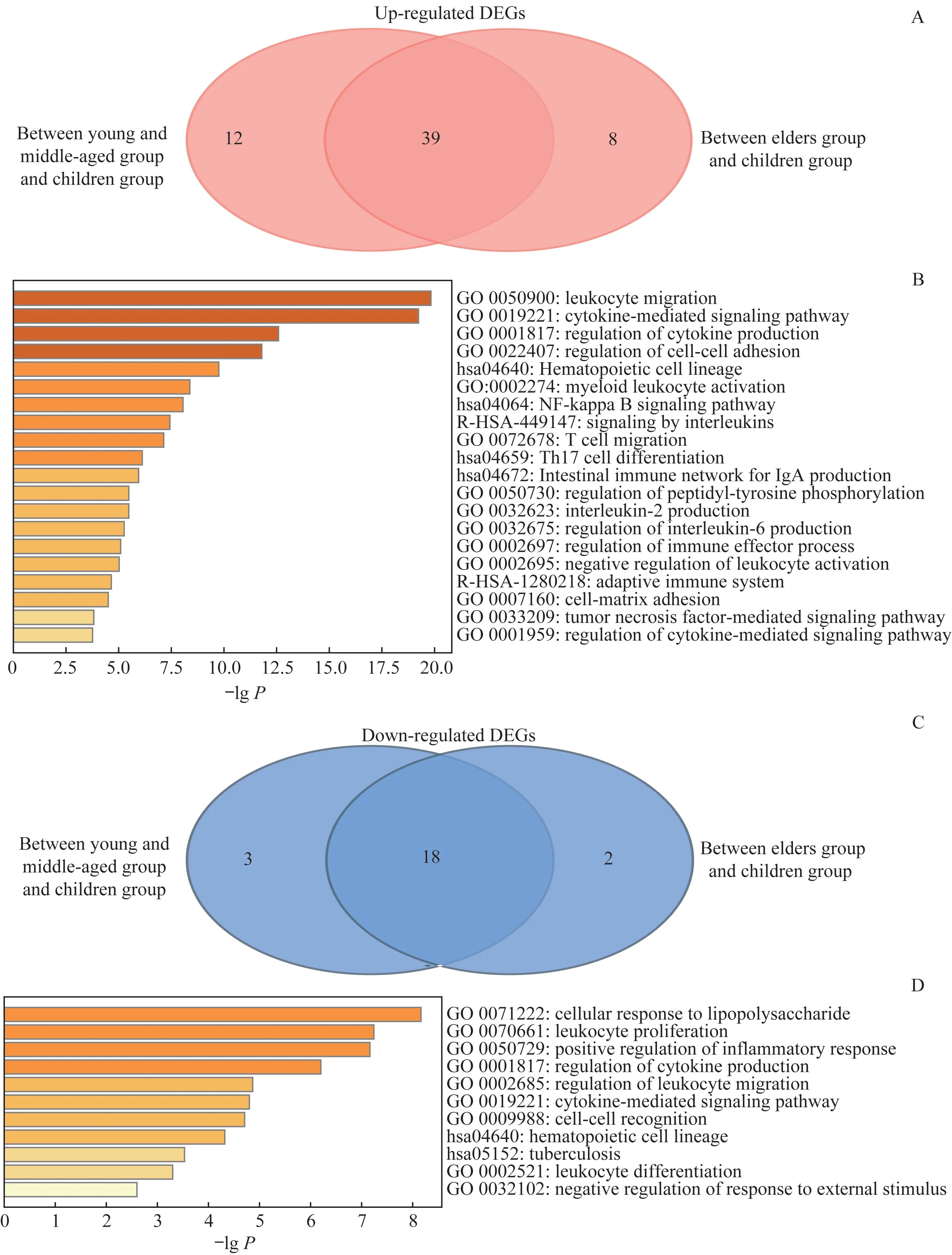
图3不同年龄分组的AML患者肿瘤免疫相关DEGs的韦恩图与富集分析Fig 3 Venn map and enrichment analysis of tumor immune-related DEGs in the AML patients of different age groups
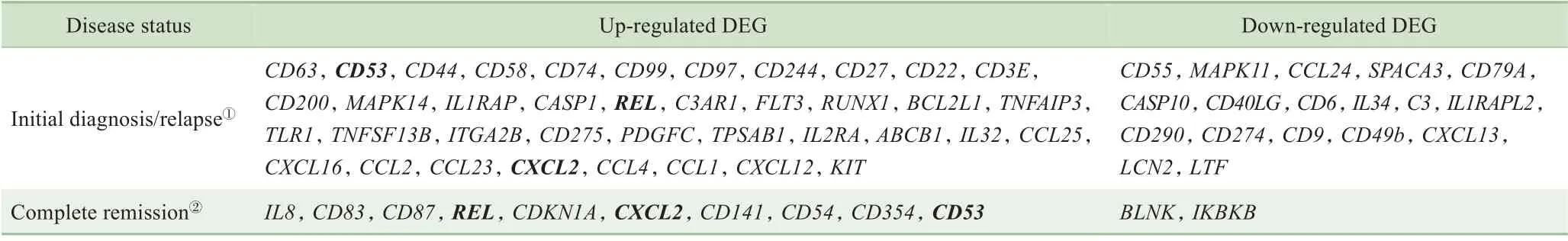
表1 AML患者在初次确诊/复发和完全缓解状态下的肿瘤免疫相关DEGs的比较Tab 1 Comparison of tumor immune-related DEGs between the AML patients in initial diagnosis/relapse and in complete remission
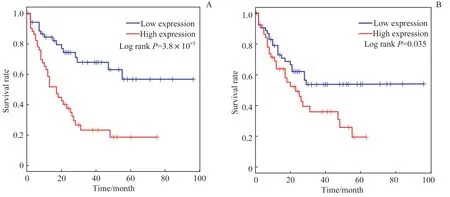
图4 IL2RA和FLT3 mRNA不同表达水平的AML患者的总体生存期分析Fig 4 Overall survival analysis of the AML patients with different levels of IL2RA and FLT3 mRNA expression
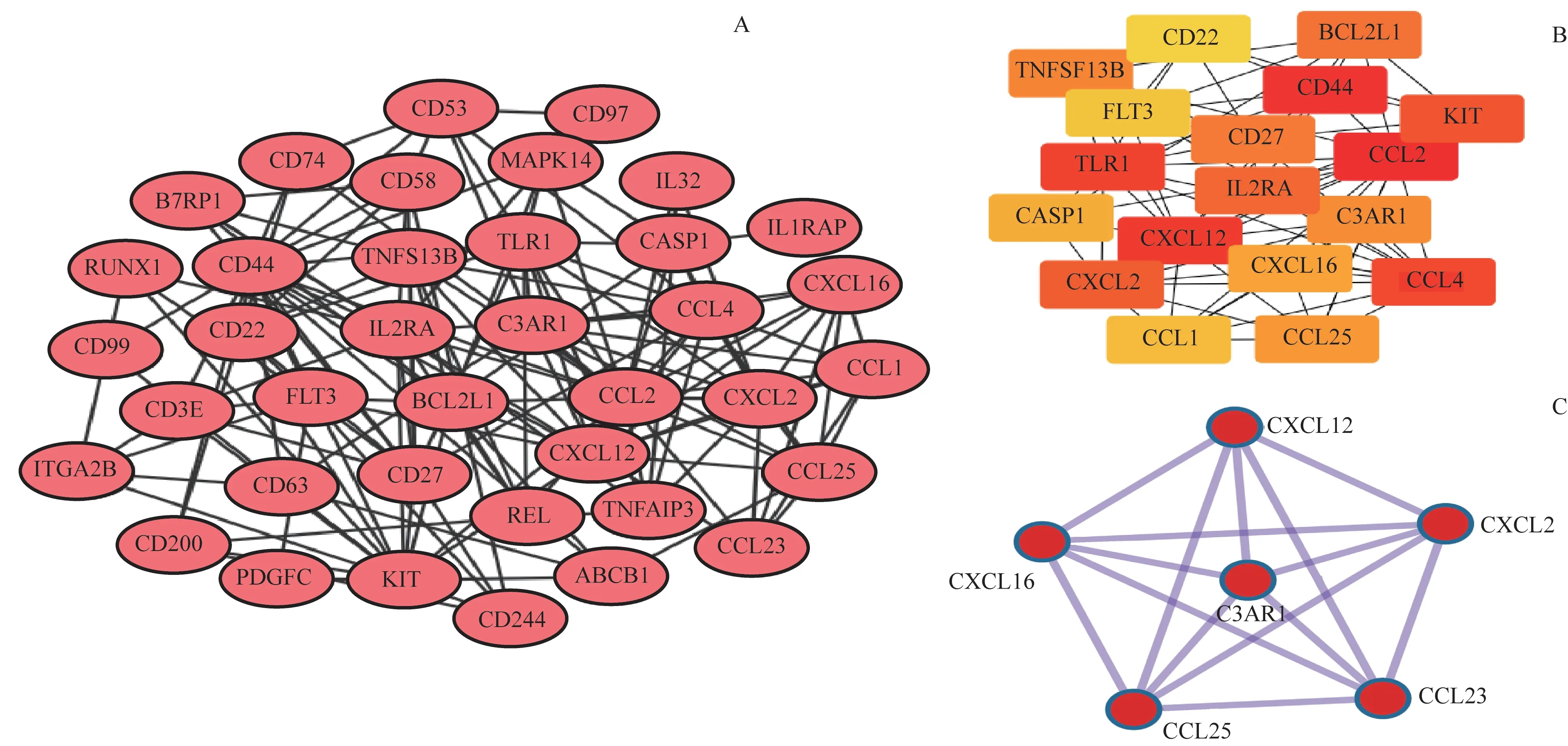
图5上调DEGs对应蛋白的PPI网络与最显著模块图Fig 5 PPI network and the most significant modules of up-regulated DEGs-corresponding proteins
2.6 关键基因的验证
综合分析生存曲线和PPI网络的关键模块,结果显示高表达IL2RA、FLT3的AML患者预后较差,而C3AR1在AML骨髓免疫的蛋白质网络里有重要作用,它们可能是对成人预后有关键影响的DEGs,因此作为关键基因。将GEPIA网站提供的AML、DLBCL和THYM患者的肿瘤样本和正常人样本的IL2RA、FLT3、C3AR1表达数据进行分析,结果显示IL2RA、FLT3、C3AR1均在AML患者样本中高表达,在正常人样本中低表达(图6A)。此外,IL2RA还在DLBCL患者样本中显著高表达,而在THYM患者样本中无明显变化(图6A)。
从Gene Expression Commons下载数据,分析关键基因在正常人骨髓和脐带血的造血干细胞、祖细胞、CD45 RA阳性祖细胞等原始造血细胞以及AML患者的肿瘤干细胞、祖细胞和芽细胞共12种低分化造血细胞中的表达情况。结果显示,IL2RA均呈低表达(图6B),提示IL2RA可能表达在分化较成熟的造血细胞上。同样,通过分析FLT3和C3AR1表达情况发现,FLT3在多数低分化造血细胞中均高表达(图6B),在DLBCL、THYM患者样本和正常人样本中均低表达(图6A),提示FLT3仅表达在未分化成熟的细胞上,在淋巴、胸腺和血液中分化相对成熟的细胞上呈低表达;而FLT3在AML肿瘤样本高表达,推测可能与肿瘤干细胞的增殖有关。C3AR1在AML患者的肿瘤干细胞、祖细胞和芽细胞中高表达,在正常的骨髓和脐带血的原始造血细胞中低表达(图6B),提示其可能具有在肿瘤干细胞特异性高表达的特征。
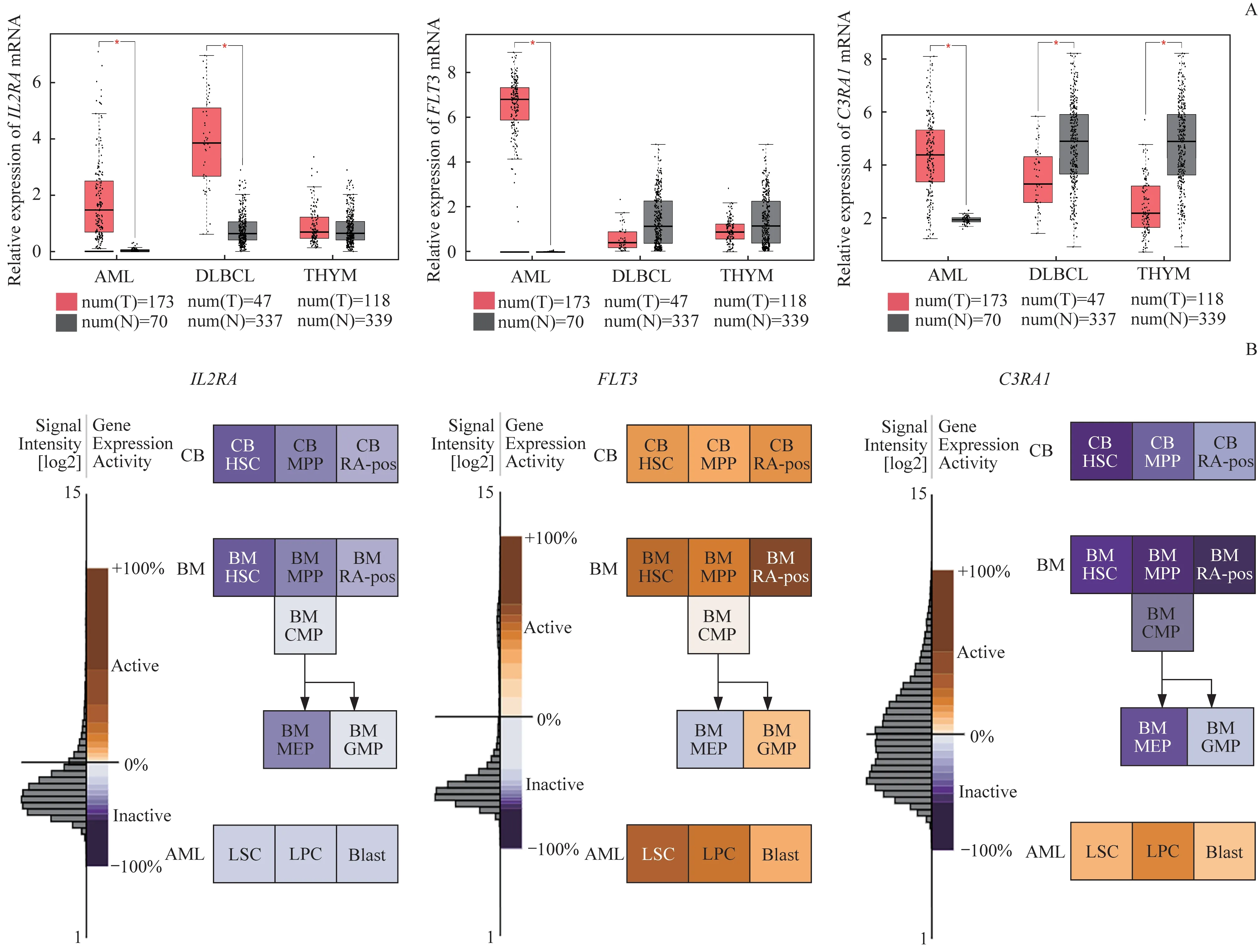
图6关键基因在3个病种肿瘤组织中以及在AML患者肿瘤干细胞与正常人造血细胞中的mRNA表达情况Fig 6 mRNA expression of key genes in the tumor tissues of the three diseases and in the tumor stem cells of the AML patients and the hematopoietic cells of normal people
3 讨论
免疫细胞与白血病干细胞、基质细胞和各种细胞因子等相互作用共同组成了骨髓肿瘤免疫抑制性微环境[15]。随着肿瘤的发展,免疫细胞的相关基因表达量也会发生动态改变[5],从免疫细胞的迁移、抑制性细胞因子释放、肿瘤表面抑制性受体上调、基质重塑和代谢异常等方面影响着肿瘤微环境[16]。AML是一种免疫细胞分化异常的肿瘤,从形态学上分为M0~M7共8个亚型[17];除了M3以外,其他亚型的预后并不理想,成人的患病率明显高于儿童而治愈率低于儿童。通过对比成人与儿童AML患者骨髓中肿瘤免疫相关基因的差异,可能有助于找到两者预后差异的原因。
本研究分析了来源于GEO的GSE134589数据集,热图结果显示成人与儿童AML患者骨髓中的多个肿瘤免疫相关基因的差异具有统计学意义。其中成人患者中高表达的IL2RA基因可通过控制调节性T细胞(regulatory T cell,Treg)的活性,抑制T细胞增殖,调节机体免疫耐受[18-19]。FLT3是原癌基因,参与调节造血祖细胞增殖,是AML患者中最常见的突变基因之一,即使在没有FLT3突变的情况下,芽细胞高表达FLT3可能与AML患者预后不良有关[20]。C3AR1属于G蛋白偶联受体的超家族成员,含7个跨膜结构,补体C3a-C3aR轴能够促进造血干细胞对Ⅰ型血管细胞黏附蛋白的黏附、增加造血干细胞对CXCL12的趋化敏感性[21]。CXCL12是已知的唯一对造血干细胞有趋化作用的因子,CXCL12-CXC趋化因子受体4(C-X-C chemokine receptor type 4,CXCR4)轴在造血干细胞的归巢和外周干细胞的动员中扮演着重要角色[22]。研究[23]表明,在肿瘤中CXCL12-CXCR4轴可激活NF-κB通路,增加癌细胞的迁徙能力和侵袭能力,进一步阐明C3a-C3aR与CXCL12-CXCR4轴之间的相互作用可为白血病干细胞在骨髓和外周的活动提供理论基础。成人AML患者发病期间特异性上调表达的基因中,CD97、IL1RAP是白血病干细胞关键的调控基因,是预后不良的指标[24-25];BCL2L1是BCL-2抗凋亡家族基因[26];IL-6可刺激骨髓瘤细胞的增殖[27];ABCB1转运体主要通过ATP水解释放能量将药物分子泵出细胞[28],导致肿瘤细胞药物蓄积减少和多药耐药性的发展;CCL2可促进肿瘤细胞发生迁移和侵袭[29]等。这些基因在肿瘤的发生、增殖和转移中发挥重要作用,它们的差异可能是成人预后比儿童差的影响因素。
利用Kaplan-Meier生存曲线和Cytoscape软件筛选了3个潜在的对AML疾病发展有重要影响的关键基因:IL2RA、FLT3和C3AR1。对GEPIA网站提供的AML肿瘤组织和正常人样本的mRNA表达数据进行分析,结果显示3个关键基因在AML肿瘤样本显著高表达,具有肿瘤组织特异性。IL2RA在正常的原始造血细胞和AML肿瘤干细胞上均低表达,推测其主要在分化较成熟的细胞上表达。然而,和正常人样本相比,IL2RA在AML和DLBCL中显著高表达,在THYM中无明显差异。DLBCL是肿瘤性大B淋巴细胞呈弥漫性生长的肿瘤,而B细胞是在骨髓中发育成熟的,推测AML患者骨髓中IL2RA的高表达可能与B细胞有关。有研究[18-19]证明B细胞可诱导Treg的分化,而Treg抑制反应性T细胞的活化和扩增,介导的免疫抑制是至关重要的肿瘤免疫逃逸机制之一。FLT3主要表达在未分化成熟的细胞上,它的高表达可能与肿瘤干细胞的异常增殖有关。而C3AR1具有在AML肿瘤干细胞中特异性高表达的特征,可作为AML的潜在靶分子开展进一步研究。
综上所述,本研究通过生物信息学的方法综合分析了不同年龄段的AML患者骨髓中肿瘤免疫相关基因,结果显示成人与儿童AML患者预后的差异与其中的DEGs有关,进而筛选出了对AML疾病发展可能有不利影响的关键基因,并预测了它们在肿瘤发展中的功能,为不同年龄段AML患者的免疫治疗和实验研究提供了一定的方向。
参·考·文·献
[1]Vago L,Gojo I.Immune escape and immunotherapy of acute myeloid leukemia[J].J Clin Invest,2020,130(4):1552-1564.
[2]Döhner H,Weisdorf DJ,Bloomfield CD.Acute myeloid leukemia[J].N Engl J Med,2015,373(12):1136-1152.
[3]Swartz MA,Iida N,Roberts EW,et al.Tumor microenvironment complexity:emerging roles in cancer therapy[J].Cancer Res,2012,72(10):2473-2480.
[4]Edgar R,Domrachev M,Lash AE.Gene Expression Omnibus:NCBI gene expression and hybridization array data repository[J].Nucleic Acids Res,2002,30(1):207-210.
[5]Vadakekolathu J,Minden MD,Hood T,et al.Immune landscapes predict chemotherapy resistance and immunotherapy response in acute myeloid leukemia[J].Sci Transl Med,2020,12(546):eaaz0463.
[6]Raney BJ,Dreszer TR,Barber GP,et al.Track data hubs enable visualization of user-defined genome-wide annotations on the UCSC Genome Browser[J].Bioinformatics,2014,30(7):1003-1005.
[7]Ritchie ME,Phipson B,Wu D,et al.Limma powers differential expression analyses for RNA-sequencing and microarray studies[J].Nucleic Acids Res,2015,43(7):e47.
[8]Zhou Y,Zhou B,Pache L,et al.Metascape provides a biologist-oriented resource for the analysis of systems-level datasets[J].Nat Commun,2019,10(1):1523.
[9]Tang Z,Li C,Kang B,et al.GEPIA:a web server for cancer and normal gene expression profiling and interactive analyses[J].Nucleic Acids Res,2017,45(W1):W98-W102.
[10]Szklarczyk D,Franceschini A,Wyder S,et al.STRING v10:protein-protein interaction networks,integrated over the tree of life[J].Nucleic Acids Res,2015,43(Database issue):D447-D452.
[11]Shannon P,Markiel A,Ozier O,et al.Cytoscape:a software environment for integrated models of biomolecular interaction networks[J].Genome Res,2003,13(11):2498-2504.
[12]Chin CH,Chen SH,Wu HH,et al.cytoHubba:identifying hub objects and sub-networks from complex interactome[J].BMC Syst Biol,2014,8(Suppl 4):S11.
[13]Bandettini WP,Kellman P,Mancini C,et al.MultiContrast Delayed Enhancement(MCODE)improves detection of subendocardial myocardial infarction by late gadolinium enhancement cardiovascular magnetic resonance:a clinical validation study[J].J Cardiovasc Magn Reson,2012,14:83.
[14]Gentles AJ,Plevritis SK,Majeti R,et al.Association of a leukemic stem cell gene expression signature with clinical outcomes in acute myeloid leukemia[J].JAMA,2010,304(24):2706-2715.
[15]Klemm F,Joyce JA.Microenvironmental regulation of therapeutic response in cancer[J].Trends Cell Biol,2015,25(4):198-213.
[16]Junttila MR,de Sauvage FJ.Influence of tumour micro-environment heterogeneity on therapeutic response[J].Nature,2013,501(7467):346-354.
[17]Bennett JM,Catovsky D,Daniel MT,et al.Proposals for the classification of the acute leukaemias[J].Br J Haematol,1976,33(4):451-458.
[18]Olkhanud PB,Damdinsuren B,Bodogai M,et al.Tumor-evoked regulatory B cells promote breast cancer metastasis by converting resting CD4⁺T cells to T-regulatory cells[J].Cancer Res,2011,71(10):3505-3515.
[19]Wejksza K,Lee-Chang C,Bodogai M,et al.Cancer-produced metabolites of 5-lipoxygenase induce tumor-evoked regulatory B cells via peroxisome proliferator-activated receptorα[J].J Immunol,2013,190(6):2575-2584.
[20]Yamamoto Y,Kiyoi H,Nakano Y,et al.Activating mutation of D835 within the activation loop of FLT3 in human hematologic malignancies[J].Blood,2001,97(8):2434-2439.
[21]Reca R,Mastellos D,Majka M,et al.Functional receptor for C3a anaphylatoxin is expressed by normal hematopoietic stem/progenitor cells,and C3a enhances their homing-related responses to SDF-1[J].Blood,2003,101(10):3784-3793.
[22]Möhle R,Bautz F,Rafii S,et al.The chemokine receptor CXCR-4 is expressed on CD34+hematopoietic progenitors and leukemic cells and mediates transendothelial migration induced by stromal cell-derived factor-1[J].Blood,1998,91(12):4523-4530.
[23]Lin Y,Ma Q,Li L,et al.The CXCL12-CXCR4 axis promotes migration,invasiveness,and EMT in human papillary thyroid carcinoma B-CPAP cells via NF-κB signaling[J].Biochem Cell Biol,2018,96(5):619-626.
[24]Martin GH,Roy N,Chakraborty S,et al.CD97 is a critical regulator of acute myeloid leukemia stem cell function[J].J Exp Med,2019,216(10):2362-2377.
[25]Zhao K,Yin LL,Zhao DM,et al.IL1RAP as a surface marker for leukemia stem cells is related to clinical phase of chronic myeloid leukemia patients[J].Int J Clin Exp Med,2014,7(12):4787-4798.
[26]Warren CFA,Wong-Brown MW,Bowden NA.BCL-2 family isoforms in apoptosis and cancer[J].Cell Death Dis,2019,10(3):177.
[27]Ferlin M,Noraz N,Hertogh C,et al.Insulin-like growth factor induces the survival and proliferation of myeloma cells through an interleukin-6-independent transduction pathway[J].Br J Haematol,2000,111(2):626-634.
[28]Di Pietro A,Conseil G,Pérez-Victoria JM,et al.Modulation by flavonoids of cell multidrug resistance mediated by P-glycoprotein and related ABC transporters[J].Cell Mol Life Sci,2002,59(2):307-322.
[29]Chiu HY,Sun KH,Chen SY,et al.Autocrine CCL2 promotes cell migration and invasion via PKC activation and tyrosine phosphorylation of paxillin in bladder cancer cells[J].Cytokine,2012,59(2):423-432.

