人源MDN1蛋白质的电镜结构研究
许云涛,李明月,雷鸣
上海交通大学医学院附属第九人民医院上海精准医学研究院,上海200125
核糖体是人体内负责蛋白质合成的重要细胞器,影响着机体生长和繁殖等一系列活动。核糖体的组装和成熟是一个高度复杂的过程,受到很多组装因子的精密调控[1-3],该过程的异常会导致心血管、神经、骨骼、造血系统等方面的疾病,如骨髓增生异常综合征(Shwachman-Diamond syndrome,SDS)、先 天 性 角 化 不 良(dyskeratosis congenita,DC)等[4-6]。在众多参与调控核糖体组装的蛋白质因子中,AAA-ATPase(ATPase associated with various activities)[7]家族是受到广泛关注的一类蛋白质,该家族成员在不同阶段参与释放或者回收不同的pre-60S核糖体组装因子,推动核糖体的成熟过程。AAA-ATPase家族成员具有2个基本的特征:其一是氨基酸序列中含有1个或多个保守的AAA结构域,AAA结构域具有ATP水解酶活性,通过水解ATP获得能量来发挥其生理功能[8];其二是大多数AAA-ATPase能够形成寡聚体,互相结合,协同作用,最常见的是形成doughnut-shaped的六聚体环状结构,这是其发挥生理功能的结构基础[9-11]。在酿酒酵母(Saccharomyces cerevisiae)中,有3种AAA-ATPase参与pre-60S核糖体成熟过程,分别为Ribosome biogenesis ATPase Rix7(Rix7)、Developmentally-regulated GTPbinding protein 1(Drg1)和Rea1[1]。Rix7是第1个与pre-60S核糖体相互作用的AAA-ATPase,主要与组装因子NOP seven-associated protein 1(Nsa1)释 放 有 关[12-13];Drg1主要在胞质中发挥作用,促进组装因子回收[14-15];而Rea1促进Pescadillo homolog(NOP7)pre-60S complex和Pre-rRNA-processing protein(Rix1)pre-60S complex中的组装因子释放,是pre-60S核糖体在核仁以及核质成熟中的关键因子[16]。
在上述3种AAA-ATPase中,Rea1作为在核内调控pre-60S核糖体组装因子释放的重要蛋白质,其敲低、敲除可致酵母死亡,在其他真核生物中对发育也有非常大的 影 响[17-21]。实 验 表 明,Rea1的metal ion-dependent adhesion site(MIDAS)结构域与pre-60S核糖体组装因子Ribosome biogenesis protein Ytm1-Erb1-Nop7(Ytm1-Erb1-Nop7)的MIDAS-interacting domain(MIDO)结构域相互作用,Rea1将Ytm1从pre-60S核糖体上释放出来[16,22];此外还发现Rea1在Rix1 pre-60S核糖体中丰度很高,并且通过其ATPase环状结构域与Rix1 pre-60S核糖体相互作用[18,22-23]。最 近发表的酵母 源(Schizosaccharomyces pombe)的结构信息显示,该蛋白质由6个AAA结构域、连接肽(linker)、天冬氨酸/谷氨酸(D/E)以及MIDAS结 构 域组成[24-25];通过AMPPNP和ATP-ribozinoindole1(ATP-Rbin1)2种不同状态的对比发现,MIDAS结构域可能通过插入AAA环与pre-60S核糖体的组分相互作用,从而介导多种组分从pre-60S核糖体释放[26]。
Rea1在人源的同源蛋白质为midasin AAA-ATPase 1(MDN1),是体内相对分子质量最大的蛋白质之一,全长包含5 596个氨基酸残基,相对分子质量为632 000。MDN1在pre-60S核糖体成熟中发挥着非常重要的作用,但是其相关结构生物学研究进展缓慢,这很大程度上限制了我们对pre-60S核糖体在核内成熟以及装配的理解。目前对MDN1结构和功能的了解,较多是基于对酵母Real的研究结论,因此,解析人源MDN1的蛋白质结构,对于阐明包括人类在内的哺乳动物MDN1调控pre-60S核糖体成熟过程的分子机制有着非常重要的意义。本研究通过哺乳细胞表达和蛋白质纯化体系,获得全长人源MDN1蛋白质样品,借助120 kV透射电子显微镜和单颗粒(singleparticle)重构技术,获得了MDN1蛋白质的负染三维结构模型,为后续解析高分辨MDN1结构奠定了坚实的基础。
1 材料与方法
1.1 实验材料
1.1.1主要试剂和仪器三羟基甲基氨基甲烷-盐酸(Tris-HCl)、氯化钠(NaCl)、氯化镁(MgCl2)、甘油(Glycerol)、二 硫 苏 糖 醇(DTT)、20×磷 酸 缓 冲 液(PBS)、吐温-20(Tween-20)均购自上海生工生物工程公司,cocktail购自瑞士Roche公司,限制性核酸内切酶、T4 DNA连接酶均购自美国Thermo Fisher公司,牛血清白蛋 白、dulbecco′s modified eagle medium(DMEM)、ANTI-FLAG® M2 Agarose Affinity Gel均购自Sigma(上海)公司,悬浮细胞培养基购自CELL-WISE公司;高速离心机、超速离心机购自美国Beckman Coulter公司,冷冻离心机购自德国Eppendorf公司,电泳仪与电泳槽购自Bio-Rad(中国)公司,细胞流式分选仪购自美国BD公司,细胞计数仪、高分辨轨道阱质谱仪均购自美国Thermo Fisher公司,激光扫描共聚焦显微镜购自德国ZEISS公司,碳膜铜网购自北京中镜科仪技术有限公司,透射电子显微镜购自美国FEI公司。
1.1.2载体、菌株和细胞pX330-mCherry载体、pUC57载体、E.coli DH5α菌种、Expi293F细胞均由本实验室保存。
1.2 实验方法
1.2.1引物合成和DNA序列测定均由上海铂尚生物技术有限公司完成。
1.2.2质粒的构建针对MDN1基因的5′端设计包含sgRNA靶位点(cac cgc aag aag tgc tcc atg acc c)的引物,体外退火后连接入pX330-mCherry载体[27],作为CRISPR/Cas9系统[28]gRNA表达质粒。同时构建一段含有MDN1基因起始密码子上游1 kb基因组序列,3×FLAG标签(tag)(DYKDHDGDYKDHDIDYKDDDDKGGSG GS)编码序列,以及下游1 kb基因组序列的供体序列,连接在pUC57载体,作为CRISPR/Cas9系统修复供体质粒。
1.2.3 FLAG-MDN1细胞株构建将CRISPR/Cas9系统的sgRNA表达质粒和修复供体质粒同时转染Expi293F细胞,采用DMEM(10% FBS)培养,48 h后进行流式分选,将mCherry阳性细胞单克隆分选至96孔板。对存活细胞进行传代培养。存活细胞培养至一定数量后,抽提基因组进行聚合酶链式反应(polymerase chain reaction,PCR)验 证(MDN1-FLAG-for,CTC GGG GTG AGG GCC CTG GGT CGC CAC CAT GGA CTA CAA GGA CCA CGA CGG;MDN1-doner-rev,ACT CAC CTG TCC CGT GGC TCA CG);全细胞裂解液用FLAG抗体进行蛋白质印迹法(Western blotting)验证。阳性细胞扩大培养至悬浮状态,悬浮细胞在无血清悬浮培养基,37℃、5% CO2、140 rpm条件下培养。在细胞密度约为3×106/mL的条件下收取,后进行蛋白质纯化。
1.2.4免疫荧光(immunofluorescence,IF)鉴定提前24 h将细胞培养在玻片上。将培养基吸掉,用4%多聚甲醛室温固定20 min,PBST(PBS+0.5%聚乙二醇辛基苯基醚,TritonX-100)处理15 min,封闭液(5% BSA+PBS+0.1% TritonX-100)封闭30 min,加FLAG一抗4℃孵育过夜。用PBST洗3次,加荧光二抗,室温孵育40 min。之后用PBST洗3次,加DAPI室温染色5 min,封片。之后在共聚焦显微镜上观察。
1.2.5 MDN1蛋白质纯化表达目的蛋白质的Expi293F细胞用1 000×g离心20 min,弃上清,用裂解缓冲液(50 mmol/L pH 7.5 Tris-HCl、400 mmol/L NaCl、1 mmol/L MgCl2、10%Glycerol、1 mmol/L DTT、cocktail)重悬,采用液氮冻存法冻存细胞,液氮研磨法裂解细胞。细胞裂解后经39 190×g离心50 min,取上清与ANTI-FLAG®M2 Agarose Affinity Gel在4℃低速旋转混合4 h,Gel用洗涤缓冲液(50 mmol/L pH 7.5 Tris-HCl、400 mmol/L NaCl、1 mmol/L MgCl2、10% Glycerol、1mmol/L DTT)洗去杂蛋白质,再用含200 ng/μL FLAG Peptide的洗脱缓冲液(50 mmol/L pH 7.5 Tris-HCl、120 mmol/L NaCl、1 mmol/L MgCl2、1 mmol/L DTT)洗脱目的蛋白质。将蛋白质样品用100 kD浓缩管浓缩至200μL,通过10%~30%甘油密度梯度离心法,用SW60转子,184 500×g,4℃,14 h分离纯化目的蛋白质。结束后每200μL收集,经十二烷基磺酸钠-聚丙烯酰胺凝胶电泳(SDS-polyacrylamide gel electrophoresis,SDS-PAGE)分析鉴定目的蛋白质。
1.2.6蛋白质组分的质谱鉴定由于许多样品中加有去污剂,需要事先沉淀洗涤处理之后才能上机。样品的处理方法步骤如下:首先沉淀样品去除样品中去污剂如TritonX-100,乙基苯基聚乙二醇(Nonidet P 40,NP40),十二烷基-beta-D-麦芽糖苷(N-Dodecyl-β-D-maltoside,DDM)等。之后重新溶解样品,还原烷基化,用胰蛋白酶,37℃酶解过夜。最后对酶解溶液进行脱盐处理,通过液相色谱-串联质谱法(liquid chromatography-tandem mass spectrometry,LC-MS/MS)进行分析;质谱原始数据提交到Proteome Discoverer 2.3软件中,使用UniProt数据库中的人(Homo sapiens)蛋白质数据库进行检索。
1.2.7负染样品制备、数据收集及模型构建因甲酸双氧铀(uranyl formate,UF)溶液渗透能力较强并且颗粒非常细腻,故选择其作为样品的负染色液。负染电镜实验步骤如下:对碳膜铜网进行亲水化处理后,取3μL蛋白质溶液滴于碳膜铜网,吸附1 min;用滤纸从碳膜铜网边缘轻轻吸去多余的溶液,滴加10μL UF负染色液(ρ=0.75%),洗2次,染色1 min;用滤纸吸去多余液体,室温晾干;晾干后将样品置于120 kV透射电镜中观察,放大倍数设置为57 000倍,欠焦值设置为-1~-2μm;选择样品颗粒分散性和均一性较好的区域进行数据收集。随后利用数据处理软件EMAN2[29]和RELION[30]对所收集的图像进行处理并产生一个三维模型。
1.2.8 MDN1蛋 白 质 结 构 预 测I-TASSER(Iterative Threading ASSEmbly Refinement)是由密歇根大学开发的在线蛋白质结构与功能预测的工具[31],预测的原理是不相似的氨基酸序列也可以对应着相似的蛋白质结构。它能从PDB(Protein Data Bank)数据库里识别结构模板,通过基于迭代模板的片段组装模拟来构建完整的结构模型。接着通过蛋白质功能数据库BioLiP对3D模型重新线程化,从而预测靶标的功能。由于MDN1蛋白质较大,只对其6个AAA结构域预测,获得相应的蛋白质三级结构预测数据。
2 结果
2.1 构建内源表达FLAG-MDN1的细胞株
人源MDN1全长包含5 596个氨基酸残基,分子量较大,进行克隆和外源表达的难度非常高,因此我们采用对Expi293F细胞内源敲入标签的方法进行内源表达。在MDN1蛋白质的N端敲入FLAG标签,用于亲和层析(图1A)。通过CRISPR/Cas9对Expi293F细胞MDN1基因组N端进行定点剪切,再通过带有FLAG标签的修复供体进行修复,然后利用流式细胞术分选mCherry阳性的单克隆Expi293F细胞进行培养。获得的单克隆细胞株抽提基因组DNA进行PCR验证,部分的细胞株样品能够获得预期的特异性PCR产物,说明相应的细胞株已经敲入目的FLAG标签(图1B)。进一步,利用FLAG抗体对全细胞裂解液进行Western blotting分析,结果显示阳性克隆细胞株能够检测到目的条带(图1C),表明带有N端FLAG标签的MDN1蛋白质可以正确表达。另外我们也采用IF技术,检测FLAG-MDN1的表达和定位(图1D)。上述3种不同的检测方法,都证实FLAG纯化标签在MDN1蛋白质N端成功敲入,获得正常表达FLAG-MDN1融合蛋白质的Expi293F细胞株。
2.2 MDN1蛋白质的纯化
对标签敲入成功的阳性细胞株进行扩大悬浮培养,收取6 L细胞(约1.8×1010个细胞)用裂解缓冲液重悬后,采用液氮研磨法破碎细胞。细胞裂解后经高速离心取上清,利用ANTI-FLAG亲和层析方法获得目的蛋白质。纯化富集的蛋白质样品采用Western blotting和银染的方法进行检测(图1E、F),证实得到的样品主要包含目的蛋白质FLAG-MDN1。将纯化样品采用体积分数为10%~30%的甘油通过密度梯度离心法进行分离纯化,进一步提高样品的纯度和均一性;密度梯度离心获得的样品按200μL/管进行组分收集,通过SDS-PAGE和银染鉴定不同的收集组分,选取纯度和均一性较好的样品用于后续的实验(图1G,孔道9~11)。
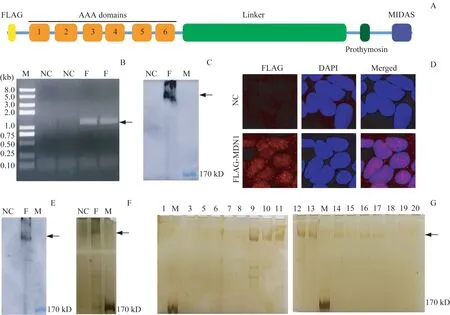
图1人源内源FLAG-MDN1蛋白质纯化Fig 1 Purification of the human endogenous FLAG-tagged MDN1 protein
2.3 MDN1蛋白质负染电镜数据收集和三维模型的构建
在获得高纯度且均一性较好的MDN1蛋白质样品后,采用负染色电镜技术和单颗粒重构技术分析目的蛋白质的三维模型。对碳膜铜网进行亲水化后,将样品吸附到碳膜铜网,经重金属甲酸双氧铀(UF)染色,通过120 kV透射电镜观察,结果表明样品在负染条件下蛋白质浓度合适,颗粒分散性和均一性较好(图2A)。大部分颗粒的形状为长条形,长度约29 nm,和预期分子量大小基本相符(图2A)。部分颗粒的形状偏向圆形,可能是MDN1蛋白质6个AAA结构域的投影。我们在57 000放大倍数条件下共收集负染照片103张,运用EMAN2软件手动挑选颗粒,共挑出16 103个颗粒,之后使用RELION软件进行二维分类平 均(2D classification)(图2B)和 三 维 分 类(3D classification)计算。结果表明MDN1蛋白质结构特征明显(图2C),用于三维重构的颗粒角度分布均匀(图2D)。
2.4 MDN1蛋白质初步结构分析
负染数据进一步经过三维优化处理(3D auto-Refine),最终获得分辨率约为45Å的人源MDN1蛋白质负染三维结构模型(图3A)。整个蛋白质大小为140Å×170Å×340Å(图3B),其整体轮廓和已报道的酵母源的MDN1蛋白质较为相似[24]。已有的研究预测MDN1蛋白质含有6个AAA结构域(图1A)[10],借助I-TASSER软件对该结构域进行模拟,结果如图所示(图3C);进一步对模拟结果通过UCSF Chimera[32]自动匹配,大致确定了AAA结构域在MDN1蛋白质负染结构模型中的位置(图3D)。将我们的负染三维模型和已报道的酵母源MDN1的蛋白质结构进行比较,发现二者的AAA结构域在整体结构中的定位是类似的(图3D)[24]。这进一步表明MDN1蛋白质结构在酵母和人之间是较为保守的。
3 讨论
由于核糖体在细胞中的重要性,其装配成熟过程一直是相关研究领域的一个热点问题。已有的研究表明,AAA-ATPase在pre-60S核糖体成熟和出核过程中发挥着重要作用[10]。尽管MDN1功能有一些研究报道,也有研究者在2018年发表了酵母源MDN1的高分辨率结构[24-25],但是人源MDN1的结构研究进展缓慢,目前尚无任何高分辨率结构的报道,这限制了研究人员对MDN1在哺乳动物pre-60S核糖体成熟过程中发挥作用的分子机制的理解。虽然氨基酸序列分析表明酵母源与人源MDN1蛋白质在某些结构域有一定的保守性,但有研究认为人源MDN1在pre-60S核糖体成熟过程中被招募的机制可能有所不同,与其相互作用的核糖体组装因子也有所差异,例如在人源细胞中MDN1能够被SUMO化的proline-,glutamic acid-and leucine-rich protein 1(PELP1)招募至pre-60S核糖体[20]。因此,解析人源MDN1蛋白质结构对于阐明哺乳动物MDN1的具体功能和相关机制有重要的意义。
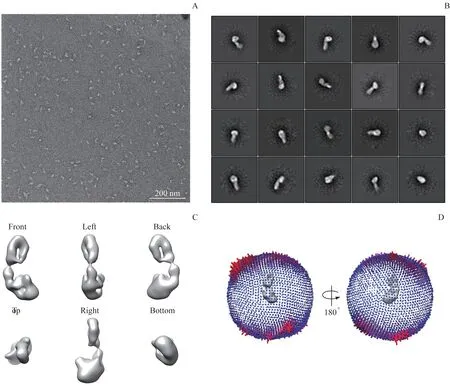
图2负染电镜分析MDN1蛋白质Fig 2 Negative-staining electron microscopy analysis of MDN1
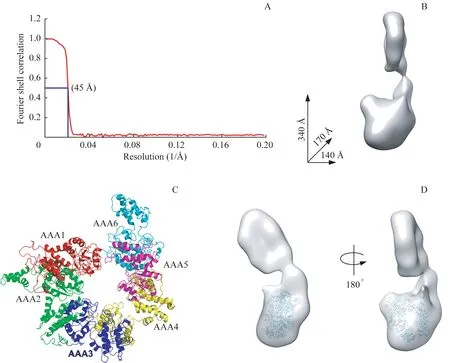
图3 MDN1蛋白质三维模型和AAA结构域的定位Fig 3 Three-dimensional reconstruction of MDN1 and localization of AAA domains
MDN1蛋白质分子量非常巨大,外源表达有一定的难度,并且整体结构也不适合采用晶体结构生物学方法进行解析,所以我们在Expi293F细胞的MDN1蛋白质的N端敲入亲和纯化标签FLAG,通过亲和层析获得少量的纯化样品进行电镜结构解析。通过负染色技术和单颗粒重构技术(single-particle),获得了MDN1蛋白质低分辨率三维结构。从整体轮廓看,MDN1的结构在人源细胞和酵母内是较为保守的。另外,我们也利用结构预测和分子对接的方法,初步确定人源MDN1的AAA结构域在整体结构中的定位。
MDN1属于AAA-ATPase家族成员,该家族蛋白质包含至少一个结构上非常保守的AAA结构域,再组装为功能活跃的环状结构。不同于其他寡聚化的AAA-ATPase复合物,MDN1蛋白质氨基酸序列包含有6个AAA结构域,能够通过自身的肽链折叠,形成相应的环状结构。事实上,我们的负染结构模型也表明人源MDN1是以单体的形式存在,并且有类似的AAA环状结构,这和酵母源的MDN1是一致的。为了得到人源MDN1高分辨率的结构,下一步我们将着重提高MDN1的纯化产量,尝试冷冻电镜分析。由于采用Expi293F内源表达的方法,获得的MDN1纯化样品相对较少,6 L细胞得到的目的蛋白量为100~120μg,我们将通过扩大细胞培养量,解决这个蛋白质产量限制问题,以期获得MDN1蛋白质的冷冻电镜高分辨结构,阐明MDN1在pre-60S核糖体成熟中发挥作用的结构基础。
参·考·文·献
[1]Kressler D,Hurt E,Bergler H,et al.The power of AAA-ATPases on the road of pre-60S ribosome maturation:molecular machines that strip preribosomal particles[J].Biochim Biophys Acta,2012,1823(1):92-100.
[2]Woolford JL,Baserga SJ.Ribosome biogenesis in the yeast Saccharomyces cerevisiae[J].Genetics,2013,195(3):643-681.
[3]Kater L,Thoms M,Barrio-Garcia C,et al.Visualizing the assembly pathway of nucleolar pre-60S ribosomes[J].Cell,2017,171(7):1599-1610.e14.
[4]Venturi G,Montanaro L.How altered ribosome production can cause or contribute to human disease:the spectrum of ribosomopathies[J].Cells,2020,9(10):2300
[5]Narla A,Ebert BL.Ribosomopathies:human disorders of ribosome dysfunction[J].Blood,2010,115(16):3196-3205.
[6]Pelava A,Schneider C,Watkins NJ.The importance of ribosome production,and the 5S RNP-MDM2 pathway,in health and disease[J].Biochem Soc Trans,2016,44(4):1086-1090.
[7]Konikkat S,Woolford JL.Principles of 60S ribosomal subunit assembly emerging from recent studies in yeast[J].Biochem J,2017,474(2):195-214.
[8]Patel S,Latterich M.The AAA team:related ATPases with diverse functions[J].Trends Cell Biol,1998,8(2):65-71.
[9]Hanson PI,Whiteheart SW.AAA+proteins:have engine,will work[J].Nat Rev Mol Cell Biol,2005,6(7):519-529.
[10]Prattes M,Lo YH,Bergler H,Stanley RE.Shaping the nascent ribosome:AAA-ATPases in eukaryotic ribosome biogenesis[J].Biomolecules,2019,9(11):715.
[11]Wendler P,Ciniawsky S,Kock M,Kube S.Structure and function of the AAA+nucleotide binding pocket[J].Biochim Biophys Acta,2012,1823(1):2-14.
[12]Gadal O,Strauss D,Braspenning J,et al.A nuclear AAA-type ATPase(Rix7p)is required for biogenesis and nuclear export of 60S ribosomal subunits[J].EMBO J,2001,20(14):3695-3704.
[13]Lo YH,Sobhany M,Hsu AL,et al.Cryo-EM structure of the essential ribosome assembly AAA-ATPase Rix7[J].Nat Commun,2019,10(1):513.
[14]Pertschy B,Saveanu C,Zisser G,et al.Cytoplasmic recycling of 60S preribosomal factors depends on the AAA protein Drg1[J].Mol Cell Biol,2007,27(19):6581-6592.
[15]Pertschy B,Zisser G,Schein H,et al.Diazaborine treatment of yeast cells inhibits maturation of the 60S ribosomal subunit[J].Mol Cell Biol,2004,24(14):6476-6487.
[16]Miles TD,Jakovljevic J,Horsey EW,et al.Ytm1,Nop7,and Erb1 form a complex necessary for maturation of yeast 66S preribosomes[J].Mol Cell Biol,2005,25(23):10419-10432.
[17]Galani K,Nissan TA,Petfalski E,et al.Rea1,a dynein-related nuclear AAAATPase,is involved in late rRNA processing and nuclear export of 60S subunits[J].J Biol Chem,2004,279(53):55411-55418.
[18]Ulbrich C,Diepholz M,Bassler J,et al.Mechanochemical removal of ribosome biogenesis factors from nascent 60S ribosomal subunits[J].Cell,2009,138(5):911-922.
[19]Li PC,Ma JJ,Zhou XM,et al.Arabidopsis MDN1 is involved in the establishment of a normal seed proteome and seed germination[J].Front Plant Sci,2019,10:1118.
[20]Finkbeiner E,Haindl M,Raman N,et al.SUMO routes ribosome maturation[J].Nucleus,2011,2(6):527-532.
[21]Li PC,Li K,Wang J,et al.The AAA-ATPase MIDASIN 1 functions in ribosome biogenesis and is essential for embryo and root development[J].Plant Physiol,2019,180(1):289-304.
[22]Bassler J,Kallas M,Pertschy B,et al.The AAA-ATPase Rea1 drives removal of biogenesis factors during multiple stages of 60S ribosome assembly[J].Mol Cell,2010,38(5):712-721.
[23]Nissan TA,Galani K,Maco B,et al.A pre-ribosome with a tadpole-like structure functions in ATP-dependent maturation of 60S subunits[J].Mol Cell,2004,15(2):295-301.
[24]Chen Z,Suzuki H,Kobayashi Y,et al.Structural insights into Mdn1,an essential AAA protein required for ribosome biogenesis[J].Cell,2018,175(3):822-834.e18.
[25]Sosnowski P,Urnavicius L,Boland A,et al.The CryoEM structure of the Saccharomyces cerevisiae ribosome maturation factor Rea1[J].Elife.2018,7:e39163.
[26]Kawashima SA,Chen Z,Aoi Y,et al.Potent,reversible,and specific chemical inhibitors of eukaryotic ribosome biogenesis[J].Cell,2016,167(2):512-524.
[27]Wu Y,Liang D,Wang Y,et al.Correction of a genetic disease in mouse via use of CRISPR-Cas9[J].Cell Stem Cell,2013,13(6):659-662.
[28]Ran FA,Hsu PD,Wright J,et al.Genome engineering using the CRISPRCas9 system[J].Nat Protoc,2013,8(11):2281-2308.
[29]Tang G,Peng L,Baldwin PR,et al.EMAN2:an extensible image processing suite for electron microscopy[J].J Struct Biol,2007,157(1):38-46.
[30]Scheres SH.RELION:implementation of a Bayesian approach to cryo-EM structure determination[J].J Struct Biol,2012,180(3):519-530.
[31]Yang JY,Zhang Y.I-TASSER server:new development for protein structure and function predictions[J].Nucleic Acids Res,2015,43(W1):W174-W181.
[32]Pettersen EF,Goddard TD,Huang CC,et al.UCSF Chimera:a visualization system for exploratory research and analysis[J].J Comput Chem,2004,25(13):1605-1612.

