酿酒酵母适应性实验室进化工具的最新进展
李祎,林振泉,刘子鹤
(1 北京化工大学生命科学与技术学院,北京 100029;2 北京化工大学北京软物质科学与工程高精尖创新中心,北京100029)
自然进化是连续且不断筛选的,具体过程包括遗传多样性的产生、选择压力的胁迫以及适应性后代的繁殖等[1]。长期以来,人类一直试图加快和控制这一过程,以快速获得具有优良表型的生物体或特定生物分子[2]。适应性实验室进化(adaptive laboratory evolution,ALE)以特定或逐渐增加的胁迫来筛选性能优良的突变体,即人为复制了自然进化的过程[3],并获得在特定条件下鲁棒性较好的突变体[4]。以特定的亲本菌株出发,在基因组水平上对遗传物质进行突变或者重组,以创建群体多样性文库;将群体文库在特定的培养条件中进行培养,筛选目标性状的菌株[5]。根据进化时间的长短可以将ALE 技术分为长期模式和短期模式[6]。长期模式通常经历数百次的传代,而短期模式往往仅需几十次甚至数十小时的传代即可完成[7]。ALE 过程可能会出现权衡(tradeoffs)现象,即在选择压力下出现相关变化沿相反方向发生的事件,而此时可通过适当减弱选择压力等方法进行规避[8]。ALE 策略已经广泛应用于构建耐高渗、耐酸性及耐热性等菌株[9-11]。
多样性文库的质量直接影响实验室适应性进化的效率,传统的实验室进化主要是通过物理/化学 诱 变 方 法[12-18]、转 座 子 突 变[19-21]、基 因 改组[22-25]、易错PCR[26-28]等方式获得多样性突变文库。近年来,随着系统生物学和合成生物学技术的发展,研究人员开发出多种基于全基因组水平的多位点多样性文库的构建策略。为了适应当前高通量甚至超高通量的研究趋势[29],同时快速地复制自然进化的全部过程,自动化连续进化策略成为进化工程重要的发展方向。
酿酒酵母具有遗传背景清晰、基因操作手段丰富、可利用碳源广泛等优势[30-32],已广泛应用于生产生物基化学品和燃料[33],如类异戊二烯、莽草酸、纤维素乙醇等[34-38]。本综述将着重介绍近年来在酿酒酵母中基于全基因组水平的多位点快速进化及体内连续进化的研究进展,通过对基因组的编辑,以产生具有遗传多样性的细胞群体,能够在短期内获得目标性状。
1 基于基因组水平的多位点进化策略
由于细胞代谢过程受到高度的调控及生物系统的复杂性,许多遗传表型不仅需要对目标途径进行有效的调控,还需要在基因组水平进行多位点的编辑调控[39]。因此,基于基因组规模的多位点进化策略能够有效提高目标性状,减少进化所需的时间和成本,是进化工程重要的研究热点之一。
本节将介绍3类在酿酒酵母中基于基因组的多位点快速进化工具(表1):重组酶介导的进化工程、基于寡核苷酸的多位点进化工具和CRISPR(clustered regularly interspaced short palindromic repeats,规律间隔成簇短回文重复序列)介导的多位点编辑。
1.1 基于重组酶介导的进化工程
随着基因组测序和基因合成技术的发展,基因组规模的分析为微生物进化的深度研究提供了有效工具,实现可在碱基对水平和基因组规模上对DNA 序列进行“读”和“写”[46]。合成酵母基因组计划(Sc2.0)是世界上首个真核基因组合成项目,旨在合成一个高适应性及通用性的人工合成酵母基因组[47]。Sc2.0 人工合成基因组中添加了loxPsym 位点[48],Cre 重组酶能够特异性识别并介导不同loxPsym 位点(对称的loxP序列)之间进行重组,建立合成型酿酒酵母基因组重排(synthetic chromosome rearrangement and modification by loxPmediated evolution,SCRaMbLE)[49]。SCRaMbLE系统(图1)通过非必需基因终止密码子后3 bp 处添加loxPsym 序列,实现全基因组范围内loxPsym位点间的DNA 片段缺失、倒置、重复和异位,以产生大量不同基因型-表型的酵母菌株,快速获得大量的酵母多样性文库[33,40]。
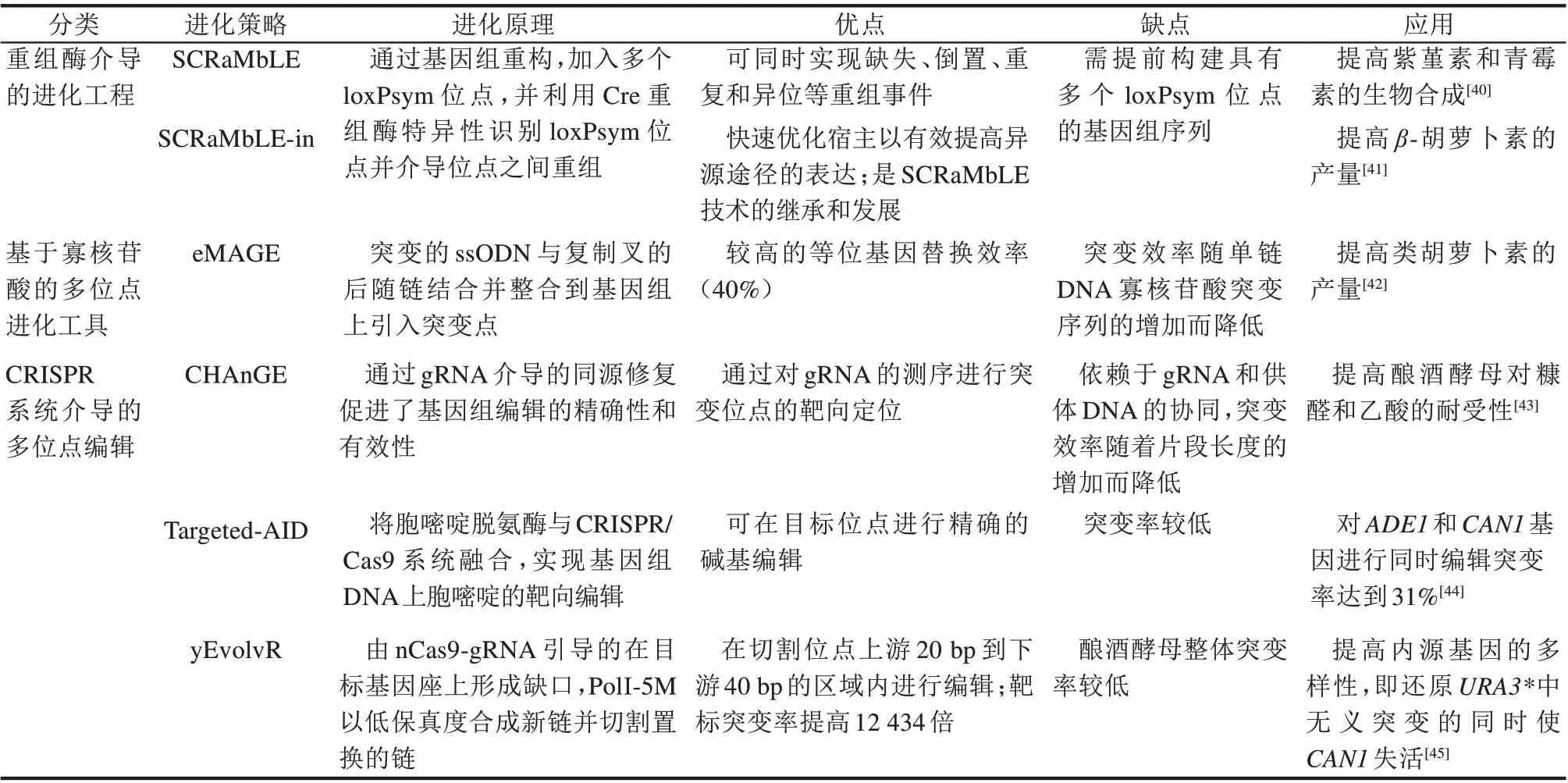
表1 酿酒酵母中基因组的多位点快速进化工具Tab.1 Multi-site rapid evolution tools for genome in S.cerevisiae
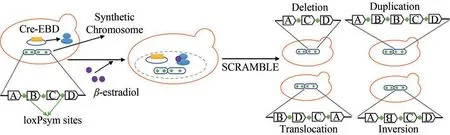
图1 SCRaMbLE技术Fig.1 Schematic for SCRaMbLE technology
1.1.1 精准控制与筛选SCRaMbLE的重排策略
通过β-雌二醇诱导Cre 重组酶表达激活SCRaMbLE 系统,Cre 重组酶的泄露表达会影响人工合成染色体的稳定性甚至致死,因此需要对Cre重组酶的表达进行严谨的调控[50]。Jia 等[51]构建了逻辑“与”门开关控制Cre的表达,通过半乳糖诱导Cre-EBD 表达,再将Cre-EBD 结合雌二醇以形成有活性的Cre重组酶,可在转录和细胞定位水平上调节单、双倍体酵母中的SCRaMbLE。除此之外,Hochrein等[52]将Cre的N末端和C末端分裂,再分别与光敏色素B 和作用因子PIF3 相融合构建L-SCRaMbLE 系统,未激活时两端处于分离状态,经红光照射后光敏色素B 和作用因子PIF3 相互作用以重构Cre 重组酶使其恢复功能。L-SCRaMbLE策略可有效控制Cre重组酶泄露表达,并通过诱导时间、光剂量和感受器浓度控制Cre 重组酶的活性,实现精准控制基因组重排。
SCRaMbLE 系统利用染色体重排产生了多种基因型的菌株,高效的筛选方法是获得目标菌株的必要途径。Luo 等[53]通过在loxP 位点的两翼设计对向排列且不携带起始密码子的营养缺陷型标记“URA3ΔATG-LEU2ΔATG”模块,构建了高效筛选重组细胞的ReSCuES 系统(SCRaMbLEd cells using efficient selection)。由于模块的侧面是两个相对的loxP位点,其中loxP位点与loxPsym位点之间不会进行重组,只有当Cre重组酶诱导基因组重排时,模块才会发生翻转(LEU2 表达的同时会关闭URA3 的表达),导致两个标记分别处于“开”和“关”的状态,通过缺陷型培养基即可筛选出发生基因组重排的突变株。利用以上策略精准控制和筛选SCRaMbLE 菌株,大大缩短了试验时间并减少了烦琐的操作,加速全基因组规模的进化。
1.1.2 基于SCRaMbLE系统的基因组工程
Wu 等[54]开发了一个基于结构变异的体外SCRaMbLE 系统,并提出“自上而下”和“自下而上”两种策略。“自上而下”策略[图2(a)]利用纯化的DNA 重组酶在体外对人工合成环形synV 染色体的loxPsym 位点间的序列进行修饰,在Cre重组酶的诱导下,随机地删除、倒置、异位或重复loxPsym 位点相邻的转录单元(transcription unit,TU)。“自下而上”策略[图2(b)]由载体和供体片段构成,且演化出两种独立的模式:第1个模式的载体具有两个loxPsym 位点,供体片段每个都带有TU 和URA3 基因(阳性选择标记);第2 个模式的载体只含有一个loxPsym 位点,一个或多个供体片段的重组模式进入该位点可将URA3 启动子和编码序列物理分离。载体、供体库与Cre重组酶在体外混合使TU 随机插入loxPsym 位点,通过TU复制数量和方向的变化实现基因重排。
为了优化异源表达中的途径和底盘适配性等问题,Liu 等[41]开发了一种基于SCRaMbLE 的组合策略SCRaMbLE-in[图2(c)]。首先在体外使用纯化的重组酶将调控元件整合到目标途径中,使目标基因表达多样化;再通过SCRaMbLE 将组装的途径随机整合到合成酵母染色体中,诱导基因组重排获得基因表达多样化和底盘优化的菌群文库,实现了快速的基因组进化。由于染色体的拓扑结构对染色体重排起着重要作用,Wang 等[55]应用SCRaMbLE 技术对环状synⅤ染色体进行基因组重排,发生了特定染色体的非整倍性重复,与线性synⅤ相比,SCRaMbLEd的环状染色体出现大量的重复现象。因此,通过对环状染色体进行基因重排可连续地产生复杂的基因型和表型,深入探索了染色体结构对生物表型多样性的重要影响。
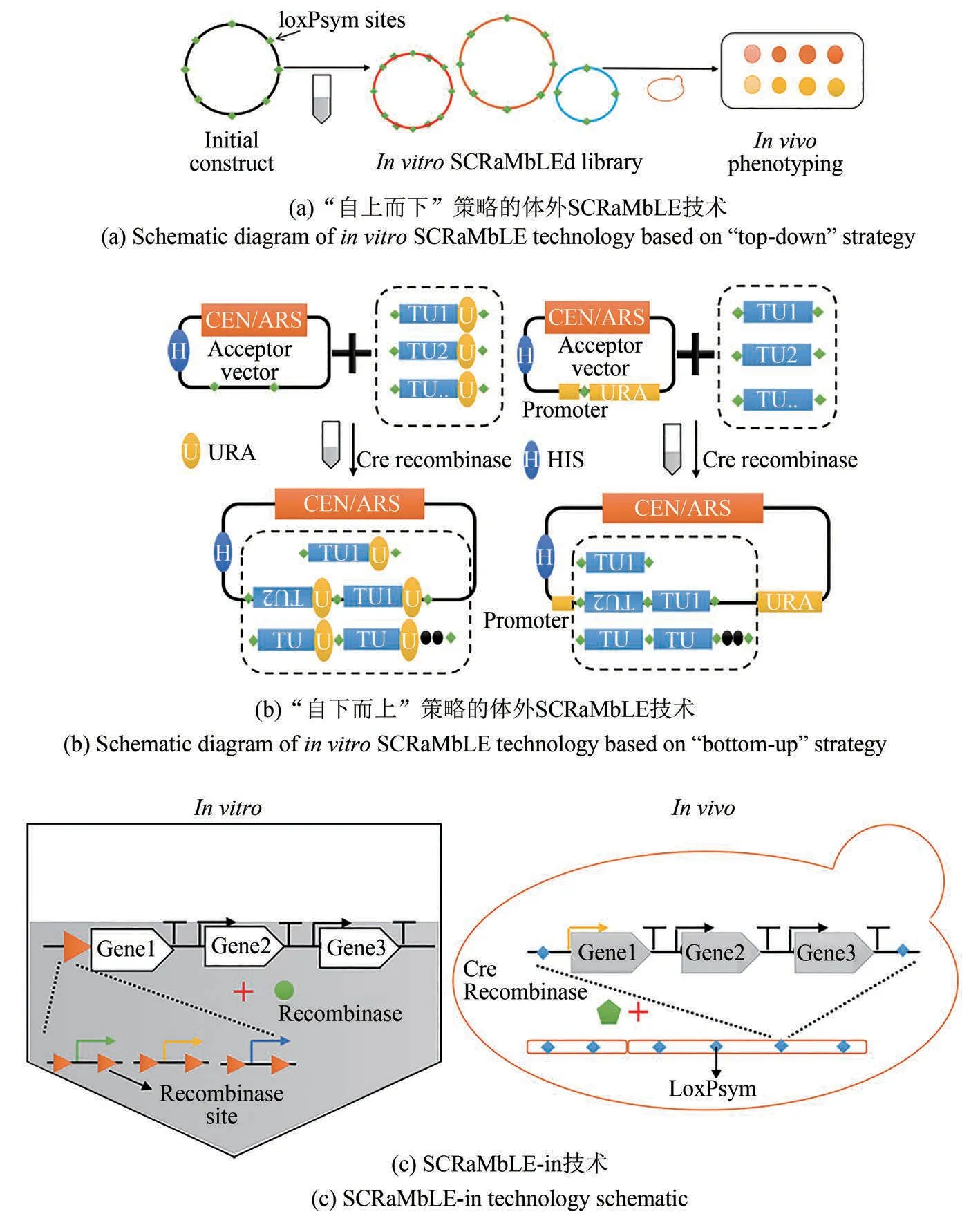
图2 体外SCRaMbLE[54]与SCRaMbLE-in技术Fig.2 Schematic for in vitro SCRaMbLE[54]and SCRaMbLE-in technology
SCRaMbLE 系统已经成功应用于提升类胡萝卜素、青霉素等产量[41,51,55-56],以及提高底盘细胞对酸、碱和高渗等耐受性[53,56-58]。由于染色体结构改变和非整数倍的研究往往基于单个基因组的静态对比,SCRaMbLE 系统提供了一个强大的平台,通过生成大量的基因组结构变异库,为深入认识染色体性状提供了有效手段[59]。
1.2 基于寡核苷酸的多位点进化工具
可以利用生物体自身同源重组介导的修复机制引入突变位点,通过引入含碱基突变的寡核苷酸建立突变文库[60]。2013 年,DiCarlo 等[61]报告了一种在酿酒酵母中以寡核苷酸介导的基因组多位点编辑技术YOGE(yeast oligo-mediated genome engineering)。通过敲除错配修复相关的基因Mlh1和Msh2、过表达DNA 重组酶Rad51 和Rad54 以及优化寡核苷酸的设计和转化量,将寡核苷酸的错配率提高到0.2%~2.0%。通过迭代循环YOGE 的方式可以逐步富集所需基因型,从而在每个循环中获得更高的频率。等位基因替换效率(allelic replacement frequencies,ARF)2%仍不足以进行多位点、规模化的基因组编辑[39]。
2017年,Barbieri等[62]利用羟基脲减慢复制叉移动速度、敲除Rad51 基因以提高ARF,在酿酒酵母DNA复制过程中建立了基于滞后链上退火合成寡核苷酸的多重基因组工程技术eMAGE(eukaryotic MAGE)。在DNA 复制起始阶段,错配的ssODN(single-stranded DNA oligodeoxynucleotides)由ssDNA退火蛋白(ssDNA annealing protein)介导,在复制叉的后随链上退火并整合到基因组上引起突变。单个碱基对的错配、插入或缺失的ARF>40%,对于30 bp 错配和100 bp 缺失的ARF 只有10%。利用多轮eMAGE 循环可提高大片段或多个寡核苷酸的ARF,在酵母中引入异源β-胡萝卜素途径,并设计了74 个精确靶向启动子、开放阅读框和终止子的ssODNs 库,经过一轮eMAGE 循环获得大量具有不同基因型-表型的菌株。此外,eMAGE可以实现自动化,为酿酒酵母多位点快速编辑提供了一种高效的编辑策略。
1.3 基于CRISPR系统介导的多位点编辑
同源定向修复(homology directed repair,HDR)和非同源末端连接(non-homologous end joining,NHEJ)是双链断裂(double strand breaks,DSB)修复的两种方式,HDR 是酿酒酵母中DSB 的主要修复方式[63]。近年来,酿酒酵母中CRISPR系统介导的基因组编辑工具得到了快速发展,其中利用CRISPR 系统介导的多位点基因编辑技术构建高通量多样性文库以进行多位点快速进化取得极大发展[64-66]。
1.3.1 基于CRISPR系统的基因组编辑
CRISPR 系统的靶向性取决于目标DNA 与gRNA 的互补配对,以及对PAM(protospacer adjacent motif)位点的特异识别,可实现基因组规模的编辑[67]。2017 年,Bao 等[43]构建了一种基于CRISPR-Cas9 和HDR 辅助修复的基因组规模工程(CRISPR-Cas9 and homology directed repair assisted genome scale engineering,CHAnGE),能够在基因组尺度上快速编辑酿酒酵母,并实现精确且可追踪的编辑。CHAnGE 技术[图3(a)]需要将gRNA 和HDR 供体组装到一个gRNA 表达盒中,该盒分别结合24 765 个特异序列,覆盖酿酒酵母中约97.8%的ORF(open reading frame,开放阅读框)以进行全基因组进化,并将此表达盒用作唯一条形码可通过高通量测序跟踪每个突变体。利用该技术获得了对糠醛和醋酸耐受性分别提高了42 倍和20 倍的突变株。通过对gRNA 的测序,鉴定出敲除SIZ1 可提高酵母的糠醛耐受性,同时敲除SIZ1 和LCB3 基因可进一步提高糠醛的耐受性。
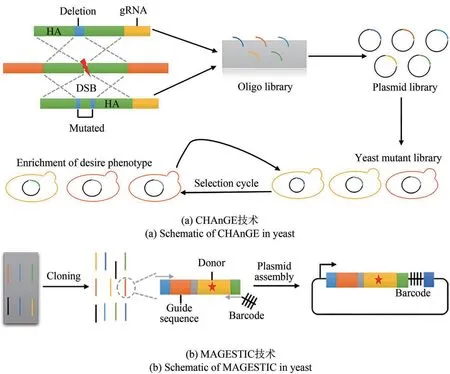
图3 CHAnGE与MAGESTIC技术Fig.3 Schematic of CHAnGE and MAGESTIC in yeast
另外,Roy 等[68]在CRISPR/Cas9 系统的基础上开发了利用短的、可追踪、可基因组整合的条形码策略MAGESTIC(multiplexed accurate genome editing with short, trackable, integrated cellular barcodes)[图3(b)],实现了基因组规模多位点精确编辑。利用寡核苷酸芯片技术合成了gRNA-同源片段的引导-供体对,质粒组装后将一个短条形码(31mer)作为标记,此标记具有基因组条形码整合功能,可防止质粒条形码丢失以实现可靠的表型分析。此外,利用LexA-Fkh1p 融合蛋白将供体DNA 募集到断裂位点,可以将基因组编辑效率提高5倍以上。
1.3.2 基于CRISPR系统的基因组规模单碱基修饰
胞嘧啶脱氨酶(activation-induced cytidine deaminase,AID)通过脱氨作用将R-loop 中单链DNA 中的胞嘧啶(C)转化为尿嘧啶(U),U-G(鸟嘌呤)错配通过DNA 复制和修复途径实现[69]。因此,AID 可对转录过程中形成的单链DNA 区域进行可诱导突变。Nishida 等[44]将来自海鳗的胞嘧啶脱氨酶PmCDA1(petromyzon marinus cytidine deaminase 1)连接到d/nCas9 蛋白C 端构建Target-AID 编辑系统[图4(a)],可实现对gRNA 非互补链中PAM 序列上游−16~−20 区域内胞嘧啶的突变。利用Target-AID 编辑器对ADE1 和CAN1 基因进行同时编辑,两种基因的独立突变频率分别为54%和51%,双表型突变的频率达到31%[44,70]。
在 此 基 础 上,2018 年,Després 等[71]通 过pDYSCKO 表达靶向目标基因(例如YFG)和负筛选标记(例如CAN1)的gRNA,利用半乳糖诱导Target-AID 载体上Cas9 不同变体的表达构建了Target-AID 双筛选系统[图4(b)]。添加刀豆氨酸进行筛选,单、双倍体的基因突变率分别提高3 倍和4.2 倍,表明双筛选策略可以富集成功编辑的 细 胞 群。2020 年,Després 等[72]构 建 基 于Target-AID 诱变和多位点基因组编辑的方法,根据酿酒酵母的必需基因设计合成大量可预测的gRNA,并将其作为突变效应的标签,对酵母必需基因进行系统的修饰,完成了1500 多个基因中的大约17 000个位点的编辑,并确定了700多个位点的突变对底盘适应性的重大影响。
此外,将大肠杆菌的高突变率DNA聚合酶PolⅠ融合到nCas9蛋白的N 端,也可实现在特定区域的碱基编辑。Halperin 等[73]在大肠杆菌中建立了一种通过CRISPR 系统突变靶基因的EvolvR 技术,由gRNA引导的nCas9在目标基因座上形成缺口,再由融合的PolⅠ以低保真度合成新链并切割置换的链。2020年,Tou等[45]使用PolⅠ-5M在酿酒酵母中建立了yEvolvR(yeast EvolvR system)系统[图4(c)],将目标位点的突变率提高了12 434 倍。yEvolvR 技术另一个优势是能够有效提高内源基因的多样性,当gRNA 双重靶向URA3*和CAN1 的条件下,可还原URA3*中的无义突变并使CAN1失活。
2 自动化连续进化技术的发展
传统的适应性实验室进化大都只针对自然进化中每个步骤的频繁干预,这往往限制了进化过程的多样性和扩展性。因此,近年来研究人员利用自动化技术与连续进化相结合,无需人工干预[74],并保持宿主细胞的稳定及样本的多样性[75]。本节将重点介绍酿酒酵母的自动化连续进化技术和连续自动化培养工具(表2)。
2.1 连续自动化进化策略
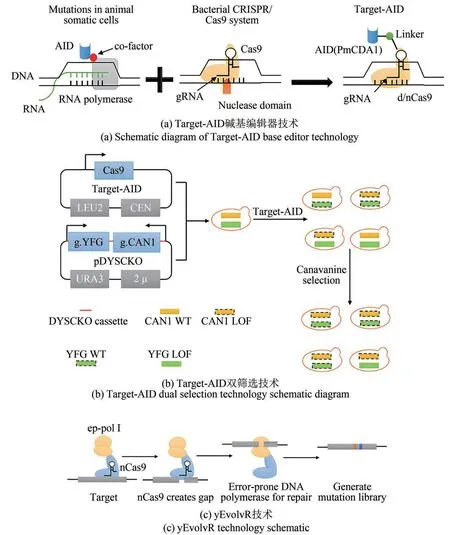
图4 Target-AID碱基编辑器、Target-AID双筛选[71]和yEvolvR技术Fig.4 Schematic for target-AID base editor,double screening selection[71]and yEvolvR technology

表2 用于酿酒酵母的自动化连续进化工具Tab.2 Automated continuous evolution tools for S.cerevisiae
在DNA 的复制过程中,DNA 聚合酶的校对和错配修复对保持生物基因组的稳定起着关键作用。研究人员在酵母中发现与基因组复制正交的质粒复制系统,通过改变正交质粒的DNA聚合酶校对和错配修复的效率能够有效提高正交质粒上DNA序列的突变效率,通过连续培养能够快速获得大量多样性群体[77,80]。正交易错复制系统(orthogonal errorprone replication system,OrthoRep)[图5(a)]是由细胞质中的线性DNA 质粒pGKL1(p1)及相应的DNA 聚合酶TP-DNAP1 组成,p1 质粒的复制受到TP-DNAP1 严格控制且不依赖于宿主DNA 聚合酶[81]。通过对TP-DNAP1进行突变,提高了TPDNAP1依赖p1质粒复制过程中的突变率,并使正交质粒上目标靶点的单碱基突变率(substitutions per base,s.p.b.)提高到4×10−8[77]。Ravikumar 等[82]将Rd3突变体与核酸外切酶结构域部分基序的突变体杂交构建了突变体TP-DNAP1-4-2,其s.p.b.提高了1000 倍(约10−5)。在酿酒酵母中利用OrthoRep 系统对二氢叶酸还原酶进行连续进化,将其针对乙胺嘧啶的耐受性提高了4 万倍。 2018 年,Arzumanyan 等[76]发 现 了pGKL2/TP-DNAP2 的 质粒/DNA 聚 合 酶 与pGKL1 质 粒/TP-DNAP1 正 交[图5(a)],研究人员将pGKL1 质粒/TP-DNAP1与pGKL2质粒/TP-DNAP2两套正交的DNA 复制系统成功导入酿酒酵母中,利用正交DNA 复制系统中不同DNA 聚合酶的突变率实现同时针对多基因进行不同突变频率的进化。利用OrthoRep 中两个正交的DNA 复制系统,能够在多种酿酒酵母(如BY4741、W303-1A、CEN.PK2-1C 和BY4743 等)进行进化[83]。由此可见,OrthoRep 可在不同的酿酒酵母中实现生物分子和细胞功能的高通量进化,为构建高效生物细胞工厂提供了新方法。
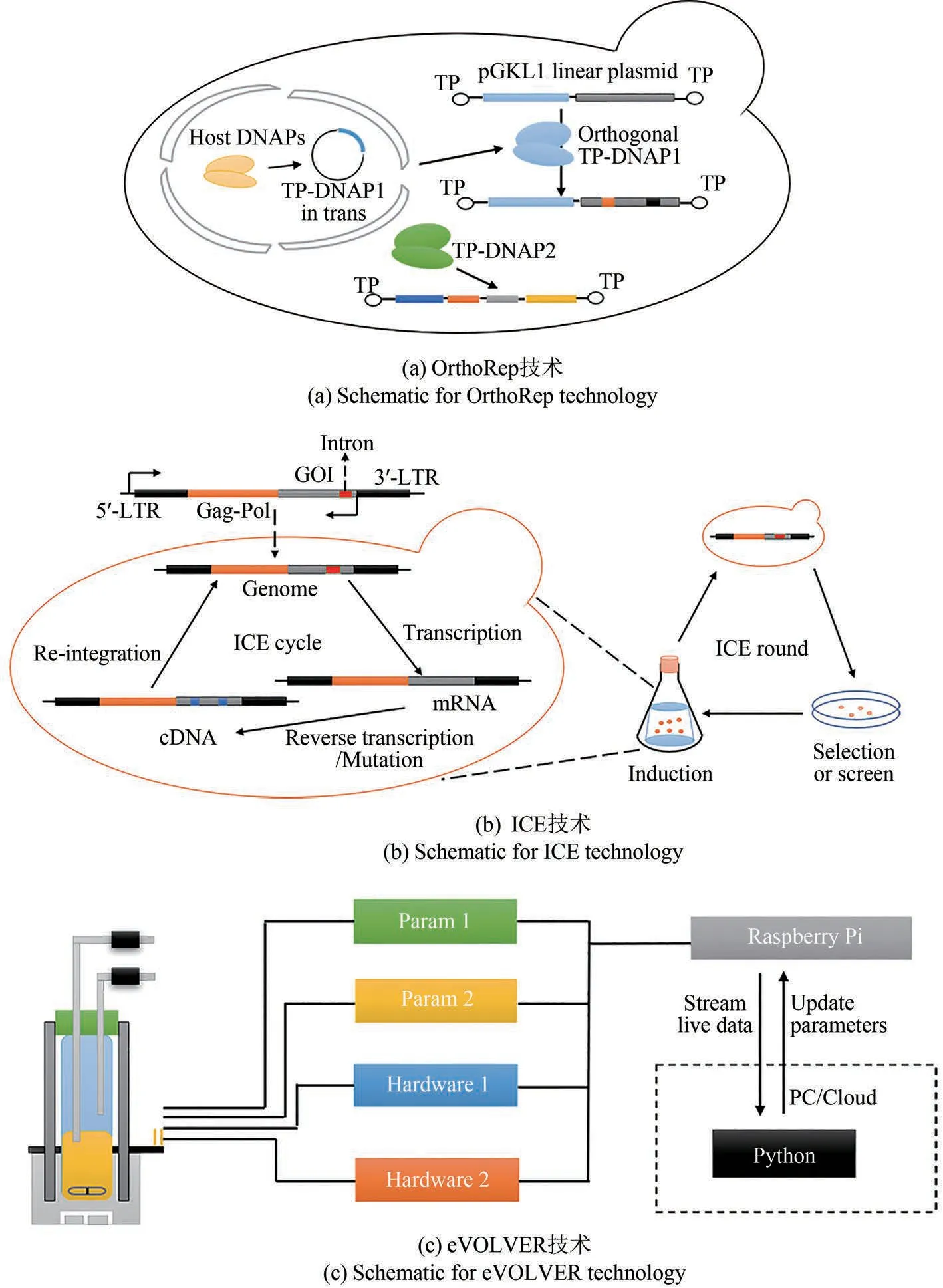
图5 OrthoRep、ICE和eVOLVER技术Fig.5 Schematic diagram of OrthoRep,ICE and eVOLVER technology
与DNA 聚合酶相比较,生物体内逆转录酶没有高效的校对活性,从而导致逆转录过程低保真复制。Crook 等[78]基于逆转录过程引起的突变开发出体内连续进化技术ICE(in vivo continuous evolution)[图5(b)]。Ty1 逆转录酶可将Ty1 识别区域的mRNA 逆转录为cDNA,再通过Ty1 整合酶整合到Ty1识别位点以进行下一轮突变,通过多轮迭代可产生大量突变的基因。使用中等强度的pTEF1 启动子控制逆转录元件的转录、敲除rrm3基因并过表达tRNAiMet可以优化连续进化系统并将目标靶点的突变率提高50 倍。利用ICE 技术在酵母中生成Spt15 文库,两次迭代后获得了在3.5%1-丁醇中耐受性提高2 倍的突变株。ICE 技术可以扩展到含有LTR(long terminal repeat)逆转录转座子的真核生物中,并随着细胞数量而扩展,但该技术依赖于逆转录的转座频率在连续的进化中定向位点的使用可能会对基因组的稳定性造成影响;依赖特定的逆转录机制限制了该技术的应用。
2.2 连续自动化培养平台
连续培养是一种定量表征工程菌株[84]以及跟踪适应性实验室进化中遗传变化的方法[85],可实现在精确控制的培养参数下进行连续进化[1]。因此,为了加速模拟自然进化的进程,连续自动化培养技术的开发在适应性实验室进化中显得尤为重要。
2018年,Wong等[86]搭建可扩展的高通量自动化细胞培养的集成平台(eVOLVER)[图5(c)],此平台利用Python 自主编程控制系统可将多个智能套管并联,通过流体控制阀门、LED+二极管进行光密度监控,比例积分微分(proportional integral derivative,PID)控制加热器用来调节温度,调整磁力控制搅拌速率,实现了实时检测光密度、温度等各种培养参数及参数的自动控制。利用eVOLVER 系统将菌体浓度维持在恒定区域内(设定光吸收的上下阈值),通过改变78 个酵母菌种群的OD 值上限和下限,可获得酵母种群的进化参数图,包括种群增长率-平均基因组复制与光密度-增长率的关系。eVOLVER系统作为自动化连续进化工具有极强的吸引力,但该系统复杂的组装和应用计算机语言设计培养程序对初学者是很大的挑战[79]。
2020 年,Zhong 等[80]将OrthoRep 系 统 和eVOLVER 系统结合起来搭建了连续进化(automated continuous evolution,ACE)平台,该平台将eVOLVER 系统的严格选择性、实时反馈和控制,与OrthoRep 系统对目标基因连续突变的特性相结合。通过ACE 平台对二氢叶酸还原酶进行连续进化,在3 mmol/L 乙胺嘧啶中只经过550 h的连续进化就达到与Ravikumar 等[82]相同的试验结果。ACE 为酿酒酵母自动化连续进化提供了前所未有的速度、深度和可扩展性,由此可见体内连续进化与体外自动化连续进化系统相结合,大大提高进化的速度。连续自动化培养平台的构建改变了适应性实验室进化的方式,通过翻译、突变、选择和复制的连续循环促进了复杂途径的进化,同时在研究适应性进化机制等方面具有广阔的前景[87]。
3 小结与展望
适应性实验室进化加速了自然进化的过程,释放出微生物在生物技术应用中的巨大潜能,并可有效改善目标表型,极大推动合成生物学细胞工厂的构建,在解决资源紧缺、生命健康、环境污染等重大难题方面提供强大的技术支撑[88]。酿酒酵母各种表型受到复杂代谢网络调控,结合系统生物学和反向代谢工程分析手段,解析酿酒酵母鲁棒性和适应性的分子基础,加深对复杂基因型-表型相互作用的理解并提高网络模型准确性,设计出符合人类需求的工程化酿酒酵母,已成为合成生物学重要的发展方向[89]。随着适应性实验室进化新策略和新工具的发展,适应性实验室进化大大加速了改造代谢途径及细胞耐受性的效率,在提高目标产物合成水平、拓展底物利用范围、提升底盘细胞的耐受性等方面得到广泛应用。由传统单一碱基、单一基因的随机进化模式到多基因、多途径的全基因组进化策略,如合成型酿酒酵母基因组重排(SCRaMbLE)、寡核苷酸介导的多位点进化工具(YOGE、eMAGE) 和基于CRISPR 系统的多位点编辑(CHAnGE、Targeted-AID 等);利用正交复制系统和逆转录系统构建体内连续进化系统,减少人为干预并加快进化速度。随着合成生物学技术与其他学科进行深度交叉融合,将基因组连续进化技术与其自动化技术相结合(eVOLVER 和ACE),加速代谢工程改造过程,实现对酿酒酵母可预测、可调控的系统性工程改造,加深了对细胞代谢网络调控及其生物过程的认知。
随着适应性试验进化技术的发展,细胞工厂的构建与筛选往往受限于目标表型的测定,研究人员还需不断改进和优化现有的技术,如利用生物传感器(转录调控因子、蛋白质传感器、核糖开关等)将难以检测的表型转化为可快速测定的表型[90-92],将连续进化技术与微流控技术、自动化技术相结合建立高通量的筛选平台[92-96],进一步优化自动连续进化系统提高其实用性,不断拓展适应性试验进化的应用。除此之外,适应性实验室进化获得的性状优良菌株往往含有大量的突变位点,其中与目标表型强关联的突变点比较有限,开发更为高效精确的生物信息学算法,降低了遗传机理解析的难度。
总之,适应性实验室进化为细胞工厂的构建提供了强大的工具,在未来的发展中将连续进化技术与先进的计算机辅助设计、自动化技术及高通量筛选技术有机结合起来,将极大促进复杂生命现象的解析,推动代谢工程、系统生物学、合成生物学快速发展。

