西瓜PME基因生物信息学分析及克隆
王学征 ,李钰婷 ,张 博 ,张雨桐 ,张晏航 ,韩文灏,朱子成 ,刘 识 ,刘宏宇
(1.农业农村部东北地区园艺作物生物学与种质创制重点实验室,哈尔滨 150030;2.东北农业大学园艺园林学院,哈尔滨 150030;3.东北农业大学研究生院,哈尔滨 150030;4.东北农业大学生命科学学院,哈尔滨 150030)
随着人们生活水平逐步提高,对生活质量特别是对农产品品质要求越来越高。农产品品质主要以商品品质、感官品质和营养品质为主。果实质地是评价西瓜商品性重要指标,果实硬度过高,难以食用;果实过于酥松,口感不佳,不耐运输和贮藏。
果胶是细胞壁主要成分之一,对果实硬度有直接影响。果胶属于多糖,结构复杂具有许多被修饰侧链。果胶甲酯酶(Pectin methylesterase,PME)广泛存在于植物和一些具有细胞壁重塑降解作用的微生物中,可催化细胞壁中果胶主链发生脱甲酯化反应,产生羧基基团,同时释放氢离子和甲醇,达到细胞壁分离目的,降低果实硬度[1]。齐秀东等研究发现,在嘎拉苹果发育过程中,PME基因表达量增加,果实硬度显著下降[2]。Segonne 等研究表明,苹果果实发育和成熟过程中PME对细胞壁重塑具有重要影响,且特定基因Md-PME2可作为苹果质地不利性状表现早期筛选指标[3]。
果胶甲酯酶不仅调控细胞壁组成、降解[4]和果实成熟[5],且在花粉萌发和花粉管生长[6]、形成层细胞分化[7-8]等植物生长发育过程发挥重要作用。Thumdee等研究发现番木瓜软化与PME相关[9]。王艳婷通过体外提取细胞壁多糖对拟南芥PMEs超大基因家族中2个新基因PME1和PME26作遗传鉴定和表征分析,结果显示PME1 和PME26 调控细胞壁果胶甲酯化程度,具有催化果胶与HG去甲酯化作用[10]。
PME是多基因家族,近年来,对于同一物种PME基因研究不断深入。熊兴鹏研究发现,BcPME37c基因敲除突变体花粉在双核时期出现异常,花粉内壁显著加厚,表明BcPME37c基因突变引起果胶中HG去甲酯化过程异常,从而影响花粉发育[11]。目前,PME基因在拟南芥中共鉴定出67 个[12]、水稻中43个[13]、大豆中16个[14]、亚麻中105个[15]、雷蒙德氏棉中81 个[16],但在葫芦科植物中研究较少。西瓜、甜瓜等葫芦科植物是主要园艺作物和经济作物,与模式植物拟南芥在果实硬度变化等方面存在差异。本试验从模式植物拟南芥和东北农业大学西甜瓜分子育种课题组西瓜转录组数据入手,通过生物信息学分析,选用BLAST 比对方法在葫芦科数据库中鉴定甜瓜、西瓜果胶甲酯酶同源基因,对其蛋白质一级、二级和三级结构预测分析,构建分子进化树。在此基础上,在西瓜中克隆一个PME基因Cla012554,分析在西瓜各部位及在不同果肉质地西瓜果实发育过程中表达情况,进一步探究PME结构及作用机制,为西瓜果胶甲酯酶基因研究、功能分析及利用提供参考及依据。
1 材料与方法
1.1 PME基因生物信息学分析
1.1.1 数据来源及BLAST比对分析
拟南芥4 个PME基因cDNA 序列来源于TAIR网站(http://www.arabidopsis.org/),西瓜2 个PME基因来源于东北农业大学西甜瓜分子育种实验室转录组数据。利用BLAST 比对分析(BLAST program为BLASTn),将上述基因核苷酸序列在葫芦科数据库(http://cucurbitgenomics.org/)甜瓜比对数据库Melon(DHL92) mRNA 和西瓜比对数据库 Water⁃melon(97103)CDS中比对,找到符合要求基因。
1.1.2PME基因各级结构分析
根据筛选出PME基因mRNA 序列,运用Prot⁃Param(http://web.expasy.org/protparam/)分析 PME 蛋白序列一级结构(分子质量、等电点和氨基酸数量等);运用ProtScale(http://web.expasy.org/protscale/)分析PME 蛋白质一级结构亲疏水性;运用SOPMA(https://npsa-prabi.ibcp.fr/cgi-bin/secpred_sopma.pl)分析PME 蛋白序列二级结构(α螺旋、β转角、链延伸结构和无规则卷曲)。运用SWISS-MODEL(http://www.swissmodel.expasy.org/)分析PME 蛋白质三级结构并构建模型;运用Cell-PLoc 2.0(http://www.csbio.sjtu.edu.cn/bioinf/Cell-PLoc-2/)对 PME 蛋白质作亚细胞定位。
1.1.3 分子进化树构建
运用MEGA 7.0软件,通过Neighbor-Joining 对筛选出PME 蛋白结构域序列作比对分析,构建系统进化树,其中矫正参数值设置为1 000。
1.2 Cla012554基因克隆及表达量测定
1.2.1 材料来源
栽培西瓜W1-1和黏籽西瓜PI186490由东北农业大学西甜瓜分子育种研究室提供,W1-1果实圆形,果肉软沙。PI186490 果实圆形,果肉坚硬。其中W1-1 为基因克隆材料,W1-1 和PI186490 为基因时空表达材料。采集黏籽西瓜PI186490 和栽培西瓜W1-1 授粉35 d 后根、茎、叶、果肉组织,3 次生物学重复。液氮处理,提取RNA,反转录cDNA备用。
1.2.2 RNA提取及cDNA合成
RNA 提取采用Trizol 提取法,利用东洋纺(上海)生物科技有限公司反转录试剂盒对经检验合格RNA 反转录为cDNA。其中 RNA 模板为 0.5~1 μg,Primer Mix 0.5 μL,5×RT Buffe 2 μL,RT Enzyme Mix 0.5 μL,水补齐至10 μL。反转录条件为37 °C 15 min,95 °C 5 min,cDNA在-20 ℃条件下保存。
1.2.3Cla012554基因克隆
根据葫芦科数据库中Cla012554 基因cDNA 序列,利用Primer 6.0 软件设计特异性上、下游引物(见表1 第一条序列),以RNA 反转录cDNA 为模板,使用TOYOBO KOD One PCR Master Mix 酶作PCR扩增。
扩增程序如下:98 ℃预变性1 min,此后33个循环,每个循环98 ℃变性10 s,58 ℃退火5 s,68 ℃延伸5 s,最终72 ℃延伸1 min。通过琼脂糖凝胶电泳检测PCR 扩增产物,选取条带位置正确且亮度高目的片段回收。通过载体转运至大肠杆菌,将菌液送往生工生物工程(上海)股份有限公司测序。
1.2.4Cla012554基因时空表达量分析
在多重Real-time 荧光定量PCR 仪下测定该基因相对表达量。引物设计采用Primer 6设计(见表1第二和第三条序列),均为特异性引物,退火温度为50~55 ℃。以西瓜Cla020175为内参基因。qPCR反应试剂为THUNDERBIRD SYBR qPCR Mix,反应体系及程序参照试剂说明书,采用2-ΔΔCt法计算待测基因相对表达量。3次生物学重复。
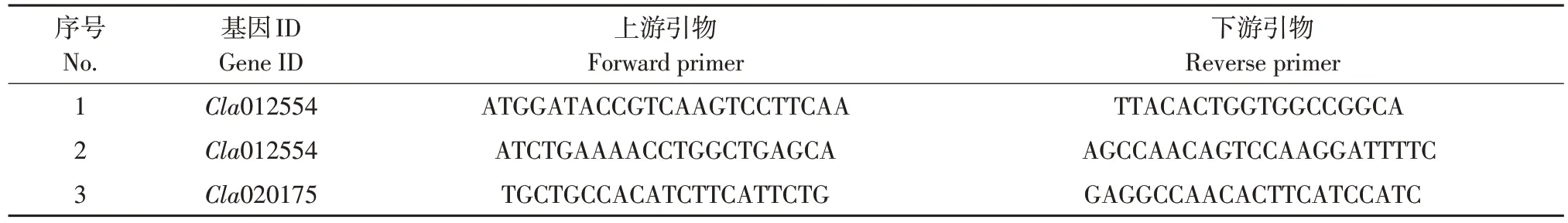
表1 Cla012554基因引物序列Table 1 Primer sequence of Cla012554 gene
2 结果与分析
2.1 PME基因生物信息学分析
2.1.1 PME蛋白质一级结构分析
基于拟南芥4个PME基因以及2个根据西甜瓜转录组数据库选定西瓜PME基因,通过BLAST 比对,在西瓜中鉴定出3 条PME同源基因,在甜瓜中鉴定出2 条PME同源基因。运用ProtParam(http://web.expasy.org/protparam/)预测筛选得到PME蛋白序列一级结构。由表2 可知,西瓜PME基因分布在5号、7号和9号染色体,其中9号染色体分布最多,有3个,其余染色体各1个。甜瓜PME基因分布在1号和4号染色体。葫芦科中PME蛋白氨基酸数目相差较少,大部分氨基酸数量约为570,其中Cla014927氨基酸数目最少(528个),Cla004251氨基酸数目最多(604个)。拟南芥PME蛋白质中,氨基酸数目相差较大,AT4G02330 氨基酸数量最少(573 个),AT3G14300 氨基酸数目最多(968 个)。葫芦科PME 蛋白质与拟南芥PME 蛋白质等电点和脂肪族指数较一致,等电点6.3~9.32,脂肪族指数75.71~89.60。葫芦科 PME 蛋白质与拟南芥 PME 蛋白质不稳定指数相差较大,葫芦科PME 蛋白质不稳定指数平均为33.05,拟南芥PME 蛋白质不稳定指数平均为26.91。
2.1.2 PME蛋白质亲疏水性预测
运用ProtScale 在线软件预测蛋白质亲疏水性,由表2 可知,Cla012554(亲水性最大值2.444,疏水性最大值3.856)和MELO3C022701T1(亲水性最大值2.444,疏水性最大值3.822)具有疏水性外,其余葫芦科PME 蛋白具有亲水性,其中,Cla015505 亲水性较强(亲水性最大值3.356,疏水性最大值2.900)。
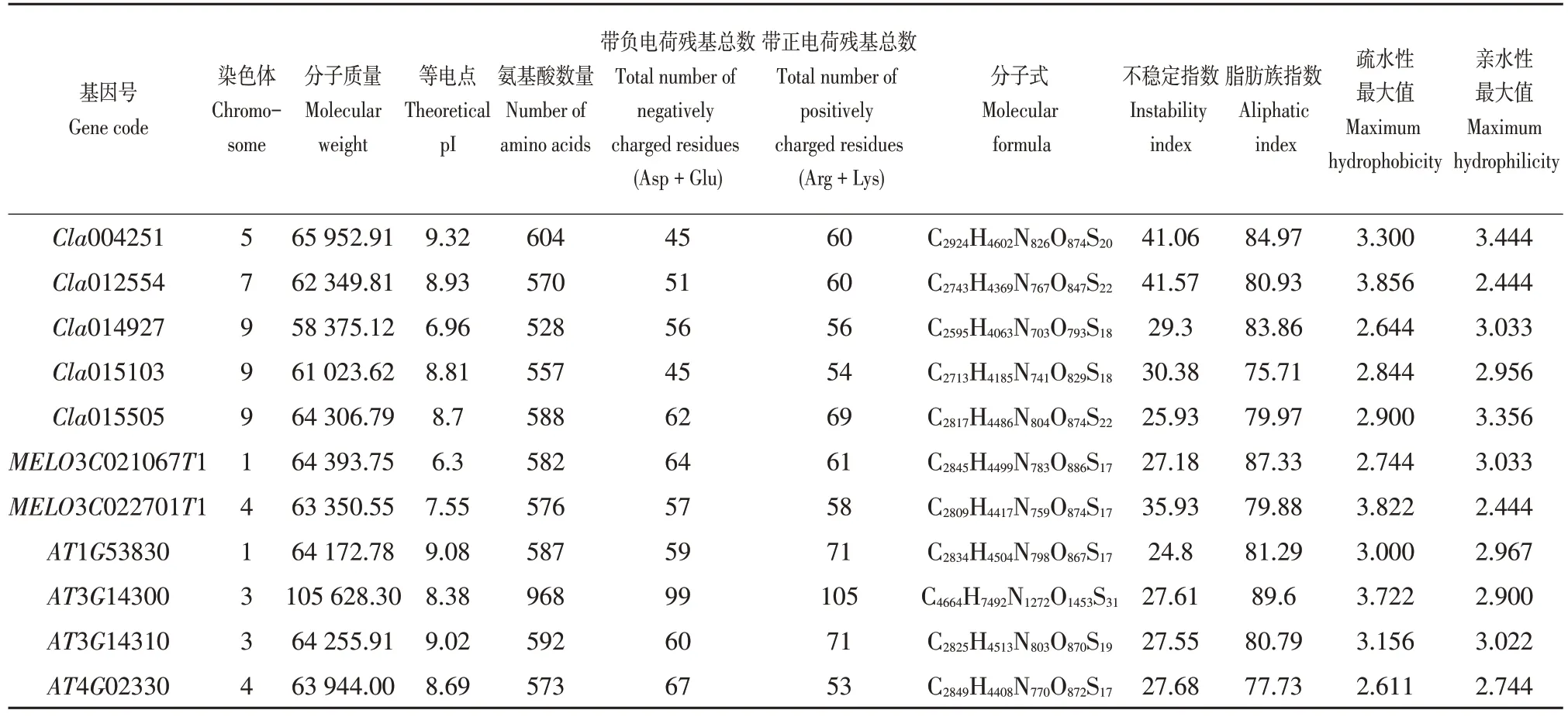
表2 PME蛋白质一级结构分析Table 2 PME protein primary structure analysis
2.1.3 PME蛋白二级结构预测
运用SOPMA在线软件预测PME蛋白序列二级结构,由表3 可知,各PME 蛋白主要是α 螺旋(35.55%)和无规卷曲(40.86%)构成,含较少链延伸结构(19.03%),含极少量β转角(5.56%)。在此基础上,AT3G14300含有较高α螺旋(41.32%),含较少无规卷曲(36.88%)和链延伸结构(15.81%);MELO3C022701T1 含较高链延伸结构(21.53%)和β转角(6.66%);AT4G02330 含较多无规则卷曲(46.07%)、较少α 螺旋(29.14%);Cla014927 含较少β转角(4.17%)。
2.1.4 PME蛋白质高级结构预测
Cla012554、Cla015505、AT3G14310 和ME⁃LO3C022701T1 有 3 种 建 模 构 型 ,Cla015103、AT1G53830 和MELO3C021067T1 有 4 种建模构型,Cla014927、Cla004251 和AT4G02330 有 5 种建模构型,AT3G14300 有6 种建模构型,如图1 所示。在葫芦科与拟南芥PME 蛋白质模型构建中,高级结构模型均含有一条链且结构相似。
2.1.5 PME蛋白亚细胞定位预测结果
运用Cell-PLoc 2.0 软件预测PME 蛋白质亚细胞定位,PME蛋白均分布于细胞壁。
2.1.6PME基因分子进化树构建
将筛选得到拟南芥及葫芦科PME 蛋白质,运用MEGA 7.0 软件,通过Neighbor-Joining 作序列比对分析且构建分子进化树。如图2所示,PME蛋白质可分为2 类。在第一类中,西瓜Cla012554、Cla014927和Cla0155051,甜瓜MELO3C021067T1和MELO3C022701T1序列存在高度相似,说明同一物种及葫芦科各物种间存在高度同源关系。第二类中,西瓜Cla015103和Cla004251,拟南芥AT4G02330蛋白质间高度相似,说明其蛋白质功能相似。
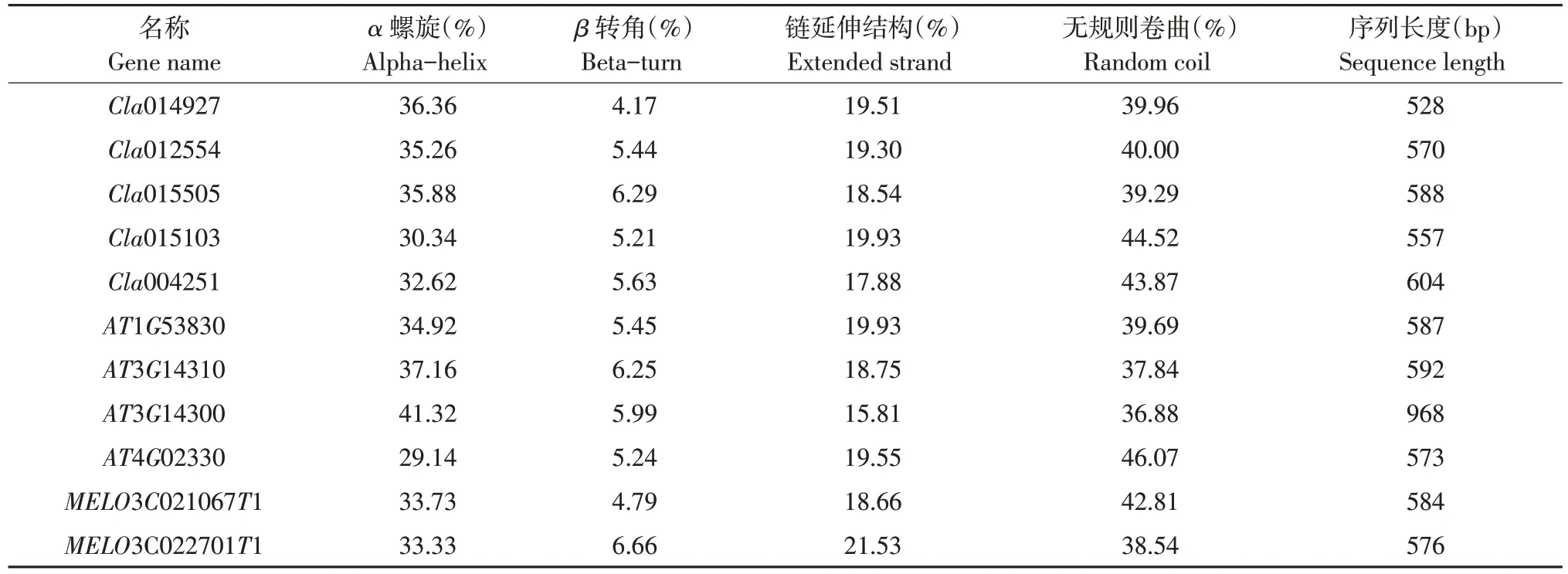
表3 PME蛋白二级结构预测Table 3 Prediction of the secondary structure of PME protein
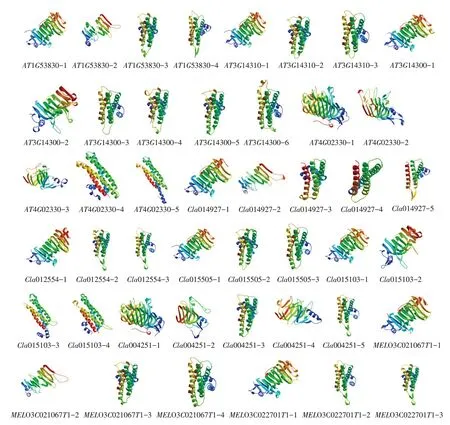
图1 拟南芥、西瓜和甜瓜PME蛋白高级结构预测Fig.1 Prediction of high-level structure of PME protein in Arabidopsis,watermelon and melon
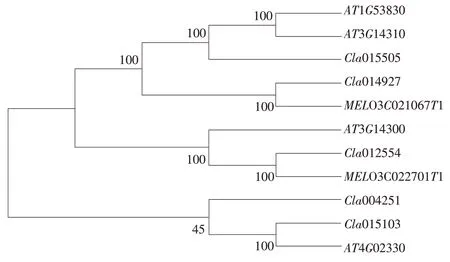
图2 分子进化树构建Fig.2 Construction of molecular evolutionary tree
2.2 Cla012554基因克隆
Cla012554 基因克隆以栽培西瓜 W1-1 的 2 叶1 心时期幼叶cDNA 为模板,作PCR 扩增,通过琼脂糖凝胶电泳检测PCR 扩增产物,选取条带位置正确且亮度高目的片段回收。通过载体转运至大肠杆菌,将菌液送往生工生物工程(上海)股份有限公司测序。菌液经测序后,利用DNAMAN 软件将测序结果与Cla012554基因CDS序列比对,两者相似度为95.62%(见图3),可能品种间存在差异。
2.3 Cla012554基因组织表达特性
qRT-PCR 分析Cla012554 基因组织表达特性,由图4 可知,在正常生长条件下Cla012554 基因在不同组织中均表达,其中,茎中表达量最高,其次为果肉和叶片,根中表达量最少。
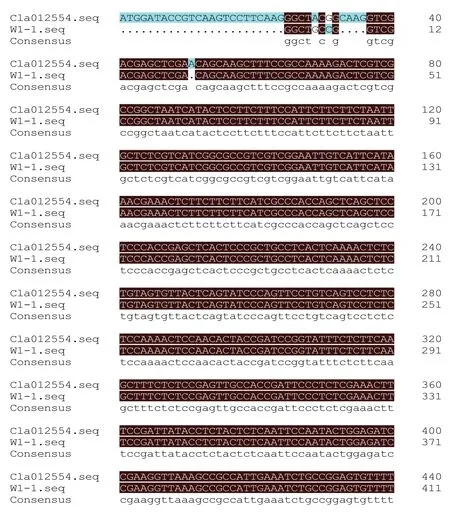


图3 Cla012554基因测序结果序列比对Fig.3 Sequence alignment of Cla012554 gene sequencing results
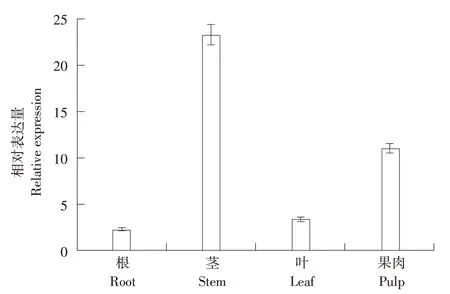
图4 Cla012554基因在W1-1不同组织中相对表达量Fig.4 Relative expression of Cla012554 gene in W1-1 different tissues
2.4 Cla012554基因在果实发育过程中表达特性
qRT-PCR结果表明,Cla012554基因随西瓜果实发育,表达量呈不同变化,结果见图5,Cla012554基因在栽培西瓜W1-1 中,随果实成熟,果肉软化,表达量逐渐上升,在28 d 果实近成熟时达到高峰。而在黏籽西瓜PI186490 果实发育过程中,果肉未软化,该基因表达量也未发生变化。因此,推测该基因与西瓜果肉成熟软化相关。
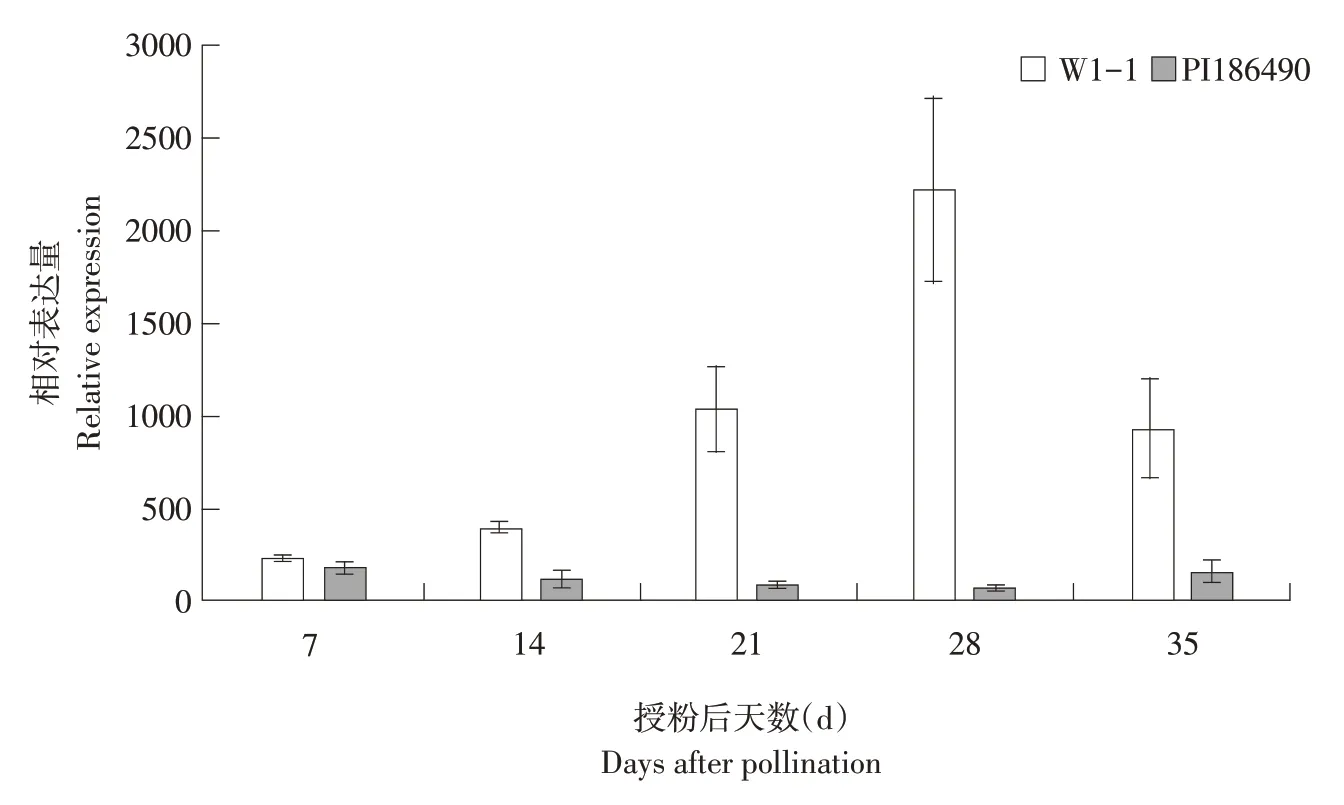
图5 Cla012554基因在两种西瓜果实发育中相对表达量Fig.5 Relative expression of Cla012554 gene during fruit development of two watermelon cultivars
3 讨 论
本研究从拟南芥4个果胶甲酯酶基因和2个来自东北农业大学西甜瓜分子育种西瓜转录组数据分析的果胶甲酯酶基因入手,通过BLAST 比对方法在葫芦科数据库中鉴定甜瓜、西瓜果胶甲酯酶同源基因,并预测分析其生物信息学。在西瓜中共鉴定出3条PME同源基因,在甜瓜中鉴定出2条PME同源基因。超过2/3西瓜、甜瓜和拟南芥PME蛋白质氨基酸数目在584、等电点8.4,脂肪族指数81.6,不稳定指数31.4,且为可溶蛋白质,具有较强亲水性。说明西瓜、甜瓜与拟南芥PME 蛋白质理化性质相似。菊欧文氏杆菌、番茄、胡萝卜以及棉花PME蛋白质结构高度相似[17-19],均具有3个内含疏水结构折叠的螺旋结构,其结构差异主要表现在半胱氨酸(Cys)残基、组氨酸(His)残基和芳香族氨基酸残基,与本研究结果一致。葫芦科作物甜瓜、西瓜PME 蛋白质二级结构主要为α-螺旋、无规卷曲和链延伸结构,含有较少链延伸结构和β转角。对蛋白质高级结构预测建模,通过构建系统发生树发现,甜瓜、西瓜与拟南芥果胶甲酯酶蛋白质均有不同程度相似性,推断各物种间PME基因家族间同源性较高。
此外,通过同源克隆方法获得西瓜Cla012554基因全长并分析该基因时空表达量,Cla012554 基因作CDS序列比对,结果发现,相似度为95.62%,表明序列间一致度较高。序列间存在差异,可能是克隆过程中由于Cla012554基因片段较长,KOD酶保真性低、切胶回收时紫外线对DNA 损伤等产生误差,也可能是Cla012554基因在葫芦科植物中存在基因多态性所致[20-21]。
本研究结果表明,Cla012554 基因在不同组织中均表达,说明PME基因在西瓜发育中发挥重要作用。Cla012554 在茎中表达量最高,其次是果肉和叶片,在根中表达量最低。Yang 等从杨树中找到果胶甲酯酶基因PtoPME35,基因表达分析表明PtoPME35 在多种组织中具有组成型表达模式,茎中表达量最高[22]。Shoko 等研究表明,拟南芥中AtPME35 在茎特异性表达最高,是控制茎机械强度必需基因,结果表明,果胶甲酯酶基因不仅控制果肉硬度,也增加茎机械强度,具有抗倒伏作用,在果胶初生细胞壁及次生细胞壁中发挥作用[23]。
果实发育过程中,Cla012554 基因在W1-1中,随果实成熟果肉软化,基因表达量不断升高。PI186490 果实发育过程中,果肉未软化且该基因表达量也未发生变化。邓佳等研究发现,在葡萄柚果实后熟软化过程中,PME基因表达量均随果实软化呈不同程度增加[24]。由此推断,PME基因家族中Cla012554基因可能与西瓜果肉成熟软化相关。通过本研究分析葫芦科PME基因家族生物信息学,初步了解家族成员,为今后葫芦科PME基因家族发掘、利用及种质资源改良提供新思路。

