印迹基因GRB10来源的环状RNA hsa_circ_0008334的特征分析及功能预测
姚文霞 董志杰 潘劲辉 林铭珍 蔡 华 梁 雪
环状RNA(circular RNA,circRNA)是一类不含有5’端帽子和3’端poly A尾巴、由3’端和5’端共价结合形成的闭合环形RNA。作为非编码RNA(noncoding RNA,ncRNA)家族的成员,circRNA广泛存在于各种真核生物中[1]。早期由于科研条件的限制,对circRNA的研究还不够深入,其被认为是RNA剪接过程中的副产物[2]。近年来随着高通量测序技术的快速发展,越来越多circRNA被发现,并被报道是充满前景的生物标记物和治疗靶点[3-7]。circRNA具有细胞特异性表达[4]、耐受核糖核酸酶R(Ribonuclease R,RNase R)消化[8]等特征,在许多生物过程以及疾病发生中发挥了重要的生物学功能。目前报道的主要功能有:充当miRNA海绵[8]、调控基因转录[9]、与RNA结合蛋白相互作用[10]甚至翻译成多肽[11]等。
基因印迹是指基因表现出亲本等位基因表达的差异性,即只表达一方的亲本等位基因。生长因子受体结合蛋白10(Growth factor receptor bound protein 10, GRB10)基因即是典型的印迹基因,其编码的蛋白是属于GRB7/GRB10/GRB14家族的衔接蛋白[9]。这些衔接蛋白可识别结合多个细胞表面受体及下游信号,从而与其他蛋白产生相互作用并调控生物学过程。具体来讲,GRB10主要参与细胞增殖、细胞凋亡和代谢的调控,调控哺乳动物生长发育、原肠运动和血糖调节等[10]。
本文研究的环状RNA hsa_circ_0008334是由GRB10基因经反向剪接而成,由转录本NM_001001555的4、5号外显子组成。根据circBase数据库[11]收录信息,人类GRB10基因可反向剪接形成多个环状RNA,其中长度为365 bp的环状RNA hsa_circ_0008334被高通量测序检测到的相关研究最多、样本分布最为广泛,提示该环状RNA具有重要的生物学功能和意义。因此,本文关注到了环状RNA hsa_circ_0008334并研究了其基本特征,包括反向剪切位点、RNase R抵抗性和胞质胞核分布。另外,通过在线数据库预测该环状RNA结合的miRNA以及miRNA的靶基因,我们构建了circRNA-miRNA-mRNA分子调控网络,构建了靶基因的蛋白交互作用网络,并对miRNA的靶基因进行基因本体论(Gene Oncology, GO)功能富集分析。通过本研究,为深入研究hsa_circ_0008334的特征和功能奠定了基础,从circRNA-miRNA-mRNA角度为 hsa_circ_0008334的功能研究提供了新的见解,并进一步扩充了GRB10基因的相关研究。
1 材料与方法
1.1 实验材料
细胞培养用试剂:DMEM(Gibco)、双抗(青霉素及链霉素)(Gibco)、FBS(Gibco)、胰蛋白酶(Gibco)、PBS(Gibco)。RNA提取及RT-PCR用试剂:TRIzol试剂(Invitrogen)、RNase-free H2O(Takara)、PrimeScript RT reagent Kit with gDNA Eraser(Takara, RR047A)、TB GreenTMPremix Ex TaqTMII(Takara, RR820A)。核酸电泳试剂:DNA marker(Omega, M10-02)、DNA loading buffer(Takara)、SYBR Green I核酸染料(Solarbio, SY1020)。其他试剂:PARIS Kit(AM1921)、RNase R(Epicentre, RNR07250)、RNeasy MinElute Cleanup Kit(Qiagen, 74204)。引物购自Invitrogen 公司。
1.2 hsa_circ_0008334反向剪接位点的PCR扩增及测序验证
根据在circBase(http://www.circbase.org/)[9]上检索得到的hsa_circ_0008334的序列,设计一对双向引物来扩增出包含反向剪接位点的序列,引物序列为:Forward Primer 5’-TGTGCACATCCTCGCTGTCA-3’;Reverse Primer 5’-GGTCCACATCATCCTCTGCT-3’。PCR扩增的模板为细胞的cDNA, 扩增产物长度为178 bp。PCR产物经2.0%琼脂糖凝胶电泳检测后,送广州艾基生物技术有限公司进行一代测序。
1.3 RNase R处理实验
1.3.1 RNA提取 收集细胞,用Trizol法提出细胞的总RNA。
1.3.2 RNase R消化 RNase R处理组和对照组(各10 μg RNA)分别加入20 U(2 U/μg)的RNase R和等量的ddH2O,37 ℃孵育10 min。
1.3.3 RNA纯化 RNase R处理后的RNA使用RNeasy MinElute Cleanup Kit试剂盒进行纯化,随后测定RNA浓度。
1.3.4 逆转录及实时定量PCR 用PrimeScript RT reagent Kit with gDNA Eraser试剂盒将纯化后的RNA逆转录成cDNA,然后通过TB GreenTMPremix Ex TaqTMII 试剂盒进行PCR扩增,反应体系共20 μl:TB Green Premix Ex Taq II 10 μL,ROX Reference Dye II 0.4 μL,正反向引物各0.8 μL,ddH2O 6 μL,cDNA 2 μL。
1.4 RNA的细胞质、细胞核表达量分析
1.4.1 细胞质、细胞核RNA分离及提取 收集细胞,用PARIS Kit试剂盒将胞质、胞核中的RNA分离并提取出来,随后进行胞质和胞核RNA浓度的测定。
1.4.2 逆转录及定量PCR 分别取细胞质和细胞核RNA各500 ng,用PrimeScript RT reagent Kit with gDNA Eraser试剂盒将其逆转录成cDNA。随后以该cDNA为模板、用TB GreenTMPremix Ex TaqTMII 试剂盒进行实时定量PCR。
1.5 miRNA结合位点的预测
利用circBank(www.circbank.cn)[12]和circInteractome(https://circinteractome.nia.nih.gov/[13]两个在线数据库来预测环状RNA上结合的miRNA,并取两个数据库预测结果的交集作为环状RNA可能靶向结合的miRNA。
1.6 miRNA靶基因的预测
利用数据库miTarBase[14]预测hsa_circ_0008334潜在结合的五个miRNA的靶基因;miTarBase是一个全面收集已被实验验证的miRNA靶标的数据库。
1.7 circRNA-miRNA-mRNA网络的构建
根据竞争内源性RNA(competing endogenous,ceRNA)理论,利用Cytoscape3.6.1[15]软件构建circRNA-miRNA-mRNA网络,并将其可视化。网络图的节点代表不同的RNA分子,连线代表节点之间的相互作用。
1.8 蛋白交互作用(Protein-protein interactions, PPI)网络的构建
靶标基因蛋白之间的相互作用,通过STRING数据库进行分析,将STRING数据库中的PPI数据导入Cytoscape3.6.1[15]软件绘图,并将其可视化。每个节点(Node)代表一个基因,两点之间的连接也就是边(Edge)表示交互作用关系。
1.9 靶基因集合的功能及通路分析
利用DAVID(https://david.ncifcrf.gov/)[16]数据库中“Functional Annotation Chart”功能模块对预测的靶基因集进行GO生物学过程(Biological Process,BP)和分子功能(Molecular Function,MF)分析。
2 结果
2.1 hsa_circ_0008334反向剪接位点的PCR扩增及一代测序鉴定
通过PCR扩增hsa_circ_0008334,琼脂糖凝胶电泳检测,结果显示成功扩增出包含反向剪接位点的大小为178 bp的片段(图1-A)。序列分析表明,hsa_circ_0008334由来源于GRB10基因的转录本NM_001001555的第5号外显子反向剪接至第4号外显子形成,共包含2个外显子(Exon 4、Exon 5)(图1-B);测序结果进一步证实这一结论,箭头左端为GRB10基因5号外显子末端序列,右侧为4号外显子的起始序列(图1-B)。
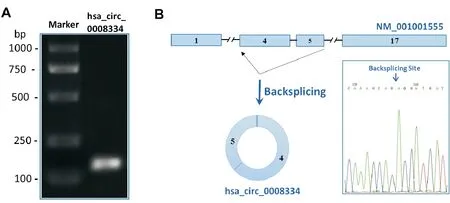
图1 A. hsa_circ_0008334反向剪接位点的PCR扩增鉴定,扩增片段长度为178 bp;B. hsa_circ_0008334的产生示意图及反向剪接位点的一代测序峰图
2.2 hsa_circ_0008334抵抗RNase R消化,且主要分布在细胞质中
环状RNA为共价闭合的环状结构,没有游离末端,故环状RNA对RNase R等RNA核酸外切酶存在一定的抵抗性。为了进一步证实hsa_circ_0008334的成环特性,本研究通过使用RNase R处理细胞总RNA,然后进行RT-qPCR来观察RNA的降解情况。实验结果显示(图2-A):与作为阳性对照的环状RNA CDR1as的结果一致,RNase R处理组的环状RNA hsa_circ_0008334基本没有降解,相比之下,GAPDH基因的线性mRNA则出现明显的降解,这表明hsa_circ_0008334耐受RNase R的消化。
由于环状RNA的功能与其亚细胞的定位有关,因此本研究将细胞的细胞质、细胞核RNA分离后,用qPCR分别检测胞质、胞核中hsa_circ_0008334的相对分布量。实验结果(图2-B)显示:对照U1 snRNA主要分布于细胞核,对照GAPDH主要分布在细胞质,说明胞质、胞核RNA分离是成功的;环状RNA hsa_circ_0008334在胞质中的表达量显著高于细胞核,即hsa_circ_0008334主要分布于细胞质。
2.3 hsa_circ_0008334上miRNA结合位点的鉴定
ceRNA发挥生物学功能的主要途径是通过与miRNA应答元件(miRNA response element, MRE)相互结合。本研究使用在线数据库circInteractome和circBank来分析环状RNA hsa_circ_0008334是否通过ceRNA机制发挥功能。CircInteractome 数据库显示环状RNA hsa_circ_0008334上结合的miRNA数量为23个(图3-A),circBank数据库显示环状RNA hsa_circ_0008334上结合的miRNA数量为46个(图3-B)。我们将circInteractome和circBank两个数据库预测结果的交集作为hsa_circ_0008334潜在靶向结合的miRNA,共5个miRNA,即hsa-miR-1265、hsa-miR-187-3p、hsa-miR-586、hsa-miR-604、hsa-miR-941。这些结果表明,hsa_circ_0008334可能通过作为miRNA的ceRNA海绵来发挥功能。
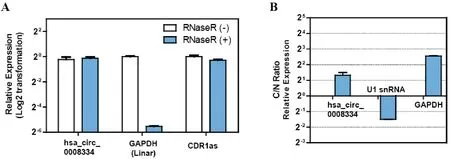
图2 A. hsa_circ_0008334抵抗RNase R的消化; B. hsa_circ_0008334、U1 snRNA和GAPDH在胞质胞核中的分布情况

图3 hsa_circ_0008334上MREs位点示意图。3个环形均代表环状RNA hsa_circ_0008334的结构模式,由两个外显子组成。红色三角代表通过CircInteractome数据库预测的hsa_circ_0008334的MRE,蓝色三角和绿色三角分别代表通过circBank中targetscan和miRanda算法预测的hsa_circ_0008334的MRE

图4 hsa_circ_0008334-miRNA-mRNA网络图。红色长方形代表circRNA,绿色长方形代表miRNA,紫色长方形代表miRNA的靶基因,线条代表不同RNA分子间的连接结合
2.4 circRNA-miRNA-mRNA网络可视化结果
环状RNA通过与miRNA相互作用后,可调控下游靶基因的表达。我们选取数据库miTarBase预测hsa_circ_0008334潜在结合的五个miRNA的靶基因。miTarBase的预测结果显示,hsa-miR-1265、hsa-miR-187-3p、hsa-miR-586、hsa-miR-604、hsa-miR-94的靶基因数量分别为72个、24个、94个、39个、55个;去除重复靶基因之后,上述五个miRNA的靶基因共为279个。通过随机选取279个靶基因中的39个靶基因,并利用Cytoscape3.6.1软件,我们构建了以hsa_circ_0008334、miRNA以及39个靶基因为核心的circRNA-miRNA-mRNA分子调控网络图。图4形象地展现了它们之间的相互关联。
2.5 靶基因的蛋白交互作用网络可视化结果
蛋白之间的相互作用对蛋白行使功能至关重要。为了评估hsa_circ_0008334下游miRNA对应的所有靶基因的功能,我们构建了279个靶标基因蛋白的PPI网络。对靶标基因蛋白之间的相互作用通过STRING数据库进行分析,并利用Cytoscape3.6.1软件绘图,得到如图5所示的PPI网络图。图5-A结果显示,279个靶标基因蛋白的PPI网络图共含有200个节点和453个边,即含有200个节点蛋白和453个交互作用关系。
关键基因/枢纽基因(Hubgene)是在生物学过程中发挥至关重要作用的基因,在相关通路中,其他基因的调控往往要受到该基因的影响,因此, Hubgene往往是重要的作用靶点和研究热点。利用Cytoscape中cytoHubba和MCODE两种算法分析上述PPI网络中的枢纽基因(Hubgene),图5-B结果显示,两种算法分析得到的前10位排名的Hubgene皆为:ASB6、CDC20、CUL3、FBXW8、FBXL13、FBXL20、KLHL25、SH3RF1、TRIM4、UBE2V1。
2.6 靶基因的蛋白交互作用网络合集的GO富集及KEGG通路分析
为了评估hsa_circ_0008334潜在结合的五个miRNA对应的所有靶基因的功能,我们通过DAVID在线分析工具对279个靶基因合集进行GO功能富集分析。GO分析结果显示(图 6A-6B),靶基因分别在12个生物学过程(Biological Process, BP)条目和8个分子功能(Molecular Function, MF)条目中显著富集。其中最显著富集的BP条目包括对凋亡、细胞程序性死亡、代谢过程等的正向调控和负向调控等,最显著的MF条目包括单链RNA结合、转录抑制因子活性、DNA结合、转录激活子活性等。
3 讨论
本研究首先分别从四个方面成功分析了印迹基因GRB10的新型环状RNA hsa_circ_0008334的特征:hsa_circ_0008334正确成环,是由GRB10基因第5号外显子反向剪接至第4号外显子形成;hsa_circ_0008334能够抵抗RNase R的消化;hsa_circ_0008334主要分布在细胞质中。随后,我们通过生物信息学方法成功建立了circRNA-miRNA-mRNA分子调控网络,GO功能富集分析表明miRNA的靶基因在对凋亡、细胞程序性死亡、代谢过程等的正向调控和负向调控的生物过程显著富集,从circRNA-miRNA-mRNA角度为 hsa_circ_0008334的功能分析提供了新的视角。
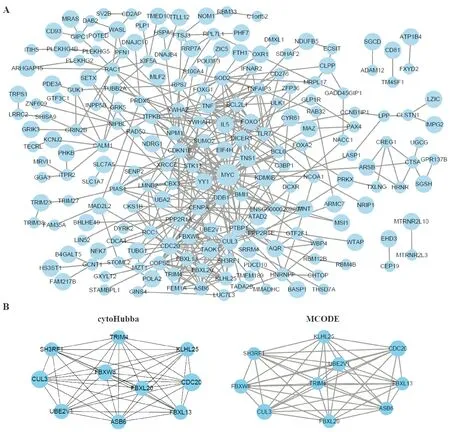
图5 靶基因的蛋白交互作用网络(A)及其关键基因(B):圆形代表节点蛋白,连线表示交互作用关系;圆形大小随相互作用蛋白的个数增加而增大;连线粗细随相互作用的增强而变粗。A. 圆形代表节点蛋白,连线表示交互作用关系。B. cytoHubba和MCODE两种算法分析得出的关键基因
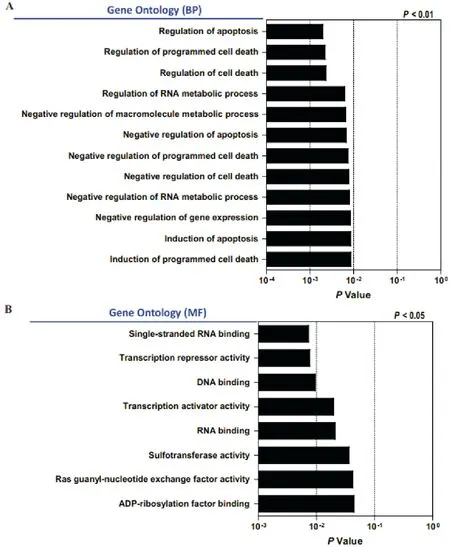
图6 A. GO富集分析:生物学过程(Biological Process, BP);B. GO富集分析:分子功能(Molecular Function, MF)
根据目前circBase数据库的收录信息,人类GRB10基因可经反向剪接产生大约30个环状RNA,其中长度为365 bp的hsa_circ_0008334样本分布最广泛,被高通量测序检测到的相关研究最多,提示该环状RNA功能相对重要,因此本文重点关注到了hsa_circ_0008334。通过PCR扩增、一代测序对hsa_circ_0008334反向剪接位点进行验证,并结合RNase R处理实验,证实hsa_circ_0008334正确成环。将hsa_circ_0008334的序列与基因组序列进行比对,结果发现其由GRB10基因转录本NM_001001555的2个外显子(exon4、exon5)经反向剪接而成,剪接点前后的序列都分别是AG和GT,符合环状RNA产生的剪接规律[17]。
环状RNA的功能与其在细胞内定位密切相关,位于细胞质中的环状RNA可以与miRNA相结合,通过充当miRNA海绵来发挥作用。本文在研究环状RNA的RNase R抵抗性时,使用的阳性对照CDRIas即是一个行使miRNA海绵体功能的典型例子[18];环状RNA CDR1as包含70多个保守的miR-7结合位点,可与miR-7吸附结合,解除miR-7对其靶基因的抑制作用,进而提高miR-7靶基因的水平。本研究中,胞质胞核分离结果显示说明hsa_circ_0008334主要分布于细胞质而非细胞核,提示hsa_circ_0008334充当miRNA海绵来行使功能。circInteractome 数据库通过targetscan软件来预测circRNA上miRNA结合位点,circBank则利用miRanda和targetscan两款软件预测circRNA和miRNA的相互作用。本研究中,我们分别使用circInteractome 和circBank数据库预测hsa_circ_0008334的miRNA结合位点,然后取两数据库预测结果的交集作为hsa_circ_0008334可能靶向结合的miRNA,即hsa-miR-1265、hsa-miR-187-3p、hsa-miR-586、hsa-miR-604、hsa-miR-94五个miRNA。miTarBase是一个全面收集已被实验证实的miRNA靶基因的数据库;本研究通过使用miTarBase数据库预测五个miRNA的靶基因并随机选取其中39个靶基因后,我们构建了circRNA-miRNA-mRNA分子调控网络,该调控网络揭示了hsa_circ_0008334可能通过作为hsa-miR-1265、hsa-miR-187-3p、hsa-miR-586、hsa-miR-604、hsa-miR-94五个miRNA的分子海绵竞争结合miRNA进而影响其下游靶基因的表达,为hsa_circ_0008334作为ceRNA发挥功能提供了证据。
本研究中功能注释结果显示,hsa_circ_0008334潜在结合的五个miRNA的靶基因在对凋亡、细胞程序性死亡、代谢过程等的正向调控和负向调控的生物过程显著富集。hsa_circ_0008334下游靶基因的这些功能与已报道的GRB10基因调控细胞增殖、细胞凋亡和代谢的调控等功能相类似,推测hsa_circ_0008334与其母基因GRB10具有部分相似功能。此外,Guo等[19-20]发现GRB10基因来源的另外一个环状RNA circ-GRB10(circBase中收录号为hsa_circ_ 0080210)参与了椎间盘退行性改变(Intervertebral Disk Degeneration, IDD)的发生、发展过程,circ-GRB10作为IDD过程中的关键circRNA具有抑制髓核细胞凋亡的重要作用,机制上circ-GRB10通过作为miRNA海绵吸附miR-328-5p调控ERBB2的表达来发挥作用。这些研究表明,GRB10基因及其衍生的某些circRNA都调控细胞增殖、细胞凋亡和代谢等。当然,对于GRB10基因衍生的circRNA的功能仍需进一步深入研究。此外,作为印迹基因,GRB10表现出亲本等位基因差异性表达;对于GRB10基因衍生的circRNA是否仍表现出亲本等位基因表达的差异性,还需进一步研究。
近年来,随着国内外学者对环状RNA关注度的提高,越来越多的环状RNA被发现,其生物学功能也被逐渐认识。然而,大量的环状RNA的功能研究还是空白的。本研究中,我们分析了hsa_circ_0008334的特征,并利用生物信息学方法成功构建了circRNA-miRNA-mRNA分子调控网络,该网络揭示了hsa_circ_0008334可能通过ceRNA机制发挥作用;本研究为进一步研究环状RNA hsa_circ_0008334的生物学功能奠定了基础。

