RHR转录因子家族起源、功能以及进化机制的研究进展
吕孟冈,刘艾嘉,李庆伟,苏鹏
综 述
RHR转录因子家族起源、功能以及进化机制的研究进展
吕孟冈1,2,刘艾嘉1,2,李庆伟1,2,苏鹏1,2
1. 辽宁师范大学生命科学学院,大连 116081 2. 辽宁师范大学七鳃鳗研究中心,大连 116081
转录因子是一类能够通过与基因特异性序列进行结合,从而调控基因转录与表达的蛋白质,对细胞的生物学活性具有重要的调节作用。RHR (Rel-homology region, RHR)转录因子家族属于IF (immunoglobulin fold)转录因子超家族最主要的成员,其成员含有保守的Rel结构域和IPT (immunoglobulin-like fold)结构域。作为古老的转录因子家族,RHR家族成员随着物种演化,通过基因的复制、突变和沉默,不断分化出新型同源基因的同时也伴随着基因的丢失。自然选择导致了各家族成员不同的进化速率,并且在一些功能结构域上展现出了特殊的进化机制。然而,目前有关RHR家族起源和分化的综述比较少见。本文综述了RHR家族各成员的分布、分类、功能及家族进化等方面的研究成果,以期为研究整个转录因子家族的演化机制和物种之间的进化关系提供参考和新的思路。
转录因子;RHR家族;进化;基因复制;同源基因
转录因子(transcription factors, TFs)即反式作用因子在基因转录过程中结合其顺式作用元件,参与靶基因的转录调控,对细胞的免疫,分化和死亡起着重要的调节作用[1]。转录因子一般包括两个主要功能域,即DNA特定序列结合域(DNA binding domain, DBD)和转录激活结构域(transcriptional activation domain, TAD)。根据DNA结合域的最新分类,将转录因子分为10个超家族,主要包含锌指结构域(zinc finger domain, ZFD)超家族、螺旋–转角–螺旋(helix-turn-helix domain, HTH)超家族、碱性结构域(basic domain, BD)超家族和免疫球蛋白折叠(immunoglobulin fold, IF)超家族等[2]。其中对于转录因子中ZFD超家族和HTH超家族的研究较为广泛[3],但是有关IF超家族的综述类研究却比较少见。RHR (Rel-homology region),也被称为RHD (Rel-homology domain,即Rel同源结构域)家族属于IF超家族最主要的成员,其都含有保守的Rel结构域和IPT (immunoglobulin-like fold, IPT)结构域[2]。它们具有结合特异性DNA序列,形成二聚体和蛋白质结合等功能[4,5]。RHR家族主要包含NF-κB (nuclear factor-κB)、NFAT (nuclear factor of activated T-cells)、EBF (early B-cell factor)和RBP (recombination signal binding protein)等亚家族。它们在真核生物中分布十分广泛,并且随着物种由简单到复杂,分别进化出多种类型的旁系同源基因(图1)。本文以RHR家族为代表,总结了其4个亚家族成员的分类、功能、起源与进化机制,以期为IF转录因子超家族的演化研究提供参考。
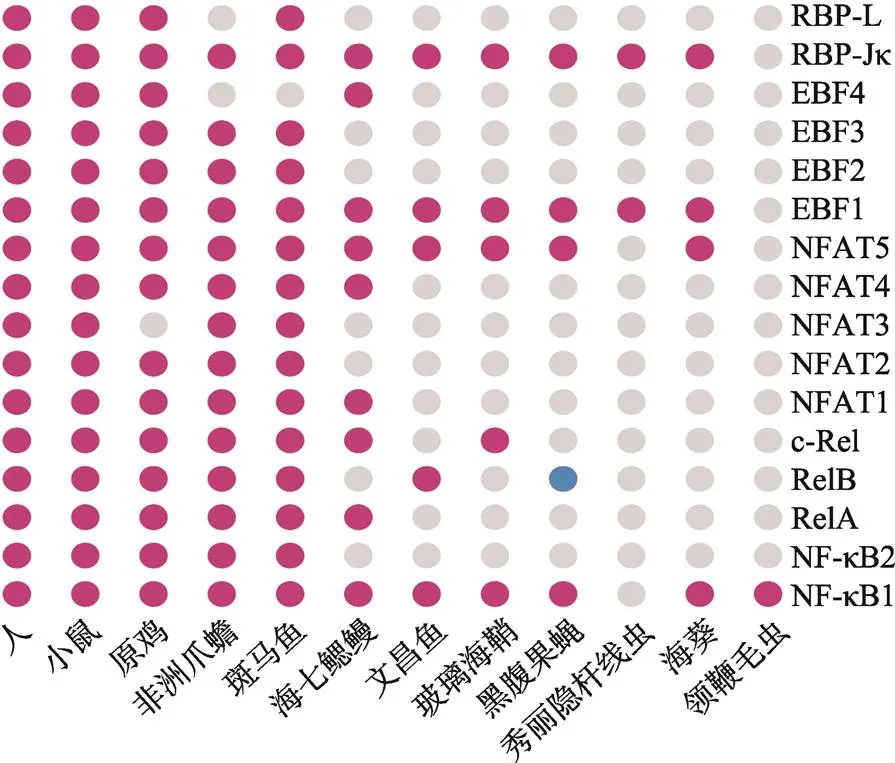
图1 RHR家族蛋白的在12种真核生物中的分布情况
根据已有文献和NCBI数据库中的数据对RHR转录因子家族蛋白绘制分布图。含有“0”、“1”、“2”蛋白个数,分别用亮灰色、玫红色和蓝色表示。
1 古老的转录因子—NF-κB
NF-κB是一类在细胞基因诱导调控中起着广泛作用的转录因子,被Sen等[6]首次发现于B淋巴细胞提取物中,因其能与κ轻链基因附近的核因子特异性结合而得名。在哺乳动物中,NF-κB家族成员主要包括:RelA(p65)、RelB、c-Rel、NF-κB1(p105)和NF-κB2(p100)[7]。该家族成员N端具有高度同源的Rel结构域[8]和IPT结构域,此外还具有核定位序列(nuclear localization sequence, NLS)。哺乳动物中NF-κB家族分为两类:第一类包括RelA(p65)、RelB和c-Rel,没有前体,除了Rel结构域之外,C末端具有转录激活结构域(TAD)。第二类包括NF-κB1 (p105)和NF-κB2(p100),其C末端区域含有ANK结构域(ankyrin repeat, ANK)[9]。ANK结构域是多个残基串联的基序,它们协同折叠成结构,通过蛋白质间相互作用来介导分子识别[10,11]。NF-κB家族成员可以形成同源二聚体或异源二聚体,并且它们通常也是以二聚体的形式发挥作用[12]。当细胞受到外界信号刺激后引发一系列反应,使IKK(inhibitor of kappa B kinase, IKK)活化。活化的IKK,诱导IκB (inhibitor of kappa B, IκB)蛋白快速降解。释放出NF-κB复合物,从细胞质转移到细胞核发挥调节转录作用[13]。
在进化过程中RelA、RelB和c-Rel具有一个高度保守的RFRYXCEG序列,能够识别DNA[14]。海葵()是已知的一种在该序列的关键位置具有Cys/Ser多态性的物种[15]。研究发现海葵中的等位基因Nv-NF-κB Cys与DNA的结合能力要高于Nv-NF-κB Ser。海葵的NF-κB这种多态性为研究NF-κB家族的Cys/Ser等位基因在自然界中是如何进化提供了可能[15]。此外,研究人员收集了403只不同种群的海葵,通过对Nv-NF-κB基因研究,鉴定出309个Cys/Cys纯合子,23个Ser/Ser纯合子和71个Cys/Ser杂合子,并计算出了遗传分化指数(st)为0.56,这表明该多态性在各亚群之间存在高度的分化[15]。研究还表明,Nv-NF-κB Ser对过氧化物和烷化剂等化合物具有更强的耐受性,这些化合物往往是在河口或者沼泽等地方含量较高,而在这种环境中分布了更多的Nv-NF-κB Ser型海葵。因此,这很可能是由环境因素导致了这两个类群的分化,体现了受适应性辐射影响的特点,同时这也是一种自然选择的结果。通过对RelA、RelB、c-Rel家族基因的进化速率的分析,它们表现出了非常相似的进化速度,RelA、RelB、c-Rel分别在0.77、0.79和1.01 PAU,其中RelB的进化速率稍快[16]。此外,如上所述,NF-κB家族成员之间可以通过相互作用形成同源二聚体和异源二聚体:c-Rel、RelA/p65、RelB、p50/p105和p52/p100,共同调节目的基因的表达[17],因此它们之间可能存在一种协同进化的关系[18]。研究表明,他们首次出现分化的证据是在果蝇()中被鉴定出了Dif和Dorsal基因,果蝇的这两个基因在同一条染色体的相邻位置,并且都具有Rel同源结构域和相似的基因结构,因此这两个基因很有可能是在进化过程中通过复制产生的[19]。Dif和Dorsal基因的产生,也伴随着新功能的获得,这体现在它们与另外一种家族成员Relish在免疫信号通路的调节上存在着差异。当行使免疫调节功能时,Dorsal和Dif被Cactus磷酸化之后,才能进行基因表达调控,而Relish的活化需要Ird5 (immune response-deficient 5, Ird5)蛋白将其C端的ANK结构域进行切割,从而释放Relish入核[20]。随后,进化至硬骨鱼阶段出现了成熟的RelA、RelB、c-Rel的基因分型[21]。在哺乳动物中,与RelA和c-Rel不同的是,RelB发挥激活转录调控功能还需要LZ (leucine zipper, LZ)结构的参与[22],这个结构域对于RelB与DNA序列的结合和蛋白质的相互作用是不可或缺的,而RelA和c-Rel并不具备这样的辅助结构域[23]。然而,有关于RelB中的LZ结构起源和演化的分子机制尚没有明确的报道。值得一提的是,NF-κB家族基因和MyD88 (myeloid differentiation primary response 88)基因在线虫()基因组中是缺失的,这提示线虫可能是通过其他转录因子和信号转导途径来行使免疫应答的[24,25]。哺乳动物的NF-κB家族中另外的两个成员NF-κB1和NF-κB2在C末端含有具有抑制性作用的ANK结构域。由于早在细菌中就发现了仅含有ANK结构域的蛋白(简称ANK蛋白)[26],表明脊椎动物NF-κB1和NF-κB2的进化是逐渐演变而来的。原始仅含有Rel结构域蛋白的NF-κB (简称Rel蛋白)和更为古老的ANK蛋白在NF-κB家族蛋白进化早期有可能发生了特异性的相互作用。这导致在单细胞原生动物中产生了极为简单的Rel-ANK融合型NF-κB蛋白[27]。随后,研究发现海葵形成了Rel蛋白和IκB蛋白,这可能是基因裂解或丢失C-末端ANK结构域的结果[28]。然而,目前还没有足够的序列信息来确定Rel蛋白的真正起源。
2 NFAT的起源与家族成员的分化
NFAT(nuclear factor of activated T cells)蛋白首次在T细胞中被发现[29,30]。NFAT家族有5位成员分别为NFAT1(NFATc2或NFATp)、NFAT2(NFATc1)、NFAT3(NFATc4)、NFAT4(NFATc3)和NFAT5[31]。NFAT家族蛋白包含两个TAD结构域,一个调节结构域(NFAT homology region,NHR),一个高度保守的Rel结构域和一个羧基末端结构域[32]。NFAT家族分为两类,分别为经典家族和非经典家族。NFAT蛋白的经典的家族成员包括:NFAT1、NFAT2、NFAT3和NFAT4,具有钙调蛋白结合位点,受Ca2+和钙调蛋白的调控[33,34]。大量研究表明,钙调神经磷酸酶与经典的NFAT蛋白结合并使其脱磷酸化,从而诱导其进入细胞核并发挥转录活性,非经典家族成员仅包括NFAT5,其并不需要钙调神经磷酸酶参与其活性[35]。NFAT5可以形成同源二聚体,其同源二聚对与DNA结合和转录活性至关重要[36]。
通过物种之间的NFAT基因同源性分析显示,在单细胞真核生物如酿酒酵母(),短毛啤酒酵母()和海绵()生物的基因组中未能发现NFAT的同源基因,而是在海葵基因组中存在一个与人NFAT5相似的NFAT基因[37],不过在线虫基因组中发生了缺失。在进化过程中,NFAT家族经过了多次基因的复制和功能的分化,在无脊椎动物和脊索动物(玻璃海鞘,文昌鱼)基因组中只含有一个NFAT同源基因,从无颌类脊椎动物的海七鳃鳗()开始,逐渐出现了更多的分化。随后在象鲨()的基因组中发现了4个旁系同源基因[38]。从鱼类如青鳉()、斑马鱼()、三刺鱼()、红鳍东方鲀()等开始[39~43],再到非洲爪蟾()和哺乳动物(大家鼠,家鼠)基因组中出现了5个完整的NFAT家族基因分型[44~47]。因此,海葵中的类NFAT5基因有可能是该家族的最原始成员。此外,Graef等[48]和Gauthier等[11]的研究显示,因为NFAT和NF-κB都含有Rel同源结构域,并在硬骨鱼NFAT的基因结构中发生了易位外显子和Rel结构域编码基因的重组,因此它们可能含有共同的祖先基因。然而,在线虫中NFAT和NF-κB都有所缺失,并且NFAT3在鸟类如原鸡()、火鸡()的基因组中也发生了丢失,而NFAT和NF-κB还表现出基因组结构多样化和功能分化的特征,因此NFAT和NF-κB的协同进化关系还需要进一步的确认[49,50]。为了研究NFAT家族基因的演化动力学特点,研究人员对这些基因构建了系统发育树。结果显示NFAT家族基因被分为3个类群:NFAT1群(包含NFAT1和NFAT2)、NFAT4群(包含NFAT3和NFAT4)和NFAT5,NFAT1群和NFAT4群仅存在于脊椎动物中。由此判断,在脊索动物出现之前,至少发生了一次基因复制,这次复制导致了NFAT5类群和NFAT1-4类群这个两个分群的产生。这次复制也使得NFAT5和NFAT1-4在结构域上产生了不同,经典的NFAT形成了钙调磷酸酶结合域,这个结构域调节它的入核,而NFAT5的核穿梭是由核输出序列(nuclear export sequence, NES)、核输出结构域(auxiliary export domain, AED)和核定位信号(NLS)共同介导的,这表示NFAT5和NFAT1-4功能上产生了差异[51]。PAML选择压力分析表明,许多NFAT家族基因分支显示了强烈的正向选择,在基因复制时加速了原始NFAT基因序列的突变,从而产生了新的分型[42]。因此,再结合全基因组复制(whole genome duplication,WGD)理论表明,完整的NFAT家族基因很有可能经历3轮全基因组复制事件[52,53]。首先,在第一次复制时产生了类NFAT5和类NFAT;随着第二次复制,类NFAT分化产生NFAT1和NFAT4;最后一次复制,NFAT1分化成了NFAT1和NFAT2,而NFAT4分化成了NFAT3和NFAT4[42]。
3 EBF的特殊结构与演化
EBF (early B-cell factor)转录因子家族,也被称为COE (collier (col) and olfactory-1/early-B-cell factor)转录因子,早期从果蝇和小鼠体内分离[54~56],具有指导多种神经元组织细胞分化的功能。哺乳动物中EBF家族成员包括EBF1、EBF2、EBF3和EBF4。在哺乳动物中EBF转录因子家族成员含有DBD结构域,IPT结构域、非经典HLH(helix-loop-helix)结构域和TAD结构域,这些结构域在进化上具有保守性[57]。其中在DBD结构域中,含有一个非典型的锌指(H-X3-C-X2-C-X5-C)基序,并且对于DNA结合起重要作用[57,58]。IPT可能通过同源二聚或异同源二聚作用参与EBF家族与其他蛋白质的结合[57]。此外,EBF家族的非经典HLH结构域,是由三个α-螺旋(H1-H2d-H2a)组成,其结构域在H1和H2之间的loop环中存在一个PXXPXXP基序,该基序在蛋白结合方面扮演重要角色[59]。
目前尚未发现EBF基因存在于真菌、植物或任何一个原生动物中。研究表明在无脊椎动物中,如海葵、线虫、果蝇、海鞘和文昌鱼中均发现了单一的EBF同源基因[55,60~63]。其中,通过对尾索动物和头索动物的EBF序列分析发现,这很有可能是脊椎动物EBF的祖先。而随着物种进化,在最原始的脊椎动物七鳃鳗中出现了EBF的同源基因的分型,分别将其命名为COE-A和COE-B,这两个同源基因在HLH结构域的核苷酸序列之间存在剪切差异[64]。进化至硬骨鱼阶段,在斑马鱼和蟾蜍()中都分化出了3个类型的EBF家族基因[65,66]。直到高等脊椎动物人和小鼠中,出现成熟的4个EBF旁系同源基因。而各物种中EBF基因的功能在某些方面也是存在差异性的,其中果蝇和线虫中EBF转录因子能够调节肽能神经元的发育,爪蟾和鸡的胚胎中的EBF转录因子具有促进神经元分化的功能,小鼠体内EBF转录因子在神经系统发育方面也扮演着不同的角色,EBF1对于面部肌肉迁移以及纹状体和视网膜神经元分化起着的关键作用,而EBF2则侧重于对小脑和嗅觉上皮神经元细胞的迁移和分化[67]。EBF转录因子DBD结构域中的锌指基序是一种非典型的基序,其所含有的H157、C161、C164和C170可以形成稳定的loop(H-X3-C-X2-C-X5-C)结构,与经典的锌指结构类似,具有识别DNA序列以及形成二聚体的作用[57]。晶体学结构分析显示,尽管EBF的DBD结构域与NF-κB和NFAT家族蛋白的Rel结构域的序列同源性不高,但是它们在三级结构上面却有着一定的相似性[59]。而不同之处在于,EBF的DBD结构域在空间上形成了不一样排列的β片层以及其独特的“伪Ig-like”折叠(即IPT结构域)[68]。除此之外,不像NF-κB和NFAT、EBF的DBD结构域并不与DNA结合,而是通过IPT结构域参与DNA的结合、形成二聚体和蛋白质-蛋白质的相互作用[69]。因此,EBF虽然在序列上存在差异,但是为了适应结合DNA等功能,从而完成基因的转录调控,在空间结构的折叠上面表现出了趋同进化的特点[68]。哺乳动物的EBF蛋白质中存在的3个α-螺旋重复序列(H1、H2d和H2a),其中每个序列都由单独的外显子(外显子E11、E12和E13,分别由内含子i11和i12分隔)编码[57]。通过分析了脊索动物之外的物种如玻璃海鞘、文昌鱼和紫海胆()等H1和H2a外显子序列,都证明它们的基因中不存在与H2d相关的编码序列。相反,通过对有颌类基因组序列分析发现,包括EBF1-3或EBF1-4,都发生了H2a-H2d的复制。值得一提的是七鳃鳗COE-B 蛋白含有H2d α-螺旋,而COE-A蛋白却没有,因此结合上述研究表明,H2a-H2d的复制在进化上非常有可能早于有颌类脊椎动物[64]。脊椎动物EBF蛋白中的H2d和H2a序列比对显示,H2a-H2d并不是由外显子E12和E13简单复制所产生的。因此,研究人员对此提出了“two-step”进化模型的假设,认为H2d的产生机制有可能是通过两步完成的(图2)。首先,脊椎动物祖先外显子E13复制之后,通过选择性剪切整合到mRNA中,形成过渡性序列。由于E13复制后的插入序列导致E12外显子下游内含子相位发生改变,这样便影响了下游序列的翻译,因此序列又经过一次剪切,使E12和E13之间的内含子恢复0相位,形成“对称”的外显子(E11-E12-E13),从而顺利完成H1、H2d和H2a的编码[70]。
4 RBP家族蛋白的起源与进化
RBP (recombination signal binding protein)家族蛋白对于神经系统的发育和造血系统的分化具有至关重要的作用[71],并且对于神经细胞分化的作用在生物进化过程中是保守的[72~75]。后生动物RBP蛋白有两种亚型,一种是RBP-Jκ (recombination signal binding protein for immunoglobulin kappa J region),其在Notch信号通路中发挥重要作用;另一种是RBP-L (recombination signal binding protein for immunoglobulin kappa J region like),其功能并不依赖于Notch信号通路[76]。RBP蛋白由3个结构域组成:N端结构域(N-terminal domain, NTD)、β折叠结构域(β-trefoil domain, BTD)和C端结构域(C-terminal domain, CTD)[77]。经过对其序列分析和结构研究证实,RBP的NTD与NF-κB转录因子家族的Rel同源结构域的N端具有相似性[78]。并且NTD以单体形式通过识别特异性序列(C/T)GTGGGAA与DNA结合,行使类似于Rel同源结构域的功能[79]。BTD结构域可以促进这种和DNA的结合,而且还能够参与蛋白质与蛋白质的相互作用,然而CTD并不与DNA发生相互作用[77,78]。除此之外,RBP分别通过与辅助激活蛋白和辅助抑制蛋白的不同相互作用,作为转录的激活因子和抑制因子发挥双重作用[80]。
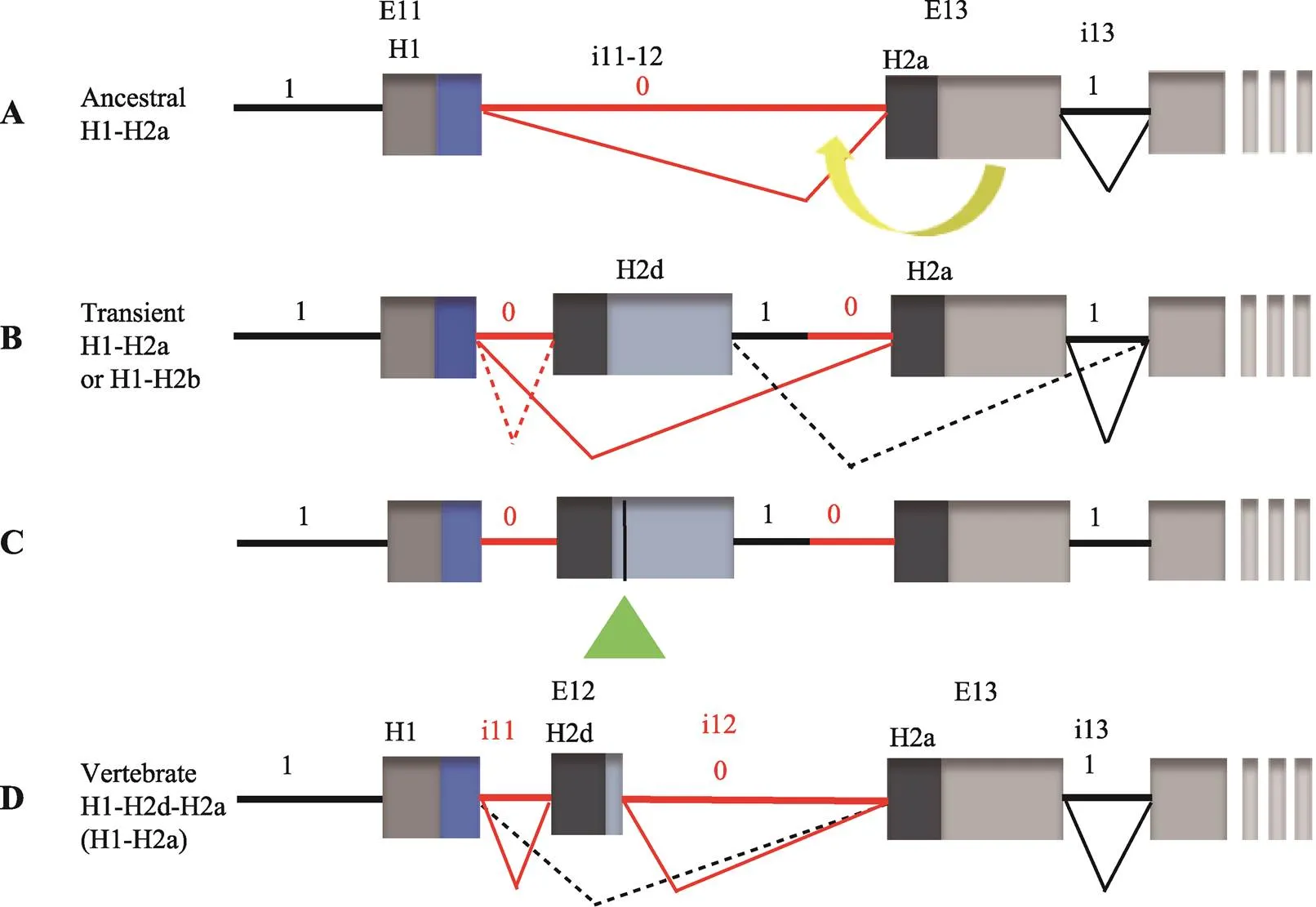
图2 脊椎动物EBF蛋白中H2复制的机制
A:EBF祖先基因中H1H2a序列的外显子–内含子的结构。0相位内含子(红色横线),1相位内含子(黑色横线)。B:H2复制的第一步是复制编码H2a的外显子。由于相位剪切规则,通过复制和选择性剪切产生了H2d的祖先基因。C:第二步是激活H2d编码区下游的隐蔽3′剪切位点(浅绿色三角)。D:产生了相同相位的i11和i12内含子,并将成熟的H2d嵌入EBF蛋白。根据文献[70]修改绘制。
通过对所有真核生物基因组中RBP的信息分析和探究,在植物和原生动物中RBP家族基因出现了缺失,但在部分真菌中发现了RBP家族同源基因,从简单的单细胞裂殖酵母()到多细胞和高度分化的灰霉菌()都有RBP家族基因的分布。由此表明后生动物和真菌中的RBP基因很有可能起源于一个共同祖先[81,82]。Převorovský等[76]对真菌基因结构分析发现,RBP祖先基因是在Rel结构域的基因序列之间插入了编码BTD结构域的DNA序列而形成的,即RBP基因是从Rel类型的转录因子中创建而来。随后,在真菌中发生了复制事件,产生了两类RBP基因,一类RBP基因具有真菌特异性,其在进化到后生动物阶段发生了丢失,因此只存在于真菌中。另一类与后生动物RBP基因存在更多的相似性,所以其有可能是后生动物RBP基因的祖先。后生动物的RBP基因也发生了明显的复制和扩增,目前的研究显示,这种复制发生于硬骨鱼阶段,并产生了RBP-L型基因和广泛存在于脊椎动物和无脊椎动物中的RBP-Jκ型基因的分化。然而随着生物进化的发展,在两栖类动物中至今还没有RBP-L型同源基因的报道,因此两栖类动物很有可能仅使用RBP-Jκ同源基因来调控体内所有RBP相关的信号通路。RBP两个家族成员RBP-Jκ型和RBP-L型之间具有相似,两者都参与转录调控以及结合DNA序列,但不同之处在于它们的相互作用的分子不同,以及它们对各种信号的反应性存在差异[83,84]。
5 结语与展望
转录因子是一类DNA结合蛋白,可以增强或抑制基因的转录,在细胞内的蛋白翻译,基因调控和组织分化等方面都发挥着重要的调节作用。根据RHR超家族的成员在整个生物进化过程中的分布可以看出,它们是以一种渐变模式进行演化的(图3)。其中,NF-κB和RBP家族是较为古老的两个成员,而随后便出现了NFAT和EBF家族。首先在低等的原生动物中发现了具有简单结构域ANK的NF-κB家族成员,这就确立了NF-κB作为古老家族成员的进化地位。但目前仍然没有足够的证据表明Rel蛋白是如何起源的。紧随其后在真菌生物中出现了RBP家族成员,并且进化至硬骨鱼阶段复制产生了两个旁系同源基因,不过对于RBP家族成员最早起源于哪一种物种仍有待于继续研究。这四个家族的成员同时出现,最早可以追溯到在腔肠动物阶段,此时NF-κB家族分化出了成熟的Rel蛋白,其可能是基因裂解或丢失C-末端ANK结构域而产生的,我们通过NCBI数据库进行检索分析发现可能在这一阶段分化出了最早含有CTD结构域的RBP家族成员。到了节肢动物阶段,NF-κB家族极有可能通过基因复制产生了Dif和Dorsal两个旁系同源基因。进化至脊索动物阶段,NFAT家族和EBF家族仍然只含有一个成员。随后在硬骨鱼阶段除了EBF家族,其他家族蛋白都形成了成熟的分型。直到高等脊椎动物阶段,EBF家族才出现了完整的家族成员分化,由此判断EBF家族在进化上相对保守。本文总结和归纳了RHR超家族成员的起源,进化方式和功能,为进一步揭示整个转录因子家族的演化机制,动力以及与物种之间的进化关系奠定基础。然而,目前对于某些家族成员的起源与分化,以及相应基序的生物学功能依然是不确定的。相信随着研究不断的拓展与深入,将会对这些问题有更加清晰的认识和理解。此外,通过生物信息学,遗传学和分子生物学对转录因子家族在不同物种中的进化机制和活性的比较研究,也会对未来转录因子相关药物研发以及临床治疗提供新的思路。
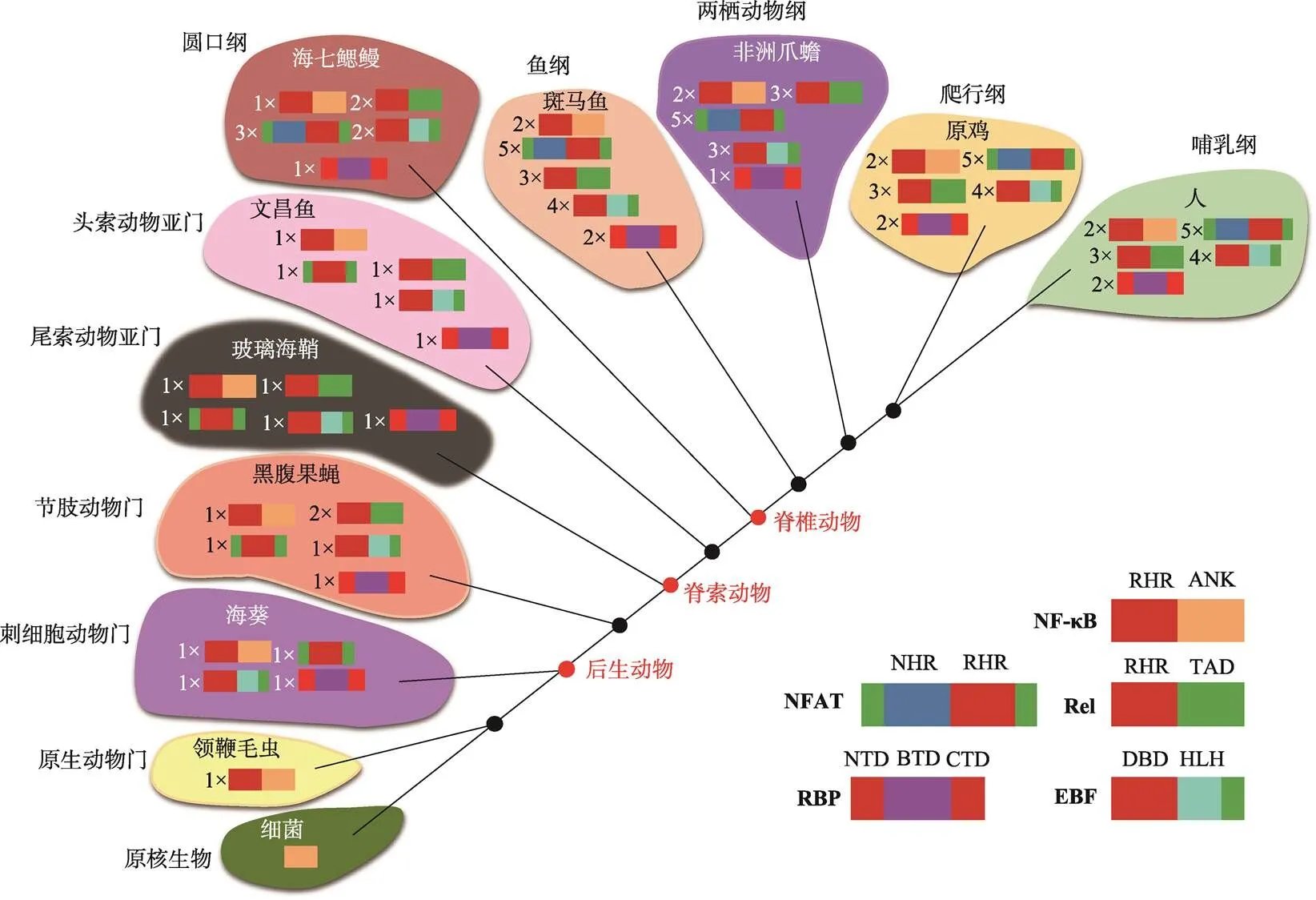
图3 RHR转录因子家族蛋白的进化与分化
[1] Lambert SA, Jolma A, Campitelli LF, Das PK, Yin YM, Albu M, Chen XT, Taipale J, Hughes TR, Weirauch MT. The human transcription factors., 2018, 175(2): 598–599.
[2] Wingender E, Schoeps T, Haubrock M, Krull M, Dönitz J. TFClass: expanding the classification of human transcription factors to their mammalian orthologs., 2018, 46(D1): D343–D347.
[3] Wang JL, Wang J, Tian CY. Evolution of KRAB- containing zinc finger proteins and their roles in species evolution., 2016, 38(11): 971–978.王进龙, 王建, 田春艳. KRAB型锌指蛋白的进化及在物种演化中的功能. 遗传, 2016, 38(11): 971–978.
[4] Laity JH, Lee BM, Wright PE. Zinc finger proteins: new insights into structural and functional diversity., 2001, 11(1): 39–46.
[5] Berisio R, Ciccarelli L, Squeglia F, De Simone A, Vitagliano L. Structural and dynamic properties of incomplete immunoglobulin-like fold domains., 2012, 19(10): 1045–1053.
[6] Sen R, Baltimore D. Multiple nuclear factors interact with the immunoglobulin enhancer sequences., 1986, 46(5): 705–716.
[7] Su P, Feng SS, Li QW. Research progress of the structure and function of NF-κB and IκB in different animal groups., 2016, 38(6): 523–531.苏鹏, 冯少姝, 李庆伟. NF-кB和IκB在不同动物类群中的结构及功能研究进展. 遗传, 2016, 38(6): 523–531.
[8] Ghosh S, Hayden MS. New regulators of NF-kappa B in inflammation., 2008, 8(11): 837–848.
[9] Bortolotto V, Cuccurazzu B, Canonico P L, Grilli M. NF-κB mediated regulation of adult hippocampal neurogenesis: relevance to mood disorders and antidepressant activity., 2014, 612798.
[10] Al-Khodor S, Price CT, Kalia A, Abu Kwaik Y. Functional diversity of ankyrin repeats in microbial proteins., 2010, 18(3): 132–139.
[11] Gauthier M, Degnan BM. The transcription factor NF- kappaB in the demosponge: insights on the evolutionary origin of the Rel homology domain., 2008, 218(1): 23–32.
[12] Zhang Q, Lenardo MJ, Baltimore D. 30 years of NF-κB: a blossoming of relevance to human pathobiology., 2017, 168(1–2): 37–57.
[13] Ghosh S, May MJ, Kopp EB. NF-kappaB and Rel proteins: evolutionarily conserved mediators of immune responses., 1998, 16: 225–260.
[14] Wang TT, Sun YX, Jin LJ, Thacker P, Li SY, Xu YP. Aj-rel and Aj-p105, two evolutionary conserved NF-κB homologues in sea cucumber (and their involvement in LPS induced immunity., 2013, 34(1): 17–22.
[15] Sullivan JC, Wolenski FS, Reitzel AM, French CE, Traylor-Knowles N, Gilmore TD, Finnerty JR. Two alleles of NF-kappaB in the sea anemoneare widely dispersed in nature and encode proteins with distinct activities., 2009, 4(10): e7311.
[16] Huguet C, Crepieux P, Laudet V. Rel/NF-kappaB transcription factors and IkappaB inhibitors: evolution from a unique common ancestor., 1997, 15(24): 2965–2974.
[17] Siggers T, Chang AB, Teixeira A, Wong D, Williams KJ, Ahmed B, Ragoussis J, Udalova IA, Smale ST, Bulyk ML. Principles of dimer-specific gene regulation revealed by a comprehensive characterization of NF-κB family DNA binding., 2011, 13(1): 95–102.
[18] De Juan D, Pazos F, Valencia A. Emerging methods in protein co-evolution., 2013, 14(4): 249– 261.
[19] Meng X, Khanuja BS, Ip YT. Toll receptor-mediatedimmune response requires Dif, an NF-kappaB factor., 1999, 13(7): 792–797.
[20] Gilmore TD, Wolenski FS. NF-κB: where did it come from and why?, 2012, 246(1): 14–35.
[21] Woods IG, Wilson C, Friedlander B, Chang P, Reyes DK, Nix R, Kelly PD, Chu F, Postlethwait JH, Talbot WS. Thegene map defines ancestral vertebrate chromosomes., 2005, 15(9): 1307–1314.
[22] Kabacaoglu D, Ruess DA, Ai J, Algül H. NF-κB/Rel transcription factors in pancreatic cancer: focusing on RelA, c-Rel, and RelB., 2019, 11(7):937.
[23] Millet P, McCall C, Yoza B. RelB: an oultlier in leukocyte biology., 2013, 94(5): 941–951.
[24] Irazoqui JE, Urbach JM, Ausubel FM. Evolution of host innate defence: insights fromand primitive invertebrates., 2010, 10(1): 47–58.
[25] Kuo CJ, Hansen M, Troemel E. Autophagy and innate immunity: insights from invertebrate model organisms., 2018, 14(2): 233–242.
[26] Li J, Mahajan A, Tsai MD. Ankyrin repeat: a unique motif mediating protein-protein interactions., 2006, 45(51): 15168–15178.
[27] Sebé-Pedrós A, de Mendoza A, Lang BF, Degnan BM, Ruiz-Trillo I. Unexpected repertoire of metazoan transcription factors in the unicellular holozoan., 2011, 28(3): 1241–1254.
[28] Wolenski FS, Garbati MR, Lubinski TJ, Traylor-Knowles N, Dresselhaus E, Stefanik DJ, Goucher H, Finnerty JR, Gilmore TD. Characterization of the core elements of the NF-κB signaling pathway of the sea anemone., 2011, 31(5): 1076–1087.
[29] Shaw JP, Utz PJ, Durand DB, Toole JJ, Emmel EA, Crabtree GR. Identification of a putative regulator of early T cell activation genes., 1988, 241(4862): 202– 205.
[30] Hogan PG. Calcium-NFAT transcriptional signalling in T cell activation and T cell exhaustion., 2017, 63: 66–69.
[31] Müller MR, Rao A. NFAT, immunity and cancer: a transcription factor comes of age., 2010, 10(9): 645–656.
[32] Lee JU, Kim LK, Choi JM. Revisiting the concept of targeting NFAT to control T cell immunity and autoimmune diseases., 2018, 9: 2747.
[33] Macian F. NFAT proteins: key regulators of T-cell development and function., 2005, 5(6): 472–484.
[34] Hogan PG, Chen L, Nardone J, Rao A. Transcriptional regulation by calcium, calcineurin, and NFAT., 2003, 17(18): 2205–2232.
[35] Feske S, Okamura H, Hogan PG, Rao A. Ca2+/calcineurin signalling in cells of the immune system., 2003, 311(4): 1117–1132.
[36] Aramburu J, López-Rodríguez C. Regulation of inflammatory functions of macrophages and T lymphocytes by NFAT5., 2019, 10: 535.
[37] Sullivan JC, Kalaitzidis D, Gilmore TD, Finnerty JR. Rel homology domain-containing transcription factors in the cnidarian., 2007, 217(1): 63–72.
[38] Venkatesh B, Lee AP, Ravi V, Maurya AK, Lian MM, Swann JB, Ohta Y, Flajnik MF, Sutoh Y, Kasahara M, Hoon S, Gangu V, Roy SW, Irimia M, Korzh V, Kondrychyn I, Lim ZW, Tay BH, Tohari S, Kong KW, Ho S, Lorente-Galdos B, Quilez J, Marques-Bonet T, Raney BJ, Ingham PW, Tay A, Hillier LW, Minx P, Boehm T, Wilson RK, Brenner S, Warren WC. Author correction:genome provides unique insights into gnathostome evolution., 2014, 505(7482): 174– 179.
[39] Kasahara M, Naruse K, Sasaki S, Nakatani Y, Qu W, Ahsan B, Yamada T, Nagayasu Y, Doi K, Kasai Y, Jindo T, Kobayashi D, Shimada A, Toyoda A, Kuroki Y, Fujiyama A, Sasaki T, Shimizu A, Asakawa S, Shimizu N, Hashimoto S, Yang J, Lee Y, Matsushima K, Sugano S, Sakaizumi M, Narita T, Ohishi K, Haga S, Ohta F, Nomoto H, Nogata K, Morishita T, Endo T, Shin-I T, Takeda H, Morishita S, Kohara Y. Thedraft genome and insights into vertebrate genome evolution., 2007, 447(7145): 714–719.
[40] Li W, Zheng NZ, Yuan Q, Xu K, Yang F, Gu L, Zheng GY, Luo GJ, Fan C, Ji GJ, Zhang B, Cao H, Tian XL. Erratum to: NFAT5-mediated CACNA1C expression is critical for cardiac electrophysiological development and maturation., 2016, 94(9): 1003–1004.
[41] Huang XD, Wei GJ, Zhang H, He MX. Nuclear factor of activated T cells (NFAT) in pearl oyster: molecular cloning and functional characterization., 2015, 42(1): 108–113.
[42] Song X, Hu J, Jin P, Chen L, Ma F. Identification and evolution of an NFAT gene involvinginnate immunity., 2013, 102(4): 355– 362.
[43] Kai W, Kikuchi K, Tohari S, Chew AK, Tay A, Fujiwara A, Hosoya S, Suetake H, Naruse K, Brenner S, Suzuki Y, Venkatesh B. Integration of the genetic map and genome assembly of fugu facilitates insights into distinct features of genome evolution in teleosts and mammals., 2011, 3: 424–442.
[44] Session AM, Uno Y, Kwon T, Chapman JA, Toyoda A, Takahashi S, Fukui A, Hikosaka A, Suzuki A, Kondo M, van Heeringen SJ, Quigley I, Heinz S, Ogino H, Ochi H, Hellsten U, Lyons JB, Simakov O, Putnam N, Stites J, Kuroki Y, Tanaka T, Michiue T, Watanabe M, Bogdanovic O, Lister R, Georgiou G, Paranjpe SS, van Kruijsbergen I, Shu S, Carlson J, Kinoshita T, Ohta Y, Mawaribuchi S, Jenkins J, Grimwood J, Schmutz J, Mitros T, Mozaffari SV, Suzuki Y, Haramoto Y, Yamamoto TS, Takagi C, Heald R, Miller K, Haudenschild C, Kitzman J, Nakayama T, Izutsu Y, Robert J, Fortriede J, Burns K, Lotay V, Karimi K, Yasuoka Y, Dichmann DS, Flajnik MF, Houston DW, Shendure J, DuPasquier L, Vize PD, Zorn AM, Ito M, Marcotte EM, Wallingford JB, Ito Y, Asashima M, Ueno N, Matsuda Y, Veenstra GJ, Fujiyama A, Harland RM, Taira M, Rokhsar DS. Genome evolution in the allotetraploid frog., 2016, 538(7625): 336–343.
[45] Vihma H, Pruunsild P, Timmusk T. Alternative splicing and expression of human and mouse NFAT genes., 2008, 92(5): 279–291.
[46] Vechetti IJ Jr, Aguiar AF, de Souza RW, Almeida FL, de Almeida Dias HB, de Aguiar Silva MA, Carani FR, Ferraresso RL, Carvalho RF, Dal-Pai-Silva M. NFAT isoforms regulate muscle fiber type transition without altering CaN during aerobic training., 2013, 34(10): 861–867.
[47] Ueno M, Shen WJ, Patel S, Greenberg AS, Azhar S, Kraemer FB. Fat-specific protein 27 modulates nuclear factor of activated T cells 5 and the cellular response to stress., 2013, 54(3): 734–743.
[48] Graef IA, Gastier JM, Francke U, Crabtree GR. Evolutionary relationships among Rel domains indicate functional diversification by recombination., 2001, 98(10): 5740–5745.
[49] International Chicken Genome Sequencing Consortium. Sequence and comparative analysis of the chicken genome provide unique perspectives on vertebrate evolution., 2004, 432(7018): 695–716.
[50] Dalloul RA, Long JA, Zimin AV, Aslam L, Beal K, Blomberg Le Ann, Bouffard P, Burt DW, Crasta O, Crooijmans RP, Cooper K, Coulombe RA, De S, Delany ME, Dodgson JB, Dong JJ, Evans C, Frederickson KM, Flicek P, Florea L, Folkerts O, Groenen MA, Harkins TT, Herrero J, Hoffmann S, Megens HJ, Jiang A, de Jong P, Kaiser P, Kim H, Kim KW, Kim S, Langenberger D, Lee MK, Lee T, Mane S, Marcais G, Marz M, McElroy AP, Modise T, Nefedov M, Notredame C, Paton IR, Payne WS, Pertea G, Prickett D, Puiu D, Qioa D, Raineri E, Ruffier M, Salzberg SL, Schatz MC, Scheuring C, Schmidt CJ, Schroeder S, Searle SM, Smith EJ, Smith J, Sonstegard TS, Stadler PF, Tafer H, Tu ZJ, Van Tassell CP, Vilella AJ, Williams KP, Yorke JA, Zhang L, Zhang HB, Zhang X, Zhang Y, Reed KM. Multi-platform next-generation sequencing of the domestic turkey (): genome assembly and analysis., 2010, 8(9): e1000475.
[51] Lee N, Kim D, Kim WU. Role of NFAT5 in the immune system and pathogenesis of autoimmune diseases., 2019, 10: 270.
[52] Meyer A, Van de Peer Y. From 2R to 3R: evidence for a fish-specific genome duplication (FSGD)., 2005, 27(9): 937–945.
[53] Hoegg S, Meyer A. Hox clusters as models for vertebrate genome evolution., 2005, 21(8):421–424.
[54] Hagman J, Belanger C, Travis A, Turck CW, Grosschedl R. Cloning and functional characterization of early B-cell factor, a regulator of lymphocyte-specific gene expression., 1993, 7(5): 760–773.
[55] de Taffin M, Carrier Y, Dubois L, Bataillé L, Painset A, Le Gras S, Jost B, Crozatier M, Vincent A. Genome-wide mapping of collierbinding sites highlights its hierarchical position in different transcription regulatory networks., 2015, 10(7): e0133387.
[56] Wang MM, Reed RR. Molecular cloning of the olfactory neuronal transcription factor Olf-1 by genetic selection in yeast., 1993, 364(6433): 121–126.
[57] Liao D. Emerging roles of the EBF family of transcription factors in tumor suppression., 2009, 7(12): 1893–1901.
[58] Fields S, Ternyak K, Gao H, Ostraat R, Akerlund J, Hagman J. The 'zinc knuckle' motif of early B cell factor is required for transcriptional activation of B cell-specific genes., 2008, 45(14): 3786–3796.
[59] Siponen MI, Wisniewska M, Lehtiö L, Johansson I, Svensson L, Raszewski G, Nilsson L, Sigvardsson M, Berglund H. Structural determination of functional domains in early B-cell factor (EBF) family of transcription factors reveals similarities to Rel DNA-binding proteins and a novel dimerization motif., 2010, 285(34): 25875–25879.
[60] Pang K, Matus DQ, Martindale MQ. The ancestral role of COE genes may have been in chemoreception: evidence from the development of the sea anemone,(Phylum Cnidaria; Class Anthozoa)., 2004, 214(3): 134–138.
[61] Stolfi A, Gainous TB, Young JJ, Mori A, Levine M, Christiaen L. Early chordate origins of the vertebrate second heart field., 2010, 329(5991): 565–568.
[62] Kim K, Colosimo ME, Yeung H, Sengupta P. The UNC-3 Olf/EBF protein represses alternate neuronal programs to specify chemosensory neuron identity., 2005, 286(1): 136–148.
[63] Mazet F, Masood S, Luke GN, Holland ND, Shimeld SM. Expression ofan amphioxus COE/EBF gene, in the developing central nervous system and epidermal sensory neurons., 2004, 38(2): 58–65.
[64] Lara-Ramírez R, Poncelet G, Patthey C, Shimeld SM. The structure, splicing, synteny and expression ofCOE genes and the evolution of the COE gene family in chordates., 2017, 227(5): 319–338.
[65] Wang Y, Chen K, Yao Q, Zheng X, Yang Z. Phylogenetic analysis ofbasic helix-loop-helix transcription factors., 2009, 68(6): 629–640.
[66] Pozzoli O, Bosetti A, Croci L, Consalez GG, Vetter ML. Xebf3 is a regulator of neuronal differentiation during primary neurogenesis in., 2001, 233(2): 495–512.
[67] Catela C, Correa E, Wen K, Aburas J, Croci L, Consalez GG, Kratsios P. An ancient role for collier/Olf/Ebf (COE)-type transcription factors in axial motor neuron development., 2019, 14(1): 2.
[68] Treiber N, Treiber T, Zocher G, Grosschedl R. Structure of an EBF1:DNA complex reveals unusual DNA recognition and structural homology with Rel proteins., 2010, 24(20): 2270–2275.
[69] Boller S, Grosschedl R. The regulatory network of B-cell differentiation: a focused view of early B-cell factor 1 function., 2014, 261(1): 102–115.
[70] Daburon V, Mella S, Plouhinec JL, Mazan S, Crozatier M, Vincent A. The metazoan history of the COE transcription factors. Selection of a variant HLH motif by mandatory inclusion of a duplicated exon in vertebrates., 2008, 8: 131.
[71] Pursglove SE, Mackay JP. CSL: a notch above the rest., 2005, 37(12): 2472–2477.
[72] Maier D. The evolution of transcriptional repressors in the notch signaling pathway: a computational analysis., 2019, 156(1): 5.
[73] Sieger D, Tautz D, Gajewski M. The role of suppressor of hairless in notch mediated signalling duringsomitogenesis., 2003, 120(9): 1083–1094.
[74] Ito M, Katada T, Miyatani S, Kinoshita T. XSu(H)2 is an essential factor for gene expression and morphogenesis of thegastrula embryo., 2007, 51(1): 27–36.
[75] Toritsuka M, Kimoto S, Muraki K, Kitagawa M, Kishimoto T, Sawa A, Tanigaki K. Regulation of striatal dopamine responsiveness by Notch/RBP-J signaling., 2017, 7(3): e1049.
[76] Prevorovský M, Půta F, Folk P. Fungal CSL transcription factors., 2007, 8: 233.
[77] Wilson JJ, Kovall RA. Crystal structure of the CSL-notch- mastermind ternary complex bound to DNA., 2006, 124(5): 985–996.
[78] Hall DP, Kovall RA. Structurally conserved binding motifs of transcriptional regulators to notch nuclear effector CSL.(), 2019, 244(17): 1520–1529.
[79] Oakley F, Mann J, Ruddell RG, Pickford J, Weinmaster G, Mann DA. Basal expression of IkappaBalpha is controlled by the mammalian transcriptional repressor RBP-J (CBF1) and its activator notch1., 2003, 278(27): 24359–24370.
[80] Collins KJ, Yuan Z, Kovall RA. Structure and function of the CSL-KyoT2 corepressor complex: a negative regulator of notch signaling., 2014, 22(1): 70–81.
[81] Hedges SB. The origin and evolution of model organisms., 2002, 3(11): 838–849.
[82] Ciccarelli FD, Doerks T, von Mering C, Creevey CJ, Snel B, Bork P. Toward automatic reconstruction of a highly resolved tree of life., 2006, 311(5765): 1283– 1287.
[83] Beres TM, Masui T, Swift GH, Shi L, Henke RM, MacDonald RJ. PTF1 is an organ-specific and notch- independent basic helix-loop-helix complex containing the mammalian suppressor of hairless (RBP-J) or its paralogue, RBP-L., 2006, 26(1): 117–130.
[84] Minoguchi S, Taniguchi Y, Kato H, Okazaki T, Strobl LJ, Zimber-Strobl U, Bornkamm GW, Honjo T. RBP-L, a transcription factor related to RBP-Jkappa., 1997, 17(5): 2679–2687.
Progress on the origin, function and evolutionary mechanism of RHR transcription factor family
Menggang Lv1,2, Aijia Liu1,2, Qingwei Li1,2, Peng Su1,2
Transcription factors are a class of proteins that regulate gene transcription and expression by binding to gene-specific sequences and play an essential role in regulating the biological activities of cells. The RHR (Rel-homology region) transcription factor family is the primary member of the IF (immunoglobulin fold) transcription factor superfamily, whose members contain the conserved Rel domain and IPT (immunoglobulin-like fold) domain. As an ancient transcription factor family, the RHR family continues differentiation on gene gain and loss through gene duplication, mutation, and silencing, accompanied with the evolution of diverse species. Natural selection has led to different rates of evolution among members of the family, and some domains of the protein family have shown unique mechanisms of evolution. However, the current reviews about the origin and differentiation of RHR family are rare. In this review, we summarize the research results on the distribution, classification, function, and evolution of the members of the RHR familyin order to provide a reference and new idea for studying the evolution mechanism of the whole transcription factor family and the evolutionary relationship among species.
transcription factors; RHR family; evolution; gene duplication; homologous gene
2020-11-25;
2021-01-18
国家自然科学基金项目(编号:31801973,31772884),大连市支持高层次人才创新创业项目(编号:2019RQ126),国家重大基础研究发展规划(973)计划课题(编号:2013CB835304),全国海洋公益项目(编号:201305016),辽宁省海洋与渔业厅科研项目(编号:201805)和大连市科技创新基金项目(编号:2018J12SN079)资助[Supported by the National Natural Science Foundation of China (Nos. 31801973, 31772884), Dalian Supports Innovation and Entrepreneurship Projects of High Level Talents(No. 2019RQ126), the Chinese Major State Basic Research Development Program (973) (No. 2013CB835304), the Marine Public Welfare Project of the State Oceanic Administration (No. 201305016), the project of Department of Ocean and Fisheries of Liaoning Province (No. 201805), and the Science and Technology Innovation Fund Research Project (No. 2018J12SN079)]
吕孟冈,在读硕士研究生,研究方向:微生物。E-mail: lvmenggang2020lr@163.com
苏鹏,博士,讲师,专业方向:细胞生物学。E-mail: sp4046@163.com
李庆伟,博士,教授,研究方向:细胞遗传学。E-mail: liqw@263.net
10.16288/j.yczz.20-332
2021/2/2 11:16:45
URI: https://kns.cnki.net/kcms/detail/11.1913.r.20210201.1110.006.html
(责任编委: 吕雪梅)

