从0到1:异源四倍体野生稻从头驯化创造全新作物
许操
热点追踪
从0到1:异源四倍体野生稻从头驯化创造全新作物
许操
中国科学院遗传与发育生物学研究所,植物基因组学国家重点实验室,种子创新研究院,北京 100101
2021年2月3日,杂志在线发表了李家洋院士团队关于四倍体野生稻从头驯化的重大突破性进展。该团队绘制了四倍体野生稻从头驯化路线图,与合作者经过多年攻关,突破了一系列限制多倍体野生稻驯化的理论难题和技术瓶颈,综合运用多维组学和基因编辑技术实现了野生四倍体水稻的从头驯化,创造了世界首例重新设计与快速驯化的四倍体水稻,将过去的“不可能”变成了现在的“一切皆有可能”。该研究突破了现有二倍体水稻作物的育种局限,实践了从0到1的突破性创新,为遗传背景复杂的野生植物重新驯化提供了范例和解决方案,将引领未来作物创造,翻开生物育种新篇章。
驯化是一个复杂的进化历程,是一种生物的繁衍逐渐受到另一种生物的影响与控制的过程[1]。农作物的栽培与人工驯化是农业文明起源的重要标志。将野生植物驯化成农作物是人类驯化活动的主要目标之一。在漫长的驯化选择历程中,植物逐渐失去其野生祖先种的遗传和生理特征,形成符合人类阶段性需求的栽培品种[2]。驯化和改良引发的农作物品种更新换代往往与人类文明的发展历程同步。20世纪40年代起,随着农业机械化和化学合成技术等工业文明成果的日臻成熟和推广应用,作物品种也随着第一次绿色革命的兴起而实现更新换代,这使得以大规模、单一品种的机械化种植,化肥及杀虫剂、除草剂等在农业生产中大量施用为特征的农业模式替代了传统的多样化品种的小规模农耕模式。这一方面提高了全球粮食产量,在一定程度上缓解了全球饥饿问题;另一方面深刻地改变了传统的农业生产模式。高产品种的单一化、同质化种植使得不同地区的特色品种逐渐消失,削弱了农业生态系统的多样性,降低了对病虫害的耐受力,农药的过度施用则又提高了病虫害的耐药性,同时也对土壤、水系造成污染[3,4]。随着世界人口快速增长和全球气候变化以及作物遗传多样性持续降低,作物改良和粮食增产遭遇瓶颈,人类粮食安全形势异常严峻。据世界粮农组织(Food and Agriculture Organization of the United Nations, FAO)最新统计[5],目前全球共有20亿粮食不安全人口,约占全球总人口的26%,其中10.3亿生活在亚洲,6.75亿在非洲。更为严峻的是,全球饥饿人口还在不断增加。2019年FAO最新统计表明[5],新冠 (COVID-19)疫情大流行发生之前,全球近6.9亿人处于饥饿状态,相当于全球人口的8.9%。自2014年以来遭受饥饿的人数新增了6000万。如果这一趋势持续下去,饥饿人口到2030年将突破8.4亿。根据最新全球经济展望所做的初步预测[5],仅COVID-19疫情可能导致2020年饥饿人口新增8300万至1.32亿,具体取决于经济增长前景。因此,为保障粮食安全和可持续发展,人类亟需创新作物育种策略,创造能智能响应环境变化,满足现代及未来社会数字化、信息化、智能化生产方式和生活方式的全新作物[6]。
我国人口多、耕地少、农产品进口依存度高,粮食安全成为关乎国计民生的头等大事,突破现有作物改良局限和技术瓶颈,创造符合我国国情、农情的全新作物,引领种子精准设计与未来作物创造,对保障我国粮食安全具有重要的战略意义[7]。近年来,我国科学家在主要农作物基础科学研究、作物改良、品种推广等方面取得重要成就,为保障粮食安全做出突出贡献。以水稻为例,我国科学家在理想株型、营养高效、优质高产、抗病稳产等领域取得一大批重要科研成果,并培育了一系列优异的水稻品种,为保障粮食安全作出重要贡献。随着研究的深入,人们对水稻产量、品质等性状的认识越来越清楚,但耐受极端气候、耐盐碱、耐旱涝等环境适应性性状,以及抵抗复杂病菌、虫害等生物适应性性状更为复杂,对其分子机理的研究进展缓慢,耐逆育种也因此遭遇瓶颈,现有品种难以满足未来农业生产和应对气候变化的需求。与此形成鲜明对比的是,大量优异的野生稻种质资源未被充分利用,尤其是异源多倍体野生稻,它们的环境适应性和生物适应性优异,具有巨大的驯化潜力和开发空间。可能原因主要包括如下几个方面:(1)野生稻受株型、花期、育性等性状影响,难以在实验室条件下驯养和表型观察;(2)野生稻的染色体倍性、杂合度、稳定性等特点,使其基因组高度复杂,遗传分析、基因注释和信息解码极具挑战;(3)野生稻受愈伤诱导、器官再生、农杆菌侵染等因素制约,难以遗传转化,无法使用基因工程、基因组编辑等现代生物学技术对其挖掘利用。如果能突破上述挑战,在保持野生多倍体水稻优异耐逆性状的前提下,导入研究相对较为清楚的产量和品质性状,将有可能实现水稻的从头驯化(domestication),突破现有的二倍体水稻的局限和育种瓶颈,创造全新的环境智能型多倍体水稻,从而有望引发新一轮绿色革命。
中国科学院遗传与发育生物学研究所李家洋院士团队于2021年2月3日在杂志报道了他们关于四倍体野生稻从头驯化的重大突破性研究成果[8]。该团队绘制了四倍体野生稻从头驯化路线图,与合作者经过多年攻关,突破了一系列限制多倍体野生稻驯化的理论难题和技术瓶颈,综合运用多维组学和基因编辑技术实现了野生四倍体水稻的从头驯化,创造了世界首例重新设计与快速驯化的四倍体水稻,将过去的“不可能”变成了现在的“一切皆有可能”(图1)。该研究突破了现有二倍体水稻作物的育种局限,实践了从0到1的突破性创新,为遗传背景复杂的野生植物重新驯化提供了范例和解决方案,将引领未来作物创造,翻开生物育种新篇章。
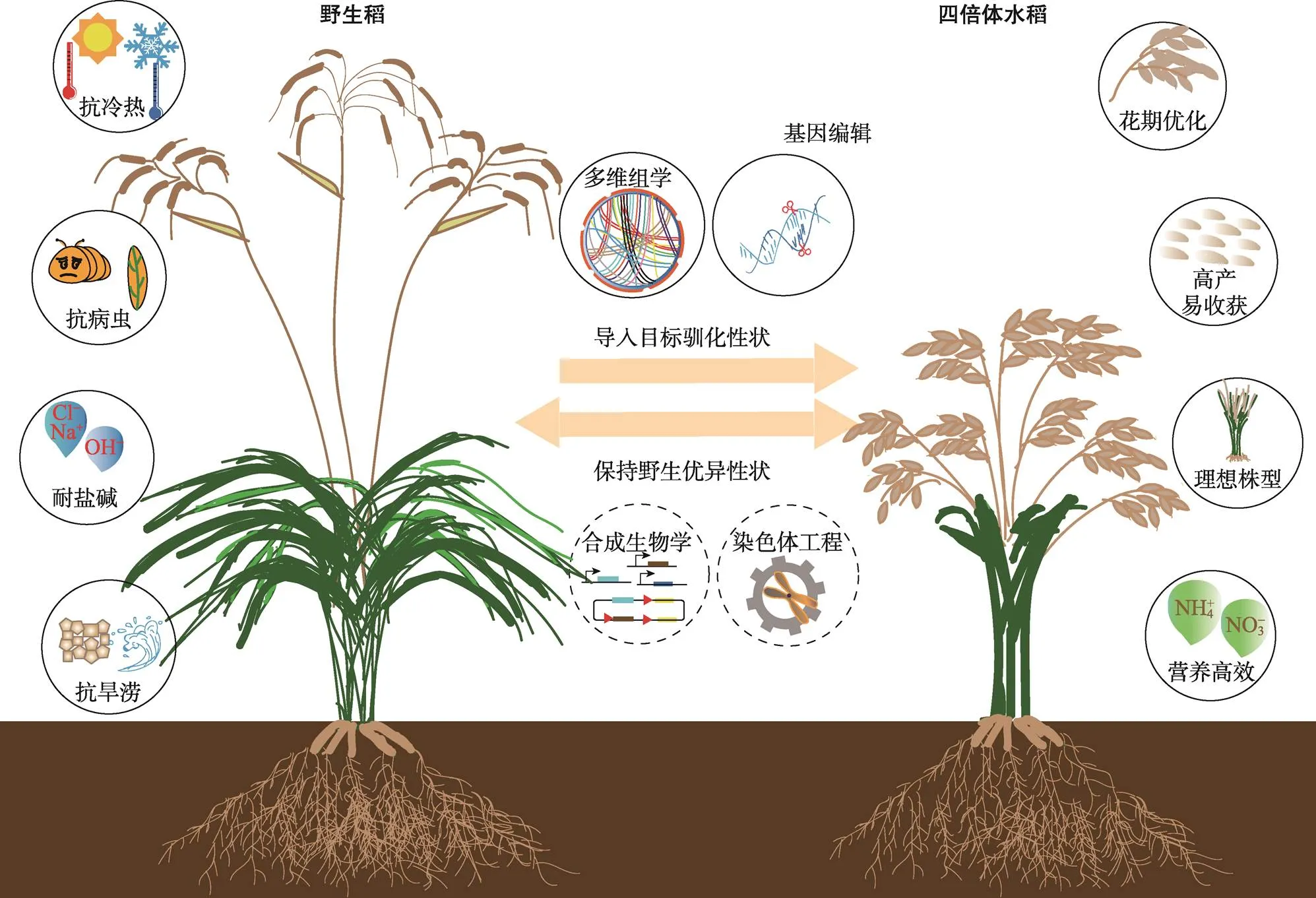
图1 四倍体野生稻从头驯化创造全新作物示意图
多倍体化是植物进化的重要机制。二倍体向多倍体的演化过程引入了新的遗传信息,提高了遗传多样性,增强了植物的环境适应性和系统稳健性。研究表明,水稻染色体在经历同源多倍化后,其抗逆性(如耐旱、耐寒及耐盐碱等)明显增强,而异源多倍体水稻则表现出更为强悍的抗逆性[9]。迄今为止,共27个稻属品种被鉴定出来,分为11个显著的基因组类型,包括6个二倍体(AA、BB、CC、EE、FF和GG)和5个异源四倍体(BBCC、CCDD、HHJJ、HHKK和KKLL)10。与栽培种相比,来源于南美的CCDD四倍体水稻的生物量较大,且对生物胁迫和非生物胁迫的抗性更强[11,12]。寻找优异野生稻种质并突破组培再生障碍是实现从头驯化的基础。为了获得合适的驯化底盘材料,突破野生稻组培再生障碍,研究人员从水稻种质资源库中收集得到28个CCDD基因型水稻,包括8个,2个和18个,系统评价了这28个品种脱分化形成愈伤的能力,筛选到再生能力相对较强的品种:(accession ID 2007-24,来源于国家野生水稻种质资源中心,南宁),并将其命名为PPR1。研究人员进而对PPR1的遗传转化及组织培养方法进行了系统优化,将其转化效率提高至80%左右,再生能力提高至40%左右,为遗传转化和基因操作铺平了道路。破译基因组并解码基因信息是实现基因操作的前提。针对PPR1异源四倍体基因组的复杂性,研究人员综合运用单分子实时测序(single-molecule real-time sequencing)、BinoNano测序,高通量染色质构象捕获测序(Hi-C)和Illumina short reads测序等多种测序技术和优化的组装算法[13],组装完成精准度高、覆盖范围大、序列完整度好的高质量野生稻基因组。结果表明,PPR1含有24条染色体,基因组大小为894.6 Mb。通过对PPR1水稻幼叶、幼根、幼茎、成熟叶、成熟根、成熟茎、种子、花序、稻穗、愈伤组织等10个代表性组织进行转录组学分析,研究人员共发现482,997条转录本。其中,99,312个基因被功能注释,99.94%的基因均匀地分布在24条染色体上。明确产量和品质等驯化性状的靶标基因是实现从头驯化的关键。该研究分析了四倍体野生稻的基因组特征和遗传变异,对PPR1和栽培种水稻基因组进行比较基因组学分析,发现栽培种中有77.87%的基因在PPR1中存在同源基因。相较于栽培种,PPR1含有相对较多的抗性基因,拥有广谱抗性潜力。研究人员对包括28个CCDD,6个EE和10个CC在内的共44个野生四倍体水稻进行了重测序。通过遗传变异分析,发现CCDD品种中碱基多态性大约为0.0049,远低于其他多倍体作物比如马铃薯(0.0111),但是高于二倍体水稻(, 0.0024;0.0030)。功能基因的保守性为筛选产量相关的驯化基因奠定了基础。多靶点精准基因编辑技术是实现快速驯化的保障。研究人员通过比较基因组学对二倍体栽培稻中已发现的株型、花期、收获方式等重要产量性状的驯化基因进行了分析,鉴定了落粒基因芒长基因,绿色革命基因、,粒长基因以及理想株型基因在PPR1中的同源基因,并使用CRISPR/Cas9技术对上述基因进行编辑,获得了含有上述基因不同突变形式的水稻材料。这些突变体表现出籽粒和梗间脱落层消失、芒长变短、株高和节间距变短,茎秆粗壮等驯化表型。通过多靶点编辑系统同时对花期相关基因、、和进行编辑, 获得突变体材料的抽穗时间均有缩短,其中有些株系抽穗期缩短至82~92天,接近现有栽培稻。花期的优化克服了野生稻抽穗的光周期敏感性,将有助于打破其生长繁育的地理位置和气候条件的限制,培育出广适型品种。
异源四倍体野生稻从头驯化路线图的绘制与实践为遗传背景复杂、驯化潜力大、农业产值高的野生植物的快速驯化开辟了道路,提供了成功范例和解决方案,并为种子精准设计与未来作物创造提供了全新策略。除了主要粮食作物,从头驯化在果蔬作物中亦取得了重要进展。2018年,中国科学院遗传与发育生物学研究所研究团队选用天然耐盐碱和抗疮痂病的野生醋栗番茄()为底盘材料,将产量和品质性状精准地导入了野生番茄,消除了野生番茄开花的光周期敏感性,驯化出株型紧凑,坐果率、果实成熟一致性和收获指数、维生素C含量显著提高的新型番茄。重要的是,从头驯化的新型番茄获得了产量和品质性状的同时,保持了野生番茄的耐盐碱和抗病性状[14]。野生稻和野生番茄的从头驯化将推动小众作物(Orphan crop)的从头驯化。小众作物是指分布在较为局限的地区,并被当地人食用的作物[15]。小众作物往往具有优良的品质或营养性状,并可以在土壤贫瘠、高海拔高、寒旱恶劣环境中生长。比如,小众作物灯笼果()具有优异的逆境耐受能力,丰富的营养成分和特别的风味物质。美国冷泉港实验室的Lippman教授团队使用基因编辑对灯笼果进行了快速驯化,改良了灯笼果的食用品质和园艺性状,为小众作物的快速驯化提供了范例[15]。随着全球人口增长和环境恶化,保障食物供应一直是很多经济不发达地区的首要问题,增加人类食谱的丰富性也是提高生活品质的重要目标,而这些小众作物能在与其原产地相似的环境恶劣地区依然保持优质高产地生长,可见其具有着十分广阔的应用潜力。对于我国而言,小众作物往往极具地域特色和特殊食用或者药用价值,对推动乡村振兴和精准扶贫具有重要意义[4]。
飞速发展的基因组编辑、合成生物学、染色体工程等现代生物技术和多维组学研究驱动的育种技术革新,在大数据、人工智能的助推下,正在开启精准设计育种的新时代,掀起“第三次绿色革命”浪潮,而异源多倍体野生植物的从头驯化无疑是这次浪潮中“最硬的骨头”、“最险的关隘”之一,作物重新设计与快速驯化领域的突破和持续进展很有可能产生“第三次绿色革命”的关键理论和核心技术。不过我们需要清醒地认识到,仍有相当一部分野生植物无法实现组培再生和基因转化,阻碍了基因编辑和合成生物学技术在其驯化中的应用;有相当一批野生植物存在生殖障碍、基因组不稳定等问题,这些问题的攻克必须依靠植物基础科学研究的重大理论创新和技术突破。因此,进一步加强作物多维组学研究,鉴定更多驯化基因,收集和发掘更多优异的野生种质资源,将为作物重新设计和快速驯化提供更多靶基因和底盘材料;加强植物再生机制和基因转化方面的研究,提高基因编辑技术的靶向效率,突破植物基因重组替换的技术瓶颈,运用合成生物学技术对作物基因信息流重编程将为从头驯化提供保障,促进新作物创造。
[1] Hancok JF. Contributions of domesticated plant studies to our understanding of plant evolution., 2005, 96: 953–963.
[2] Diamond J. Evolution, consequences and future of plant and animal domestication., 2002, 418: 700–707.
[3] Hardin LS. Bellagio 1969: the green revolution., 2008, 455: 470–471.
[4] Yang XP, Yu Y, Xu C.domestication to create new crops., 2019, 41(9): 827–835.杨新萍, 于媛, 许操. 重新设计与快速驯化创造新型作物. 遗传, 2019, 41(9): 827–835.
[5] FAO, IFDA, UNICEF, WFP, WHO. The state of food security and nutrition in the world, transforming food systems for affordable healthy diets.,2020, doi:10.4060/ ca9692zh.
[6] Yu H, Li J. Short and long term challenges in crop breeding., 2021.doi: 10.1093/nsr/nwab002.
[7] Tian Z, Wang J, Li J, Han B. Designing future crops: challenges and strategies for sustainable agriculture., 2020, doi:10.1111/tpj.15107.
[8] Yu H, Lin T, Meng X, Du H, Zhang J, Liu G, Chen M, Jing Y, Kou L, Li X, Gao Q, Ling Y, Liu X, Fan Z, Liang Y, Cheng Z, Chen M, Tian Z, Wang Y, Chu C, Zuo J, Wan J, Qian Q, Han B, Zuccolo A, Wing RA, Gao C, Liang C, Li J.A route todomestication of wild allotetraploid rice., 2021,doi:10.1016/j.cell.2021.01.013.
[9] Angeles-Shim R B, Shim J, Vinarao RB, Lapis RS, Singleton JJ. A novel locus from the wild allotetraploid rice speciesDesv. Confers bacterial blight (pv) resistance in rice ()., 2020, 15(2):e0229155.
[10] Wing RA, Purugganan MD, Zhang Q. The rice genome revolution: from an ancient grain to Green Super Rice., 2018, 19(8): 505–517.
[11] Ammiraju JS, Fan C, Yu Y, Song X, Cranston KA, Pontaroli AC, Lu F, Sanyal A, Jiang N, Rambo T, Currie J, Collura K, Talag J, Bennetzen JL, Chen M, Jackson S, Wing RA. Spatio-temporal patterns of genome evolution in allotetraploid species of the genus.,2010, 63(3): 430–442.
[12] Prusty MR, Kim SR, Vinarao R, Entila F, Egdane J, Diaz MGQ, Jena KK. Newly identified wild rice accessions conferring high salt tolerance might use a tissue tolerance mechanism in leaf., 2018, 9: 417.
[13] Du H, Liang C. Assembly of chromosome-scale contigs by efficiently resolving repetitive sequences with long reads., 2019, 10: 5360.
[14] Li T, Yang X, Yu Y, Si X, Zhai X, Zhang H, Dong W, Gao C, Xu C. Domestication of wild tomato is accelerated by genome editing., 2018, 36: 1160–1163.
[15] Lemmon ZH, Reem NT, Dalrymple J, Soyk S, Swartwood KE, Rodriguez-Leal D, Van Eck J, Lippman ZB. Rapid improvement of domestication traits in an orphan crop by genome editing., 2018, 4(10): 766–770.
2021-02-04;
2021-02-02
许操,研究员,研究方向:小肽信号与植物发育、作物重新驯化、蛋白质相分离。E-mail: caoxu@genetics.ac.cn
10.16288/j.yczz.21-056
2021/2/5 15:28:37
URI: https://kns.cnki.net/kcms/detail/11.1913.R.20210205.1057.003.html

