中国人群肝功能检测指标全基因组关联分析研究
孙一丹,田子钊,周伟,李沫,怀聪,贺林,秦胜营
研究报告
中国人群肝功能检测指标全基因组关联分析研究
孙一丹,田子钊,周伟,李沫,怀聪,贺林,秦胜营
上海交通大学Bio-X研究院,上海 200030
肝功能检测(liver function test, LFTs)指标是受遗传和环境影响的复杂性状,具有个体差异性。为系统性研究中国人群全基因组范围内单核苷酸多态性(single nucleotide polymorphism, SNP)与肝功能指标之间的联系,本研究利用英国生物银行(UK Biobank)中1653名中国人的基因分型数据和表型数据为研究对象,利用PLINK软件进行全关联分析研究(genome-wide association study, GWAS),发现229个SNP与中国人群血液中的总胆红素(total bilirubin, TB)相关,27个SNP与中国人群血液中碱性磷酸酶(alkaline phosphatase, ALP)相关,36个SNP与中国人群血液中的γ-谷氨酰转肽酶(γ-glutamyl transpeptidase, GGT)相关,1个SNP与中国人群血液中的门冬氨酸氨基转移酶(aspartate transaminase, AST)相关,最显著的位点中有11个位点是新的LFTs关联位点。通过功能基因组分析,发现这些位点的临床意义(如吉尔伯特综合征),确定了候选基因(,,),为从遗传角度理解中国人群LFTs的个体差异性和肝功能指标临床精准检测提供了前期研究基础。
肝功能检测;总胆红素;碱性磷酸酶;γ-谷氨酰转肽酶;全基因组关联分析
肝功能检测(liver function test, LFTs)是临床上常见的检测肝病的手段,其中血清酶活性测定在临床上被用于检测肝损伤[1]:当肝脏发生炎症或其他损伤时,肝细胞代谢异常、细胞膜的通透性改变,导致肝酶释放进入血液,因此通过检测血清中的肝酶和胆红素等指标可以评估肝脏受损程度和肝病的严重程度[2]。LFTs是检测药物性肝损伤的临床一线指标,通常也可以指示多器官复杂疾病——在心脑血管疾病,糖尿病,或代谢综合征中能观察到血清中肝酶活性的异常[3~5]。常见的LFTs指标包括:丙氨酸氨基转移酶(alanine aminotransferase, ALT),门冬氨酸氨基转移酶(aspartate transaminase, AST),碱性磷酸酶(alkaline phosphatase, ALP),γ-谷氨酰转肽酶(γ-glutamyl transpeptidase, GGT),胆红素(bilirubin)等[2]。但是有研究表明,即使是同一个人,LFTs在短期内也会存在差异,超过30%的第一次检测出异常结果的成年人在重新进行LFTs后,肝酶和胆红素水平被重新归类为正常[6]。
LFTs的检测指标——肝酶和胆红素是受多基因和环境因素决定的复杂性状。其中,环境因素包括年龄、性别、饮食习惯、身体质量指数(body mass index, BMI)、吸烟、饮酒等[7];而国内外的众多研究也提示了基因多态性对肝功能指标的影响。一项基于丹麦双生子的研究发现ALT、GGT和胆红素等肝功能指标具有高遗传力(35%~61%),且这些指标的遗传力独立于饮酒和BMI等环境因素[8]。几项全基因组关联研究(genome-wide association studies, GWAS)也发现了与外周血中肝酶和胆红素水平相关的基因,如影响GGT水平,与ALP水平相关,和可能影响ALT,与胆红素相关,与AST和ALT有关。同时,这些基因对肝功能指标的影响呈现明显的种族差异 性[5,9,10]。中国是肝脏疾病的高发地区,为了更好地使用LFTs指标辅助疾病诊断,有必要加深对影响肝功能指标的非疾病因素的理解,因此深入研究影响中国人群的肝功能指标的遗传特征非常重要。本研究利用英国生物银行(UK Biobank)中的中国人基因分型数据进行全基因组关联分析研究,旨在评估中国人群中与肝功能指标相关的单核苷酸多态性(single nucleotide polymorphism, SNP)风险位点。
1 对象与方法
1.1 实验对象
本研究入组了UK Biobank[11]中的1653名中国人,经申请,获得了他们的基因信息和表型信息,申请号为(Application 34716)。基因型由UK Biobank通过Affymetrix UK BiLEVE Axiom和Affymetrix UK Biobank Axiom两种芯片获得超过800,000个SNP,覆盖率超过95%[12]。表型信息包括电子病历所记录的患病史和血液生化指标检测等。
1.2 表型分析
通过表型信息对样本进行筛选,排除了患有肝胆疾病的病例,包括ICD10和ICD9编码的肝病和参与者自我报告的肝病,这些疾病包括:酒精性肝病、肝衰竭、慢性肝炎、肝纤维化、肝硬化、病毒性肝炎、肝癌等。最后筛选获得1508名无肝病史的中国人入组本研究。
1.3 数据分析
从UK Biobank获取SNP基因分型数据后,使用PLINK (version1.9, http://www.pngu.mgh.harvard. edu/purcell/plink/)软件对数据进行质控,质控标准为去除缺失率大于5%、哈迪–温伯格平衡(hardy- Weinberg equilibrium, HWE)值偏离1.00×10–6、最小等位基因频率(minor allele frequency, MAF)小于1%的SNP;使用Minimac4 (https://imputationserver. sph.umich.edu/)对数据进行基因型填充(参考单倍型为1000 Genomes Phase 3东亚人群)。填充结果用PLINK对数据进行质控,质控标准为除缺失率大于1%、杂合率偏离3倍的标准差、HWE值偏离1.00×10–6的样本;将通过质控的样本与1000人基因组样本(1000 Genomes Phase 3)一同进行多维尺度分析(multidimensional scaling, MDS)来对人群分层进行可视化[13],通过主成分分析(principal components analysis, PCA)去除偏离整体目标群体的游离样本,去除重复和具有亲缘关系的样本。选取通过质控的样本的常见变异(MAF > 1%)后进行卡方关联分析(含Bonferroni矫正),和线性模型矫正协变量的关联分析,协变量矫正(年龄、性别、吸烟)[14,15],其中协变量包括PCA的前六个主成分、年龄、性别、吸烟和饮酒。使用R软件qqman包绘制经协变量矫正后的关联分析的曼哈顿图和QQ图,使用Locus Zoom (http://locuszoom.org/)绘制LocusZoom图展示显著SNP附近的局部信息[16]。将得到的易感SNP (<5.00×10–8)使用VarNote (http://mulinlab.org/varnote/ index.html)进行注释[17,18],在ClinVar (https://www. ncbi.nlm.nih.gov/clinvar/)数据库中查询易感SNP的临床数据,在RegulomeDB (https://www.regulomedb. org/regulome-help/)中对SNP进行功能基因组学打分,估算SNP的调控能力[19]。使用SMR (https:// cnsgenomics.com/software/smr/)在GTEx数据库中查询SNP在肝中的数量表达性状位点信息(expression quantitative trait loci, eQTL)[20]。
2 结果与分析
2.1 中国人群筛选和芯片数据质控结果
在UK Biobank共获取1508名未患肝病的中国人的肝功能指标数据,其中1482个样本有基因分型数据,经PLINK质控共得到784,256个SNP位点进行填充。使用Minimac4进行填充后共获得6,977,555个SNP位点。使用PLINK软件对填充过后的位点进行质控,并与1000人基因组样本进行MDS分析,大部分样本与东亚血统重合,但也有少部分可能为欧美血统(图1 A),根据PCA分析保留东亚血统后,MDS分析结果显示已去除可能具有欧美血统的样本,剩余样本全部为东亚血统(图1 B)。最终得到1361人,6,906,867个SNP分型结果。样本杂合率偏离均值不超过3倍的标准差,无亲缘关系(IBD> 0.1875);SNP位点缺失率小于5%,检出率大于99%,HWE值大于1.00×10–6,MAF大于1%。
2.2 全基因组关联分析筛选差异位点
使用PLINK软件对6,906,867个SNP进行卡方关联分析,通过Bonferroni矫正,观察到了与总胆红素(total bilirubin, TB)水平相关的228个易感SNP (<0.05);最显著的易感SNP为rs1983023、rs6431625、rs2018985和rs4477910 (=3.89×10–38),位于已报道的胆红素相关调控基因基因簇区域内[5];观察到了与ALP水平相关的28个易感SNP(<0.05),其中最显著的易感SNP为rs550057、rs579459、rs651007、rs507666、rs600038和rs532436 (=8.27×10–5),位于已报道过的ALP相关调控基因内[7]。观察到了与GGT水平相关的36个易感SNP (<0.05),其中最显著的易感SNP为rs5760500,位于已报道过的GGT相关调控基因内[10]。未观察到经Bonferroni矫正后的AST和ALT卡方关联分析显著结果。
由于肝功能指标为连续变量,因此使用PLINK软件对这些位点用线性回归模型进行协变量矫正后的全基因组关联分析。将套用线性模型且经协变量矫正后进行GWAS,观察到与TB相关的229个SNP达到全基因组显著(<5.00×10–8),rs1983023、rs6431625、rs7576166、rs4477910和rs2018985最为显著(=4.07×10–44)。;观察到与ALP相关的27个SNP达到全基因组显著(<5.00×10–8),rs2519093最为显著(=1.18×10–10);观察到与GGT相关的36个SNP达到全基因组显著(<5.00×10–8),rs5760500最为显著(=7.80×10–11);观察到与AST相关的1个SNP达到全基因组显著(<5.00×10–8),为rs150693807 (=1.34×10–8)。QQ图证明全基因组范围内存在大量SNP与TB (图2 A),ALP (图2 B),GGT (图2 C)和AST (图2 D)有显著的相关性。曼哈顿图(图3)显示了经矫正后的关联分析结果,依然在2号染色体上发现了具有全基因组显著性的SNP与TB水平相关(图3 A),在9号染色体上发现具有全基因组显著性的SNP与ALP水平相关(图3 B),在3号和22号染色体上发现了具有全基因组显著性的SNP与GGT水平相关(图3 C),在2号染色体上发现了具有全基因组显著性的SNP与AST水平相关(图3 D)。在两项研究中,人群分层得到了矫正,观察到了较低的膨胀系数(λTB=1.01, λALP=1.00, λGGT=1.01, λAST=1.01)。LocusZoom图(图4)展示了具有全基因组显著性的SNP附近位点,并且注释了这些SNP所处的基因。
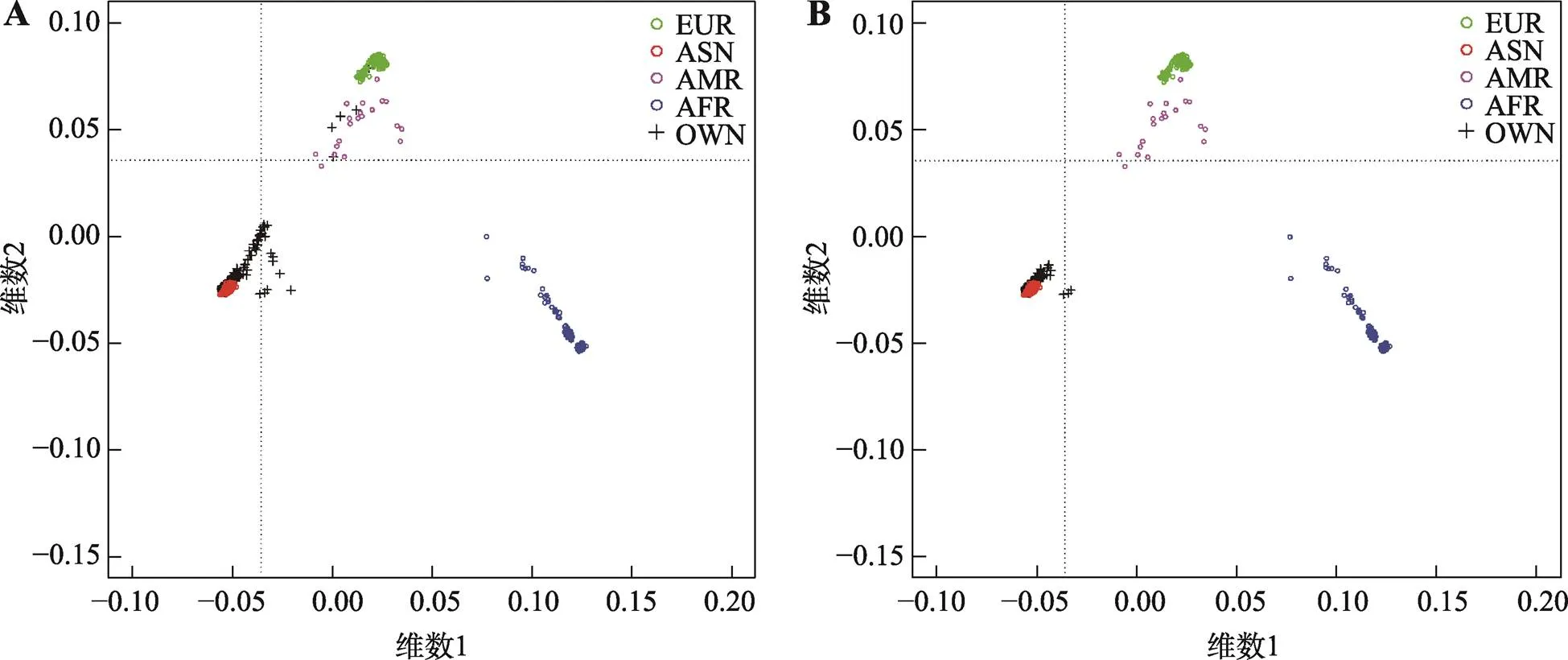
图1 多维尺度分析显示的群体结构图
A:质控前MDS结果;B:质控后MDS结果。绿圈为欧洲血统,红圈为东亚血统,粉圈为美洲血统,蓝圈为非洲血统,加号为本课题样本。

图2 中国人群肝功能检测指标的Q-Q图
A: TB-GWAS; B: ALP-GWAS; C: GGT-GWAS; D: AST-GWAS。
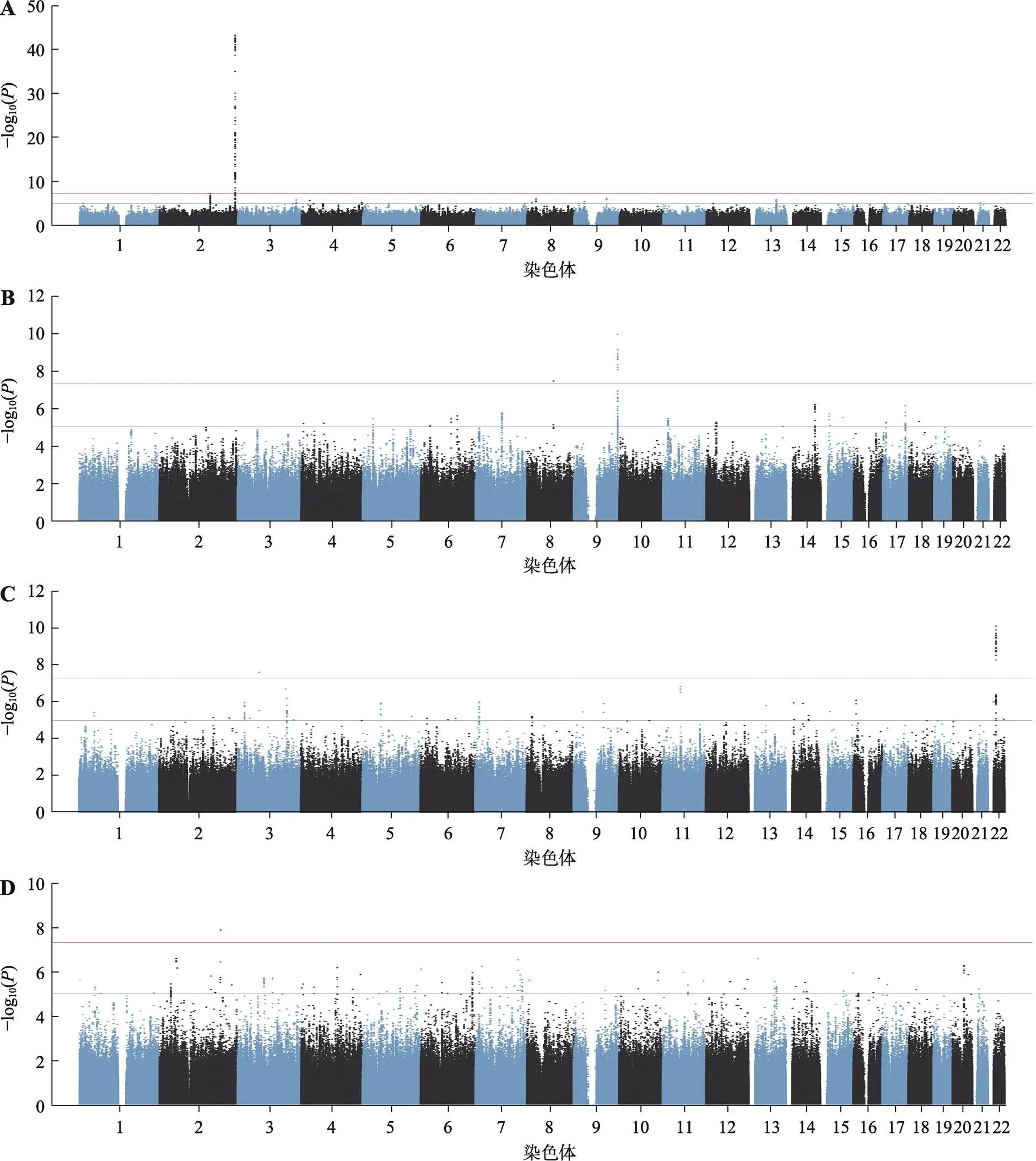
图3 中国人群肝功能检测指标的曼哈顿图
A: TB-GWAS; B: ALP-GWAS; C: GGT-GWAS; D: AST-GWAS。

图4 中国人群肝功能检测指标关联区段局部图
A:TB-GWAS 2号染色体;B:ALP-GWAS 9号染色体;C:ALP-GWAS 8号染色体;E:GGT-GWAS 22号染色体;F:GGT-GWAS 3号染色体;G:AST-GWAS 2号染色体。紫色菱形为最显著SNP位点,灰色圆点为SNP位点,蓝色曲线为重组率,下方为注释基因箭头表示转录方向。
2.3 相关位点功能分析
在VarNote中进行SNP和基因的注释,在ClinVar查询所有达到全基因组显著SNP (<5.00× 10–8)的临床信息,结果如表1所示。共获得与TB相关的4个SNP存在临床意义,包括与药物反应相关,是吉尔伯特综合征、Lucey-Driscoll综合征和Crigler-Najjar综合征的风险位点;获得了和与ALP相关的1个SNP存在临床意义,与血型相关。
在RegulomeDB中对所有达到全基因组显著SNP (<5.00×10–8)进行调控能力的打分,共获得与TB相关的12个SNP,与ALP相关的3个SNP,和与GGT相关的3个SNP具有80%以上的可能性为调控SNP,其所在基因和区域如表2所示,证明了基因簇与TB、与ALP、与GGT之间的关系。除此之外还发现了一个的同义突变(rs13009407)与TB相关。
使用SMR在GTEx数据库中查询到39个与TB相关的SNP影响了在肝组织中的表达(<1.00×10–5)。
3 讨论
LFTs常用来评估肝功能和疾病,血液中高水平肝酶和胆红素与多种疾病相关,如黄疸,吉尔伯特综合征和多种伤害引起的肝损伤,包括酒精滥用,病毒感染,自身免疫,药物毒性和代谢综合征等[21,22]。药物引起的肝损伤是常见的药物不良反应,肝功能异常也是终止临床治疗的常见原因,2019年中华医学会结核病学分会出台的抗结核药物诊疗指南也根据血清中的ALP、ALP和TB对肝损伤进行诊断和严重程度分级[23,24]。但在对正常人进行血液生化指标常规检查时,也会出现错误检测出异常值的情况[6]。除了受环境影响外,所有的肝功能指标都有显著的遗传基础,了解肝功能指标的潜在遗传贡献有助于解释个体之间的广泛差异[25]。尽管已有相关基因报道,但人种之间遗传特征不同。如未在东亚人群中重复出之前欧洲人种中发现的与ALP相关的、、、和[7]。本研究对UK Biobank中的中国人群进行全基因组关联分析,确定了在中国人群中与LFTs相关的293个SNPs,并且查询了所有达到全基因组显著位点的临床意义,预估其调控能力,查询其eQTL信息,旨在寻找影响中国人LFTs的潜在遗传标记。

表1 中国人群肝功能指标相关位点临床意义
Ref:原始碱基;Alt:突变碱基。
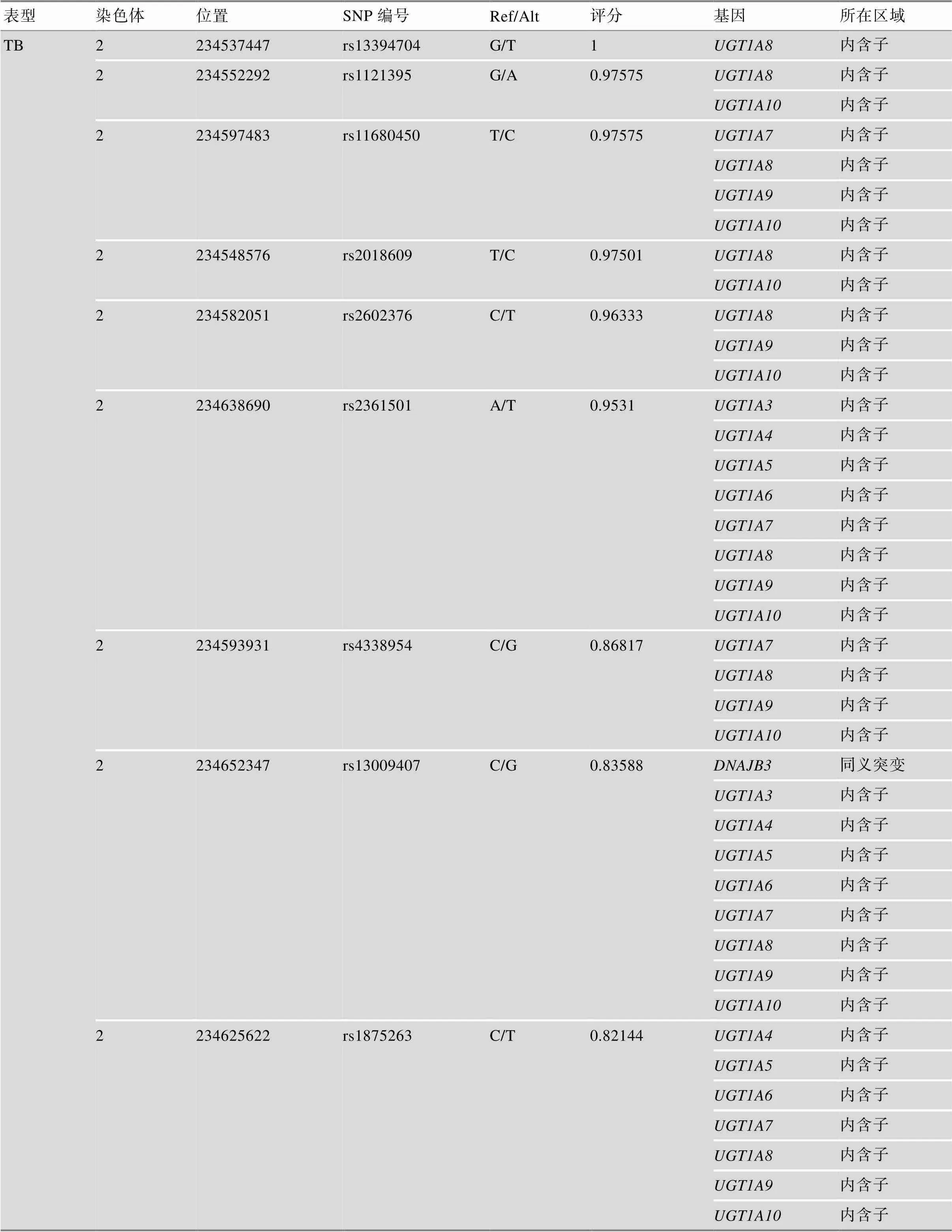
表2 中国人群肝功能指标相关位点调控能力预测结果
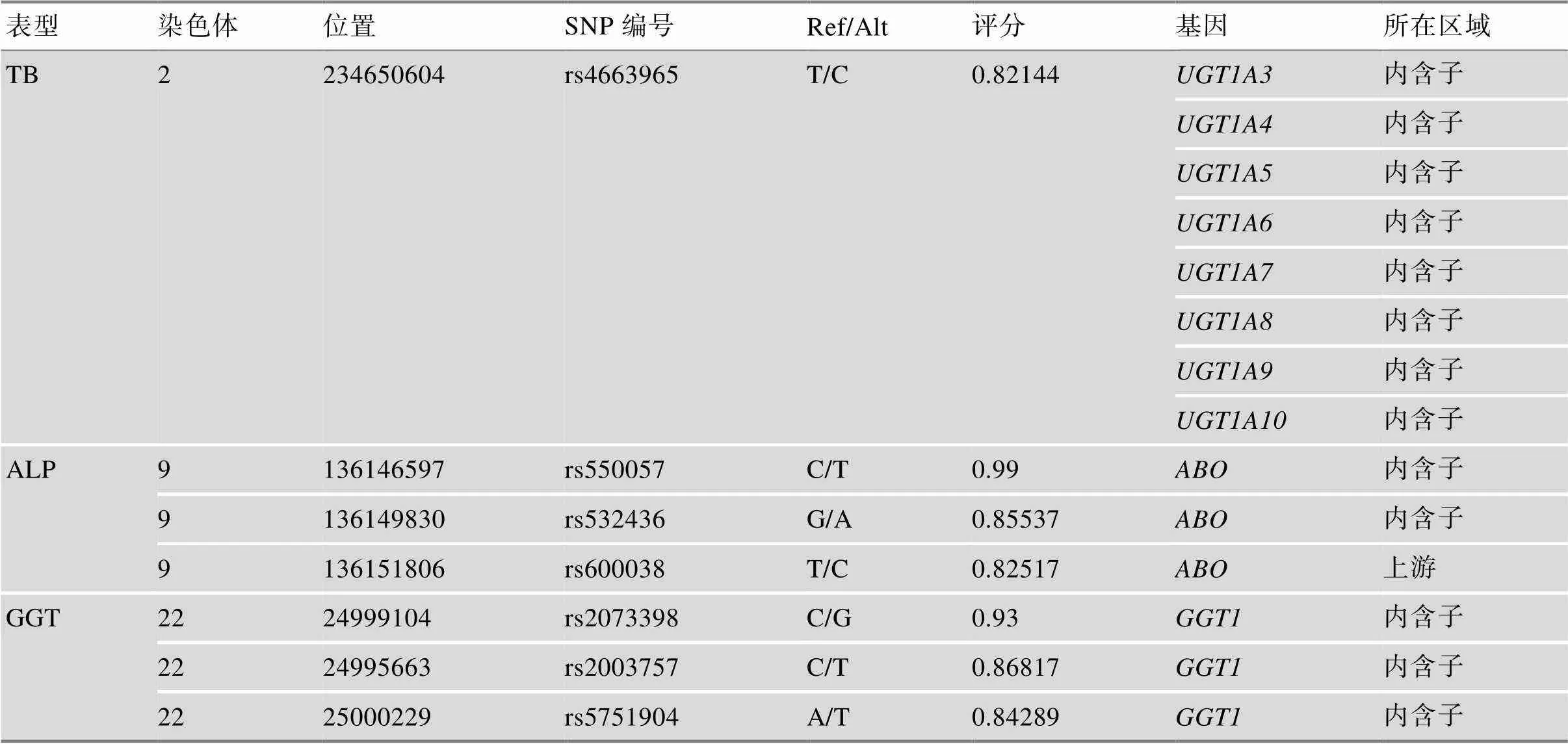
续表
Ref:原始碱基;Alt:突变碱基。评分,范围为0~1,越接近1,该位点为调控SNP的可能性越大。
胆红素是血红素分解的最终产物,血清中的胆红素会因黄疸等疾病升高,并且胆红素浓度受到基因调控。在欧美人群和东亚人群中,基因座和基因座与血清中的高水平胆红素相关[5]。本研究所发现的最显著位点之一的rs2018985在西班牙人群中被证实与TB相关[26],首次报道了rs1983023、rs6431625、rs7576166和rs4477910与中国人群TB之间的关联。显著SNP所在的基因簇编码UDP-葡糖醛酸糖基转移酶(UGT)家族,是药物代谢酶之一[27],在胆红素代谢中起到重要作用[28]。该区域包含9个相似的蛋白编码基因和4个相似的非蛋白编码基因[29]。其中的遗传缺陷被证明和高胆红素相关,而与葡萄糖醛酸化率相关,影响胆红素代谢[28]。葡萄糖醛酸与胆红素的结合影响胆红素代谢,导致血液中存在高胆红素,例如吉尔伯特综合征,Crigler-Najjar综合征,和Lucey-Driscoll综合征[30],本研究所发现的rs4148323和rs887829被报道过与中国人群胆红素水平相关[31]。本研究首次证明了与吉尔伯特综合征相关的rs4124874和rs35350960也与中国人群胆红素水平升高有关。除此之外本研究发现的rs13009407是一个同义突变,有研究证明表达的降低与二型糖尿病有关[32],胆红素升高与二型糖尿病有密切的联系[33]。
基因座和血清中的ALP水平之间的关联已在包括中国人在内的人群中都有报道过,本研究所发现的最显著的rs579459、rs651007已在欧洲、韩国和日本人人群中报道过[5,21,22],首次发现了rs550057、rs507666、rs600038、rs532436和rs2519093与中国人群ALP之间的关联。基因编码糖基转移酶,其催化活性有助于形成血型的抗原结构[34]。基因表达与ALP水平之间的关系尚不明确,本研究所发现的rs1053878与A型血的一个亚型相关[35],rs550057、rs532436和rs600038都有可能调控基因的表达,因此推测ALP水平可能与不同血型同工酶变化有关。虽然发现8号染色体上发现了一个ALP相关的SNP,但经注释rs143653948位于基因间区。
参与谷胱甘肽的代谢,是最常见的GGT相关基因[5]。本研究首次发现了rs5760500与中国人群GGT之间的关联,rs2073398、rs2003757和rs5751904可能具有调控的作用。本研究还发现在3号染色体上的rs9846667与GGT相关,位于内含子区域,但该基因在大脑高度表达[36]。与AST相关的rs150693807,经注释位于基因间区,并未查询到其相关临床信息和调控作用。并未发现达到全基因组显著的与ALT相关的位点。
本研究的优势之一是使用UK Biobank所收集的中国人群基因分型数据和电子病历中的肝功能数据进行关联分析,分析结果复现了既往研究中的显著SNP、基因的同时也得到新的发现,TB与的关联、ALP与的关联、GGT和的关联和之前的研究高度一致,证明了电子病历记录数据的有效性和价值。数据共享和生物银行的重要性在全球卫生研究中日益到认可,虽然这些项目在分析过程中需要进行更为精细地处理,如人群分层,但生物银行的建立无疑将为疾病的诊断、预防和治疗提供强大的研究资源。若能建立中国人群为主的数据库,必将极大推进中国人群的健康研究。
综上所述,本研究通过对1653名中国人群进行肝功能指标的全基因组关联分析的系统性研究,发现rs1983023、rs6431625、rs7576166和rs4477910与中国人群TB最为相关,发现rs550057、rs507666、rs600038、rs532436和rs2519093与中国人ALP水平最为相关,发现rs5760500与中国人GGT水平最为相关,发现rs150693807与中国人AST水平最为相关,同时结合显著位点的临床意义、eQTL和调控能力为解释肝功能指标的个体差异性和在临床上检测肝功能指标提供了前期研究基础。
[1] Liu FY, Evaluation of clinical application effect of liver function test., 2015, 13(36): 54–55.柳富云, 肝功能化验在临床中的应用效果评价. 中国医药指南, 2015, 13(36): 54–55.
[2] Song GP. I. liver function tests -- clinical significance of serum enzymology test., 2003, 19(4): 195– 197.宋国培. Ⅰ. 肝功能——血清酶学检测的临床意义. 临床肝胆病杂志, 2003, 19(4): 195–197.
[3] 陈成伟. 药物性肝损害诊断标准的评价. 诊断学理论与实践, 2009, 8(2): 121–124.Chen CW. Evaluation of diagnostic criteria of drug-induced liver injury., 2009, 8(2): 121–124.
[4] Bénichou C. Criteria of drug-induced liver disorders. Report of an international consensus meeting., 1990, 11(2): 272–276.
[5] Seo JY, Lee JE, Chung GE, Shin E, Kwak MS, Yang JI, Yim JY. A genome-wide association study on liver enzymes in Korean population., 2020, 15(2): e0229374.
[6] Lazo M, Selvin E, Clark JM. Brief communication: Clinical implications of short-term variability in liver function test results., 2008, 148(5): 348– 352.
[7] Li J, Gui LX, Wu C, He YF, Zhou L, Guo H, Yuan J, Yang BY, Dai XY, Deng QF, Huang SL, Guan L, Hu D, Deng SY, Wang T, Zhu J, Min XW, Lang MJ, Li DF, Yang HD, Hu FB, Lin DX, Wu TC, He MA. Genome-wide association study on serum alkaline phosphatase levels in a Chinese population., 2013, 14: 684.
[8] Bathum L, Petersen HC, Rosholm JU, Petersen PH, Vaupel J, Christensen K. Evidence for a substantial genetic influence on biochemical liver function tests: results from a population-based Danish twin study., 2001, 47(1): 81–87.
[9] Kamatani Y, Matsuda K, Okada Y, Kubo M, Hosono N, Daigo Y, Nakamura Y, Kamatani N. Genome-wide association study of hematological and biochemical traits in a Japanese population., 2010, 42(3): 210– 215.
[10] Yuan X, Waterworth D, Perry JRB, Lim N, Song K, Chambers JC, Zhang WH, Vollenweider P, Stirnadel H, Johnson T, Bergmann S, Beckmann ND, Li Y, Ferrucci L, Melzer D, Hernandez D, Singleton A, Scott J, Elliott P, Waeber G, Cardon L, Frayling TM, Kooner JS, Mooser V. Population-based genome-wide association studies reveal six loci influencing plasma levels of liver enzymes., 2008, 83(4): 520–528.
[11] Sudlow C, Gallacher J, Allen N, Beral V, Burton P, Danesh J, Downey P, Elliott P, Green J, Landray M, Liu B, Matthews P, Ong G, Pell J, Silman A, Young A, Sprosen T, Peakman T, Collins R. UK Biobank: An open access resource for identifying the causes of a wide range of complex diseases of middle and old age., 2015, 12(3): e1001779.
[12] Howie B, Fuchsberger C, Stephens M, Marchini J, Abecasis GR. Fast and accurate genotype imputation in genome-wide association studies through pre-phasing., 2012, 44(8): 955–959.
[13] Marees AT, de Kluiver H, Stringer S, Vorspan F, Curis E, Marie-Claire C, Derks EM. A tutorial on conducting genome-wide association studies: Quality control and statistical analysis., 2018, 27(2): e1608.
[14] Das S, Forer L, Schönherr S, Sidore C, Locke AE, Kwong A, Vrieze SI, Chew EY, Levy S, McGue M, Schlessinger D, Stambolian D, Loh PR, Iacono WG, Swaroop A, Scott LJ, Cucca F, Kronenberg F, Boehnke M, Abecasis GR, Fuchsberger C. Next-generation genotype imputation service and methods., 2016, 48(10): 1284– 1287.
[15] Purcell S, Neale B, Todd-Brown K, Thomas L, Ferreira MAR, Bender D, Maller J, Sklar P, de Bakker PIW, Daly MJ, Sham PC. PLINK: A tool set for whole-genome association and population-based linkage analyses., 2007, 81(3): 559–575.
[16] Pruim RJ, Welch RP, Sanna S, Teslovich TM, Chines PS, Gliedt TP, Boehnke M, Abecasis GR, Willer CJ. LocusZoom: regional visualization of genome-wide association scan results., 2010, 26(18): 2336–2337.
[17] Huang DD, Yi XF, Zhou Y, Yao HC, Xu H, Wang JH, Zhang SJ, Nong WY, Wang PW, Shi L, Xuan CH, Li MX, Wang JW, Li WD, Kwan HS, Sham PC, Wang K, Li MJ. Ultrafast and scalable variant annotation and prioritization with big functional genomics data., 2020, 30(12): 1789–1801.
[18] Huang DD, Yi XF, Zhang SJ, Zheng ZY, Wang PW, Xuan CH, Sham PC, Wang JW, Li MJ. GWAS4D: multidimensional analysis of context-specific regulatory variant for human complex diseases and traits., 2018, 46(W1): W114–W120.
[19] Boyle AP, Hong EL, Hariharan M, Cheng Y, Schaub MA, Kasowski M, Karczewski KJ, Park J, Hitz BC, Weng S, Cherry JM, Snyder M. Annotation of functional variation in personal genomes using RegulomeDB., 2012, 22(9): 1790–1797.
[20] Zhu ZH, Zhang FT, Hu H, Bakshi A, Robinson MR, Powell JE, Montgomery GW, Goddard ME, Wray NR, Visscher PM, Yang J. Integration of summary data from GWAS and eQTL studies predicts complex trait gene targets., 2016, 48(5): 481–487.
[21] Chambers JC, Zhang WH, Sehmi J, Li XZ, Wass MN, Van der Harst P, Holm H, Sanna S, Kavousi M, Baumeister SE, Coin LJ, Deng GH, Gieger C, Heard-Costa NL, Hottenga JJ, Kühnel B, Kumar V, Lagou V, Liang LM, Luan JA, Vidal PM, Leach IM, O'Reilly PF, Peden JF, Rahmioglu N, Soininen P, Speliotes EK, Yuan X, Thorleifsson G, Alizadeh BZ, Atwood LD, Borecki IB, Brown MJ, Charoen P, Cucca F, Das D, de Geus EJC, Dixon AL, Döring A, Ehret G, Eyjolfsson GI, Farrall M, Forouhi NG, Friedrich N, Goessling W, Gudbjartsson DF, Harris TB, Hartikainen AL, Heath S, Hirschfield GM, Hofman A, Homuth G, Hyppönen E, Janssen HLA, Johnson T, Kangas AJ, Kema IP, Kühn JP, Lai S, Lathrop M, Lerch MM, Li Y, Liang TJ, Lin JP, Loos RJF, Martin NG, Moffatt MF, Montgomery GW, Munroe PB, Musunuru K, Nakamura Y, O'Donnell CJ, Olafsson I, Penninx BW, Pouta A, Prins BP, Prokopenko I, Puls R, Ruokonen A, Savolainen MJ, Schlessinger D, Schouten JNL, Seedorf U, Sen-Chowdhry S, Siminovitch KA, Smit JH, Spector TD, Tan WT, Teslovich TM, Tukiainen T, Uitterlinden AG, Van der Klauw MM, Vasan RS, Wallace C, Wallaschofski H, Wichmann HE, Willemsen G, Würtz P, Xu C, Yerges-Armstrong LM, Alcohol Genome-wide Association (AlcGen) Consortium, Diabetes Genetics Replication and Meta-analyses (DIAGRAM+) Study, Genetic Investigation of Anthropometric Traits (GIANT) Consortium, Global Lipids Genetics Consortium; Genetics of Liver Disease (GOLD) Consortium, International Consortium for Blood Pressure (ICBP-GWAS), Meta-analyses of Glucose and Insulin-Related Traits Consortium (MAGIC), Abecasis GR, Ahmadi KR, Boomsma DI, Caulfield M, Cookson WO, van Duijn CM, Froguel P, Matsuda K, McCarthy MI, Meisinger C, Mooser V, Pietiläinen KH, Schumann G, Snieder H, Sternberg MJE, Stolk RP, Thomas HC, Thorsteinsdottir U, Uda M, Waeber G, Wareham NJ, Waterworth DM, Watkins H, Whitfield JB, Witteman JCM, Wolffenbuttel BHR, Fox CS, Ala-Korpela M, Stefansson K, Vollenweider P, Völzke H, Schadt EE, Scott J, Järvelin MR, Elliott P, Kooner JS. Genome-wide association study identifies loci influencing concentrations of liver enzymes in plasma., 2011, 43(11): 1131–1138.
[22] Namjou B, Marsolo K, Lingren T, Ritchie MD, Verma SS, Cobb BL, Perry C, Kitchner TE, Brilliant MH, Peissig PL, Borthwick KM, Williams MS, Grafton J, Jarvik GP, Holm IA, Harley JB. A gwas study on liver function test using emerge network participants., 2015, 10(9): e0138677.
[23] Watkins PB. Idiosyncratic liver injury: challenges and approaches., 2005, 33(1): 1–5.
[24] Guidelines for the diagnosis and treatment of antituberculosis drug-induced liver injury (2019 Edition)., 2019, 42(5): 343–356.抗结核药物性肝损伤诊治指南(2019年版). 中华结核和呼吸杂志, 2019, 42(5): 343–356.
[25] Rahmioglu N, Andrew T, Cherkas L, Surdulescu G, Swaminathan R, Spector T, Ahmadi KR. Epidemiology and genetic epidemiology of the liver function test proteins., 2009, 4(2): e4435.
[26] Coltell O, Asensio EM, Sorli JV, Barragán R, Fernández- Carrión R, Portolés O, Ortega-Azorín C, Martínez-LaCruz R, González JI, Zanón-Moreno V, Gimenez-Alba I, Fitó M, Ros E, Ordovas JM, Corella D. Genome-wide association study (GWAS) on bilirubin concentrations in subjects with metabolic syndrome: sex-specific gwas analysis and gene- diet interactions in a mediterranean population., 2019, 11(1): 90.
[27] Jinno H, Tanaka-Kagawa T, Hanioka N, Saeki M, Ishida S, Nishimura T, Ando M, Saito Y, Ozawa S, Sawada JI. Glucuronidation of 7-ethyl-10-hydroxycamptothecin (SN-38), an active metabolite of irinotecan (CPT-11), by human UGT1A1 variants, G71R, P229Q, and Y486D., 2003, 31(1): 108–113.
[28] Peters WHM, te Morsche RHM, Roelofs HNJ. Combined polymorphisms in UDP-glucuronosyltransferases 1A1 and 1A6: implications for patients with Gilbert's syndrome., 2003, 38(1): 3–8.
[29] Tang W, Fu YP, Figueroa JD, Malats N, Garcia-Closas M, Chatterjee N, Kogevinas M, Baris D, Thun M, Hall JL, De Vivo I, Albanes D, Porter-Gill P, Purdue MP, Burdett L, Liu LY, Hutchinson A, Myers T, Tardón A, Serra C, Carrato A, Garcia-Closas R, Lloreta J, Johnson A, Schwenn M, Karagas MR, Schned A, Black A, Jacobs EJ, Diver WR, Gapstur SM, Virtamo J, Hunter DJ, Fraumeni JF, Chanock SJ, Silverman DT, Rothman N, Prokunina- Olsson L. Mapping of the UGT1A locus identifies an uncommon coding variant that affects mRNA expression and protects from bladder cancer., 2012, 21(8): 1918–1930.
[30] Memon N, Weinberger BI, Hegyi T, Aleksunes LM. Inherited disorders of bilirubin clearance., 2016, 79(3): 378–386.
[31] Lin R, Wang Y, Wang Y, Fu WQ, Zhang DD, Zheng HX, Yu T, Wang Y, Shen M, Lei R, Wu H, Sun AJ, Zhang RF, Wang XF, Xiong MM, Huang W, Jin L. Common variants of four bilirubin metabolism genes and their association with serum bilirubin and coronary artery disease in Chinese Han population., 2009, 19(4): 310–318.
[32] Arredouani A, Diane A, Khattab N, Bensmail I, Aoude I, Chikri M, Mohammad R, Abou-Samra AB, Dehbi M. DNAJB3 attenuates metabolic stress and promotes glucose uptake by eliciting Glut4 translocation., 2019, 9(1): 4772.
[33] Abbasi A, Deetman PE, Corpeleijn E, Gansevoort RT, Gans ROB, Hillege HL, van der Harst P, Stolk RP, Navis G, Alizadeh BZ, Bakker SJL. Bilirubin as a potential causal factor in type 2 diabetes risk: a mendelian randomization study., 2015, 64(4): 1459–1469.
[34] Greenwell P. Blood group antigens: Molecules seeking a function?, 1997, 14(2): 159–173.
[35] Paterson AD, Lopes-Virella MF, Waggott D, Boright AP, Hosseini SM, Carter RE, Shen EQ, Mirea L, Bharaj B, Sun L, Bull SB, Diabetes Control and Complications Trial/ Epidemiology of Diabetes Interventions and Complications Research Group. Genome-wide association identifies the ABO blood group as a major locus associated with serum levels of soluble E-selectin., 2009, 29(11): 1958–1967.
[36] Tang YT, Emtage P, Funk WD, Hu TH, Arterburn M, Park EEJ, Rupp F. TAFA: a novel secreted family with conserved cysteine residues and restricted expression in the brain., 2004, 83(4): 727–734.
Genome-wide association study on liver function tests in Chinese
Yidan Sun, Zizhao Tian, Wei Zhou, Mo Li, Cong Huai, Lin He, Shengying Qin
Liver function tests (LFTs) measure a series of complex traits that are affected by genetic and environmental factors. Previous studies demonstrated that there exist significant differences in LFTs across individuals, which may be the results of the unique genetic profile of each individual. Therefore, we performed a genome-wide association study (GWAS) to evaluate the influence of single nucleotide polymorphisms (SNPs) in LFTs and identify the significant association between genotypes and phenotypes using 1653 Chinese subjects from the UK Biobank database. We successfully identified 229 SNPs significantly associated with total bilirubin (TB) levels, 27 SNPs significantly associated with alkaline phosphatase (ALP), and 36 SNPs significantly associated with γ-glutamyl transpeptidase (GGT) in serum. In addition, one SNP significantly associated with aspartate transaminase (AST) in serum was also found. Among the most significant ones, the topmost 11 SNPs were found to be associated with LFTs for the first time. Through functional genomic analysis, the clinical significance of these SNPs could be associated with diseases (e.g. Gilbert's syndrome) and the respective candidate genes (,,). This study provides a preliminary rationale for research to understand the associations of individual genetic differences and clinical liver functions in the Chinese population.
Liver function tests; total bilirubin; alkaline phosphatase; γ-glutamyl transpeptidase; genome-wide association study
2020-12-16;
2021-02-01
国家自然科学基金项目(编号:81773818,82003856)资助[Supported by the National Nature Science Foundation of China (Nos. 81773818, 82003856)]
孙一丹,在读硕士研究生,专业方向:生物学。E-mail: yidan_sun@sjtu.edu.cn
秦胜营,教授,研究方向:药物基因组学与个体化医学。E-mail: chinsir@sjtu.edu.cn
10.16288/j.yczz.20-435
2021/3/5 17:27:10
URI: https://kns.cnki.net/kcms/detail/11.1913.R.20210305.0938.002.html
(责任编委: 周钢桥)

