前噬菌体研究进展
陈学梅,魏云林,季秀玲
综 述
前噬菌体研究进展
陈学梅,魏云林,季秀玲
昆明理工大学生命科学与技术学院,昆明 650500
噬菌体是地球上最多的生物实体,一直被认为是细菌的天敌。然而随着基因组学和分子生物学等技术的快速发展,人们发现噬菌体与宿主之间存在微妙而复杂的关系。前噬菌体是指溶原性细菌内存在的整套噬菌体DNA基因组,广泛分布在细菌基因组中,对调节细菌宿主生理具有重要作用,如参与调节宿主的毒力、影响生物膜形成、赋予宿主免疫力等。有趣的是,前噬菌体可以通过“监听”细菌的群体感应来调节自身的溶原–裂解状态。近年来,一些细菌中由前噬菌体编码的抗CRISPR蛋白的发现引起了人们对前噬菌体研究的关注。因此,对前噬菌体的研究可以为改造宿主和前噬菌体提供基础理论参考。本文对前噬菌体的预测、分布、分类及功能进行了综述,以期为进一步研究噬菌体与宿主间的关系提供基础。
前噬菌体;毒力;抗CRISPR蛋白;溶原转换;群体感应
噬菌体广泛存在于自然界中,是能够感染细菌、真菌、放线菌等微生物的病毒。噬菌体是地球上最多的生物实体,在许多环境中,它们的数量超过宿主约10倍[1]。噬菌体往往遵循两个生命周期之一:裂解或溶原(图1)[2]。烈性噬菌体采用裂解生命周期,它们感染细菌宿主,利用宿主细胞的资源制造更多的噬菌体颗粒,最终裂解细胞,将后代病毒粒子释放到环境中。溶原噬菌体将它们的基因组整合到宿主染色体中的特定附着位置,并与宿主保持稳定的联系,其中整合在细菌基因组中的噬菌体基因组称为前噬菌体;而带有前噬菌体基因组的细菌称为溶原性细菌。某些前噬菌体能够自发地、或被某些理化和生物因素诱导,从而脱离宿主菌基因组进入溶菌周期,形成成熟噬菌体,最终导致细菌裂解;前噬菌体既可以整合到宿主的染色体上,也可以以游离质粒的形式长期存在于宿主细胞中[3](图1)。
由于基因水平转移等作用,前噬菌体广泛存在于细菌中。几乎所有测序的致病和非致病细菌基因组中都发现了噬菌体样元件,而在真菌基因组鲜有报道,仅有一些RNA病毒。基因组研究揭示噬菌体基因组约占某些细菌基因组的20%[4]。越来越多的研究表明:在宿主进化过程中选择了某些前噬菌体基因,而它们编码的产物在一定条件下对宿主有利。如大肠杆菌() K-12的隐蔽前噬菌体与几种宿主表型有关,包括生物膜形成、压力敏感性和抗生素抗药性[5];大多数细菌染色体包含“隐蔽的”前噬菌体,它们丢失了生产噬菌体后代所需的基因,但保留了功能未知的基因,这些基因对调节细菌宿主生理具有重要作用。因此对前噬菌体的研究有助于全面理解噬菌体,同时也有利于完善基因组注释数据。本文将从前噬菌体的预测、分布、前噬菌体元件的分类及功能等方面展开综述。
1 噬菌体预测工具
随着计算机生物信息学技术的广泛运用、新测序细菌基因组数量的不断增加以及人们对前噬菌体序列在细菌DNA中所占比例的认识,研究者开发出了预测细菌基因组中前噬菌体的软件,见表1。早期开发的PhiSpy软件通过监测与测序基因组没有相似性的噬菌体特征来识别噬菌体[6],VirSorter利用已知病毒数据库中的比对和相似性搜索预测宏基因组数据中病毒的重叠群[7];VirFinder用于从宏基因组学数据中识别原核病毒序列,其基于k-mer特征的机器学习来预测重叠群是否为噬菌体[8];MetaPhinder通过与全基因组噬菌体序列数据库进行比较,在宏基因组学数据库中识别噬菌体起源的组装基因组片段(即重叠群)[9];PHASTER用于快速识别和注释细菌基因组和质粒中的噬菌体序列[10];2018年,Amgarten等[11]开发了一种预测宏基因组库中双链DNA噬菌体序列的工具MARVEL,并将MARVEL的性能与VirSorter和VirFinder进行比较,发现尽管3个程序都有很好的特异性,但MARVEL灵敏度更高,能够在宏基因组文库中鉴定出比以前更多的噬菌体序列;近年来开发了Prophage Hunter,它提供一站式web服务,从细菌基因组中提取前噬菌体基因组,评估前噬菌体的活性,识别系统发育相关的噬菌体,并注释噬菌体蛋白质的功能[12]。

图1 噬菌体的不同生命循环周期
噬菌体采用不同的生命周期:烈性噬菌体遵循其复制的裂解周期,并在周期结束时导致宿主细胞的裂解;温和噬菌将其基因组整合到宿主细菌染色体中。前噬菌体DNA与细菌染色体一起复制,并传给子细胞。在某些条件下,包括造成DNA损伤的各种压力,前噬体会被切除并进入裂解循环,而一些噬菌体也会以质粒的形式存在。根据参考文献[2]修改绘制。
2 前噬菌体的分布
目前正在研究的许多噬菌体是从溶原性细菌释放后分离出来的,因此前噬菌体普遍存在。Stevens等[13]检测了61株粪肠球菌,发现19株含有ɸEf11这种温和噬菌体。噬菌体研究者从38株海洋中共诱导出30株前噬菌体[14]。Schmieger等[15,16]对173株肠炎沙门氏菌(伤寒沙门氏菌血清型)的研究发现136株释放了功能性噬菌体;其他肠炎沙门氏菌血清型鼠伤寒中还发现Fels-1、Fels-2、GIFSY-1、GIFSY-2、GIFSY-3、E34和ST64B等前噬菌体;Schicklmaier等[16]发现107株大肠杆菌中有83株至少释放一种功能性噬菌体类型;Osawa等[17]发现从27个大肠杆菌菌株中释放出51个不同功能的噬菌体;通常用于实验室研究的肠球菌LT2分离物携带四个完整的、功能完整的前噬菌体[18~20];研究表明丝裂霉素C可以诱导170株耶尔森氏菌中的7株合成功能性噬菌体[21]和68株革兰氏阳性乳链球菌中的38株噬菌体或噬菌体样颗粒[22];最近研究者检测了15株柑橘黄龙病菌的基因组,发现每株菌的基因组中包含至少一个前噬菌体序列[23]。当然,这些研究都只能找到最少数量的功能前噬菌体,因为它们依赖于成功诱导和研究清楚的指示菌株。前期研究中,我们通过生物信息学预测发现低温荧光假单胞菌W-6中含有前噬菌体序列,并与侵染W-6的长尾噬菌体VW-6S具有同源性。这些证据表明前噬菌体分布广泛,并且一株细菌可能整合了多种前噬菌体序列。
一些噬菌体基因组序列本身具有独特的特性。例如,某些前噬菌体与它们宿主的基因组具有不同的G+C含量、寡核苷酸频率或密码子[24~26],但这种类型的分析还没有进展到可以明确识别前噬菌体序列的程度。因此,必须通过与已知噬菌体基因的相似性来识别细菌基因组序列中的前噬菌体。将不同菌株的DNA与噬菌体或前噬菌体DNA探针杂交,表明在同一物种的其他分离株中通常存在相关的前噬菌体[27,28]。在对用于相关细菌分离物分型序列的一部分搜索中发现了前噬菌体序列,例如弯曲杆菌属()[29]、奈瑟氏菌属()[30]和乳杆菌()[31]。显然,前噬菌体在种类繁多的细菌中普遍分布。
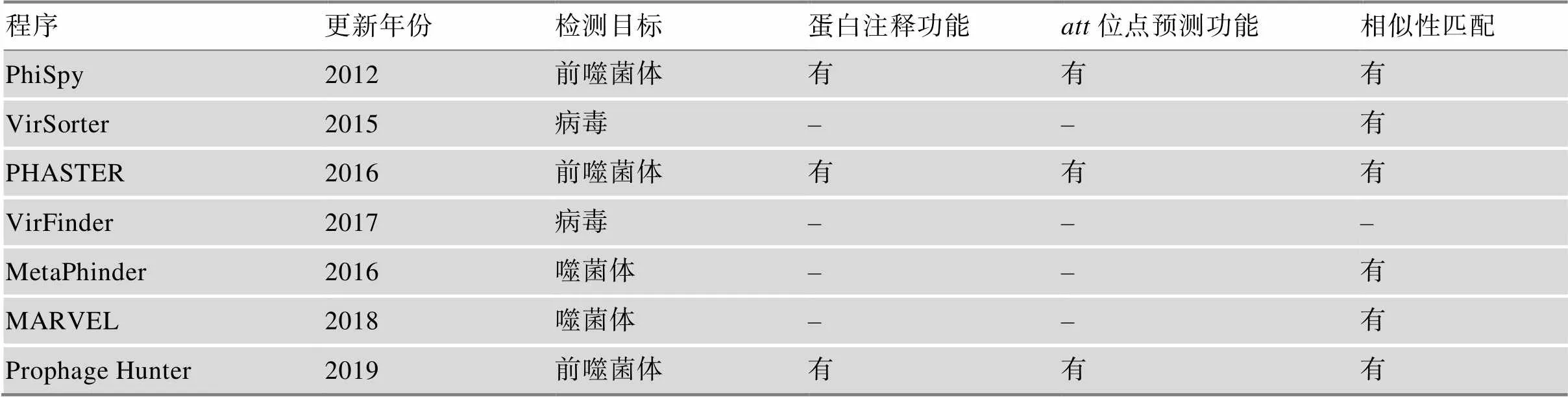
表1 不同噬菌体预测工具及其部分特征
“–”:代表无。
3 前噬菌体样元件分类
整合于细菌基因组内的具有前噬菌体序列特征的DNA片段,依据其序列完整性及是否具有前噬菌体功能,可分为前噬菌体样元件和功能性前噬菌体两类[32]。功能性前噬菌体是指可被诱导的噬菌体,从溶原态转为裂解态,从而具有完整噬菌体功能。
除了可以诱导裂解生长的功能完全的前噬菌体外,还鉴定了其他与前噬菌体相关的实体,即有缺陷的前噬菌体(defective prophages)、卫星前噬菌体(satellite prophages)、细菌素(prophage tail-like bacteriocins)和基因转移剂(gene transfer agent)。Rac是一个在进化过程中丢失了原始序列近60%组分的λ类前噬菌体,也是最早被鉴定的缺陷型前噬菌体。缺陷型前噬菌体的基因序列缺失或基因功能减弱,虽然拥有部分噬菌体功能基因,却不能进入裂菌生长阶段,因此也称隐匿性前噬菌体。K-12的前噬菌体包括Rac[33]、e14[34]、CP4-6、DLP12、cp4- 44、cps-53、cpz-55以及cp4-57等[5],嗜热链球菌M17PTZA496中的隐匿噬菌体,枯草芽孢杆菌的前噬菌体PBSX[35]等均属于隐匿性前噬菌体。卫星前噬菌体携带自主复制模块,缺乏形态发生和结构病毒粒子编码基因,但它们在其他方面是功能性噬菌体;利用另一种“辅助”病毒提供的结构蛋白来组装自己的病毒粒子进而进行繁殖和传播。大肠杆菌的P4噬菌体缺乏编码衣壳蛋白的基因,需要其他辅助噬菌体的作用才能裂解细菌,因此宿主中辅助噬菌体P2基因组存在与否对P4的存在状态起决定性作用[36]。除此之外,霍乱弧菌中的RS1[37]、噬菌体ФAH14a和ФAH14b[38]、噬菌体诱导的染色体岛状元件(phage-inducible chromosomal island-like element)[39],以及最近发现的噬菌体诱导的染色体岛(phage- inducible chromosomal islands)[40]元件家族都属于此类。噬菌体尾状细菌素(prophage tail-like bacteriocins, PTLB)广泛存在于细菌中,是一种与各种噬菌体的尾部结构相似且在进化上与之相关的高分子量杀菌蛋白颗粒。PTLB分为收缩性肌病毒科噬菌体尾巴有关的R型PTLB,和与非收缩性剑叶病毒科噬菌体尾巴有关的F型PTLB[41]。铜绿假单胞菌R型和F型细菌素为典型的噬菌体尾样细菌素[42]。1972年Lotz等[43]对羽扇豆根瘤菌的R型PTLB进行了研究。基因转移因子样前噬菌体是一种尾状噬菌体颗粒,包装细菌基因组的随机片段;由于绝大多数粒子不携带编码GTA的基因,因此这些粒子不能以病毒的形式传播,而且这些粒子含有一个DNA片段,但该片段太短而不能包含全部GTA基因[44];这些类似病毒离子的粒子可以将它们的DNA有效载荷传递到同一物种的另一种细菌中。最早发现的GTA是由荚膜红杆菌()产生的,它是一种紫色的非硫α蛋白细菌,已用作生理学各个方面的模型生物[45]。
4 前噬菌体的功能
目前在许多已测序的细菌基因组中都发现了前噬菌体和前噬菌体残留物,这表明这组可移动的遗传元件在细菌中广泛存在,是形成遗传多样性和菌株变异的主要来源。前噬菌体通过与宿主基因组中的其他噬菌体或感染性噬菌体发生重组促进病毒多样性,但是在这些重组或基因转移之前,前噬菌体在产生新基因过程中的作用未知[46]。前噬菌体基因可以调节宿主菌毒力和生物膜的形成;噬菌体片段还可以赋予宿主免疫力或排除性,保护携带者菌株免受双重感染。Gentile等[47]研究发现一种新的病毒防御系统,在该系统中分枝杆菌噬菌体Sbash前噬菌体与其耻垢分枝杆菌宿主合作,对无关的分枝杆菌噬菌体交叉感染产生高度特异性的防御。
4.1 前噬菌体调节细菌生物膜的形成
细菌生物膜(bacterial biofilm)是指细菌为了达到生存的目的,在黏附于有生命或无生命物体表面后,被胞外自身产生的多聚物基质包裹的有组织的细菌群体。细菌生物膜与大部分菌感染相关,有助于病原菌抵抗外部不利的环境,比如抗生素和抗噬菌体等。生物膜的形成受细菌密度调控[48],前噬菌体的存在也会影响生物膜的形成。研究表明胞外DNA (extracellular deoxyribonucleic acid, eDNA)的抑制降低了粪肠球菌生物被膜的形成,增加了粪肠球菌对NaOCl的敏感性[49]。同样,相比不含前噬菌体的菌株,前噬菌体的存在是生物膜形成增强的原因。例如放线菌中线性质粒样前噬菌体Xhp1促进生物膜形成[50]。相比被敲除前噬菌体的菌株,含有ɸEf11前噬菌体的溶原粪肠球菌生物膜形成增强约50%[13]。噬菌体在细菌生物膜的形成过程中起着重要的作用,在含有丝状噬菌体的菌株中,生物膜形成较稳定[51]。由VP882编码的芽孢杆菌VqmA蛋白同源物是群体敏感物质3,5-二甲基吡嗪2-醇的响应者。宿主的VqmA变异体通过刺激小调控RNA的转录,在高细胞密度下抑制生物膜形成和毒力基因的表达[52]。
4.2 前噬菌体对致病菌毒力的影响
前噬菌体编码许多细菌性病原体的关键毒力因子。病原体中前噬菌体的消除可以降低毒力,而非致病菌株不携带各自的前噬菌体。溶原菌中的前噬菌体可以诱导溶原转化,改变宿主细菌的毒力,所以溶原性转换在毒力菌株中扮演重要作用。前噬菌体还可以对已经进化了的细菌的特性进行“改进”,因此是对其编码的适应性因子进化功能的试金石。
一些溶原噬菌体携带增强细菌宿主毒力的基因,这些基因会导致细菌宿主致病,例如大肠杆菌O157菌株中的噬菌体编码志贺毒素,白喉杆菌中的噬菌体编码白喉毒素和肉毒杆菌中的神经毒素。噬菌体编码的毒素、宿主细胞入侵因子和血清抗性蛋白促进细菌病原体进行感染过程的多个方面。这些基因一旦整合到细菌基因组中,就能使无害的细菌释放出可致病的毒素。志贺毒素是研究最多的毒力因子,其基因位于肠出血性大肠杆菌的λ噬菌体基因组上。许多抗生素促进志贺毒素转化前噬菌体的诱导,前噬菌体被诱导后刺激这些毒素的产生和释放[53]。产毒霍乱弧菌的基因组含有一种产霍乱毒素的前噬菌体—丝状噬菌体(CTXφ),CTXφ被两个卫星噬菌体RS1和TLC包围,而且三者还能以复制形式存在于宿主菌中。在脓毒症和心内膜炎动物模型中,多溶原性粪肠球菌菌株比无前噬菌体的同基因菌株显示出更强的毒力[54]。柑橘黄龙病菌携带的前噬菌体的基因与其毒力相关[23]。单增李斯特菌的致病性与其毒力因子的作用具有重要相关性,同时,毒力基因的调控也是单增李斯特菌在腐生菌和致病菌这两种不同状态之间切换的重要机制,提高了其在不同环境下的生长适应水平。研究表明铜绿假单胞菌中的丝状噬菌体pf4突变体毒力低于野生型菌株[50]。同样,禽致病性大肠杆菌的前噬菌体phiv205-1缺失突变体毒力明显下降[55]。
大多数前噬菌体的基因组不仅包含编码与噬菌体复制、结构和组装有关的功能性基因,而且还包含辅助基因。例如,MG1655噬菌体编码I型、II型和IV型毒素/抗毒素(TA)系统。TA系统是广泛分布于原核生物中的遗传模块。TA基因通常编码杀死细胞或抑制细胞生长的毒素和中和毒素毒性的同原抗毒素,可作为稳定和自我保存噬菌体的手段[56]。根据其作用机理和抗毒素是蛋白质或sRNA,将TA系统分为六类[57]。各种细菌菌株中TA系统的丰富性可能表明了重要的进化作用。TA系统的功能包括持久性、DNA稳定、噬菌体抑制、应激反应和生物膜形成[58]。研究表明Pf4噬菌体编码的II型TA能够控制PAO1中丝状噬菌体的产生,前噬菌体中的毒素抑制了铜绿假单胞菌中丝状噬菌体的产生,同时有助于细胞抵抗Pf4噬菌体感染[59]。RAC噬菌体中毒素ralR的产物是一种DNA酶,I型RalR/Rala TA增加了细胞对磷霉素的耐药性[60]。自发前噬菌体诱导机制有助于肺炎链球菌血清型19A ST320菌株的竞争优势,祖先菌株通过前噬菌体的切除和整合来调节ychF的表达进而增加细菌的粘附[61]。
4.3 温和噬菌体窃听宿主的群体感应(quorum sensing, QS)信号,控制自身的溶原–裂解开关
宿主细胞密度影响噬菌体裂解-溶原的转换,表明温和噬菌体可能利用特定的机制来感知宿主细胞密度。早期细胞生理学和最适感染复数(multiplicity of infection, MOI)有利于溶原性,这表明可能在共感染噬菌体之间发生一些细胞内通讯,以做出有利于两种入侵噬菌体的选择。但是,共同感染的噬菌体如何在裂解和溶原这两个相反的生存途径上折中仍然是个难题。Ghosh等[62]首次证明依赖细胞密度的前噬菌体诱导与群体感应系统有关。宿主细胞内的噬菌体具有进化的机制以感知其宿主密度,并在溶原-裂解之间做出协调决定,以便噬菌体可以根据宿主细胞密度估计后代噬菌体的成功感染率,从而最大限度地繁殖和避免耗尽敏感宿主。
枯草芽孢杆菌噬菌体SPbeta的温和型噬菌体可以感知近亲的种群水平,利用一种基于宿主裂解时释放的特定六肽的噬菌体QS——称为“仲裁系统”来影响裂解–溶原决定[63]。噬菌体可产生不同的特异性肽,通过感知特异性肽的局部浓度,新感染的噬菌体能够对以前的裂解事件在空间和时间上的邻近程度做出估计,从而调整自身关于裂解的决定;特异性肽水平高,表明可用的宿主很少,则进行溶原。当然这些机制都是有限的,因为噬菌体只能间接地或通过利用噬菌体衍生的信号来感知细胞外环境。Silpe和Bassler等[52]展示了霍乱弧菌的温和噬菌体VP882是如何窃听宿主的QS系统,直接读出宿主的种群大小作为裂解/溶原决定的输入。噬菌体的繁殖取决于细菌宿主细胞,因此对于温和噬菌体来说,响应宿主细胞的密度调节其繁殖策略至关重要,进而阐明了宿主的QS控制弧菌噬菌体做出的依赖宿主细胞密度的裂解–溶原性决定的机制。因此在溶原过程中(而不仅仅是在裂解感染过程中)会产生信号的类似仲裁系统,以告知噬菌体局部溶原物的丰度的说法引起了科学家的关注。
5 前噬菌体的进化策略
噬菌体通常被认为是细菌的天敌。然而,对噬菌体-宿主动力学的进化分析表明,捕食者和猎物经常以一种避免完全灭绝的方式共同进化。因此,在噬菌体与宿主共进化过程中,部分噬菌体迫于进化压力,将自身基因整合到宿主菌中,随宿主DNA一起复制而得以生存,人们将这种方式成为噬菌体的溶源转换。溶源转化只是温和噬菌体影响细菌适合性5种方式之一:(1)作为基因组重排的锚点;(2)通过基因破坏;(3)通过防止裂解感染;(4)通过原噬菌体诱导竞争菌株的裂解;(5)通过引入新的适合性因子(溶原转化、转导)。
在这场进化战争中,细菌从噬菌体感染的第一步吸附入手,发展了几种吸附阻断机制,例如,用各种蛋白质掩蔽噬菌体受体,产生细胞外基质或竞争抑制剂,一些已知的系统(CRISPR系统)可以阻止噬菌体DNA进入。除此之外,在噬菌体复制过程中存在抗病毒防御机制、限制修饰系统(R-M)和流产感染等。作为对噬菌体感染的细菌战的回应,噬菌体反过来编码细菌免疫系统的抑制剂以增强裂解宿主菌或整合到其基因组中的能力。噬菌体编码的“抗CRISPR”(anti-CRISPR, ACR)蛋白的第一个例子来自铜绿假单胞菌中的I型I-F和I-E系统[64,65]。在铜绿假单胞菌中,I型抗CRISPR是从整合的噬菌体基因组也就是前噬菌体中表达的,并导致宿主CRISPR- Cas系统的结构性失活[65]。目前已发现10个I-F型抗CRISPR基因和4个I-E型抗CRISPR基因[64],它们都编码功能未知的独特的小蛋白(50~150个氨基酸)。Bondy-Denomy等[65]对4种I-F抗CRISPR蛋白的生化研究表明,它们直接与CRISPR-Cas复合物中的不同Cas蛋白相互作用,来防止靶DNA的识别或切割。单增李斯特菌前噬菌体编码几种独特的II-A型CRISPR-Cas9抑制剂蛋白,抗CRISPR蛋白灭活李斯特菌CRISPR-Cas9。研究表明ACR噬菌体需要协同才能克服CRISPR免疫宿主的部分抗性,如果ACR噬菌体的初始滴度低于临界阈值会导致ACR噬菌体灭绝,但当它们的滴度超过这个阈值时,它们就会放大[66]。溶原过程中噬菌体在宿主内保持休眠状态,通常将自身整合到宿主染色体中,这使得噬菌体可以作为细菌基因组的一部分进行复制,并赋予细胞免疫力,使其免受二次感染。当相互作用时,细菌和噬菌体可以进化,这种关系被描述为对抗性共进化,但这种模式并不适合所有的模式;研究发现当宿主菌和噬菌体共同生长时,有利于细菌而不是共同进化。
6 结语与展望
前噬菌体在宿主生物膜、毒力以及噬菌体与宿主的进化等方面扮演重要作用。虽然这里以单一作用的形式逐次描述,但这些作用之间往往相互关联,例如前噬菌体增强了病原菌生物膜的形成,从而增强了病原菌的毒性。生物膜的形成给致病菌带来了更强的耐药性,这使得临床治疗和面临巨大的挑战。因此对前噬菌体的全面了解,有利于探寻清除生物膜的途径,从而为医疗手段提供新思路。
随着分子生物学及生物信息学等多学科的发展,前噬菌体受到越来越多的关注,部分研究者已经注意到前噬菌体给致病菌带来抗逆境作用以及生态方面的意义,但这方面的研究还相对较少。另一方面前噬菌体编码监听宿主菌的群体感应通信,来给裂解噬菌体传达信号,对这种噬菌体之间的交流也才刚刚起步。温和噬菌体和裂解噬菌体之间在交流什么,怎么交流,是否还存在别的交流方式?这需要进一步深入的研究。在各学科和多种手段的结合下,相信这些问题都会有答案。
[1] Bondy-Denomy J, Qian J, Westra ER, Buckling A, Guttman DS, Davidson AR, Maxwell KL. Prophages mediate defense against phage infection through diverse mechanisms., 2016, 10(12): 2854–2866.
[2] Fortier LC, Sekulovic O. Importance of prophages to evolution and virulence of bacterial pathogens., 2013, 4(5): 354–365.
[3] Shen P, Chen XD. Microbiology 8th Edition. Beijing: Higher Education Press, 2016, 176.沈萍陈向东. 微生物学第8版. 北京: 高等教育出版社. 2016, 176.
[4] Fernández L, Rodríguez A, García P. Phage or foe: an insight into the impact of viral predation on microbial communities., 2018,12(5): 1171–1179.
[5] Wang XX, Kim Y, Ma Q, Hong SH, Pokusaeva K, Sturino JM, Wood TK. Cryptic prophages help bacteria cope with adverse environments., 2010, 1: 147.
[6] Akhter S, Aziz RK, Edwards RA. PhiSpy: a novel algorithm for finding prophages in bacterial genomes that combines similarity- and composition-based strategies., 2012, 40(16): e126.
[7] Roux S, Enault F, Hurwitz BL, Sullivan MB. VirSorter: mining viral signal from microbial genomic data., 2015, 3: e985.
[8] Ren J, Ahlgren NA, Lu YY, Fuhrman JA, Sun FZ. VirFinder: a novel k-mer based tool for identifying viral sequences from assembled metagenomic data.. 2017, 65(1): 69.
[9] Jurtz VI, Villarroel J, Lund O, Voldby Larsen M, Nielsen M. MetaPhinder-Identifying bacteriophage sequences in metagenomic data sets., 2016, 11(9): e0163111.
[10] Arndt D, Marcu A, Liang YJ, Wishart DS. Phast, Phaster and Phastest: Tools for finding prophage in bacterial genomes., 2019, 20(4): 1560–1567.
[11] Amgarten D, Braga LPP, da Silva AM, Setubal JC. Marvel, a tool for prediction of bacteriophage sequences in metagenomic bins., 2018, 9: 304.
[12] Song WC, Sun HX, Zhang C, Cheng L, Peng Y, Deng ZQ, Wang D, Wang Y, Hu M, Liu W, Yang HM, Shen Y, Li JH, You LC, Xiao MF. Prophage Hunter: an integrative hunting tool for active prophages., 2019; 47(W1): W74–W80.
[13] Stevens RH, Zhang HM, Sedgley C, Bergman A, Manda AR. The prevalence and impact of lysogeny among oral isolates of Enterococcus faecalis., 2019, 11(1): 1643207.
[14] Ma RJ. Citromicrobium bathyomarinum: diversity, activity and interaction with hosts., 2018.马瑞洁. 海洋Citromicrobium bathyomarinum前噬菌体的多样性、生理生态特性及其与宿主的相互作用. 厦门大学, 2018.
[15] Schmieger H, Schicklmaier P. Transduction of multiple drug resistance of Salmonella enterica serovar typhimurium DT104., 1999, 170(1): 251–256.
[16] Schicklmaier P, Moser E, Wieland T, Rabsch W, Schmieger H. A comparative study on the frequency of prophages among natural isolates of Salmonella and Escherichia coli with emphasis on generalized transducers., 1998, 73(1): 49–54.
[17] Osawa RO, Iyoda S, Nakayama SI, Wada A, Yamai S, Watanabe H. Genotypic variations of Shiga toxin-converting phages from enterohaemorrhagicO157: H7 isolates., 2000, 49(6): 565–574.
[18] Yamamoto N. Genetic evolution of bacteriophage. I. Hybrids between unrelated bacteriophages P22 and Fels 2.,1969, 62(1): 63–69.
[19] McClelland M, Florea L, Sanderson K, Clifton SW, Parkhill J, Churcher C, Dougan G, Wilson RK, Miller W. Comparison of theK-12 genome with sampled genomes of apneumoniae and three salmonella enterica serovars, Typhimurium, Typhi and Paratyphi., 2000, 28(24): 4974–4986.
[20] Figueroa-Bossi N, Bossi L. Inducible prophages contribute to Salmonella virulence in mice., 1999, 33(1): 167–176.
[21] Popp A, Hertwig S, Lurz R, Appel B. Comparative study of temperate bacteriophages isolated from Yersinia., 2000, 23(4): 469–478.
[22] Huggins AR, Sandine WE. Incidence and properties of temperate bacteriophages induced from., 1977, 33(1): 184–191.
[23] Dominguez-Mirazo M, Jin R. Weitz JS. Functional and Comparative Genomic Analysis of Integrated Prophage-LikeSequences in “Liberibacter asiaticus”., 2019, 4(6): e00409– e00419.
[24] Castillo D, Middelboe M. Genomic diversity of bacteriophages infecting the fish pathogen., 2016, 363(24):fnw272.
[25] Petrov VM, Ratnayaka S, Nolan JM, Miller ES, Karam JD. Genomes of the T4-related bacteriophages as windows on microbial genome evolution., 2010, 7: 292.
[26] Blaisdell BE, Campbell AM, Karlin S. Similarities and dissimilarities of phage genomes.,1996,93(12): 5854–5859.
[27] Agron PG, Walker RL, Kinde H, Sawyer SJ, Hayes DC, Wollard J, Andersen GL. Identification by subtractive hybridization of sequences specific forserovar enteritidis., 2001, 67(11): 4984–4991.
[28] Chang CC, Gilsdorf JR, DiRita VJ, Marrs CF. Identification and genetic characterization ofinfluenzae genetic island 1., 2000, 68(5): 2630–2637.
[29] Dep MS, Mendz GL, Trend MA, Coloe PJ, Fry BN, Korolik V. Differentiation betweenandusing genotypic and phenotypic analyses.,2001, 51(Pt 3): 819– 826.
[30] Klee SR, Nassif X, Kusecek B, Merker P, Beretti JL, Achtman M, Tinsley CR. Molecular and biological analysis of eight genetic islands that distinguishmeningitidis from the closely related pathogengonorrhoeae., 2000, 68(4): 2082–2095.
[31] Brandt K, Tilsala-Timisjärvi A, Alatossava T. Phage-related DNA polymorphism in dairy and probiotic., 2001, 32(1): 59–65.
[32] Hu FQ, Li S. Prophage., 2009, 36(3): 432– 438.胡福泉, 黎庶. 前噬菌体. 微生物学通报, 2009, 36(3): 432–438.
[33] Krishnamurthi R, Ghosh S, Khedkar S, Seshasayee ASN. Repression of YdaS toxin is mediated by transcriptional repressor RacR in the cryptic rac prophage ofK-12.,2017, 2(6): e00392–17.
[34] Mehta P, Casjens S, Krishnaswamy S. Analysis of the lambdoid prophage element e14 in theK-12 genome., 2004, 4: 4.
[35] Jing TT, Fu ZW, Shen P, Chen XD. General investigation of PBSX-like defective prophages resident instrains., 2016, 43(6): 1244–1252.晋婷婷, 付正伟, 沈萍, 陈向东. 枯草芽孢杆菌携带PBSX类缺陷性原噬菌体的普遍性调查. 微生物学通报, 2016, 43(6): 1244–1252.
[36] Ruzin A, Lindsay J, Novick RP. Molecular genetics of SaPI1--a mobile pathogenicity island in., 2001, 41(2): 365–377.
[37] Rezaie N, Bakhshi B, Najar-Peerayeh S. The role of CTX and RS1 satellite phages genomic arrangement intoxin production in two recent cholera outbreaks (2012 and 2013) in IR Iran., 2017, 112: 89–94.
[38] Dziewit L, Radlinska M. Two inducible prophages of an antarctic. ANT_H14 use the same capsid for packaging their genomes - characterization of a novel phage helper-satellite system., 2016, 11(7): e0158889.
[39] Dieterle ME, Fina Martin J, Durán R, Nemirovsky SI, Rivas CS, Bowman C, Russell D, Hatfull GF, Cambillau C, Piuri M. Characterization of prophages containing “evolved” Dit/Tal modules in the genome ofBL23., 2016, 100(21): 9201–9215.
[40] Crestani C, Forde TL, Zadoks RN. Development and application of a prophage integrase typing scheme for group B., 2020, 11: 1993.
[41] Chen J, Zhu Y, Yin M, Xu Y, Liang X, Huang YP. Characterization of maltocin S16, a phage tail-like bacteriocin with antibacterial activity againstand.,2019, 127(1): 78–87.
[42] Nakayama K, Takashima K, Ishihara H, Shinomiya T, Kageyama M, Kanaya S, Ohnishi M, Murata T, Mori H, Hayashi T. The R-type pyocin ofis related to P2 phage, and the F-type is related to lambda phage., 2000, 38(2): 213–231.
[43] Lotz W, Mayer F. Isolation and characterization of a bacteriophage tail-like bacteriocin from a strain of., 1972, 9(1): 160–173.
[44] Lang AS, Zhaxybayeva O, Beatty JT. Gene transfer agents: phage-like elements of genetic exchange., 2012, 10(7): 472–482.
[45] Marrs B. Genetic recombination in., 1974, 71(3): 971–973.
[46] Wahl LM. Pattenden T. Prophage provide a safe haven for adaptive exploration in temperate viruses., 2017, 206(1): 407–416.
[47] Gentile GM, Wetzel KS, Dedrick RM, Montgomery MT, Garlena RA, Jacobs-Sera D, Hatfull GF. More evidence of collusion: a new prophage-mediated viral defense system encoded by mycobacteriophage Sbash., 2019, 10(2): e00196–19.
[48] Chen YF, Liu SY, Liang ZB, Lu MF, Zhou JL, Zhang LH. Quorum sensing and microbial drug resistance., 201638 (10): 881–893.陈昱帆, 刘诗胤, 梁志彬, 吕明发, 周佳暖, 张炼辉. 群体感应与微生物耐药性. 遗传, 2016, 38(10): 881–893.
[49] Yu MK, Kim MA, Rosa V, Hwang YC, Del Fabbro M, Sohn WJ, Min KS. Role of extracellular DNA inbiofilm formation and its susceptibility to sodium hypochlorite., 2019, 27: e20180699.
[50] Shen MY, Yang YH, Shen W, Cen LJ, McLean JS, Shi WY, Le S, He XS. A linear plasmid-like prophage of actinomyces odontolyticus promotes biofilm assembly., 2018, 84(17): e01263–18.
[51] Rice SA, Tan CH, Mikkelsen PJ, Kung V, Woo J, Tay M, Hauser A, McDougald D, Webb JS, Kjelleberg S. The biofilm life cycle and virulence ofare dependent on a filamentous prophage., 2009, 3(3): 271–282.
[52] Silpe JE, Bassler BL. A host-produced quorum-sensing autoinducer controls a phage lysis-lysogeny decision., 2019, 176(1–2): 268–280
[53] Loś JM, Loś M, Węgrzyn G. Bacteriophages carrying Shiga toxin genes: genomic variations, detection and potential treatment of pathogenic bacteria., 2011, 6(8): 909–924.
[54] Rossmann FS, Racek T, Wobser D, Puchalka J, Rabener EM, Reiger M, Hendrickx AA, Diederich AK, Jung K, Klein C, Huebner J. Phage-mediated dispersal of biofilm and distribution of bacterial virulence genes is induced by quorum sensing., 2015, 11(2): e1004653.
[55] Liu Y, Gong QW, Qian XJ, Li DZ, Zeng H, Li YH, Xue F, Ren JL, Zhu Ge XK, Tang F, Dai JJ. Prophage phiv205-1 facilitates biofilm formation and pathogenicity of avian pathogenicstrain DE205B., 2020, 247: 108752.
[56] Cui P, Xu T, Zhang WH, Zhang Y. Molecular mechanisms of bacterial persistence and phenotypic antibiotic resistance., 2016, 38(10): 859–871.崔鹏, 许涛, 张文宏, 张颖. 细菌持留与抗生素表型耐药机制. 遗传, 2016, 38(10): 859–871.
[57] Unterholzner SJ, Poppenberger B, Rozhon W. Toxin- antitoxin systems: biology, identification, and application., 2013, 3(5): e26219.
[58] Zander I, Shmidov E, Roth S, Ben-David Y, Shoval I, Shoshani S, Danielli A, Banin E. Characterization of PfiT/PfiA toxin-antitoxin system ofthat affects cell elongation and prophage induction., 2020, 22(12): 5048–5057.
[59] Li YM, Liu XX, Tang KH, Wang WQ, Wang YX, Wang XX. Prophage encoding toxin/antitoxin system PfiT/PfiA inhibits Pf4 production in., 2020. 13(4): 1132–1144.
[60] Guo YX, Quiroga C, Chen Q, McAnulty MJ, Benedik MJ, Wood TK, Wang XX. RalR (a DNase) and RalA (a small RNA) form a type I toxin-antitoxin system in., 2014, 42(10): 6448–6462.
[61] Chen YY, Wang JT, Lin TL Gong YN, Li TH, Huang YY, Hsieh YC. Prophage excision inpneumoniae serotype 19A ST320 promote colonization: insight into its evolution from the ancestral clone taiwan 19F-14 (ST236)., 2019, 10: 205.
[62] Ghosh D, Roy K, Williamson KE, Srinivasiah S, Wommack KE, Radosevich M. Acyl-homoserine lactones can induce virus production in lysogenic bacteria: an alternative paradigm for prophage induction., 2009, 75(22):7142–7152.
[63] Erez Z, Steinberger-Levy I, Shamir M, Doron S, Stokar-Avihail A, Peleg Y, Melamed S, Leavitt A, Savidor A, Albeck S, Amitai G, Sorek R. Communication between viruses guides lysis-lysogeny decisions.,2017, 541(7638): 488–493.
[64] Pawluk A, Staals RHJ, Taylor C, Watson BNJ, Saha S, Fineran PC, Maxwell KL, Davidson AR. Inactivation of CRISPR-Cas systems by anti-CRISPR proteins in diverse bacterial species., 2016, 1(8): 16085.
[65] Bondy-Denomy J, Pawluk A, Maxwell KL, Davidson AR. Bacteriophage genes that inactivate the CRISPR/Cas bacterial immune system., 2013, 493(7432): 429– 432.
[66] Landsberger M, Gandon S, Meaden S, Rollie C, Chevallereau A, Chabas H, Buckling A, Westra ER, Houte S. Anti- CRISPR phages cooperate to overcome CRISPR-Cas immunity., 2018, 174(4): 908–916.
Research progress of prophages
Xuemei Chen, Yunlin Wei, Xiuling Ji
,,,
As the most abundant biological entities on earth, bacteriophages (phages) were considered as the antagonists of bacteria. With the rapid development of genomics and molecular biology technologies, a subtle and complex relationship between phages and their host bacteria has been uncovered. Prophage refers to an intracellular form of a bacteriophage, which is usually integrated into the hereditary material of the host. Prophage is ubiquitously distributed in bacterial genomes. It reproduces when the host does and can affect important biological properties of their bacterial hosts, such as virulence, biofilm formation and host immunity. Interestingly, prophages were also involved in regulating the lysogeny-lytic state by “monitoring” the quorum sensing of bacteria. Recently, anti-CRISPR proteins encoded by prophages were found, which attracts a lot of attention. In this review, we summarized the prediction, distribution, classification and functions of prophages to lay a foundation for further studying interactions between phages and bacteria.
prophage; virulence; anti-CRISPR protein; lysogenic conversion; quorum sensing
2020-11-27;
2021-01-12
国家自然科学基金项目(编号:31860147,31700324)资助[Supported by the National Natural Science Foundation of China (Nos. 31860147, 31700324)]
陈学梅,在读硕士研究生,专业方向:低温微生物。E-mail: 1179823820@qq.com
季秀玲,博士,教授,研究方向:低温微生物。E-mail: jixiuling1023@126.com
10.16288/j.yczz.20-355
2021/1/18 10:50:43
URI: https://kns.cnki.net/kcms/detail/11.1913.R.20210115.1427.002.html
(责任编委: 张天宇)

