蛋白质结构域划分方法及在线服务综述
(华中科技大学 a.生命学院; b.软件学院, 湖北 武汉 430074)
随着大量物种全基因组测序的完成,以测定蛋白质结构为目的的结构基因组学和以研究蛋白质功能为目的的蛋白质组学成为当前研究热点之一.根据蛋白质三级结构的测定和功能研究,有利于增深对疾病发生的分子机制理解,从而有助于开发新的手段与方法来预防、诊断疾病和新药研发[1-2].
结构域是蛋白质的一个结构层次, 可以看作是蛋白质结构、折叠、功能、进化和设计的基本单位.根据PDB数据库统计[3-4],已知结构蛋白质中约40%为多结构域蛋白[5].结构域的不同组合使多结构域蛋白质具有不同的三级结构和功能.准确识别蛋白质结构域对结构基因组学选择目标序列、结构解析至关重要,也是预测和理解蛋白质功能关键的一步.自1973年以来,若干研究者就蛋白质结构域划分问题进行研究,可归纳为从实验测定三维结构着手的结构域划分方法和不依赖三维结构仅从序列出发的结构域划分方法.前者的代表性工作包括 Wetlaufer[6]首次提出的基于原子间接触密度划分结构域的方法,以及后期Domain Parser[7-9]、PDP等方法[10];后者的代表性工作包括DROP[11-12]、Dompro[13]、DOBO[14]、ThreaDom等[15].
目前已建立一些结构域数据和在线预测的服务系统,例如,Pfam[16-17]、SMART[18-19]、SCOP[20-21]、CATH[22-23]、InterPro[24]、ThreaDomEx[25]等.据2016年2月份的数据统计,当前最完整的蛋白质序列数据库(UniProt)中去掉重复序列后有74 897 059条序列,该数据库的结构域注释主要来自Pfam、SMART、SCOP、CATH以及InterPro等结构域数据库,其中只有36 449 183(48.7%)的序列有结构域注释.其中被研究工作者熟知、并广泛使用的Pfam结构域数据库注释了33 529 428条序列.究其主要原因:已解析三级结构的蛋白质及其近同源蛋白质序列只占有较小的比例,当前技术还无法较大规模地从序列注释远同源蛋白质结构域.本文从蛋白质结构域识别问题的提出、结构域边界预测、不连续结构域检测及相关在线服务情况进行介绍,供相关研究者参考.
1 结构域识别问题
蛋白质结构域识别问题不仅要准确识别蛋白质结构域划分边界,还要准确检测出组成蛋白质结构域的序列片段(即不连续结构域).
以多结构域蛋白4-α-葡聚糖转移酶(PDB:1LWH)为例来说明结构域识别过程,从序列出发的结构域识别过程包括结构域边界预测和不连续结构域检测2个步骤.图1a 是4-α-葡聚糖转移酶的蛋白质结构图,图1b是该蛋白结构域示意图.从图1a可以看出该蛋白包含3个结构域:[1~93(紫红色)|159~391(红色)]、[94~158(黄色)]、[392~441(蓝色)].识别该蛋白的结构域的过程:首先确定结构域边界HIS93、ASN158、ARG391,这3个残基将该蛋白分为4段; 然后检测不连续结构域.对该蛋白, 第1段[1~93]与第3段[159~391]构成不连续结构域,这从图1b中可以更清晰的看出,A1[1~93]和A2[159~391]在序列上不临近,但在三级结构上是一个结构域(即不连续结构域).

图1 4-α-葡聚糖转移酶结构与结构域示意图Fig.1 Schematic diagram of structure and domain of 4-α-glucanotransferase
一个优秀的结构域划分工具需要准确的判断出在氨基酸序列位置93(94)、158(159)、391(392)3个位置附近存在结构域划分边界,即把序列划分为(1~93))(94~158)(159~391)(392~441)4个片段;同时要应该具有将片段(1~93)和片段(159~391)组装成一个结构域的能力(不连续结构域检测).对不具备这2种能力的结构域划分的工具来说,至少是不完美的.
结构域划分问题又分为从结构出发的结构域划分和从序列出发的结构域划分.对从结构出发的结构域划分是根据序列对应的3D结构进行空间上的结构域划分;对于从序列出发的结构域划分,则不使用3D结构,只根据序列信息进行预测或检测,以进行结构域划分.多数基于结构的结构域划分空间考虑了不连续结构域划分,而只有少数几个从序列出发的结构域划分工具考虑了不连续结构域检测问题.
2 从结构出发的蛋白质结构域划分方法
从通过实验获得蛋白质三维结构开始,通过把蛋白质分子划分为小的域进行研究,可以降低研究的复杂程度.而多数情况下,蛋白质域的定义是指从结构上讲的域,即结构域.结构域是一个具有以下特征的蛋白质结构单元[26]:①是紧密的;②是稳定的;③含有一个疏水核心;④可以独立蛋白质的其他部分进行单独折叠;⑤可以跟其它结构域结合并出现在其他蛋白质中;⑥行使特定的功能.根据这一定义,不仅有专家手工定义的结构域划分数据库如SCOP[20,27-28]、CATH[29]等,还有其他自动划分工具.Rossman等[30]根据给出结构Cα-Cα距离图进行结构域划分;Crippen[31]采用聚类的方法进行结构域划分;Rose[32]采用将3D空间投影到2D空间的方法进行结构域划分;Wodak等[33]通过发现2个结构域间最小接触界面进行结构域划分;Holm等[34]使用刚体震动构建的接触矩阵开发PUU方法;Swindells[35]通过构建疏水核心进行结构域划分;Islam等[36]采用发现结构域间最小接触进行结构域划分;Siddiqui等[37]通过计算结构域内外最大比值进行结构域划分;Sowdhamini等[38]通过二级结构域聚类的方法进行结构域划分;Taylor[39]采用残基间空间接近度模型进行划分;Wernisch 等[40]利用Kernighan-lin图启发式算法,发现结构域间最小接触进行结构域划分;Xu等[8]利用图论中最大流和最小割方法,发现结构域最小接触进行切割;Xuan等[41]使用模糊聚类对基础片段组装的方法进行结构域划分;Alexandrov等[10]利用结构域接触最小数量进行结构域划分;Berezovsky[42]使用原子间范德华接触进行聚类的方法进行结构域划分;Kundu等[43]利用高斯网络模型进行结构域划分.这些方法可以归纳为自下向上的方法或自上向下的方法,指用从小的基本片段开始组装,或者总体进行划分,再由某种准则判断划分.很明显,通过原子间接触作为量度,成为从结构进行结构域划分的主要手段.图2 给出了Xu等[8]开发的Domain Parser以原子间相互作用为量度的从上到下的一种方法,其将蛋白质结构用一个网络表示,网络的节点为氨基酸残基,边表示残基间相互作用,然后用最大流最小割的方法进行结构域划分.2个氨基酸相互作用的强度可以视为边的容量,并是如下量的函数:残基间原子的接触数量、残基间主链接触数、跨β折叠的相互作用、是否属于同一个β折叠.
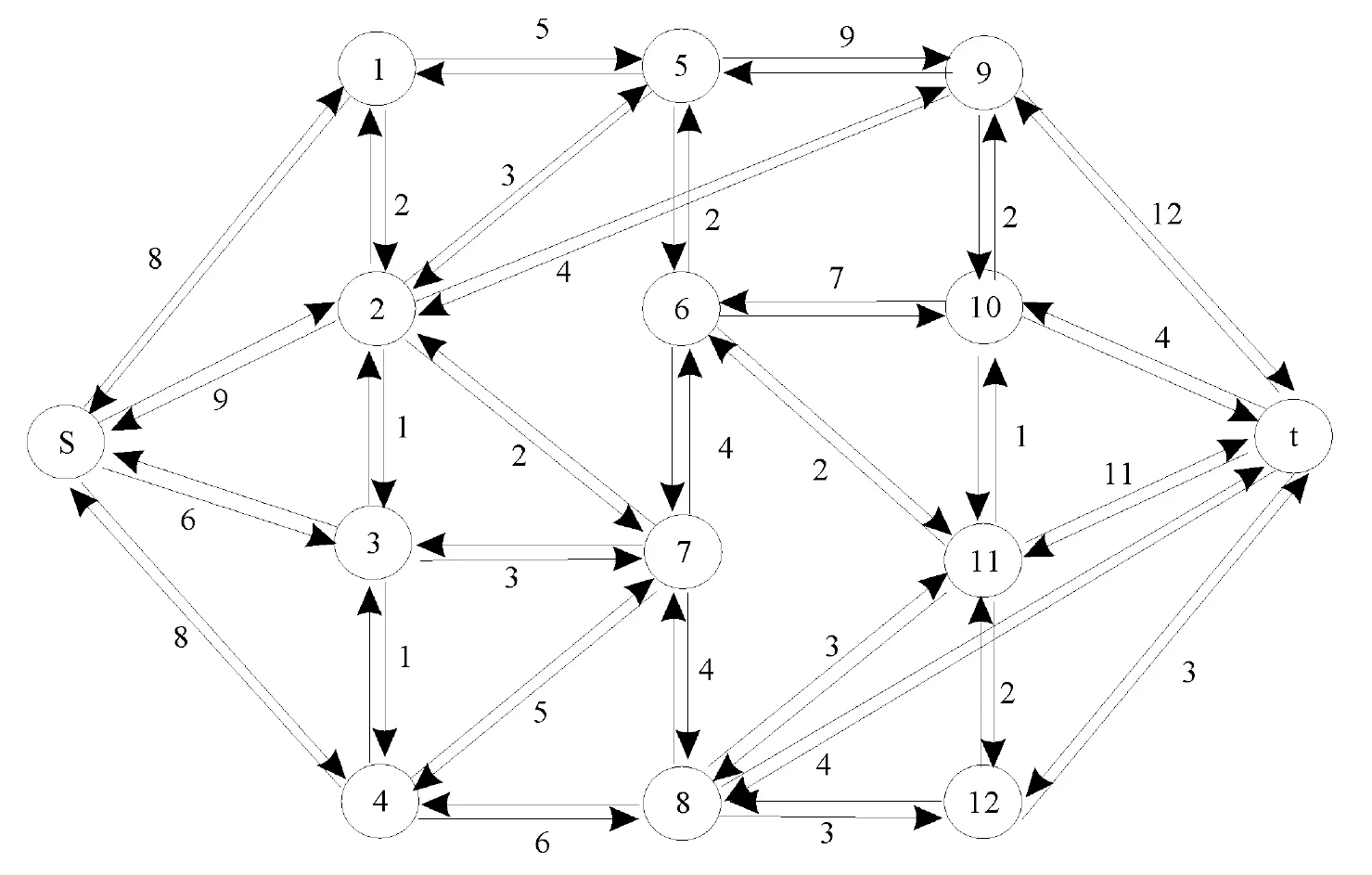
图2 Domain Parser 蛋白质图表示
Fig.2 Protein representation based on graph in Domain Parser
采用最大流最小割的方法进行的基本过程:给图增加一个虚拟的源s和槽节点t,采用最大流最小割原理寻找将蛋白质分成2个结构域的瓶颈边,然后删除这些边,网络被表示为一个跟源s相连接的网络,一个跟槽t相连接的网络,并各自代表了被划分出来的结构域.重复此过程,直到满足终止条件结束.
基于结构的蛋白质结构域边界划分思路和方法,也可以结合预测特征应用到从序列划分结构域问题.
3 从序列预测结构域边界
从序列预测/划分结构域边界主要包括基于同源比对、三级结构预测和机器学习等3类方法.
同源比对方法是识别结构域边界的最基本方法.例如Pfam等采用多序列比对,搜索序列间相似度高的蛋白质结构域家族,从而确定结构域边界.再如FIEFDOM通过PSI-blast搜索已知结构近同源模板,推知结构域边界[44].该类方法在不存在已知结构的近同源蛋白的情况下,无法完成结构域识别.Xue等[15]开发的ThreaDom,探索基于远同源比对方法进行蛋白质结构域预测研究,取得较好的结果.
采用预测的三级结构识别结构域,是一种很直观的方法,可以采用诸如Modeller[45-46]、I-Tasser[47]等三级结构建模软件进行结构建模,再使用诸如Domain parser等从结构出发的结构域划分工具进行结构域划分,如SnapDRAGON[48]、 RosettaDom[49]、 OPUS-DOM[50]等.此类方法不仅依赖于找到近同源模板,而且会受到目标序列长度的限制.况且,结构域识别的重要应用之一就是支持结构预测,因而这种方法也有诸多限制.
在没有近同源模板的情况下,研究者主要使用基于统计或机器学习的方法预测蛋白质结构域的边界.DGS根据序列长度估计蛋白质结构域位置[51];DomCut利用结构域边界的连接区域的倾向性统计[52],判断结构域边界;Armadillo利用氨基酸在结构域及结构域边界出现的倾向性预测边界[53].自2005年开始,研究集中在采用机器学习的方法预测蛋白质结构域,如DROP[11-12]、DOMPro[13]、DOBO[14]、PPRODO[54]和DomNet[55]等.这些方法利用局部或整体的残基的统计特征,及psi-blast序列比对的特异矩阵等构成特征向量,再使用神经网络、支持向量机、随机森林等方法进行学习分类.我国吉林大学Zou等[56-57]采用距离最大熵和支持向量机的方法,上海大学Li等[58]结合最大相关最小冗余特征选择方法,同济大学Zhang等[59]采用条件随机场的开发的DomHR方法等都属于这一类.还有一些方法,组合多个或多种方法对蛋白质结构域进行综合预测.例如DOMAC组合了基于同源比对方法和机器学习方法[60];Meta_DP使用了10 个预测器[61],根据“多数”的投票原则,给出一致性预测.这些方法是对没有近同源模板序列进行结构域预测有价值的探索.
这些从序列出发的方法中,通过高序列相似度的同源模板拷贝结构域划分边界具有较高的可信度.ThreaDom是一个使用多个远同源比对的结构域划分方法[15],较以往方法有较大的性能优势.经过benchmark测试,在缺乏序列相似度>30%模板的情况下,ThreaDom的边界预测准确性较以往同源比对或机器学习方法均有明显的提高.
ThreaDom流程及边界划分见图3.

图3 ThreaDom 流程及边界划分示意图Fig.3 The flowchart and boundary decision of ThreaDom
从图3a可见,输入序列通过LOMET进行远同源比对后,得到多个已知3D结构的蛋白质序列模板,然后对这些模板与标准数据库进行位置映射,再通过计算结构域边界或比对缺失惩罚分数计算出结构域保守分数,最后通过全局门槛值方法确定结构域边界的位置(图3b).在对用户提交的序列的预测结果分析发现,ThreaDom在针对“Hard”类型、长序列及包含不连续结构域的结构域预测方面存在不足,在方法的模板选择、保守分数的设计、决策规则设计等领域还有很多未解决的问题.
4 从序列检测不连续结构域
根据PDB数据统计,约45%的多结构域蛋白质包括一个或多个不连续结构域.在基于结构划分结构域的方法中,已经有多个方法可以划分不连续结构域,如Domain Parser、PDP等.然而从序列出发的不连续结构域检测严重依赖于发现高序列相似度模板.三级结构建模的方法,在没有高序列相似度模板的情况下,很难完成对包含不连续结构域的多结构域蛋白的建模.基于统计与机器学习的方法更侧重于结构域边界的预测,很少涉及到不连续结构域检测.目前,Sikder等[62]采用基于预测原子接触方法、Xue等[5,15]的Threadom和DomEx,以及ThreaDomEx[25]是少数几个具备从序列检测不连续结构域的方法.
Sikder等基于预测的原子间接触间接预测不连续结构域,只能对极少部分蛋白质是否包含不连续结构域进行判断,无法报告准确的不连续结构域及边界.
ThreaDom具有检测不连续结构域的功能,是通过将LOMET返回模板的结构域边界聚类来实现的,该方法简单、直观,但依然依赖于Threading程序给出的远同源模板的准确性.ThreaDom检测不连续结构域的步骤如下:①检测输入序列是否含有不连续结构域.如果LOMET返回的模板中有超过30%的模板包括1或多个不连续结构域,则认为该输入序列含有1个不连续结构域;②对不连续结构域模板进行聚类.对具有相同结构域连续序列片段的数量和相似的边界的模板聚成一类,以边界误差在5个氨基酸以内为界定义边界的相似性;③边界优化与边界替换.根据结构域保守分数预测结构域边界和边界聚类中的第一个聚类结果融合.如果预测结构域的边界与第一个聚类中的结构域边界误差在20个残基内,这个预测结构域边界将合并入第一类聚类相应结构域中;同时,如果预测的边界结构域边界与聚类边界有很好的吻合度,且第一类结构域数量多于预测的结构域,将采用第一个聚类边界替换预测的结构域.
DomEx提出了组装序列对称比对的思想,以进行不连续结构域检测.可以使用任何结构域边界预测工具预测边界,进行不连续结构域检测.DomEx有3个基本假设: ⓐ同源的蛋白结构域可以使用profile-profile比对的方法检测到; ⓑ同源的结构域之间应该有相似的长度;ⓒ组装拼接的不连续结构域,再拼接点的两侧有相似的比对长度和序列相似性,即具有关于组装点两侧的对称性.
DomEx设计了模板相似分数、对称指数和profile-profile比对分数,用于不连续结构域检测.DomEx结合ThreaDom边界检测的流程图如图4所示.其关键步骤包括:①利用ThreaDom或者其他结构域边界预测工具预测结构域边界,把序列分成多个片段;②将空间上不连续的序列片段组装为候选的不连续结构域;③使用psi-blast搜索候选不连续结构域的同源结构域;④利用模板相似分数、对称指数、长度相似度评价组装结构域是一个不连续结构域的可能性;⑤利用profile-profile 比对进一步确认不连续结构域;⑥检测冲突并给出最终结果.
DomEx在组装候选不连续结构域后,使用psi-blast搜索单结构域nr数据库.该库的结构域主要来源于CATH、SCOP和PFam.对通过在PFam中找到的模板,还需要采用profile-profile比对方法进行进一步确认.通过实验观测,定义了一个参数b用于对不连续检出率MCC进行训练,具有较好的鲁棒性.
DomEx方法在不连续结构域检测方面与ThreaDom有互补性.在ThreaDom不能检测出的结果中,DomEx可以检测出26.7%的不连续结构域,且准确率在72%以上.当前基于对称比对和序列组装方法,需要在以下2方面重点突破:①在3个及3个以上序列片段组成的不连续结构域检测方面需要进一步扩展;②需要与Threading等远同源序列比对方法深度融合,提高不连续结构域的检出率和准确性.
ThreaDomEx 则组合了ThreaDom及DomEx的优点,其不连续结构域检测能力比ThreaDom更为优秀.
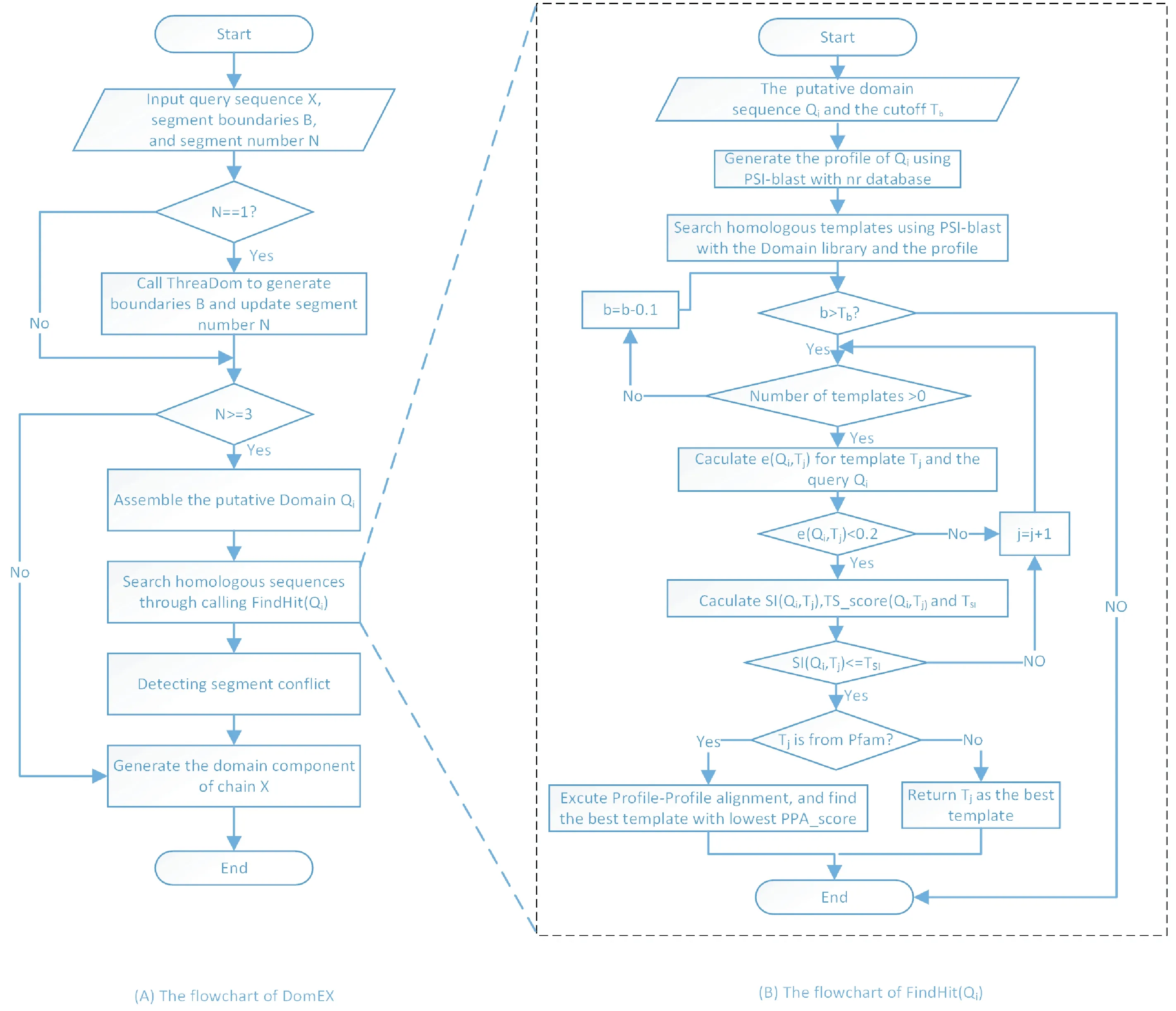
图4 DomEx不连续结构域检测流程Fig.4 Discontinuous domain detection of DomEx
5 结构域在线预测服务
蛋白质结构域的在线服务主要包括数据库和在线预测网站2类.Pfam、SMART、SCOP、CATH、InetrPro 和PROSITE是常用的结构域数据库[63].
Pfam、SMART都是基于隐马尔科夫模型进行近同源序列比对方法构建.PROSITE是使用profile和相关规则构建的结构域相关数据,其与Pfam相比,更侧重功能标注.这类数据库是建立在高序列相似度的近同源序列比对的基础上,无法深层次识别远同源序列蛋白质结构域.主要的不足是无法对于找不到近同源模板的序列进行结构域识别.
SCOP、CATH等仅仅对已知结构的蛋白质数据库进行整理、分类、标定等,不包含未知三级结构的蛋白质序列.这类数据库不提供对未知结构的蛋白质序列的结构域识别.InterPro通过整合多个结构域数据库的不同的结构域特征,作为结构域的预测模型,该数据库自身并不生成结构域的识别模型,只提供多个数据库的整合信息.
与蛋白质结构域数据库并存的是结构域在线预测服务,例如,DOBO、ThreaDom、ThreaDomEx等,提供了在线从序列预测结构域边界的能力.例如,ThreaDom自2013年7月上线以来,已经为来自世界各地的研究者提供9 600余次在线服务.
ThreaDomEx继承了ThreaDom和DomEx的优势,是其中具有代表性的在线预测服务.ThreaDomEx不仅能预测结构域边界、检测序列中存在不连续结构域,而且在线服务器用户界面友好,允许用户根据个人知识使用系统提供中间结果,进行可视化交互修改、保存预测结果.图5给出了ThreaDomEx在线服务预测界面的结果,用户可以根据系统预测的结果用鼠标进行拖拽操作,可以参考预测二级结构和溶液可及性进行修改;可以在增加删除结构域片段后,提交服务器再次进行不连续结构域检测.
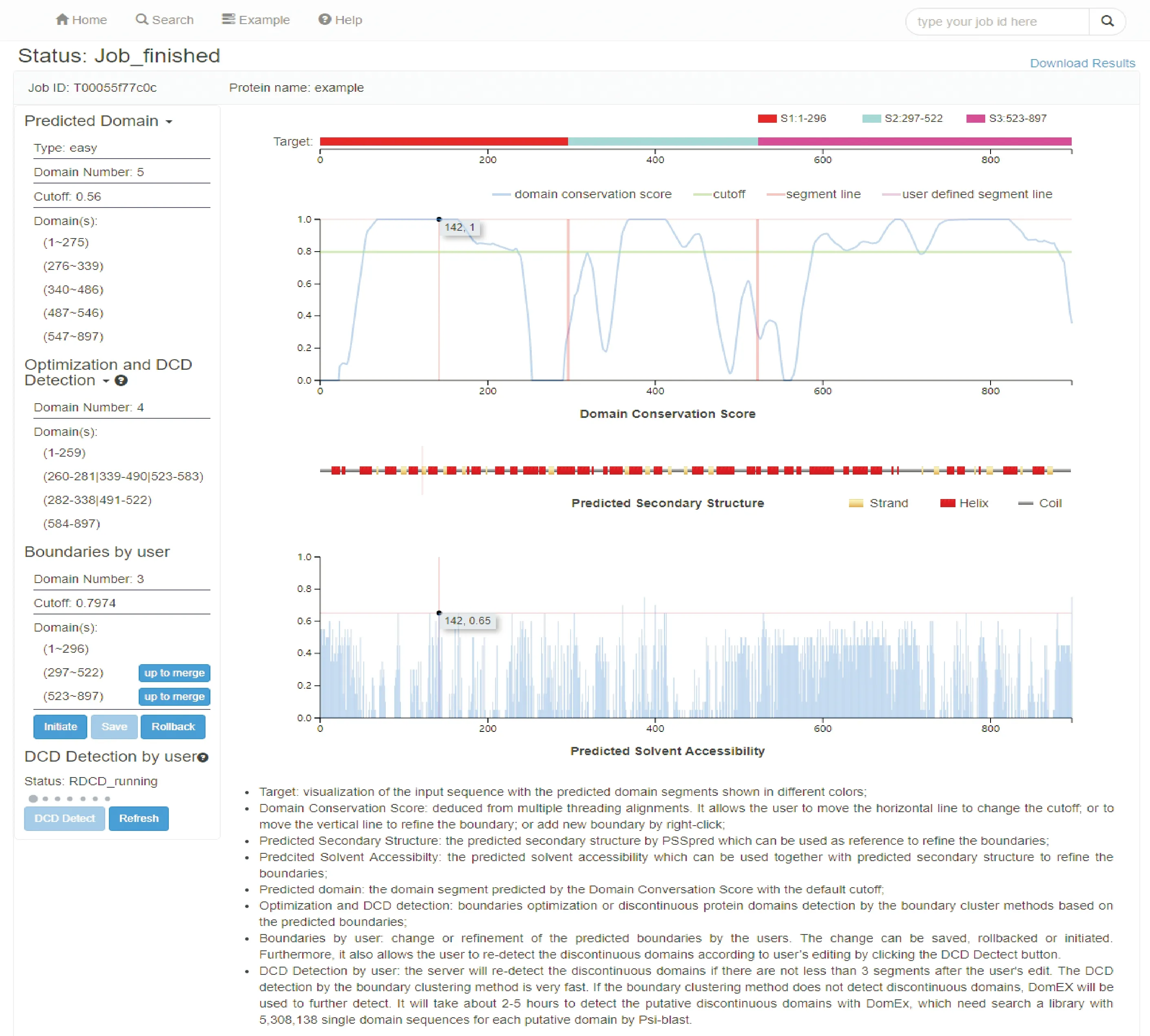
图5 ThreaDomEx在线服务界面Fig.5 The oneline service page of ThreaDomEx
6 结 论
总之,以往的研究对蛋白质结构域识别做了积极而有意义的探索,但依然存在大于50%的非重复蛋白质序列亟需标注结构域信息.在缺少近同源模板的情况下,通过基于Threading检测远同源模板方法,成为提高结构域标注准确性和标注比例最为有效和可能的途径.另外,由于基因插入、融合造成大量存在的不连续结构域,也要求有新的方法和手段提高不连续结构域的标注比例.建议相关研究者在使用PFam、CATH、SCOP等数据库无法得到满意结果的情况下,使用ThreaDom、ThreaDomEx进行常识性结构域划分;同时,亟需开发能从序列识别远同源蛋白质结构域的新方法及相应的数据库,进而注释这些蛋白质的家族与功能,为研究者提供更加丰富、便捷的蛋白质结构域数据库系统和工具.
——紫 苏

